凡纳滨对虾(Litopenaeus vannamei)亦称南美白对虾、太平洋白对虾,原产于中南美太平洋海岸水域,主要分布于秘鲁北部至墨西哥湾中部,与斑节对虾(Penaeus monodon)和中国明对虾(Fenneropenaeus chinensis)并列为世界上养殖产量最高的三大优良虾种(Meehan et al, 2003)。凡纳滨对虾生长快、营养丰富、肉味鲜美、抗应激能力强且易进行集约化养殖,随着淡水养殖技术的推广,近年来其养殖面积和产量的增长尤其迅速。目前,全球范围内有52个国家养殖凡纳滨对虾,亚洲的中国、新加坡、印度、越南、印度尼西亚及南美洲的厄瓜多尔、墨西哥等国的养殖量较大,其中,亚洲占了约85%的养殖产量。中国的对虾养殖总产量占全球对虾养殖总量的40%以上,而其中的80%以上为凡纳滨对虾(王昌翰, 2013),主要集中在广东、福建、浙江、江苏、山东、辽宁等沿海地区。凡纳滨对虾的淡水养殖在我国的珠江三角洲、广西、江西和湖南等地区也已经有了相当规模。
全球凡纳滨对虾种虾资源主要分布在美国(夏威夷、迈阿密、关岛)、新加坡、泰国和印度尼西亚等地区,美国是凡纳滨对虾种质资源的拥有国,新加坡、泰国及印度尼西亚的种虾资源均为从美国转移过去的。2012年,我国凡纳滨对虾养殖总产量为117万t,约占全球凡纳滨对虾产量的40%,近年呈逐年上升的趋势。相对于如此巨大的凡纳滨对虾养殖产业,种虾的供应极为短缺,基本处于“无种有业”状态。目前,国内所用的亲虾,少部分是从当年养殖的大规格虾中直接挑选,未经过选育,大量亲虾都依赖于每年从国外引进。进口亲虾与国产亲虾的价格尽管相差悬殊,却仍供不应求。而我国进口的亲虾大部分遗传背景不明,是否具备作为进一步选育的遗传资源是目前面临的问题。因此,需要了解国内外不同地区凡纳滨对虾群体间的遗传多样性等背景方面的差异,为凡纳滨对虾的生产养殖和良种选育提供遗传背景信息。
目前,关于凡纳滨对虾遗传多样性的分析多采用限制性片段多态性(RFLP)、线粒体DNA标记(mtDNA)、扩增片段长度多态性(AFLP)、简单序列重复(SSR)等分子生物学标记技术(Valles-Jimenez et al, 2006; Dutta et al, 2014; Shen et al, 2007; Pébrez et al, 2004、Pérez et al, 2005; Gorbach et al, 2009)。由于动物mtDNA具有严格的母系遗传、DNA分子量小、拷贝数高、结构和组织简单而高度保守、缺乏重组、DNA突变率高等一系列优点(Wolstenholme, 1992),因而,近年来通过测定、分析mtDNA全序列或部分基因片段来探讨物种进化关系已成为研究水产动物系统进化和群体遗传学分析的理想手段。其中,有关线粒体12S rRNA、16S rRNA、COI、D-loop等基因片段的研究较多(Bouchon et al, 1994; Machado et al, 1993; Lavery et al, 2004; McMillen-Jackson et al, 2004)。
控制区(D-loop)是线粒体上的非编码区,进化速度快,适合群体水平的变异分析(Zhang et al, 1992)。本研究利用线粒体控制区测序技术,对美国塞班岛、美国关岛、中国广东茂名三个地区的6个人工养殖凡纳滨对虾群体进行了遗传多样性和差异性分析,以期为凡纳滨对虾的生产养殖、良种选育和种质资源评估等提供参考。
1 材料与方法 1.1 样品来源及基因组DNA的提取本研究的样本分别取自美国塞班岛凡纳滨对虾养殖基地(S1、S2、Sg群体)、美国关岛凡纳滨对虾养殖基地(G1、G2群体)和中国广东茂名凡纳滨对虾养殖基地(K1群体),其中S1、S2和Sg群体购自不同种虾公司的仔虾,养殖在塞班基地,S2群体于不同时间分3次购自同一种虾公司;G1、G2群体来自关岛大学不同家系的繁殖后代。实验样本中亲虾或成虾取附肢,仔虾或幼虾取完整个体,无水乙醇密封于样本瓶中,带回实验室后于4℃冰箱保存备用。其中,S1、K1群体全部样本和S2群体的部分样本有雌雄信息记录,S2群体的前两批次的样本有体长、体重的记录,具体取样信息见表 1。本研究全部样本使用海洋动物组织基因组DNA提取试剂盒[天根生化科技(上海)有限公司]提取基因组总DNA。提取所得到的DNA样品经1.0%的EB-琼脂糖凝胶电泳检测纯度,微量紫外可见分光光度计(Thermo Scientific)检测记录部分样品DNA浓度后,全部DNA样品分装,保存于−20℃冰箱备用。
|
|
表 1 凡纳滨对虾6个群体样品来源与取样信息 Table 1 Sample source and sampling information of 6 populations of L. vannamei |
参考NCBI的GenBank中已公布的凡纳滨对虾线粒体DNA全序列设计控制区部分片段扩增引物,上下游引物序列分别为:F-276(5′-CCTAAAATTGAA-AGAATAAGCTAGG-3′)和R-1089(5′-AGTGTCTTCT-TTTTGTATGAAACTTTAATC-3′),上游引物位于保守区,下游引物在可变区,引物由生工生物工程(上海)股份有限公司合成。首先,优化PCR条件,通过设置不同退火温度(53℃、55℃、57℃、59℃和61℃)进行PCR部分梯度扩增,扩增产物经1.0%的EB-琼脂糖凝胶电泳检测,最终确定55℃扩增能够获得稳定清晰的条带,可用于全部样品的扩增。PCR反应体系为50 μl:DNA模板2.0 μl;上、下游引物(10 μmol/L)各1 μl;2×PCR Master Mix (上海莱枫生物科技有限公司) 25 μl;补充灭菌双蒸水至终体积为50 μl。PCR反应条件为:94℃预变性3 min;94℃变性1 min,55℃退火1 min,72℃延伸1 min,35个循环;最后72℃延伸5 min;15℃保存。PCR扩增产物经1.0%的EB-琼脂糖凝胶电泳检测,扩增效果符合测序要求的,直接送生工生物工程(上海)股份有限公司进行双向测序。
1.3 mtDNA控制区序列的整理与分析将测序所得mtDNA控制区序列用序列编辑软件BioEdit v7.0.5.2进行比对整理并辅以人工校正,去除两端多余序列;用ClustlW软件程序进行多重比对分析,使用DNAsp v5.1软件计算序列保守位点、变异位点和简约信息位点,并计算基因单倍型多样性(Haplotype diversity, Hd)、核苷酸多样性(Nucleotide diversity, π)和平均核苷酸差异数(Average number of nucleotide differences, k);用Arlequin v3.1软件统计碱基组成、多态位点数、转换(Transitionsal, Si)、颠换(Transversional, Sv)和插入/缺失(Insertion deletion, Indel)等分子多态性指数,并对6个群体样本的遗传多样性进行Tajima’s D值中性检验,对群体内和群体间的遗传变异进行AMOVA(Analysis of Molecular Variance)分子方差分析和遗传分化固定系数Fst (Fixation Index statistics)检验。用MEGA v5.0软件中的Kimura Two-Parameter程序计算群体内和群体间遗传距离,采用非加权组平均法(Unweighted pair-group method with arithmetic means, UPGMA)构建基于遗传距离的群体间的分子系统进化树和基于单倍型结果的单倍型NJ(Neighbor Joining)聚类图,并对系统树各分支的支持率进行1000次重复抽样检验。
2 结果 2.1 凡纳滨对虾mtDNA控制区序列的变异分析PCR扩增所得凡纳滨对虾mtDNA控制区序列产物,采用双向测序的方法,其中,S2组部分样本直接测序结果不理想,采用纯化后克隆测序的方法,共计得到6个凡纳滨对虾群体的285个不同凡纳滨对虾样本的mtDNA控制区序列。引物预计扩增长度813 bp,实际测序所得片段长度平均为783 bp (730–813 bp),使用BioEdit软件比对分析并去除两端冗余序列后,序列平均有效长度为603 bp (602–604 bp)。将所得序列通过BLASTn与NCBI中GenBank公布的凡纳滨对虾mtDNA序列同源性进行比对分析,其同源相似性比例平均为94% (87%–100%),初步确认所得序列为凡纳滨对虾的mtDNA控制区的目标序列。使用DNAsp v5计算保守位点、变异位点和简约信息位点,其中,基因保守位点477个,基因变异位点127个,简约信息位点121个,主要变异位点集中在S2、K1两个群体中。所测得的所有序列中,T、C、A、G的平均碱基含量分别为47.6%、10.4%、34.4%、7.6%,A+T的含量为82.0%,明显高于G+C的含量(18.0%)。在285条序列中,共检测到133个多态位点,约占总序列长度的22.1%,这些多态位点共定义了94个转换、50个颠换和12个插入缺失,转换与颠换比值(R=Si/Sv)为1.88。
2.2 凡纳滨对虾单倍型及遗传多样性在6个凡纳滨对虾群体的285个样品中,共检测到146个单倍型,其中,S1群体的两个单倍型(H1、H2)与群体S2共享,其他144个单倍型均为单个群体特有单倍型。6个凡纳滨对虾群体的单倍型多样性和核苷酸多态性分析结果显示(表 2),单倍型多样性(Hd)为(0.42±0.08)–(0.99±0.01),核苷酸多样性(π)为(0.00±0.00)–(0.08±0.04)。其中,单倍型多样性最高的是K1群体(Hd=0.99±0.01),最低的是S1群体(Hd=0.42±0.08);核苷酸多样性指数最高的为S2群体(π=0.08±0.04),最低的为S1群体(π=0.00);综合考虑,S1群体的遗传多样性最低(Hd=0.42±0.08, π=0.00),S2群体的遗传多样性水平最高(Hd=0.88±0.02, π=0.08±0.04)。对6个群体遗传多样性进行Tajima’s D值中性检验(表 2),所有群体的Tajima’s D中性检验范围为−1.493–5.081,G1群体中性检验值(−1.493)为负值,但由于6个群体的中性检验结果均未达到显著水平(P > 0.05),因此,不支持这些群体曾经历过群体扩张或群体快速增长。
|
|
表 2 6个凡纳滨对虾的遗传变异参数统计 Table 2 Statistics of genetic variation parameters of 6 populations of L. vannamei |
利用mtDNA控制区序列比较6个凡纳滨对虾群体的遗传分化,根据软件Arlequin 3.1的AMOVA分析结果显示,来自于群体间的遗传差异(46.98%)略低于来自群体内的遗传差异(53.02%),说明主要的变异来源于各群体内部。群体间遗传差异的Fst指数分析结果显示,6个养殖群体之间均存在不同程度的遗传分化(Fst值均为正),除了G1和G2群体间属于较大的遗传分化(Fst=0.173),其他各群体间遗传分化程度均为极大(Fst > 0.25)。采用MEGA 5.0软件中的Kimura Two-Parameter程序计算群体间遗传距离(表 3),结果表明,群体G1、G2和Sg三者之间的遗传关系最近;S1群体和关岛的Sg群体之间的遗传距离最远,约为0.2。其中,S1和S2两个群体与关岛G1、G2、Sg群体间及中国广东茂名K1群体间遗传距离均明显较远,数值均在0.1以上。
|
|
表 3 6个凡纳滨对虾群体间遗传分化Fst值(对角线下)和群体遗传距离(对角线上) Table 3 Genetic differentiation Fst values(down diagonal) and genetic distance(up diagonal) among 6 populations of L. vannamei |
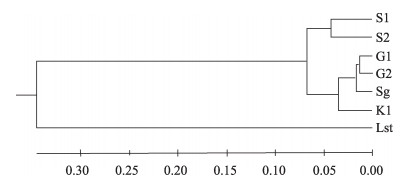
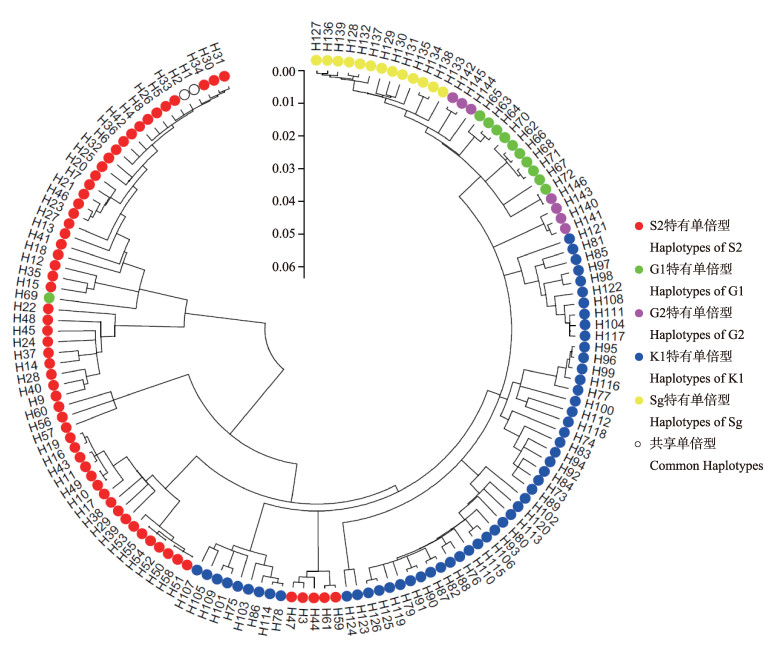
根据遗传距离构建群体之间的UPGMA系统进化树,以及单倍型结果构建单倍型NJ聚类图,本研究采用与凡纳滨对虾同属不同种的南美蓝对虾(Litopenaeus stylirostris, Lst)为外延生物,从遗传距离UPGMA进化树(图 1)可知,除去外延生物,6个凡纳滨对虾群体分别聚为两支,群体S1和S2遗传距离较近,聚为一支,其他4个群体聚成另外一支。其中,G1和G2群体首先聚在一起,然后再与Sg群体聚在一起,之后再与K1群体聚在一起。从单倍型NJ聚类图(图 2)结果中得到,进化树主要分为两个大的分支:S1和S2群体共享单倍型H1、H2,且群体S1没有独立单倍型存在,两群体的遗传关系较近,基本聚在一起;S2群体的部分单倍型与K1群体距离较近,且与K1群体有单倍型交叉现象,而G1、G2、Sg群体的单倍型明显聚在一起。可见,基于单倍型的聚类结果与基于遗传距离的聚类结果基本相似。
|
图 1 基于遗传距离建立的6个凡纳滨对虾群体间的UPGMA进化树 Figure 1 The UPGMA evolutionary tree of 6 populations ofL. vannameibased on the genetic distance of mitochondrial control region sequences |
|
图 2 基于Kimura Two-Parameter模型构建的线粒体控制区序列的单倍型NJ聚类 Figure 2 The NJ haplotype clustering diagram of the mitochondrial control region sequences constructed based on Kimura Two-Parameter model |
考虑到本研究所选用的线粒体标记为母系遗传,为了解所得到的分析结果在雌雄个体间是否存在差异,进一步对S1、S2、K1群体中的雌雄个体分别组群,进行了遗传多样性指数计算和遗传分化指数Fst检验,并对遗传多样性进行Tajima’s D值检验,检验结果采取1000次自举检验验证。结果表明,同一群体的雌雄样本群体间遗传多样性水平相近(表 4)。遗传分化Fst指数显著性检验结果显示,S1、S2和K1三个群体的雌虾群体、雄虾群体与各自群体间的Fst值均为负值,即没有遗传分化;S1群体的雌虾群体和雄虾群体间的Fst值也为负值,没有遗传分化,而S2和K1群体的各自雌虾群体和雄虾群体间有极小的遗传分化(Fst分别为0.01和0.04)。3个群体S1、S2、K1的雌雄群体间交叉Fst值均为正且数值大小都在0.20以上,即均存在较大程度的遗传分化,且分化差异为显著(P < 0.05)(表 5)。考虑到S1群体的样本量比较小,代表性不充分,可以初步认为同一群体内的雌雄群体间线粒体控制区序列有较大的变异,导致了少量遗传分化的产生。
|
|
表 4 3个凡纳滨对虾群体雌雄群体间的遗传变异参数统计 Table 4 Statistics of genetic variation parameters between male and female in 3 populations of L. vannamei |
|
|
表 5 3个凡纳滨对虾群体雌雄群体间遗传分化Fst值 Table 5 Genetic differentiationFstvalues between male and female individuals of 3 populations ofL. vannamei |
S2群体购自同一家种虾公司,分别为一年时间跨度内分3次购入,为了了解同一公司进口的虾不同批次间是否存在较大的遗传差异,对S2群体不同购买时间的虾分别进行了遗传多样性指数计算和遗传分化Fst检验,并对遗传多样性进行Tajima’s D值检验,检验结果采取1000次自举检验验证。同时,对S2群体在养殖中生长较快和生长较慢的个体也进行了相同的遗传分析检验,目的是了解同一批虾苗不同生长速率的个体之间是否存在遗传差异。结果显示(表 6),S2群体中同一批次的大小群体间遗传多样性参数相近,3批群体中,第3批Ss群体的遗传参数水平与前两批(N7 & N8与F3 & I3)偏差较大。遗传分化Fst值检验结果显示(表 7),S2群体的3个批次中,前两批(N7 & N8与F3 & I3)之间遗传分化较小,Fst值仅为0.037,第3批与前两批之间均存在很大的遗传差异,Fst值分别为0.457、0315。第1批次群体中,个体较大群体N7和个体较小群体N8之间Fst为负(−0.015),表示N7和N8之间没有遗传分化差异;而第2批次中,个体较大群体I3和个体较小群体F3之间有一定程度的遗传分化差异,Fst值为0.081且差异显著性检验为显著。从上述结果可以看出,同一批虾苗在养殖过程中通过多次筛选,生长较快的群体(如I3)同生长较慢的群体(如F3)之间可能存在一定程度的遗传差异;同一家种虾公司的虾苗,不同购买时间的群体间存在遗传分化,该遗传分化随时间间隔的加长而呈增加趋势。
|
|
表 6 S2群体不同批次及不同个体大小群体的遗传参数统计 Table 6 Statistics of genetic variation parameters of different batches and different size of individuals in populations S2 |
|
|
表 7 S2群体不同批次、不同个体大小群体间Fst值 Table 7 Genetic differentiationFst values of different batches and different size of individuals in populations S2 |
本研究初步分析并比较了6个凡纳滨对虾不同群体的mtDNA控制区部分序列的差异。由于线粒体基因组是独立于核基因组的遗传物质,它普遍存在于真核细胞中,线粒体内包含有DNA和转录与转译系统,是具有一定自主性的细胞器;线粒体DNA分子小、拷贝数高,结构和组织简单而高度保守,母系遗传,缺乏重组;加之凡纳滨对虾DNA突变率较高(Shen et al, 2007),结果显示,凡纳滨对虾具有较高的遗传杂合水平。mtDNA控制区序列的碱基组成中,G含量最低(7.7%),AT含量(82.0%)明显高于GC含量(18.0%),该结果与其他对虾类的研究结果相似(Peregrino-Uriarte et al, 2009; Klinbunga et al, 1999),也与线粒体控制区基因碱基组成中普遍存在的AT含量高现象一致(Williams et al, 2001)。从原核生物到真核生物,其基因组中碱基偏向性的现象广泛存在,且不同物种间碱基的不均衡程度也各不相同。这一现象的产生可能与基因的表达水平、翻译起始效应、碱基组分、基因长度以及密码子与反密码子间结合能力的大小有关(武伟等, 2007; Perna et al, 1995)。在DNA进化过程中,碱基转换(Si)发生的频率要比颠换(Sr)高得多,本研究结果与此相符。利用转换/颠换比值R,可以估计序列的饱和度,一般分歧时间越长、亲缘关系越远的分类单元之间核苷酸发生颠换的频率越高,即Si/Sr值越小,由此可初步判断所研究的分类单元之间的进化关系(Tamura et al, 1992; Yang et al, 1999)。本研究中转换与颠换比值(R=Si/Sv)较小,为1.88(R < 5),说明本研究中的凡纳滨对虾群体存在一定程度的进化隔离,亲缘关系较远。
3.2 凡纳滨对虾的遗传多样性和遗传分化分析Liu等(2006)研究表明,群体中mtDNA的单倍型多样性(Hd)和核苷酸多样性(π)是衡量群体多态程度和群体遗传分化的重要指标之一,Hd值、π值越大,表示群体多态程度越高。本研究中单倍型结果表明,DNA单倍型作为一种DNA条码(Hebert et al, 2003)能够区分多数不同群体(G1、G2、K1和Sg),而少数群体不具有独特的单倍型(S1和S2群体共享单倍型H1、H2)。Sg群体具有较高的单倍型多样性(Hd=0.98±0.04),但核苷酸多样性(π=0.01±0.01)却明显偏低,说明Sg群体可能是由一个较小的有效种群迅速增长,虽然通过变异积累了单倍型的多态性,但却还未能积累核苷酸序列的多样化(You et al, 2008)。S1群体的单倍型多样性和核苷酸多样性均较低(Hd=0.420±0.082, π=0.001±0.001),表明该群体在进化过程中可能经历过长期的瓶颈效应(Effects of bottlenecks)或奠基者效应(Founder effect)影响(Leberg, 1992)。本研究中,多个凡纳滨对虾群体具有较高的单倍型多样性及核苷酸多样性,表明凡纳滨对虾具有较丰富的遗传多样性。
遗传分化的固定系数(Fst)常用来表示两个群体间的遗传分化程度,在0–l的范围内,Fst值越大,两群体间的分化程度越高(杨金权等, 2008)。Fst值为负,表示两群体间没有遗传分化;Fst为0–0.05,表示两群体间有极小的遗传分化;Fst为0.05–0.15,表示两群体间有中等程度的遗传分化;Fst为0.15–0.25,表示两群体间有较大的遗传分化;Fst值为0.25以上,表示两群体间有极大的遗传分化(Balloux et al, 2002)。比较本研究中凡纳滨对虾两两群体间的Fst值发现,除了关岛群体G1和G2间(0.173)是较大的遗传分化外,凡纳滨对虾其他各群体间均出现了极大的遗传分化,且遗传分化检验为显著,可以作为未来选育的基础群体。关岛的G1和G2两个群体是分别来自于较小保存家系后代,遗传分化未能达到极大差异水平,未来选育工作中可以考虑合并为1个基础群体。
3.3 凡纳滨对虾的遗传距离和部分群体雌雄样本间的遗传差异分析分别基于遗传距离构建UPGMA系统进化树和基于单倍型结果构建群体之间的单倍型NJ聚类图,UPGMA系统进化树结果表明,6个凡纳滨对虾养殖群体可以分为两个进化支(塞班岛S1、S2两群体聚为一支;Sg群体与关岛G1、G2两个群体聚在一起后再与广东茂名K1群体聚为另一支),由于Sg群体仔虾来源于关岛的核心家系,只是在塞班岛进行养殖,与G1和G2聚为一支,也侧面反映了进化支的分布和地理位置之间的对应性。6个群体单倍型的聚类关系与遗传距离的进化关系类似,各群体特有单倍型也基本聚在一起,部分有交叉现象。产生这些结果的原因可能是:这些群体在选育过程中是以经济性状为目标,几乎都是通过从不同地区引进的凡纳滨对虾群体之间的杂交选育而来,各养殖群体在选育过程中部分亲本的遗传背景可能存在交叉(Francisco et al, 2005)。
对凡纳滨对虾3个养殖群体S1、S2、K1的雌雄样本群体间的遗传差异进行分析,结果表明,同一群体的雌雄群体间遗传多样性水平相近(Hd、π值均相近)。Fst检验结果表明,S1的雌雄群体间没有遗传分化差异,但S2群体的雄虾群体S2M与雌虾群体S2F间的Fst值为正(0.014),表明S2群体中雌雄群体之间存在轻微的遗传分化(Fst < 0.05)。而K1群体与S2群体类似,雌雄群体间也出现了轻微的遗传分化差异。Lee等(2011)研究发现,不同群体的遗传分化差异来源是多方面的,对于本研究凡纳滨对虾mtDNA控制区序列分析中,短暂的隔离作用、较低的基因流、分化时间的早晚、地理位置差异及外界环境的差异等都可能是遗传分化差异的影响因子。此外,养殖环境的差异导致其累积了一定程度的遗传变异,也会产生遗传分化(Chow et al, 1991),也可能与不同群体的遗传背景本身可能存在较大差异有关(Freitas et al, 2007)。
3.4 凡纳滨对虾S2群体不同批次及不同个体大小群体间的遗传差异分析由于S2群体的样本是分为3个批次从同一公司购买而来(第1批购买时间为2012年11月,第2批为2013年1月,第3批为2013年8月),且3批中前两批次样本每隔一段时间均进行个体大小筛选,优先选取一定数量生长较快、个体较大的样本(N7、I3),剩余的归为个体较小的样本(N8、F3)。对个体较大群体和较小群体的遗传比较分析发现,通过选择确实能将本来存在遗传差异的个体筛选出来,如I3(较大个体群)与F3(较小个体群)间观察到遗传分化的出现;而相似的情况在N7和N8间没有得到类似结果,遗传分化不明显。这可能与塞班当地在养殖过程中的筛选不够严格和彻底有关。至少在有限的数据中发现,通过筛选分开的不同生长速率群体之间是存在遗传分化的,说明只要有严格的筛选机制,可以从同一批次虾苗中选出具备较好生长遗传特性的个体,即严格选择在良种选育中是有效的。
在进口种虾和虾苗时,对种虾公司所提供的产品无法直接获得相关的遗传背景数据,在未来的国产种虾培育过程中,很难确定哪些是可以留种,而哪些是需要舍弃的。在本研究中,对同一公司的不同批次群体间是否存在遗传分化进行了相关遗传差异Fst分析检验,发现S2群体中前两批群体间(购入时间间隔为两个月)遗传分化差异较小(0.037),第3批Ss群体与前两批群体之间(购入时间间隔分别为9个月和7个月)的遗传分化差异明显偏大(0.457、0.315),说明随着时间间隔的增加,即使是同一家公司的产品之间遗传分化也在加大,遗传分化的差异可能会大于不同公司的产品,如S2与K1群体分别来自两家不同的种虾公司,它们之间的Fst为0.228,小于S2第3批次与前两批次间的遗传分化值。这为购买进口种虾和虾苗来培育国产种虾提供了思路,可以尽可能获得遗传分化最大的群体。
本研究仅是从线粒体DNA控制区序列结果进行分析,由于线粒体自身特殊的遗传机制,结果反映的是凡纳滨对虾母系遗传过程中的一些特征,在进一步研究中,需要考虑细胞核DNA分子标记的分析结果,下一步将配合使用微卫星标记(SSR)对本研究中的凡纳滨对虾群体进行相关遗传性分析,将细胞核DNA标记技术和核外mtDNA标记技术结合起来共同分析,以期能对凡纳滨对虾的遗传背景和遗传多样性进行更加全面、准确的分析,为遗传育种提供指导。
| 王昌翰. 南美白对虾种业与市场分析. 海洋与渔业·水产前沿 , 2013 (7) : 81-83 | |
| 武伟, 刘洪斌, 张泽, 等. 节肢动物线粒体基因组碱基组成特征分析. 生物信息学 , 2007, 5 (3) : 102-105 | |
| 杨金权, 胡雪莲, 唐文乔, 等. 长江口邻近水域刀鲚的线粒体控制区序列变异与遗传多样性. 动物学杂志 , 2008, 43 (1) : 8-15 | |
| Balloux F, Lugon-Moulin N. The estimation of population differentiation with microsatellite markers. Mol Ecol , 2002, 11 (2) : 155-165 DOI:10.1046/j.0962-1083.2001.01436.x | |
| Bouchon D, Souty-Grosset C, Raimond R. Mitochondrial DNA variation and markers of species identity in two penaeid shrimp species: Penaeus monodon Fabricius and P. japonicus Bate. Aquaculture , 1994, 127 (94) : 131-144 | |
| Chow S, Sandifer PA. Differences in growth, morphometric traits, and male sexual maturity among Pacific white shrimp (Penaeus vannamei)from different commercial hatcheries. Aquaculture , 1991, 92 : 165-178 DOI:10.1016/0044-8486(91)90018-3 | |
| Dutta S, Biswas S, Mukherjee K, et al. Identification of RAPD-SCAR marker linked to white spot syndrome virus resistance in populations of giant black tiger shrimp, Penaeus monodon Fabricius. J Fish Dis , 2014, 37 (5) : 471-480 DOI:10.1111/jfd.2014.37.issue-5 | |
| Francisco AKD, Junior PMG. Genetic distance between broodstocks of the marine shrimp Litopenaeus vannamei (Decapoda, Penaeidae) by mtDNA analyses. Genet Mol Biol , 2005, 28 (2) : 258-261 DOI:10.1590/S1415-47572005000200014 | |
| Freitas PD, Calgaro MR, Galetti Jr PM. Genetic diversity within and between broodstocks of the white shrimp Litopenaeus vannamei (Boone, 1931)(Decapoda, Penaeidae) and its implication for the gene pool conservation. Braz J Biol , 2007, 67 (4) : 939-943 DOI:10.1590/S1519-69842007000500019 | |
| Gorbach DM, Hu ZL, Du ZQ, et al. SNP discovery in Litopenaeus vannamei with a new computational pipeline. Anim Genet , 2009, 40 (1) : 106-109 DOI:10.1111/age.2009.40.issue-1 | |
| Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes. Proc Royal Soc London B: Biol Sci , 2003, 270 (1512) : 313-321 DOI:10.1098/rspb.2002.2218 | |
| Klinbunga S, Penman DJ, Mcandrew BJ, et al. Mitochondrial DNA diversity in three populations of the giant tiger shrimp Penaeus monodon. Mar Biotechnol , 1999, 1 (2) : 113-121 DOI:10.1007/PL00011758 | |
| Lavery S, Chan TY, Tam YK, et al. Phylogenetic relationships and evolutionary history of the shrimp genus Penaeus s.l. derived from mitochondrial DNA. Mol Phylogenet Evol , 2004, 31 (1) : 39-49 | |
| Leberg PL. Effects of population bottlenecks on genetic diversity as measured by allozyme electrophoresis. Evolution , 1992, 46 (2) : 477-494 DOI:10.2307/2409866 | |
| Lee CR, Mitchell-Olds T. Quantifying effects of environmental and geographical factors on patterns of genetic differentiation. Mol Ecol , 2011, 20 (22) : 4631-4642 DOI:10.1111/mec.2011.20.issue-22 | |
| Liu RY, Xia XL, Lei CZ, et al. Genetic diversity of mitochondrial DNA D–loop sequences in cattle breeds in Guizhou. Hereditas(Beijing) , 2006, 28 (3) : 279-284 | |
| Machado EG, Dennebouy N, Suarez MO, et al. Mitochondrial 16S rRNA gene of two species of shrimps: sequence variability and secondary structure. Crustaceana , 1993, 65 (3) : 279-286 DOI:10.1163/156854093X00711 | |
| McMillen-Jackson AL, Bert TM. Genetic diversity in the mtDNA control region and population structure in the pink shrimp Farfantepenaeus duorarum. J Crustacean Biol , 2004, 24 (1) : 101-109 DOI:10.1651/C-2372 | |
| Meehan D, Xu ZK, Zuniga G, et al. High frequency and large number of polymorphic microsatellites in cultured shrimp, Penaeus (Litopenaeus) vannamei. Mar Biotechnol , 2003, 5 (4) : 311-330 DOI:10.1007/s10126-002-0092-z | |
| Peregrino-Uriarte A, Varela-Romero A, Muhlia-Almazán A, et al. The complete mitochondrial genomes of the yellowleg shrimp Farfantepenaeus californiensis and the blue shrimp Litopenaeus stylirostris (Crustacea: Decapoda). Comp Biochem Phys Part D: Genomics and Proteomics , 2009, 4 (1) : 45-53 DOI:10.1016/j.cbd.2008.10.003 | |
| Pérez F, Erazo C, Zhinaula M, et al. A sex-specific linkage map of the white shrimp Penaeus (Litopenaeus) vannamei based on AFLP markers. Aquaculture , 2004, 242 (1–4) : 105-118 | |
| Pérez F, Ortiz J, Zhinaula M, et al. Development of EST-SSR markers by data mining in three species of shrimp: Litopenaeus vannamei, Litopenaeus stylirostris, and Trachypenaeus birdy. Mar Biotechnol , 2005, 7 (5) : 554-569 DOI:10.1007/s10126-004-5099-1 | |
| Perna NT, Kocher TD. Patterns of nucleotide composition at fourfold degenerate sites of animal mitochondrial genomes. J Mol Evol , 1995, 41 (3) : 353-358 DOI:10.1007/BF01215182 | |
| Shen X, Ren J, Cui Z, et al. The complete mitochondrial genomes of two common shrimps (Litopenaeus vannamei and Fenneropenaeus chinensis) and their phylogenomic considerations. Gene , 2007, 403 (1–2) : 98-109 | |
| Tamura K. Estimation of the number of nucleotide substitutions when there are strong transition-transversion and G+C-content biases. Mol Biol Evol , 1992, 9 (4) : 678-687 | |
| Valles-Jimenez R, Gaffney PM, Perez-Enriquez R. RFLP analysis of the mtDNA control region in white shrimp (Litopenaeus vannamei) populations from the eastern Pacific. Mar Biol , 2006, 148 (4) : 867-873 DOI:10.1007/s00227-005-0122-2 | |
| Williams ST, Knowlton N, Weigt LA, et al. Evidence for three major clades within the snapping shrimp genus alpheus inferred from nuclear and mitochondrial gene sequence data. Mol Phylogenet Evol , 2001, 20 (3) : 375-389 DOI:10.1006/mpev.2001.0976 | |
| Wolstenholme DR. Animal mitochondrial DNA: structure and evolution. Int Rev Cytol , 1992, 141 : 173-216 DOI:10.1016/S0074-7696(08)62066-5 | |
| Yang ZH, Yoder AD. Estimation of the transition/transversion rate bias and species sampling. J Mol Evol , 1999, 48 (3) : 274-283 DOI:10.1007/PL00006470 | |
| You EM, Chiu TS, Liu KF, et al. Microsatellite and mitochondrial haplotype diversity reveals population differentiation in the tiger shrimp (Penaeus monodon) in the Indo-Pacific region. Anim Genet , 2008, 39 (3) : 267-277 DOI:10.1111/age.2008.39.issue-3 | |
| Zhang YP, Shi LM. Mitochondrial DNA polymorphisms in animals: a review. Zool Res , 1992, 13 (3) : 289-298 |



