2. 农业部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;
3. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266200;
4. 中国海洋大学水产学院 青岛 266003
2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071 ;
3. Function Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266200 ;
4. College of Aquaculture, Ocean University of China, Qingdao 266003
Sox (SRY -related HMG-box)基因家族是一类SRY (Sex determination region of Y chromosome)相关基因构成的基因家族,属于HMG-box超家族成员(Laudet et al , 1993)。最早发现的Sox 基因是在哺乳动物中鉴定出来的睾丸决定因子SRY (Gubbay et al , 1990),根据一段长约79个氨基酸的DNA结合基序HMG-box的保守性,将在HMG-box区与SRY 产物具有50%以上的氨基酸序列相似性的编码基因定义为Sox 基因家族成员(Chardard et al , 1993)。随着种内及种间越来越多的Sox 基因被发现,Bowles等(2000)提出了新的Sox 基因定义标准,即以HMG结构域的第5–10个氨基酸残基(RPMNAF)作为识别Sox 基因的标志性基序,其对除SRY 外的所有Sox 基因都是保守的。目前,根据HMG-box序列的系统发生分析、蛋白结构特征及功能的不同,Sox 基因分属A–J共10个亚族(Schepers et al , 2002)。此外,青鳉中Sox 基因全基因组范围的分析将Olsox32 定义为K亚族(Cui et al , 2011)。广泛存在的Sox 基因在软骨形成、血细胞生成、神经系统发育、晶状体形成以及性别决定和分化等早期胚胎发育过程中发挥着重要的作用(常重杰等, 2002)。
鉴定基因家族的方法主要有分子生物学和生物信息学两种,随着越来越多物种的基因组测序和基因注释的完成,利用生物信息学方法对不同基因家族成员进行鉴定和分析,这对于筛选重要性状相关功能基因具有重要意义。目前,基于这种生物信息学分析的方法对感兴趣的基因家族进行分析已在完成基因组测序的动植物中应用(于雪等, 2014; 梁小娟等, 2015; 丁安明等, 2014),从而成为在全基因组层面鉴定和研究基因家族的重要手段。Sox 基因家族分析不仅有整体上的结构和胚胎发育时期的表达研究(Bowles et al , 2000; Schepers et al , 2002; Cui et al , 2011),同时也有基因家族成员或亚族在生理及生化层面上的功能分析(Barrionuevo et al , 2010; Azim et al , 2009; Hosking et al , 2009)。杨超(2013)1)进行罗非鱼Sox 基因家族分离鉴定及生物信息学分析,并综合分析了性腺转录组数据。实验不仅对Sox 基因在不同性别、不同性腺发育时期、关键阶段的表达模式和动态变化规律进行了全面分析,也为整个基因家族在性别决定和性腺发育过程中潜在作用的研究奠定了基础。而家蚕Sox 基因家族的鉴定和研究(魏玲, 2011)2)不仅揭示了BmsoxE 在家蚕生殖腺发育中的重要作用及调控机理,并且有助于家蚕性别决定与分化及生殖腺发育的分子机制研究。
半滑舌鳎不仅是重要的海水养殖鱼类,也是性别决定机制研究的理想鱼类模型,具有很高的经济价值和理论研究价值。而基因组测序的完成(Chen et al , 2014)也为半滑舌鳎Sox 基因家族的全基因组生物信息学分析奠定了基础。性别决定与分化一直是Sox 基因功能研究的经典领域,同时也是半滑舌鳎研究中亟待解析的分子机制之一。迄今,有关半滑舌鳎Sox 基因的研究主要有Sox10 的cDNA克隆和组织、胚胎与幼鱼的表达分析以及Sox9a 的克隆与表达分析等(Deng et al , 2008; Dong et al , 2011),尚未见有关半滑舌鳎Sox 基因家族的全基因组分析的研究报道。鉴于Sox 基因家族在半滑舌鳎早期发育及性别调控中的重要功能,运用生物信息学的方法在基因组中进行分离、鉴定及分析,可为Sox 基因在半滑舌鳎性别调控和胚胎发育中的功能研究提供参考。
1 材料与方法 1.1 基因组及功能注释信息从NCBI(http://www.ncbi.nlm.nih.gov/)下载半滑舌鳎基因组数据及基因注释文件并进行数据转化,用于构建本地BLAST基因组数据库。构建进化树的人(Homo sapiens )、斑马鱼(Danio rerio )、青鳉(Oryzias latipes )、罗非鱼(Oreochromis niloticus )、河豚(Takifugu rubripes )和三棘刺鱼(Gasterosteus aculeatus )的基因组和蛋白编码基因序列则来自Ensemble (http://asia.ensembl.org/index.html)。
1.2 Sox 蛋白序列鉴定以Sox 基因家族HMG-box一致序列(DHVKR PMNAFMVWSRGERRKIAQQNPDMHNSEISKRLGKRWKLLSESEKRPFIEEAERLRAQHMKDYPDYKYRPRRKKK)为种子序列(汪锐等, 2002),将其编译为Fasta格式并在半滑舌鳎本地BLAST基因组数据库中进行氨基酸的同源性比对,根据比对得到的结果筛选出Sox 基因氨基酸序列的候选片段。同时,利用保守域预测的在线软件Pfam (http://pfam.xfam.org/)和SMART (http://smart.embl-heidelberg.de/)确保上述候选氨基酸片段含有HMG-box保守域(Finn et al , 2015; Letunic et al , 2015),完成半滑舌鳎Sox 基因家族成员的分离鉴定。
1.3 蛋白保守结构域分析将种子序列与半滑舌鳎Sox 基因蛋白序列BLAST后得到的一段79 aa序列定义为Sox 基因蛋白保守结构域(HMG结构域),全部保守结构域序列经ClustalX软件比对后,利用GeneDoc软件实现比对结果的可视化。
1.4 基因结构分析及染色体定位为分析半滑舌鳎Sox 基因家族各个成员的基因结构,利用在线GSDS(Gene Structure Display Server) 2.0软件(http://gsds.cbi.pku.edu.cn/index.php)显示外显子、内含子等结构特征(Hu et al , 2015)。同时,实现基因家族成员间的结构比对。
结合半滑舌鳎基因组数据及Sox 基因编码信息对Sox 家族基因进行染色体定位。
1.5 基因进化分析分析Sox 基因家族成员的进化关系,进一步将半滑舌鳎Sox 基因家族成员与人、斑马鱼、青鳉、尼罗罗非鱼、河豚及三棘刺鱼等模式动物的Sox 基因构建系统发育树。使用ClustalX软件对Sox 基因氨基酸序列进行多序列联配,将联配的结果导入MEGA 6.06软件(Tamura et al , 2013)中,采用邻接树法(Neighbor-Joining)以MEGA软件构建进化树,设置Bootstrap为10000次重复检测其置信度。将进化树导入FigTree软件中优化,获取最终的系统进化树图。
1.6 基因表达分析半滑舌鳎各个时期及组织的转录组数据来源于NCBI(National Center for Biotechnology Information)的SRA数据库,主要分析雌雄鱼及伪雄鱼性腺组织和变态前后幼鱼的基因表达谱。半滑舌鳎发育过程中基因表达谱数据来源组织包括精巢(ZZ, SRX106096)、卵巢(ZW, SRX106097)、伪雄鱼性腺(ZW, SRX106098)、变态前幼鱼全身(ZW, SRX106100)及变态后幼鱼全身(ZW, SRX106101)。其中,精巢、卵巢和伪雄鱼性腺为1龄左右,精巢和伪雄鱼性腺较小,黑色,外覆一层白膜,成熟的精巢里有白色液体;卵巢较大,透明,外覆一层无色膜;幼鱼变态前后,形态差别较大。变态前幼鱼为左右对称状,浮游生活;而变态后幼鱼眼部发生偏转,呈左右不对称的扁平状,且开始底栖生活。参考Chen等(2014)的基因表达值计算方法,用RPKM(Reads Per Kilobase of exon model per Million mapped reads)表示基因表达值大小。利用R语言的pheatmap程辑包对半滑舌鳎Sox 基因在不同性腺组织及变态前后的基因表达数据进行数据聚类分析和作图。
2 结果与分析 2.1 半滑舌鳎Sox 基因家族成员的分离鉴定基于半滑舌鳎基因组数据,以HMG-box区种子序列为探针进行同源比对搜索,结合基因注释信息以及在线软件的保守结构域验证结果,本研究从半滑舌鳎基因组中共分离鉴定出23个Sox 基因。所有的Sox 基因家族成员分属7个亚族(表 1),分别为B1亚族CseSox2 、CseSox3 、CseSox19 )、B2亚族(CseSox14a 、CseSox14b 、CseSox21 )、C亚族(CseSox4a 、CseSox4b 、CseSox11 )、D亚族(CseSox5 、CseSox6a 、CseSox6b 、CseSox13 )、E亚族(CseSox8a 、CseSox8b 、CseSox9a 、CseSox9b 、CseSox10a 、CseSox10b )、F亚族(CseSox18 )和K亚族(CseSox32 )。
|
|
表 1 半滑舌鳎Sox 基因家族成员 Table 1 The members of Sox family in C. semilaevis |
通过对半滑舌鳎Sox 共79 aa的HMG结构域进行分析,结果显示,半滑舌鳎23个Sox 基因的HMG的结构域是高度保守的(图 1)。本研究分离到的半滑舌鳎Sox 基因除CseSox32 外均含有Sox 基因的保守识别基序(黑色线框标注区域),即满足HMG结构域第5–10个氨基酸残基(RPMNAF)的保守识别基序对除SRY 外的所有基因都保守的原则,而CseSox32 保守识别基序第3个为L。CseSox32 HMG结构域上第11–13个氨基酸残基为IIW,这也与其他Sox 基因家族成员高度保守的MVW氨基酸残基不同。此外,全部的23个Sox 基因在HMG结构域其他位置上也存在多个高度保守区域。

|
图 1 半滑舌鳎Sox 基因家族保守结构域分析 Figure 1 Conserved domains analysis of Sox gene family in C. semilaevis |
经基因结构分析,内含子和外显子结构分布显示,半滑舌鳎Sox 基因家族根据基因结构可以大体分为两大类。第一类为单外显子类,无内含子,主要为CseSox1b 和CseSox19 以外的B、C亚族的家族成员。第二类为多外显子类,这类基因按照结构又可细分为两种,一种是内含子不多于3个的CseSox1b 、CseSox19 及E、F和G亚族的家族成员;另一种是内含子数目显著多于3个且都多于10个的D亚族家族成员(图 2)。

|
图 2 半滑舌鳎Sox 基因家族基因结构示意 Figure 2 Gene structure of Sox genes in C. semilaevis |
结合基因组及编码信息获取半滑舌鳎Sox 基因家族相关染色体定位信息,结果显示,23个半滑舌鳎Sox 基因分布于15条染色体中,呈不均匀分布(表 1)。所有Sox 基因均定位到常染色体上,未在性染色体上发现Sox 基因家族成员的存在。其中,3、5、6、9、10、11、13、15、18和20号染色体上只存在1个Sox 基因,1、2号染色体上存在两个Sox 基因,而8、16和17号染色体则存在3个。
2.5 半滑舌鳎Sox 基因进化分析选取不同物种Sox 基因编码的蛋白序列,利用MEGA 6.06软件,采用邻接法构建系统发育树(图 3)。结果显示,半滑舌鳎Sox 基因与人、斑马鱼、青鳉、尼罗罗非鱼、河豚和三棘刺鱼一起被划分至7个主要的亚族(B-K),其具体的亚族分类保持着高度的一致性。
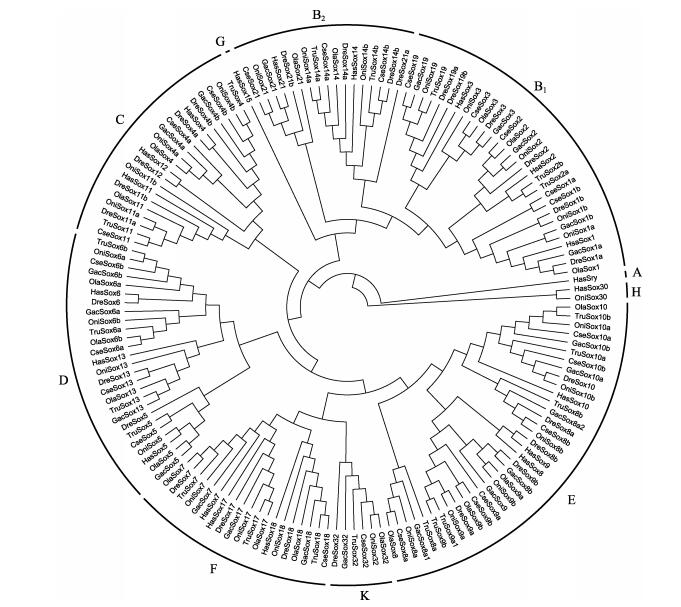
|
图 3 半滑舌鳎、人、斑马鱼、青鳉、尼罗罗非鱼、河豚、三棘刺鱼Sox 家族进化树 Figure 3 Phylogenetic tree of Cynoglossus semilaevis, Homo sapiens , Danio rerio, Oryzias latipes, Oreochromis niloticus, Takifugu rubripes and Gasterosteus aculeatus Sox family Cse:半滑舌鳎; Has:人; Dre:斑马鱼; Ola:青鳉; Oni:罗非鱼; Tru:河豚; Gac:三棘刺鱼 Cse: Cynoglossus semilaevis ; Has: Homo sapiens ; Dre: Danio rerio ; Ola: Oryzias latipes ; Oni: Oreochromis niloticus ; Tru: Takifugu rubripes ; Gac: Gasterosteus aculeatus |
利用R语言的pheatmap程辑包对半滑舌鳎Sox 家族基因表达模式进行分析,绿-黑-红3个颜色表示基因的相对表达强度逐渐增大。本研究对Sox 基因家族在精巢、卵巢、伪雄鱼性腺以及幼鱼变态前后的表达数据进行聚类分析(图 4),结果显示,半滑舌鳎的Sox 基因主要被划分为两大类。一类在幼鱼变态前后基因有较高的表达,如图 4中聚类的前15个Sox 基因,其中,CseSox4a 、CseSox4b 、CseSox9a 及CseSox9b 等基因变态前后表达差异显著,CseSox4 基因变态前基因表达高于变态后,而CseSox9 和CseSox9b 则相反。另一类则在幼鱼变态前后表达较低,但在性腺中却有较高的表达,且在不同种类的性腺中具有表达差异存在,这类的基因主要有CseSox1a 、CseSox14b 、CseSox32 、CseSox18 及CseSox13 等。
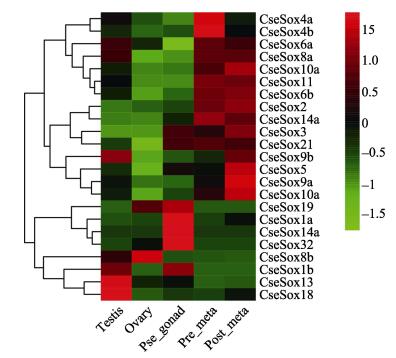
|
图 4 半滑舌鳎Sox 基因表达模式 Figure 4 The expression profiles of C. semilaevis Sox genes Testis:精巢; Ovary:卵巢; Pse_gonad:伪雄鱼性腺; Pre_meta:幼鱼变态前; Post_meta:幼鱼变态后 Pse_gonad: gonad of Pseudo-male; Pre_meta: pre-metamorphosis; Post_meta: post-metamorphosis |
基于完成测序的基因组数据,从全基因组层面对基因家族进行分离鉴定、分类、序列特征、结构及进化等方面的分析已成为生物学研究领域的热点和要点。Sox 基因家族在动物特别是脊椎动物体内广泛存在,且参与多个发育过程,而Sox 基因资源的挖掘也为其进一步的功能研究提供了参考借鉴。迄今为止,通过分子克隆或基因组序列筛选等方法,在线虫、果绳等无脊椎动物中共鉴定出约10个Sox 基因,而在脊椎动物中己经鉴定出大约40个左右Sox 基因(Koopman et al , 2004)。本研究从目前公开的半滑舌鳎基因组中,使用生物信息学的方法一共鉴定出23个半滑舌鳎Sox 基因。由于全基因组序列组装过程中存在可能的gaps(Cui et al , 2011),使得这一数目只是初步研究的结果,随着Sox 基因的克隆和基因组的组装、注释工作的不断完善或有增减。
与其他硬骨鱼类相似,半滑舌鳎的基因家族成员被划分至B–K共7个亚族。蛋白结构域及基因结构分析发现,其HMG结构域在种间及种内绝大部分成员中都是高度保守的,而半滑舌鳎CseSox32 基因的保守结构域虽然与其他家族成员有着一定的差异性,但与其他物种的K亚族基因却保持着高度的一致性(杨超, 2013)1)。D亚族基因相较于其他亚族含有更多内含子,使其基因结构具有显著特点,这可能是由于D亚族有着特殊的进化历程所导致的。基因结构示意图显示(图 2),CseSox13 基因跨越长度接近12 kb,而CseSox5 、CseSox6a 及CseSox6b 基因的跨越长度更是超过50 kb。与B、C亚族内的单外显子基因相比,D亚族基因在进化过程中趋于保守使大量内含子得以保留,而那些单外显子基因则在进化过程中发生内含子丢失现象(Roose et al , 1999)。此外,半滑舌鳎Sox 基因染色体定位发现,Sox 基因在染色体上的分布是散乱无规律的,没有在特定染色体上发生优先主导现象,同时表明其没有出现基因簇,这一结果与其他物种上Sox 基因家族的染色体分布情况相似(Pevny et al , 1997)。
半滑舌鳎Sox 基因系统进化分析表明,Sox 基因不同亚族之间在进化方向上表现出了差异性,这也预示着其基因功能的多样性。多物种联合构建的系统发育树显示,不同物种间及种内的Sox 基因在大体上呈现出进化的趋同性,但在不同物种中的Sox 基因亚族类型及数目还是存在差异的。在6种硬骨鱼类中都存在Sox32 基因,而人类中却不存在K亚族的Sox 基因,但A亚族的Sry 和G亚族的Sox15 基因却是这7种物种中人所独有的。无脊椎动物Sox 基因家族虽然成员数目较少,但其亚族分类与脊椎动物Sox 基因的亚族分类却是一致的(Crémazy et al , 2000)。由此推测,在整个动物进化过程中的某一时刻,Sox 基因家族亦随着动物的进化出现爆发性的拓群现象。在随后的一段稳定期内,HMG基序的序列可能发生重复,或者出现某一个多余的拷贝并进化为一个新的基因,又或者从基因组中消失(Force et al , 1999)。半滑舌鳎Sox 基因表达信息分析结果显示,Sox 基因家族在半滑舌鳎性腺发育及早期幼鱼变态过程中具有一定的特异性。Dong等(2011)对半滑舌鳎Sox9a 基因及表达的克隆分析发现,CseSox9a 功能与性反转、性别分化、胚胎发育中细胞分化及生精细胞形成相关联。Kobayashi等(2008)研究表明,尼罗罗非鱼Sox9a 基因在性腺分化及性反转过程中亦发挥重要作用。使用促性腺激素释放激素类似物(GnRHa)处理黑鲷导致Sox3 及DRMT1 的表达上调,表明Sox3 和DMRT1 参与了黑鲷精巢发育过程(Shin et al , 2009)。此外,在小鼠性别决定过程中,利用双敲除的方法验证了Sox8 和Sox9 基因在精巢分化中起着关键性作用,且Sox8 对Sox9 表达具有增强作用(Chaboissier et al , 2004)。上述研究表明,Sox 基因家族在性别决定及性腺分化的过程中可能发挥重要作用,而其在半滑舌鳎中的具体功能还有待进一步的实验验证。
4 结论综上所述,本研究首次运用生物信息学方法对半滑舌鳎Sox 基因家族进行了全基因组层面的鉴定、保守结构域分析、染色体定位、基因结构分析、进化及表达分析。结果不仅表明了半滑舌鳎Sox 基因家族在性别决定及早期发育过程中可能发挥着重要的作用,也为其进一步的基因功能验证及日益丰富的水产基因组资源的挖掘利用提供参考。
| 丁安明, 李凌, 屈旭, 等. 番茄PPR基因家族的鉴定与生物信息学分析. 遗传 , 2014, 36 (1) : 77-84 | |
| 于雪, 于红, 孔令锋, 等. 太平洋牡蛎酪氨酸酶基因家族的系统发生分析. 遗传 , 2014, 36 (2) : 135-144 | |
| 汪锐, 程汉华, 郭一清, 等. 脊椎动物Sox基因家族的系统发生分析. 遗传学报 , 2002, 29 (11) : 990-994 | |
| 常重杰, 杜启艳, 邵红伟. Sox基因家族研究的新进展. 遗传 , 2002, 24 (4) : 470-476 | |
| 梁小娟, 王志萌, 孟琼, 等. 猪14-3-3蛋白家族基因生物信息学分析. 畜牧与兽医 , 2015, 47 (5) : 31-37 | |
| Azim E, Jabaudon D, Fame RM, et al. SOX6 controls dorsal progenitor identity and interneuron diversity during neocortical development. Nat Neurosci , 2009, 12 (10) : 1238-1247 DOI:10.1038/nn.2387 | |
| Barrionuevo F, Scherer G. SOXE genes: SOX9 and SOX8 in mammalian testis development. Int J Biochem Cell B , 2010, 42 (3) : 433-436 DOI:10.1016/j.biocel.2009.07.015 | |
| Bowles J, Schepers G, Koopman P. Phylogeny of the SOX family of developmental transcription factors based on sequence and structural indicators. Dev Biol , 2000, 227 (2) : 239-255 DOI:10.1006/dbio.2000.9883 | |
| Chaboissier MC, Kobayashi A, Vidal VI, et al. Functional analysis of Sox8 and Sox9 during sex determination in the mouse. Development , 2004, 131 (9) : 1891-1901 DOI:10.1242/dev.01087 | |
| Chardard D, Chesnel A, Gozé C, et al. Pw Sox-1: the first member of the Sox gene family in Urodeles. Nucleic Acids Res , 1993, 21 (15) : 35-76 | |
| Chen SL, Zhang GJ, Shao CW, et al. Whole-genome sequence of a flatfish provides insights into ZW sex chromosome evolution and adaptation to a benthic lifestyle. Nature Genet , 2014, 46 (3) : 253-260 DOI:10.1038/ng.2890 | |
| Crémazy F, Berta P, Girard F. Sox neuro, a new Drosophila Sox gene expressed in the developing central nervous system. Mech Develop , 2000, 93 (1-2) : 215-219 DOI:10.1016/S0925-4773(00)00268-9 | |
| Cui J, Shen X, Zhao H, et al. Genome-wide analysis of Sox genes in Medaka (Oryzias latipes ) and their expression pattern in embryonic development. Cytogenet Genome Res , 2011, 134 (4) : 283-294 DOI:10.1159/000329480 | |
| Deng SP, Chen SL. cDNA cloning, tissues, embryos and larvae expression analysis of Sox10 in half-smooth tongue-sole, Cynoglossus semilaevis. Mar Genom , 2008, 1 (3-4) : 109-114 DOI:10.1016/j.margen.2008.10.003 | |
| Dong XL, Chen SL, Ji XS, et al. Molecular cloning, characterization and expression analysis of Sox9a and Foxl2 genes in half-smooth tongue sole (Cynoglossus semilaevis). Acta Oceanol Sin , 2011, 30 (1) : 68-77 DOI:10.1007/s13131-011-0092-9 | |
| Finn RD, Coggill P, Eberhardt RY, et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Res , 2016, 44 (7) : 279-285 | |
| Force A, Lynch M, Pickett FB, et al. Preservation of duplicate genes by complementary, degenerative mutations. Genetics , 1999, 151 (4) : 1531-1545 | |
| Gubbay J, Collignon J, Koopman P, et al. A gene mapping to the sex-determining region of the mouse Y chromosome is a member of a novel family of embryonically expressed genes. Nature , 1990, 346 (6281) : 245-250 DOI:10.1038/346245a0 | |
| Hosking B, Fran ois M, Wilhelm D, et al. Sox7 and Sox17 are strain-specific modifiers of the lymphangiogenic defects caused by Sox18 dysfunction in mice. Development , 2009, 136 (14) : 2385-2391 DOI:10.1242/dev.034827 | |
| Hu B, Jin J, Guo AY, et al. GSDS 2.0: an upgraded gene feature visualization server. Bioinformatics , 2015, 31 (8) : 1296-1297 DOI:10.1093/bioinformatics/btu817 | |
| Kobayashi T, Kajiura-Kobayashi H, Guan GJ, et al. Sexual dimorphic expression of DMRT1 and Sox9a during gonadal differentiation and hormone-induced sex reversal in the teleost fish Nile tilapia (Oreochromis niloticus). Dev Dynam , 2008, 237 (1) : 297-306 DOI:10.1002/(ISSN)1097-0177 | |
| Koopman P, Schepers G, Brenner S, et al. Origin and diversity of the SOX transcription factor gene family: genome-wide analysis in Fugu rubripes. Gene , 2004, 328 (3) : 177-186 | |
| Laudet V, Stehelin D, Clevers H. Ancestry and diversity of the HMG box superfamily. Nucleic Acids Res , 1993, 21 (10) : 2493-2501 DOI:10.1093/nar/21.10.2493 | |
| Letunic I, Doerks T, Bork P. SMART: recent updates, new developments and status in 2015. Nucleic Acids Res , 2015, 43 (7) : 257-260 | |
| Pevny LH, Lovell-Badge R. Sox genes find their feet. Curr Opin Genet Dev , 1997, 7 (3) : 338-344 DOI:10.1016/S0959-437X(97)80147-5 | |
| Roose J, Korver W, Boer R, et al. The Sox-13 gene: structure, promoter characterization, and chromosomal localization. Genomics , 1999, 57 (2) : 301-305 DOI:10.1006/geno.1999.5779 | |
| Schepers GE, Teasdale RD, Koopman P. Twenty pairs of sox: extent, homology, and nomenclature of the mouse and human sox transcription factor gene families. Dev Cell , 2002, 3 (2) : 167-170 DOI:10.1016/S1534-5807(02)00223-X | |
| Shin HS, An KW, Park MS, et al. Quantitative mRNA expression of sox3 and DMRT1 during sex reversal, and expression profiles after GnRHa administration in black porgy, Acanthopagrus schlegeli. Comp Biochem Phys B Biochem Mol Biol , 2009, 154 (1) : 150-156 DOI:10.1016/j.cbpb.2009.05.013 | |
| Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0. Mol Biol Evol , 2013, 30 (12) : 2725-2729 DOI:10.1093/molbev/mst197 |



