2. 上海海洋大学 上海 201306;
3. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. Shanghai Ocean University, Shanghai 201306 ;
3. Function Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071
黄头病毒(Yellow Head Virus,YHV)属于单链RNA病毒的套式病毒目、杆套病毒科、头甲病毒属(Cowley et al,2012)。黄头病毒群分为黄头病毒(YHV-1型)、鳃联病毒(Gill-associated virus,GAV)(YHV-2型)及其他至少4种基因型(YHV-3型-YHV-6型)(Wijegoonawardane et al,2009)。
2010年起,越南、马来西亚、泰国和我国海南等地养殖的凡纳滨对虾(Litopenaeus vannamei)相继出现不明原因的疫病,并导致较高的死亡率。目前,已经确定对虾早期死亡综合征能感染凡纳滨对虾、斑节对虾(Penaeus monodon)、中国明对虾(Fenneropenaeus chinensis)等。Tran等(2013)证实,一种高毒力的副溶血弧菌是导致早期死亡综合征(EMS)或急性肝胰腺坏死病(AHPND)的病原。2012年在疑似EMS的中国明对虾样品中用PCR方法检测出了YHV(刘群等,2014)。随后,3年持续的流行病学监测表明,我国沿海对虾养殖省市都存在YHV感染,通过ORF1、ORF3部分序列的比对发现,我国对虾中检出的YHV与现有YHV、GAV的基因型不同。这为我国对虾新发疫病的病原学研究提供了重要信息,该结果也表明我国养殖对虾正面临着新的病害威胁。
研究者已报道了世界各地的患病对虾中YHV的6个基因型。根据刘群等(2014)发表的部分基因序列,将此YHV的基因型编号为YHV-9。我国各地检出的YHV与这些已知基因型的YHV在进化上的亲缘关系如何,感染的对虾种类和地区分布均尚无报道。本研究在流行病学调查及YHV-9基因测序的基础上,对我国不同地区YHV样品ORF1、ORF2和ORF3序列与已知YHV株系的同源性进行分析,以期阐明我国流行的YHV株系与已知YHV株系在进化上的关系,丰富对YHV基因组变异和进化的认识,为YHV的有效防治提供基础理论知识。
1 材料与方法 1.1 样品采集与保存2012-2014年从天津、河北、山东和浙江等地的对虾养殖场采集或送检的凡纳滨对虾、中国明对虾、罗氏沼虾(Macrobrachium rosenbergii)、日本囊对虾(Marsupenaeus japonicus)等样品共计299份(表 1)。用于核酸检测的样品采用RNALater保存或冻存于-80℃冰箱,用于组织病理学检查的样品采用RNA Friendly固定液(Hasson et al,1997)和Davidson’s AFA固定液,固定方法参考Lightner(1996)。
|
|
表 1 样品信息 Table 1 The information of samples |
取RNALater保存的或冰冻虾肝胰腺和鳃丝10- 50 mg,加入800 μl TRIzol试剂(Invitrogen)。用RNase- free研磨棒将样品匀浆,加入160 μl氯仿,振荡混匀后室温静置15 min,然后12000 r/min 4℃离心15 min,取上层水相,加入等体积异丙醇,混匀后放置5 min,12000 r/min 4℃离心10 min,弃上清液,于沉淀中加入1 ml冰冷75%乙醇,轻轻混匀,静置5 min,12000 r/min 4℃离心10 min,沉淀晾干,加入50 μl RNase-free水溶解,经核酸蛋白测定仪(NanoDrop 2000,美国)测定其浓度与纯度,置于-80℃保存。
1.3 YHV的套式RT-PCR检测 1.3.1 cDNA的合成根据世界动物卫生组织(OIE)《水生动物诊断手册》(OIE,2012)推荐的YHV套式RT-PCR检测方法对样品进行检测。取2 μl样品模板,加入50 μmol/L引物GY1和GY5(表 2)各1.0 μl、0.5 μl 10 mmol/L dNTP和2.5 μl无RNase水,混匀,70℃反应5 min后,置于冰上2 min,向体系内加入2 μl 5×MMLV Buffer、0.5 μl 40 U/μl RNase inhibitor(TaKaRa)和0.5 μl 200 U/L MMLV反转录酶(TaKaRa),42℃保温60 min,72℃反应10 min,以此合成cDNA作为模板。
|
|
表 2 PCR扩增引物序列 Table 2 Sequences of primers used in PCR amplification |
第一步PCR用25 μl反应体系,含2.5 μl 10× Ex Taq PCR Buffer(Mg2+-free,TaKaRa)、1.5 μl 25 mmol/L MgCl2、0.5 μl 10 mmol/L dNTP、50 μmol/L引物GY1和GY4各0.35 μl(表 2)、0.25 μl 5 U/μl Ex Taq DNA 聚合酶(TaKaRa),85℃预热3 min,加入1.0 μl cDNA,18.55 μl ddH2O。扩增反应程序为:95℃ 30 s,66℃ 30 s,72℃ 45 s,35个循环;72℃ 7 min;4℃保存。
1.3.3 第二步PCR反应第二步PCR采用25 μl反应体系,含2.5 μl 10×Ex Taq PCR Buffer(Mg2+-free,TaKaRa)、1.5 μl 25 mmol/L MgCl2、0.5 μl 10 mmol/L dNTP、50 μmol/L引物GY2和Y3各0.35 μl(表 2)、0.25 μl 5 U/μl Ex Taq DNA聚合酶(TaKaRa),85℃预热3 min,加入1.0 μl第一步PCR反应产物。扩增反应程序为:95℃ 30 s,66℃ 30 s,72℃ 30 s,35个循环;72℃ 7 min;4℃保存。反应产物于1%琼脂糖凝胶电泳,并采用凝胶成像仪(培清JS-380)拍照。
1.4 ORF1b部分基因片段的扩增根据Wijegoonawardan等(2008)和已测序YHV序列(编号20120706,未发表)设计ORF 1b扩增引物(表 2)。
1.4.1 cDNA的合成所用反转录的引物为10 μmol/L1b-F1/R1(表 2),其余操作方法与1.3.1相同。
1.4.2 第一步PCR反应第一步PCR反应采用25 μl反应体系,含2.5 μl 10×LA Taq PCR Buffer(Mg2+-free,TaKaRa)、2.5 μl 25 mmol/L MgCl2、4.0 μl 10 mmol/L dNTP、10 μmol/L引物1b-F1/R1各0.5 μl(表 2)、0.25 μl 25 U/μl LA Taq DNA聚合酶,1.0 μl cDNA,13.75 μl ddH2O。扩增反应程序为:94℃ 3 min;94℃ 30 s、46℃ 30 s、72℃ 100 s,30个循环;72℃ 7 min;4℃保存。
1.4.3 第二步PCR反应第二步PCR反应体系与第一步相同,取1.0 μl第一步PCR产物作为模板,引物为1b-F2/R2(表 2)(10 μmol/L)。扩增反应程序为:94℃ 3 min;94℃ 30 s、58℃ 30 s、72℃ 60 s,35个循环;72℃ 7 min,4℃保存。反应产物于1%琼脂糖凝胶电泳,并采用凝胶成像仪拍照。
1.5 ORF3部分基因片段的扩增根据Gangnonngiw等(2009)和Senapin等(2010)以及已测序YHV-9(20120706)序列(未发表)设计ORF3扩增引物(表 2)。
1.5.1 cDNA合成方法参照1.3.1,引物为116-F1/R1(表 2)。
1.5.2 套式RT-PCRPCR反应体系参照1.4.2,第一步扩增反应中,引物为116-F1/R1(表 2),扩增反应退火温度为55℃,其余扩增反应参数与1.4.2中反应程序相同。第二步扩增反应中,引物为116-F2/R2(表 2),退火温度为50℃,扩增35个循环,其余扩增反应参数与1.4.3相同。反应产物于1%琼脂糖凝胶电泳,并采用凝胶成像仪拍照。
1.6 ORF 2片段的扩增根据Wijegoonawardan等(2008)和已测序YHV-9(20120706)序列(未发表)设计引物(表 2)。
cDNA合成参照1.3.1,引物为p20-S/A(表 2)。PCR反应体系与1.4.2相同扩增反应,退火温度为60℃,扩增35个循环,其余扩增反应参数与1.4.3相同。产物经1.5%琼脂糖凝胶电泳。
1.7 DNA的回收和连接转化采用胶回收试剂盒(QIAGEN)回收电泳后的PCR扩增目的产物片段,按照试剂盒说明书操作,加10 μl无菌水,溶解后离心,取2 μl胶回收产物于1%琼脂糖凝胶电泳,取条带明亮、单一的样品进行连接转化。NanoDrop 2000测定浓度,取pMD18-T载体(TaKaRa)1 μl,胶回收产物(0.1-0.3 pmol)4 μl,SolutionⅠ 5 μl,16℃连接30 min,置于冰上,加入50 μl E coli DH5α感受态细胞,冰浴转化30 min,于42℃热激45 s,置于冰上2 min,接入LB液体培养基于37℃摇床培养。
1.8 挑取单克隆菌落及菌落PCR摇菌1 h后,取50 μl菌液涂布于含氨苄青霉素(Amp)的LB平板,倒置培养16 h后,挑取单克隆,每个样品挑选10个平行于含Amp的LB液体培养基37℃培养,2 h后取2 μl作为模板进行菌落PCR。体系与1.3.2相同,加入10 μmol/L通用引物M13-47和RV-M各0.5 μl,反应程序为:94℃ 10 min;94℃ 30 s、55℃ 30 s、72℃ 60 s,30个循环;72℃ 7 min。反应产物于1%琼脂糖凝胶电泳,挑选3份平行的单克隆样品至上海生工生物工程有限公司测序。
1.9 序列比对和系统发育分析采用Bioedit软件对核酸序列进行拼接、比对,采用Geneious软件进行多序列比对分析,并构建NJ系统发育树。
2 结果 2.1 阳性样品RT-PCR检测结果299份样品两步法共检出阳性样品33个,有6份样品第一步RT-PCR产物电泳就能出现明亮的794 bp的目的条带(图 1-a,b),其中,两份来自山东的中国明对虾样品阳性强,其他4份样品阳性较弱。6份样品编号分别为20130301-ZJ(广东,凡纳滨对虾)、20130902-JX(浙江,罗氏沼虾)、20120706-TH(河北,中国明对虾)、20140707-JM(山东,中国明对虾)、20131018-JM(山东,中国明对虾)和20120628-HG(天津,凡纳滨对虾)。

|
图 1 YHV套式RT-PCR检测结果 Figure 1 Results of nested-RT-PCR of YHV a: 样品第一步PCR结果Result of first-step PCR; b: 样品第二步PCR结果Result of second-step PCR M: Marker; B: 空白对照Blank control; P: 阳性对照Positive control; 1: 20130301-ZJ; 2: 20130902-JX; 3: 20120706-TH; 4: 20140707-JM; 5: 20121018-JM; 6: 20120628-HG |
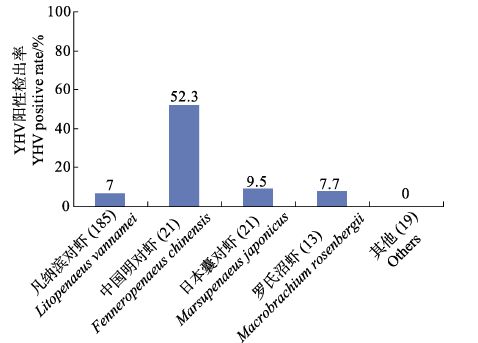
对来自辽宁、河北、天津、山东、江苏、浙江、福建、广东、广西、海南等省市的凡纳滨对虾、中国明对虾、日本囊对虾、罗氏沼虾、斑节对虾、脊尾白虾、克氏原螯虾、梭子蟹、卤虫等样品进行了套式RT-PCR检测。结果显示,在凡纳滨对虾、中国明对虾、日本囊对虾和罗氏沼虾部分样品中检测到YHV阳性,其中,中国明对虾的阳性检出率最高,21份样品中有11份为阳性,185分凡纳滨对虾中有13份为阳性(图 2)。在2.1中的6份第一步RT-PCR样品中,有3份是中国明对虾,提示YHV可能对我国中国明对虾养殖存在严重风险。
|
图 2 YHV在不同宿主样品中的检出情况(2013.03-2014.12) Figure 2 Detection of YHV in different hosts (March 2013-December 2014) 从阳性样品的分布地区来看,我国沿海省市均检出括号内为检测样品总数,其他包括斑节对虾、脊尾白虾、克氏原螯虾、梭子蟹、卤虫等 The number in parentheses is the total number of samples. “Others” includes Penaeus monodon,Palaemon carinicauda,Procambarus clarkia,Portunidae and Artemia sp. etc |
YHV阳性样品,新疆也检出YHV阳性样品,说明YHV在我国已经广泛蔓延(表 3)。YHV虽然在我国多个省市检出,但作为主要养殖品种的凡纳滨对虾检出率低于平均水平且样品多采集健康状态不佳的对虾,其他品种受采样数量等因素的影响,实际检出率可能更低。
|
|
表 3 不同省市的样品中YHV检出情况(2012.05-2014.12) Table 3 Detection of YHV in samples from different provinces and cities(May 2012-December 2014) |
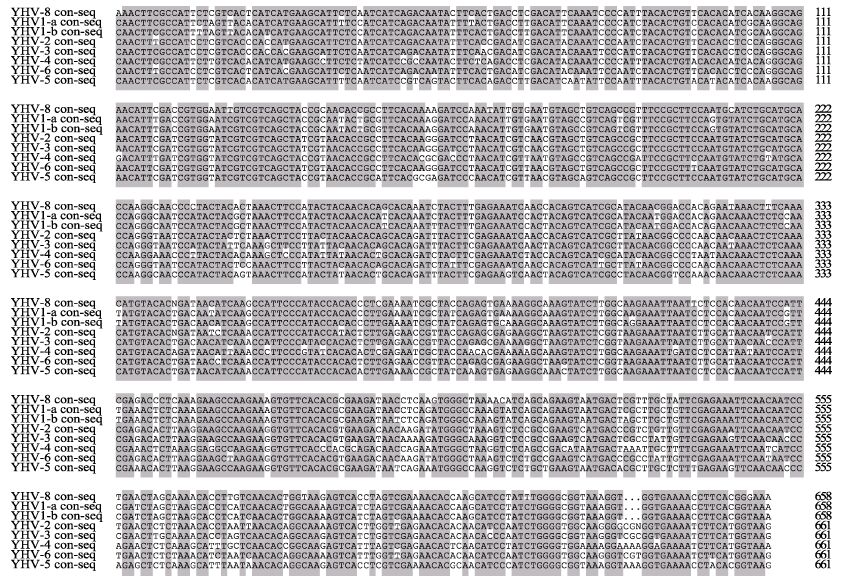
对上述6份阳性样品ORF1b部分片段第二步扩增所得的1002 bp产物进行测序。其序列与YHV已知6个基因型24份样品的ORF1b片段进行多重序列比对,结果显示,6份样品的ORF1b序列长度与YHV-1的相同,比YHV-2-YHV-6少3 bp。6份样品间差异很小,相似度为97.6%-98.5%。6份样品共有序列与YHV-1-YHV-6型共有序列相似度为90.5%-80.9%,与YHV-1相似度较高(图 3)。
|
图 3 ORF1b片段共有序列比对 Figure 3 Alignment of ORF1b fragment consensus sequence |
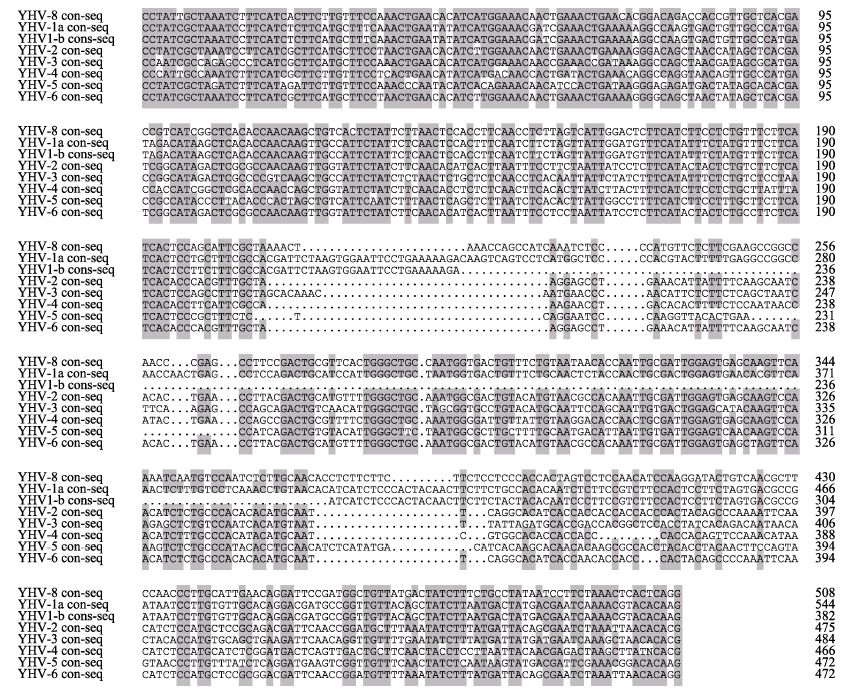
6份样品ORF3片段套式PCR扩增目的条带经测序显示,产物大小为509 bp,与YHV-1a 545 bp、YHV-1b 383 bp、YHV-2 476 bp均不同。序列与YHV已知6个基因型20份样品进行多重序列比对显示(图 4),6份样品间相似度为97.2%-99.4%。6份样品共有序列与YHV-1a、YHV-1b、YHV-2、YHV-3、YHV-4、YHV-5和YHV-6型共有序列的相似度分别为67.9%、63.2%、62.5%、60.8%、62.8%、62.5%和62.1%,相似度均较小。
|
图 4 ORF3片段共有序列比对 Figure 4 Alignment of ORF3 fragment consensus sequence |
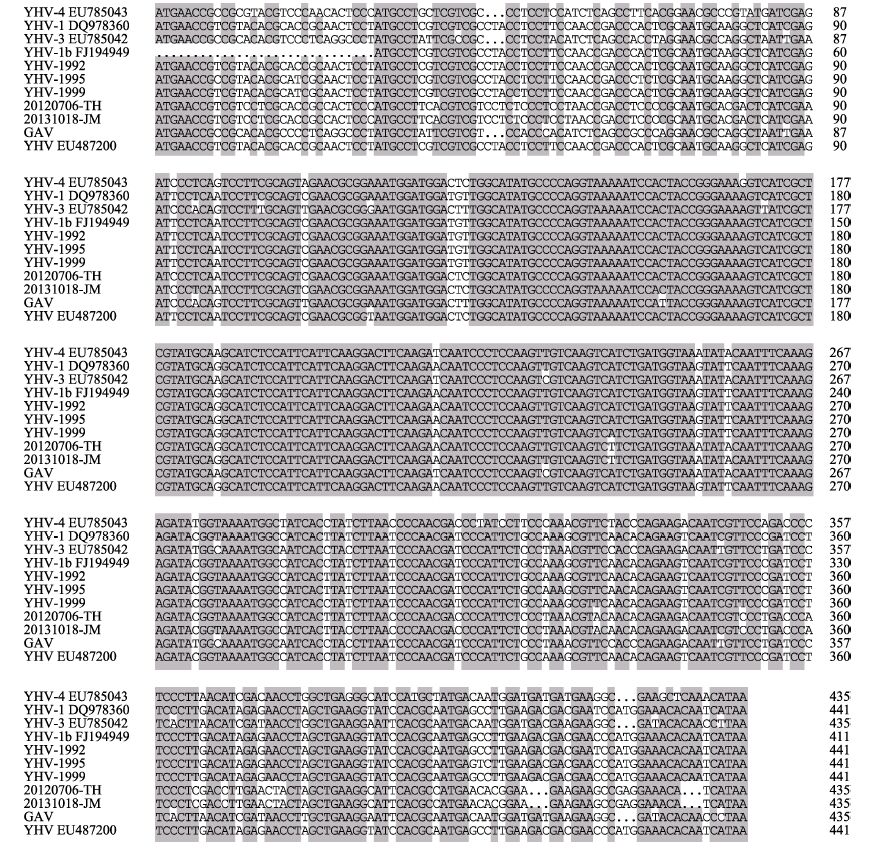
两份样品ORF2的套式PCR扩增目的条带经测序得到445 bp产物序列,共编码144 aa,与YHV- 2-YHV-5型长度相同,比YHV-1型(146 aa)短。多重序列比对结果显示(图 5),两份样品序列相似性为99.8%,蛋白序列完全相同,与YHV-1-YHV-4共有序列相似度分别为85.9%、80.7%、80.6%、82.4%,与YHV-1相似度较高。
|
图 5 ORF2序列比对 Figure 5 Alignment of ORF2 consensus sequence |
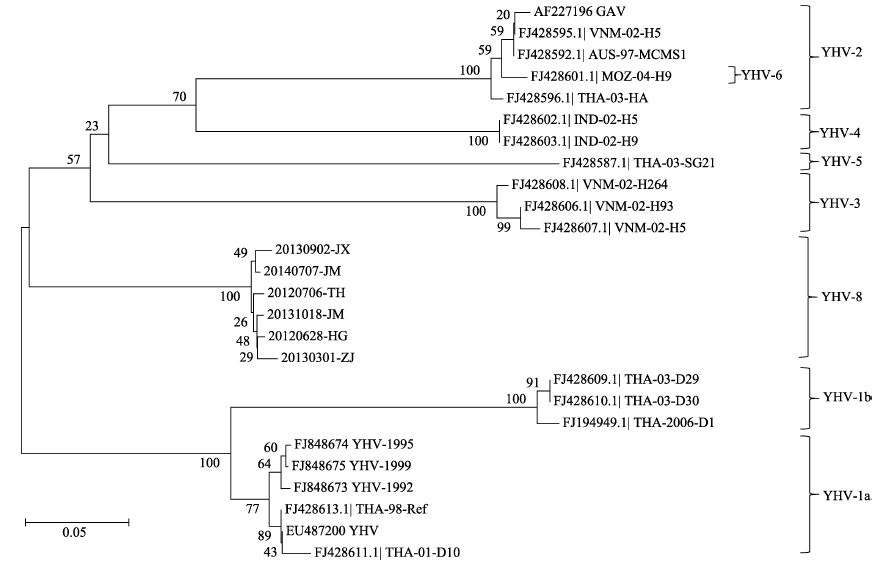
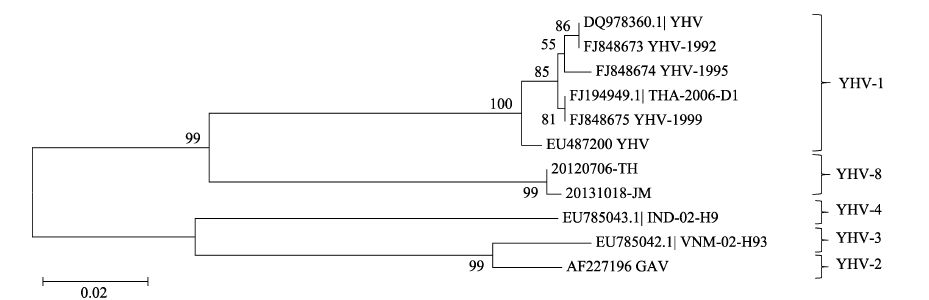
采用Geneious(v4.8.5)软件对ORF1b、ORF3部分片段和ORF2片段序列与NCBI中部分已知序列构建系统发育树(图 6-图 8)。

|
图 6 基于YHV ORF1b片段序列多重比对后构建的系统发育树 Figure 6 Phylogenetic tree based on the sequences of ORF1b fragment multiple sequence alignment |
|
图 7 基于ORF3片段序列多重比对后构建的系统发育树 Figure 7 Phylogenetic tree based on the sequences of ORF3 fragment multiple sequence alignment |
|
图 8 基于ORF2序列多重比对后构建系统发育树 Figure 8 Phylogenetic tree based on the sequences of ORF2 fragment multiple sequence alignment |
ORF1b部分片段的系统发育树显示(图 6),6份样品处于同一分支,都属于YHV-9的基因型,该分支的各样品在年代和地域上没有明显聚合规律。YHV-9与YHV-1聚为一个较大的分支,与YHV其他5个基因型分支较远;YHV-2和YHV-6亲缘关系较近,先聚为同一支,再与YHV-3聚合;前述这5个基因型再聚为一个大簇,最后与YHV-4及YHV-5汇聚。该基因片段无法有效对YHV-1a和YHV-1b进行区分。
ORF3部分片段的系统发育树分析显示(图 7),6份样品相似性高,与20120706TH一起均属于YHV-9,处于同一分支;YHV-2与YHV-4先聚为一支,再依次与YHV-5、YHV-3、YHV-9、YHV-1等各大分支聚合。YHV-9与YHV已知6个基因型分支距离较为均等。YHV-1a和YHV-1b则在分支上被清晰地区分出来。
ORF2片段只有20120706-TH和20131018-JM这两份样品得到有效扩增和测序,序列位于YHV-9的同一分支,该分支再与YHV-1聚于同一大的分支;而在该片段上,YHV-2和YHV-3的亲缘关系很近,这两个基因型再与YHV-4汇聚为一大分支;表现出YHV-1、YHV-9和YHV-2、YHV-3、YHV-4两大分支的结构(图 8)。由于报道序列较少,YHV-5和YHV-6均未能在该片段的系统发育树上绘出。
3个片段进化树的分析显示,YHV-9与YHV-1的进化关系较近,ORF1b和ORF2片段的进化关系比ORF3片段的进化关系近,说明YHV中国株虽然与YHV-1进化关系较近,但不同片段的进化关系仍存在显著差别。
3 讨论黄头病是养殖对虾的主要病害之一,是OIE收录的甲壳动物疫病,我国将其列为二类动物疫病。黄头病毒(YHV)最早在泰国被发现(Limsuwan,1991),澳大利亚于1995年在养殖对虾内发现另一致病型YHV(Spann et al,1995)。我国台湾地区很早就在养殖的斑节对虾、日本囊对虾中检测到YHV的存在,但为非致病型(Wang et al,1996)。我国于2005年从泰国进口的斑节对虾内检测到YHV,但我们国内养殖对虾中未曾检测到YHV阳性(熊炜等,2006)。2012年,在疑似发生EMS养殖场的中国明对虾内检测到YHV阳性(刘群等,2014)。由于我国长期从国外引进亲虾,东南亚及澳洲地区作为YHV的主要疫病区和水产品出口地,我国通过水产品进口或引进携带YHV亲虾并垂直传播的可能性较高。本研究对2012-2014年间采集自国内的299份对虾样品进行了YHV检测,套式RT-PCR检出32份YHV阳性样品,其中,6份样品在第一步RT-PCR检出阳性。
前人研究表明,YHV病毒包括至少6种基因型YHV,其中,YHV-1是对虾的主要病原,GAV是YHV-2,与其他基因型(YHV-3-YHV-6)多存在于东南亚、澳洲及东非等地养殖的健康斑节对虾体内,很少引起疫病(Walker et al,2001; Wijegoonawardane et al,2008、2009)。根据2012年在我国发现的YHV的RT-PCR检测的目的片段的序列,推测我国新发现的YHV是一个新的基因型(刘群等,2014)。本研究对我国更多地区采集的样品的YHV的检测和YHV基因组的ORF1b、ORF3和ORF2片段的序列测定和比对进一步确证了这一推测,这一新的基因型在其基因组的ORF1b、ORF3和ORF2区域与YHV的已知基因型均表现出了遗传差异,均属于YHV的同一个亚型。为避免在今后的研究中出现亚型命名混乱的问题,本研究仍采用了澳大利亚对我国该亚型的命名方式,称为YHV-9。本研究检出的6份YHV-9阳性样品在不同片段上也存在亚型内的差异,由于YHV为RNA病毒,依赖于RNA的RNA聚合酶缺乏3'→5'的外切活性,无法修复复制中的错误,导致RNA复制过程中变异较大,YHV核酸序列的差异变化符合RNA病毒变异较快的特点。
在对多亚型间和亚型内的各片段序列比对中可以看出,YHV基因组的ORF3序列表现出较高的亚型间的变异性和亚型内的稳定性,能更好地用于YHV亚型的分型;而ORF2序列表现出较高的亚型间的相似性,适用于对各种YHV变异型的检出。由于这些序列的差异,OIE《水生动物诊断手册》中的YHV检测的RT-PCR检测法对YHV-9型的检测灵敏度可能有一定影响,表明还需根据YHV全基因序列的比对结果,设计新的RT-PCR引物,以实现对多种YHV的同时检测或分型检测(OIE,2012)。
YHV对一些重要养殖对虾种类,如斑节对虾、凡纳滨对虾、褐对虾(Penaeus aztecus)、墨吉明对虾(Fenneropenaeus merguiensis)、细角滨对虾(Litopenaeusstylirostris)、刀额新对虾(Metapenaeus ensis)、白滨对虾(Litopenaeus setiferus)等都具有感染性(Jatuyosporn et al,2014)。本研究的流行病学调查显示,我国养殖的中国明对虾、凡纳滨对虾、日本囊对虾以及罗氏沼虾中均检出了YHV,而且在中国明对虾中的检出率最高,阳性也最强,说明中国明对虾是YHV的一个新发现的自然宿主,罗氏沼虾也是本研究新发现的一个自然宿主,这对增补YHV的宿主范围有重要意义(OIE,2012)。近年来,陆续在国内多个省市检测到YHV病毒的存在,说明YHV已经在我国养殖区域扩散,但由于发病样品较少,且受养殖品种、采样区域等因素限制,病原的源头尚未确定。新型YHV的出现表明我国对虾养殖产业已面临了新的疫病威胁,建立高效、系统、全面防控疫病的引入、传播和扩散风险的生物安保体系对于对虾健康养殖具有重要意义。
| 刘群, 黄倢, 杨昊霖, 等. 疑患EMS/AHPNS对虾中检出黄头病毒的一种新株型. 海洋与湖沼 , 2014, 45 (4) : 703-709 | |
| 熊炜, 邱璐, 李健, 等. 上海检验检疫局从泰国进境草虾中检出虾黄头病毒. 检验检疫科学 , 2006, 16 (6) : 61-62 | |
| Cowley JA, Walker PJ, Flegel TW, et al. Family Roniviridae. In King A, ed, Virus Taxonomy, IXth Report of the International Committee on Taxonomy of Viruses. London: Elsevier, Academic Press, 2012 : 797 -801. | |
| Gangnonngiw W, Anantasomboon GOW, Sang-oum W, et al. Non-virulence of a recombinant shrimp nidovirus is associated with its non-structural gene sequence and not a large structural gene deletion. Virology , 2009, 385 (1) : 161-168 DOI:10.1016/j.virol.2008.10.044 | |
| Hasson KW, Hasson J, Aubert H, et al. A new RNA-friendly fixative for the preservation of penaeid shrimp samples for virological detection using cDNA genomic probes. J Virol Methods , 1997, 66 (2) : 227-236 DOI:10.1016/S0166-0934(97)00066-9 | |
| Jatuyosporn T, Supungul P, Tassanakajon A, et al. The essential role of clathrin-mediated endocytosis in yellow head virus propagation in the black tiger shrimp Penaeus monodon. Dev Comp Immunol , 2014, 44 (1) : 100-110 DOI:10.1016/j.dci.2013.11.017 | |
| Lightner DV. A handbook of shrimp pathology and diagnostic procedures for diseases of cultured penaeid shrimp. Baton Rouge, LA: World Aquaculture Society, 1996 | |
| Limsuwan C, Handbook for cultivation of black tiger prawns. Tansetakit Co. Ltd, Bangkok, 1991 | |
| OIE. Manual of diagnostic tests for aquatic animals. World Organization for Animal Health, 2012 | |
| Senapin S, Thaowbut Y, Gangnonngiw W, et al. Impact of yellow head virus outbreaks in the whiteleg shrimp, Penaeus vannamei (Boone), in Thailand. J Fish Dis , 2010, 33 (5) : 421-430 DOI:10.1111/jfd.2010.33.issue-5 | |
| Spann KM, Vickers JE, Lester RJG. Lymphoid organ virus of Penaeus monodon from Australia. Dis Aquat Organ , 1995, 23 (2) : 127-134 | |
| Tran L, Nunan L, Redman RM, et al. Determination of the infectious nature of the agent of acute hepatopancreatic necrosis syndrome affecting penaeid shrimp. Dis Aquat Organ , 2013, 105 (1) : 45-55 DOI:10.3354/dao02621 | |
| Walker PJ, Cowley JA, Spann KM, et al. Yellow head complex viruses: transmission cycles and topographical distribution in the Asia-Pacific region. The New Wave: Proceedings of the Special Session on Sustainable Shrimp Culture, Aquaculture, 2001, 227-237 | |
| Wang CS, Tang KFJ, Kou GH, et al. Yellow head disease-like virus infection in the Kuruma shrimp Penaeus japonicus cultured in Taiwan. Fish Pathol , 1996, 31 (4) : 177-182 DOI:10.3147/jsfp.31.177 | |
| Wijegoonawardane PKM, Cowley JA, Phan T, et al. Genetic diversity in the yellow head nidovirus complex. Virology , 2008, 380 (2) : 213-225 DOI:10.1016/j.virol.2008.07.005 | |
| Wijegoonawardane PKM, Sittidilokratna N, Petchampai N, et al. Homologous genetic recombination in the yellow head complex of nidoviruses infecting Penaeus monodon shrimp. Virology , 2009, 390 (1) : 79-88 DOI:10.1016/j.virol.2009.04.015 |



