2. 上海海洋大学省部共建水产种质资源发掘与利用教育部重点实验室 上海 201306
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Shanghai Ocean University, Ministry of Education, Shanghai 201306
条斑紫菜(Pyropia yezoensis)为主要分布于我国北方及韩国和日本等地的重要紫菜栽培物种(张美如等,2012)。近年来,全球海水温度不断上升,对人工栽培紫菜的品质和产量产生了较大影响。为此,研究者通过人工选育的方法获得了稳定高产且抗逆性好的条斑紫菜优良品系(王华芝等,2012; Yan,2000; Zhang et al,2011)。条斑紫菜品系间的准确种质鉴定,不仅可以加快优良品种的选育速度,而且也是推广紫菜良种的重要基础工作。
目前,紫菜的种质鉴定主要有形态学、细胞学、生理生化和分子标记辅助等鉴定方法(张庆杰,2012)1)。在以往的条斑紫菜种质鉴定中,主要是依据传统的形态学和细胞学特征(Miura,1984、1988; Niwa,2005)。Hebert等(2003a、b)首次提出了基因条形码(DNA barcoding)的概念;随后,基因条码在紫菜种质鉴定中也得到了较广泛的应用(赵玲敏等,2009; Niwa,2003、2008、2009; Saunders,2005)。Wang等(2009)采用直接PCR技术对8个紫菜种的RUBISCO Spacer序列进行比较分析,表明该序列的同源性与紫菜种的来源呈相关关系,说明RUBISCO Spacer序列适用于紫菜种的分类。Milstein等(2012)对巴西3个紫菜种的25个品系的UPA、Cox1和Cox2-3基因条码进行了分析,并依据这3种序列的同源性成功命名了1个新的紫菜种(Pyropia sp. 77),同时确认了Pyropia drewiana和Pyropia spiralis是同一紫菜种。谢小雪等(2013)测定了坛紫菜11个品系的UPA、Cox2-3、Cox1和ITS-5.8S共4种基因条码序列,发现只有ITS-5.8S序列可以完全区分各个坛紫菜品系。
1) 张庆杰. 坛紫菜“申福2 号”SSR 分子标记的研究和性别相关AFLP 反应体系的建立. 上海海洋大学硕士研究生学位论文, 2012, 1–38
本研究利用4种基因条码(RUBISCO Spacer、UPA、Cox2-3和ITS-5.8S)对条斑紫菜6个品系的亲缘关系进行了比较分析,旨在找到一种适合于条斑紫菜品系间种质鉴定的方法。
1 材料与方法 1.1 实验材料本研究所用条斑紫菜6个品系(LS、B4、HT、TM18、GT和ROS-2)均以自由丝状体的形式被保存于实验室内,其保存和培养方法同Kato等(1984),其来源、代码及特性见表 1。
|
|
表 1 本研究所用的6个条斑紫菜品系的代码、来源及特性 Table 1 The codes,origins and characteristics of the six strains of Pyropia yezoensis in this study |
条斑紫菜各品系的自由丝状体用灭菌ddH2O清洗3次,用灭菌吸水纸吸去水分,取0.1 g自由丝状体,经液氮快速充分研磨后,使用DP320新型植物基因组试剂盒(TIANGEN,北京)提取基因组DNA,再用1%琼脂糖凝胶电泳检测基因组DNA的完整性,利用NanoDrop2000测定其基因组DNA的浓度和纯度,DNA保存在-20℃备用。
1.2.2 基因条码的扩增和测序用于RUBISCO Spacer、UPA、Cox2-3和ITS-5.8S基因条码扩增的特异性引物分别依据Wang等(2009)和谢小雪等(2013),由生工(上海)生物工程有限公司合成,具体的扩增位点及复性温度列入表 2。
|
|
表 2 4种基因条码的扩增引物序列 Table 2 Primers used for the amplification of the four DNA barcodings |
PCR反应体系均为25 μl,其中,ddH2O 13.5 μl,10×Loading buffer 2.5 μl,MgCl2 2 μl,dNTP 2.5 μl,正反向引物各0.5 μl,1 U Taq酶0.5 μl,基因组DNA 3.0 μl。
RUBISCO Spacer、UPA、Cox2-3和ITS-5.8S序列的PCR反应程序:94℃ 5 min;35个循环:94℃45 s,退火依据各引物的复性温度,72℃延伸45 s;最后72℃延伸10 min,4℃下保存。PCR产物经纯化后由生工(上海)生物工程有限公司进行克隆测序。
1.2.3 序列分析及系统树的构建用Blast软件进行同源性检测,利用DNAMAN软件进行序列比对及同源性分析,使用MEGA 5.0软件通过邻接法(NJ)构建4种基因条码的系统发育树。
2 结果与分析 2.1 条斑紫菜6个品系丝状体基因组DNA的提取条斑紫菜6个品系丝状体基因组DNA的分子量约为23 kb(图 1),OD260 nm/OD280 nm在1.8-2.0之间,可满足后续的实验需要。

|
图 1 条斑紫菜6个品系的基因组DNA的琼脂糖凝胶电泳 Figure 1 Agarose electrophoresis of the total DNA extracted from the six strains of P. yezoensis M: λDNA/HindⅢ digest DNA marker; 1-6: LS,B4,HT,TM18,GT,and ROS-2 respectively |
经PCR反应,从条斑紫菜6个品系的基因组DNA中分别扩增出4种基因条码的特异性产物,分子量分别约为500 bp、365 bp、365 bp和1100 bp(图 2)。PCR反应产物条带单一,经克隆测序后,4种基因条码的NCBI收录号列入表 3。
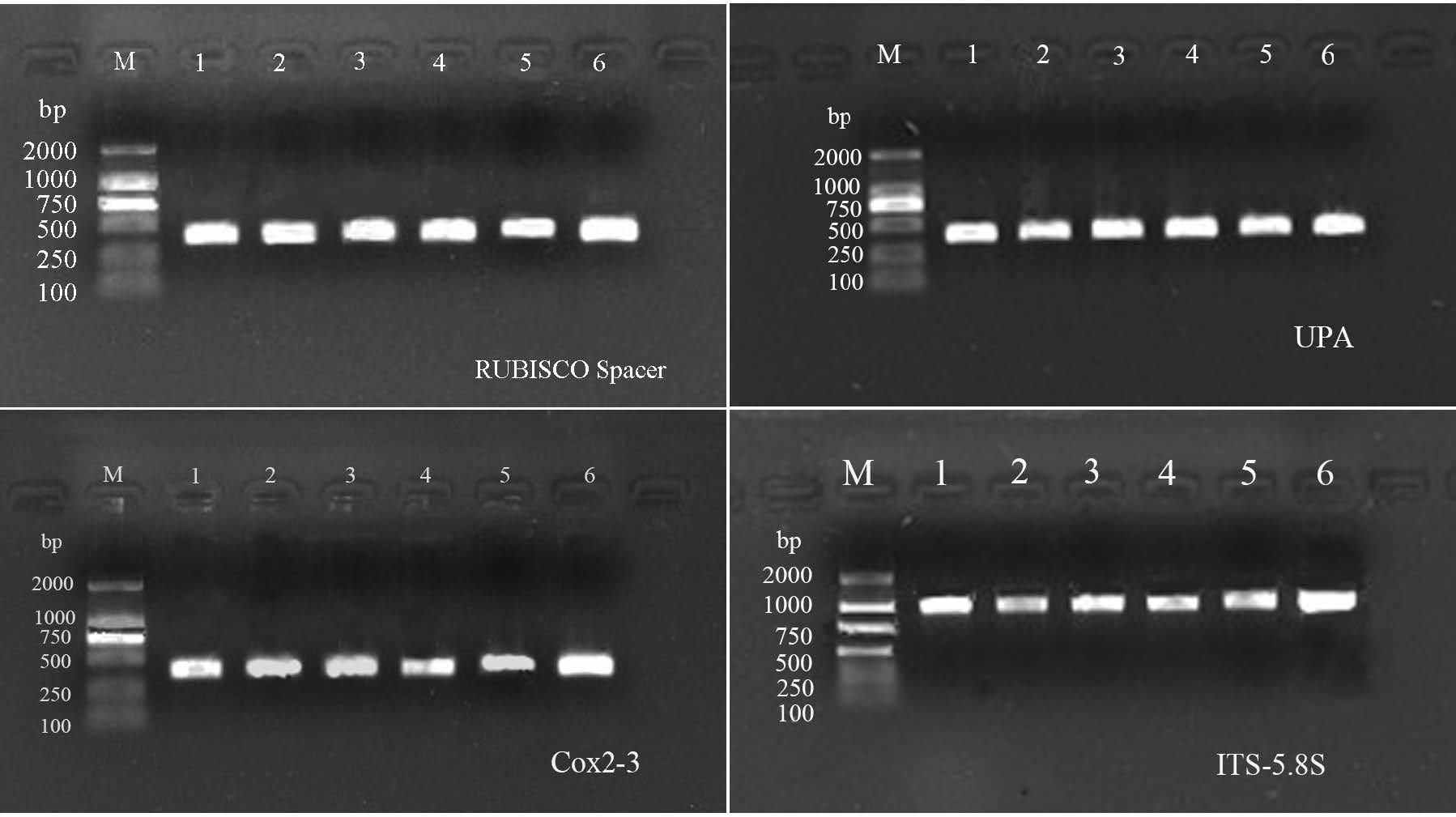
|
图 2 条斑紫菜6个品系的4种基因条码的琼脂糖凝胶电泳 Figure 2 Agarose electrophoresis of the four DNA barcodings of the six strains of P. yezoensis M: DL2000 DNA Marker; 1-6: LS,B4,HT,TM18,GT,and ROS-2 respectively |
|
|
表 3 条斑紫菜6个品系4种基因条码的NCBI收录号 Table 3 Accession numbers of the four DNA barcodings of the six strains of P. yezoensis |
条斑紫菜6个品系的RUBISCO Spacer序列产物包括大亚基rbcL、小亚基rbcS以及基因间隔区RUBISCO Spacer。根据基因间隔区RUBISCO Spacer序列计算的同源性如表 4所示,条斑紫菜6个品系具有完全相同的RUBISCO Spacer序列,同源性为100%。
|
|
表 4 基于RUBISCO Spacer和UPA序列的条斑紫菜6个品系同源性分析 Table 4 Sequence homology analysis of the six strains of P. yezoensis based on RUBISCO Spacer and UPA |
条斑紫菜6个品系的UPA序列的同源性在99.2%- 100%之间,其中LS、B4、HT和TM18这4个品系的UPA序列完全相同,序列同源性为100%。ROS-2和GT品系在4个位点发生了突变,与LS、B4、HT和TM18这4个品系的序列同源性均为99.2%。
Cox2-3序列由COX2和COX3的部分编码序列以及基因间隔区3个部分组成。条斑紫菜6个品系的Cox2-3序列同源性在97.8%-99.2%之间(表 5)。GT与ROS-2品系和HT与B4品系的序列同源性最高,均达到99.2%,HT与ROS-2品系序列同源性最低,只有97.8%。
|
|
表 5 基于Cox2-3和ITS-5.8S序列的条斑紫菜6个品系同源性分析 Table 5 Sequence homology analysis of the six strains of P. yezoensis based on Cox2-3 and ITS-5.8S |
ITS-5.8S序列包括核糖体18S、28S部分序列以及ITS-5.8S完整序列。条斑紫菜6个品系ITS-5.8S序列同源性较高,同源性在98.4%-99.8%之间。其中,GT与ROS-2品系、B4与TM18品系的ITS-5.8S序列同源性最高,均为99.8%。B4与ROS-2品系的序列同源性最低,为98.6%。
2.4 条斑紫菜6个品系4种基因条码的系统发育树基于UPA、Cox2-3和ITS-5.8S基因条码,通过邻接法(NJ)构建的系统发育树如图 3-图 5所示。UPA和ITS-5.8S两种序列均将中国产的4个品系与日本产的两个品系分别聚成两个分支,其聚类结果与6个品系的原有地域亲缘关系是一致的。利用ITS-5.8S序列均能对中国产的4个品系和日本产的两个品系进行再区分,而利用UPA序列却无法对中国产的4个品系进行再区分,只能区分日本产的两个品系。依据Cox2-3序列将中国产的两个品系(LS和TM18)和日本产的两个品系(GT和ROS-2)聚集成一个大分支,其聚类结果与实际亲缘关系不一致。以上结果表明,在本研究的6个条斑紫菜品系的种质鉴定中,ITS-5.8S序列的区分效果最好。

|
图 3 基于UPA序列通过邻接法构建的条斑紫菜6个品系的系统发育树 Figure 3 Phylogenetic tree of UPA of the six strains of P. yezoensis constructed with the neighbor-joining analysis |
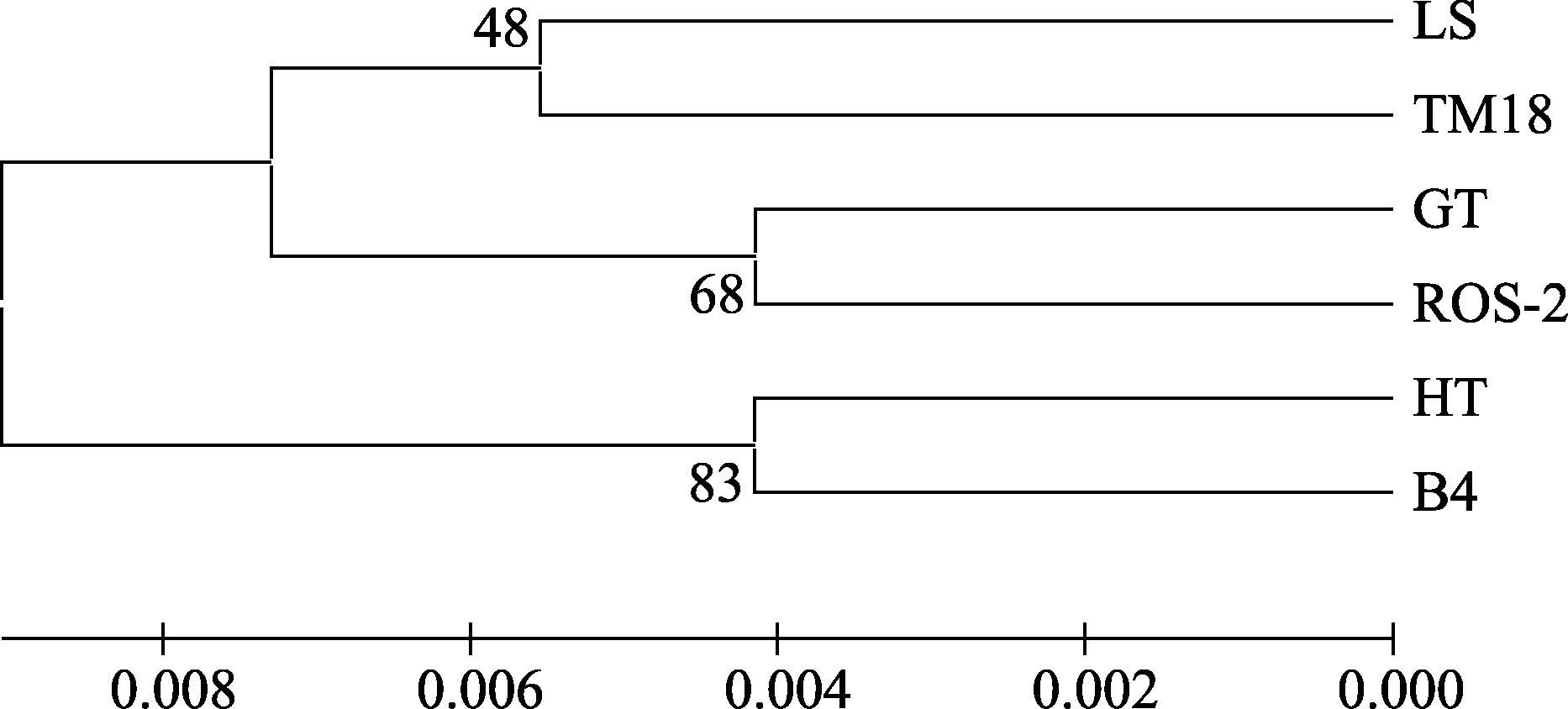
|
图 4 基于Cox2-3序列通过邻接法构建的条斑紫菜6个品系的系统发育树 Figure 4 Phylogenetic tree of Cox2-3 of the six strains of P. yezoensis constructed with the neighbor-joining analysis |
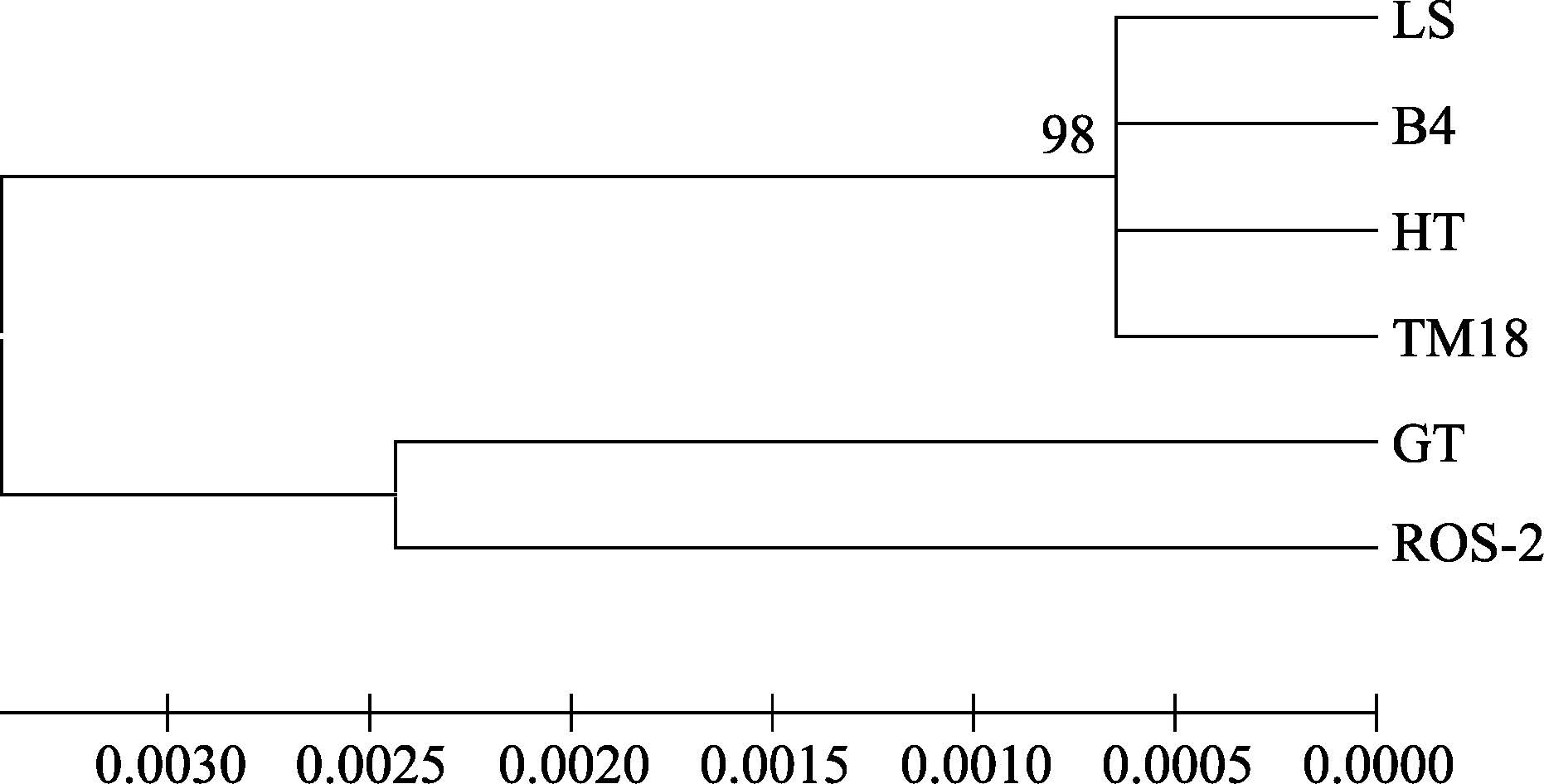
|
图 5 基于ITS-5.8S序列通过NJ法构建的条斑紫菜6个品系的系统发育树 Figure 5 Phylogenetic tree of ITS-5.8S of six strains of P. yezoensis constructed with the neighbor-joining analysis |
目前,DNA分子标记技术主要有两种:一是依据PCR反应扩增产物的有无和长短,对生物物种的遗传多样性进行研究,如SSR、AFLP、SRAP等。这类分子标记技术已经在紫菜的种质鉴定中得到了较广泛的应用(杨锐等,2005; 袁昭岚等,2006; 纪德华等,2008; 张鹏等,2009; 谢潮添等,2010; 贾威等,2013; 王婷等,2013);二是以基因条码(DNA barcoding)即基因组中DNA片段的序列信息为基础,对生物物种的遗传多样性进行分析。作为基因组的一部分,在个体发育的不同时期和处于环境胁迫条件下,基因条码的序列不会发生改变,避免了传统形态学和细胞学鉴定中易受环境因子影响的弊端,使得鉴定的结果更加真实可靠(徐涤等,2001; 袁昭岚等,2006)。
本研究利用4种基因条码(RUBISCO Spacer、UPA、Cox2-3和ITS-5.8S)对中国产和日本产的6个条斑紫菜品系的亲缘关系进行了研究,其结果证实,6个条斑紫菜品系具有完全相同的RUBISCO Spacer序列,序列同源性均为100%,中国产的4个条斑紫菜品系(LS、B4、HT和TM18)的UPA序列完全相同,与日本产的两个品系(GT和ROS-2)相比,存在4个碱基的差别,序列同源性均为99.2%,上述结果与 孙雪等(2006)对坛紫菜的RUBISCO Spacer以及Milstein等(2012)对紫菜的UPA序列的研究结果类似,说明RUBISCO Spacer和UPA的序列在紫菜种间或品系间均是很保守的。另外,本研究也发现ITS-5.8S序列的同源性虽然为98.4%-99.8%,但分子量为160 bp的5.8S rDNA的序列则完全相同。利用UPA和ITS-5.8S两种序列均能将中国产的4个品系与日本产的两个品系分别聚成两个分支,其聚类结果与6个品系的原有地域亲缘关系是一致的,表明生长环境的差异也许是导致以上两种序列聚类结果的原因。但是,利用ITS-5.8S序列均能对中国产的4个品系和日本产的两个品系进行再区分,而利用UPA序列却无法对中国产的4个品系进行再区分,只能区分日本产的两个品系。依据Cox2-3序列则将中国产的两个品系(LS和TM18)和日本产的两个品系(GT和ROS-2)聚集成一个大分支,其聚类结果与实际亲缘关系不一致,这与谢小雪等(2013)对坛紫菜Cox2-3序列的研究结果相似。依据本研究结果,在6个条斑紫菜品系间的种质鉴定中,ITS-5.8S序列的区分效果最好,这个结果与坛紫菜品系间分子鉴定的相关研究结果类似(谢小雪等,2013)。
| 王华芝, 严兴洪, 李琳. 条斑紫菜(Porphyra yezoensis)耐高温品系的筛选及特性分析. 海洋与湖沼 , 2012, 43 (3) : 363-369 | |
| 王婷, 徐燕, 谢潮添, 等. 基于SCAR标记的坛紫菜“闽丰1号”多重PCR鉴定技术的建立. 水产学报 , 2013, 37 (5) : 688-695 | |
| 孙雪, 杨锐, 刘必谦, 等. 坛紫菜rbcS及rbcL-rbcS基因间隔区的序列分析. 水产科学 , 2006, 25 (1) : 23-26 | |
| 纪德华, 谢潮添, 陈昌生, 等. 坛紫菜ISSR反应体系的建立与优化. 海洋科学 , 2008, 32 (6) : 20-24 | |
| 严兴洪, 田中次郎, 有贺佑胜. 条斑紫菜色彩突变体的诱导、分离和特性分析. 水产学报 , 2000, 24 (3) : 221-228 | |
| 杨锐, 刘必谦, 骆其君, 等. 利用AFLP技术研究条斑紫菜的遗传变异. 海洋学报 , 2005, 27 (3) : 159-162 | |
| 张美如, 陆勤勤, 许广平. 条斑紫菜产业现状及对其健康发展的思考. 中国水产 , 2012 (11) : 15-19 | |
| 张鹏, 张源, 王铁杆, 等. 坛紫菜不同品系亲缘关系的SSR标记分析. 中国水产科学 , 2009, 16 (6) : 842-849 | |
| 赵玲敏, 谢潮添, 陈昌生, 等. 5.8S rDNA-ITS区片段的序列分析在坛紫菜种质鉴定中的应用. 水产学报 , 2009, 33 (6) : 940-948 | |
| 袁昭岚, 黄鹤忠, 沈颂东, 等. 条斑紫菜5个栽培品系的ISSR分析. 海洋科学 , 2006, 30 (7) : 9-14 | |
| 贾威, 黄林彬, 严兴洪. 条斑紫菜6个品系的SRAP分析. 水产学报 , 2013, 37 (10) : 1495-1502 | |
| 徐涤, 宋林生, 秦松, 等. 五个紫菜品系间遗传差异的RAPD分析. 高科技通讯 , 2001, 11 (12) : 1-4 | |
| 谢小雪, 陈昌生, 徐燕, 等. DNA序列标记在坛紫菜种质鉴定中适用性的比较分析. 应用海洋学学报 , 2013, 32 (3) : 404-410 | |
| 谢潮添, 陈昌生, 纪德华, 等. 坛紫菜种质材料DNA指纹图谱的构建. 水产学报 , 2010, 34 (6) : 913-920 | |
| Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes. Proc Biol Sci , 2003a, 270 (1512) : 313-322 DOI:10.1098/rspb.2002.2218 | |
| Hebert PDN, Ratnasingham S, dewaard JR. Barcoding animal life: cytochrome Coxidase I divergences among closely related species. Proc Roy Soc B Biol Sci , 2003b, 270 (1524) : S96-S99 | |
| Kato M, Aruga Y. Comparative studies on the growth and photosynthesis of the pigmentation mutants of Porphyra yezoensis in laboratory culture. J Phycol , 1984, 32 : 333-347 | |
| Milstein D, Medeiros AS, Oliveira EC, et al. Will a DNA barcoding approach be useful to identify Porphyra species (Bangiales, Rhodophyta). J Appl Phycol , 2012, 24 (4) : 837-845 DOI:10.1007/s10811-011-9702-3 | |
| Miura A. A new variety and a new form of Porphyra (Bangiales, Rhodophyta) from Japan: Porphyra tenera Kjellman var. tamatsuensis Miura, var. nov. and P. yezoensis Ueda form. narawaensis Miura, form. nov. J Tykyo Univ Fish , 1984, 71 : 1-37 | |
| Miura A. Taxonomic studies of Porphyra species cultivated in Japan, referring to their transition to the cultivated variety. J Tokyo Univ Fish , 1988, 75 : 311-325 | |
| Niwa K, Aruga Y. Rapid DNA extraction from conchocelis and ITS-1 rDNA sequences of seven strains of cultivated Porphyra yezoensis (Bangiales Rhodophyta). J Appl Phycol , 2003, 15 (1) : 29-35 DOI:10.1023/A:1022998823920 | |
| Niwa K, Iida S, Kato A, et al. Genetic diversity and introgression in two cultivated species (Porphyra yezoensis and Porphyra tenera) and closely related wild species of Porphyra (Bangiales, Rhodophyta). J Phycol , 2009, 45 (2) : 493-502 DOI:10.1111/jpy.2009.45.issue-2 | |
| Niwa K, Kato A, Kobiyama A, et al. Comparative study of wild and cultivated Porphyra yezoensis (Bangiales, Rhodophyta) based on molecular and morphological data. J Appl Phycol , 2008, 20 (3) : 261-270 DOI:10.1007/s10811-007-9242-z | |
| Niwa K, Kikuchi N, Aruga Y. Morphological and molecular analysis of the endangered species Porphyra tenera (Bangiales, Rhodophyta). J Phycol , 2005, 41 (2) : 294-304 DOI:10.1111/(ISSN)1529-8817 | |
| Saunders GW. Applying DNA barcoding to red macroalgae: a preliminary appraisal holds promise for future applications. Philos Trans R Soc Lond B Biol Sci , 2005, 360 (1462) : 1879-1888 DOI:10.1098/rstb.2005.1719 | |
| Wang C, Dong D, Wang GC, et al. An improved PCR method for direct identification of Porphyra (Bangiales, Rhodophyta) using conchocelis based on a RUBISCO intergenic spacer. Chin J Oceanol Limnol , 2009, 27 (3) : 513-518 DOI:10.1007/s00343-009-9169-0 | |
| Zhang BL, Yan XH, Huang LB. Evaluation of an improved strain of Porphyra yezoensis Ueda (Bangiales Rhodophyta) with high-temperature tolerance. J Appl Phycol , 2011, 23 (5) : 841-847 DOI:10.1007/s10811-010-9587-6 |



