2. 中国水产科学研究院黄海水产研究所 青岛 266071;
3. 烟台海益苗业有限公司 烟台 265608
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071 ;
3. Yantai Haiyi Seeds Co., Ltd, Yantai 265608
刺参(Apostichopus japonicus),又称仿刺参,在我国主要分布于辽宁、山东、河北等北方沿海地区,其营养价值高且药用价值广泛(廖玉麟,1997),是我国重要的海水养殖品种。刺参体色变化较大,背面一般为黄褐色或棕褐色,腹面为浅黄褐色,此外还有黄绿色、赤褐色、灰白色(于东祥等,2005),而只有极少数刺参背腹面均为纯白色,呈白化特征,被称为“白刺参”(赵鹤凌,2011)1)。野生白化刺参十分稀少,其经济和营养价值不容小觑,受到研究人员的广泛关注。
1) 赵鹤凌. 刺参Apostichopus japonicus (Selenka)白化特征发生机理的基础研究.中国科学院研究生院(海洋研究所)博士研究生学位论文, 2011, 1-74
DNA甲基化(DNA methylation)是一种表观遗传(Epigenetic)现象,即在生物发育过程中,虽然核苷酸序列没有发生变化,但基因表达调控发生改变,它可以遗传,也可以逆转(Henikoff et al,1997)。DNA甲基化在基因沉默、基因印迹、X染色体失活、染色体完整性的维持、细胞分化、发育、DNA修复及重组等多个生物学过程中发挥着重要作用(宫时玉等,2005)。目前,DNA甲基化检测的方法较多,其中,甲基化敏感扩增多态性(Methylation-sensitive amplified polymorphism,MSAP)技术由于其简便、高效、可靠且对全基因组和片段基因都可以进行检测等优点而得到广泛应用。MSAP分析是基于选择性PCR的一种新技术,是AFLP技术的延伸(Reyna-Lopez et al,1997),其独特之处是利用两种对DNA甲基化敏感程度不一样的内切酶(HpaⅡ和MspⅠ),分别与一种对DNA甲基化不敏感的内切酶(EcoRⅠ)一起对同一识别序列CCGG进行不同的切割,从而产生不同大小的酶切片段,再连接相应的接头和引物进行特异性扩增,以辨别哪些位点存在甲基化。
目前,MSAP技术已广泛应用于检测生物在胁迫条件下基因组DNA甲基化的变化(Vandegehuchte et al,2009; Labra et al,2004; Aina et al,2004; Wang et al,2011; 朱华平等,2013),杂种优势与杂交种基因组DNA甲基化的关系(Sun et al,2014; Zhang et al,2007; Tsaftaris et al,1998),DNA甲基化对发育相关基因表达的调控作用(BrÖske et al,2009; Rai et al,2006; Beaujean et al,2004; Feng et al,2010),生物不同组织基因组DNA甲基化水平分析(曹哲明等,2007、2009; 姜群等,2014; 杜盈等,2013)等方面。郭婷婷等(2013)通过对刺参4种组织基因组甲基化状态的MSAP分析,证实了甲基化是刺参基因组DNA的一种普遍特征,而且不同组织间甲基化模式存在显著差异。目前,有关白刺参DNA甲基化方面的研究,尚未见报道。本研究运用MSAP技术比较分析了普通刺参和白刺参的体壁、呼吸树、消化道3种组织的基因组DNA甲基化水平和模式,初步探讨了DNA甲基化差异性调控对组织间基因特异性表达的影响,以期为白刺参的选育提供基础数据。
1 材料与方法 1.1 材料来源普通刺参和白刺参采自烟台海益苗业有限公司,其中,普通刺参为人工选育的快速生长的F2代刺参,白刺参为人工选育的F2代白刺参。两个群体各取10头刺参,普通刺参平均体质量为(49.58±0.78)g,白刺参平均体质量为(51.02±0.97)g。刺参在实验室驯养7 d后,清空消化道,分别取其等量体壁、呼吸树和消化道于-80℃保存备用。
1.2 基因组DNA提取DNA提取采用CTAB法(战爱斌等,2006),略有改动。取材料约100 mg,用液氮研磨至粉末,加入裂解液600 μl(2% CTAB,1.4 mol/L NaCl,0.1 mol/L Tris-HCl,20 mmol/L EDTA-Na2,0.2% β-羟基乙醇,pH=8.0),15 μl 20 mg/ml的蛋白酶K,混合均匀,60℃水浴锅中3-5 h,期间每隔片刻缓慢晃动离心管,用氯仿:异戊醇=24:1抽提两次后,用酚:氯仿:异戊醇=25:24:1抽提两次,再加入等量异丙醇,然后用预冷的无水乙醇沉淀,风干,后用灭菌ddH2O溶解。通过1%琼脂糖电泳检测DNA是否降解,检测OD260nm/280nm以确定DNA的纯度,并稀释至300 ng/μl,-20℃中保存备用。
1.3 MSAP分析参考Xiong等(1999)的MSAP分析方法,并作适当改动,MSAP反应程序和条件如下:
1.3.1 酶切和连接反应体系对于每份DNA,同时设置EcoRⅠ+ MspⅠ和EcoRⅠ+HpaⅡ两种酶切反应。酶切反应体系20 μl:300 ng DNA,5 U EcoR I(TaKaRa),5 U Hpa II/Msp I(TaKaRa),2 μl 10×Buffer(TaKaRa),加灭菌ddH2O至20 μl。酶切体系经过37℃水浴6 h、70℃变性10 min后进行1%琼脂糖电泳检测酶切产物,于-20℃保存备用。连接反应体系:5 μl酶切产物,50 pmol HM接头,10 pmol E接头,5 U T4 DNA Ligase(TaKaRa),4 μl 5×T4 DNA Ligase Buffer,补水至20 μl,16℃连接过夜,产物稀释10倍后用于预扩增。
1.3.2 预扩增反应体系预扩增反应体系:5 μl稀释连接产物,20 pmol预扩引物E0和HM0,0.1 μl Taq酶,2 μl dNTPs,2 μl buffer(含Mg2+),加入灭菌ddH2O至20 μl。混匀后进行预扩增反应,PCR预扩增反应为25个循环,每个循环包括94℃变性30 s,56℃退火1 min,72℃延伸1 min,首次循环前94℃预变性5 min;最后一次循环结束后72℃延伸10 min。产物于4℃保存,1%琼脂糖检测扩增产物后,稀释20倍用于选择性扩增,-20℃保存备用。
1.3.3 选择性扩增反应体系选择性扩增反应体系:3 μl稀释预扩增产物,20 pmol选扩引物HMn和0.5 pmol选扩引物En,2 μl buffer(含Mg2+),2 μl dNTPs,0.1 μl Taq酶,加入灭菌ddH2O至20 μl。混匀后进行选择性扩增反应,PCR选择性扩增反应条件为:94℃预变性5 min;94℃变性30 s,65℃退火30 s(每循环降低0.7℃),72℃延伸1 min,13个循环;95℃变性30 s,56℃退火30 s,72℃延伸1 min,27个循环;72℃延伸10 min;-20℃保存备用。本研究所用的引物信息见表 1。
|
|
表 1 本研究所用的接头及引物序列 Table 1 Sequence of adapters and primers used in this study |
变性聚丙烯酰胺凝胶电泳检测:选扩引物与上样缓冲液按4:1.5均匀混合,95℃变性10 min,迅速冰浴5 min,6%聚丙烯酰胺凝胶电泳(功率为60 W)2.5 h,电泳结束后采用银染法显色。
1.4 MSAP条带统计及数据分析同一DNA分别用HpaⅡ/MspⅠ与EcoRⅠ组合酶切后选扩,因此,MSAP图谱上对应有两个泳道,分别是H和M泳道,根据扩增产物在两个泳道内有无的情况,可以将甲基化模式分为4种(吕佳等,2013):Ⅰ型,H泳道无带,M泳道有带,为双链内甲基化,代表全甲基化位点;Ⅱ型,H泳道有带,M泳道无带,为单链外甲基化,代表半甲基化位点;Ⅲ型,两个泳道均有条带,代表去甲基化位点;Ⅳ型,H和M泳道均没有带,代表双链外甲基化的位点,Ⅳ型位点不做统计。统计MSAP电泳图每个泳道中大小为100-1500 bp的条带,同一位点上有带记为1,无带记为0,并计算每种组织的全甲基化率、半甲基化率及总甲基化率,利用SPSS 19.0进行方差分析。
2 结果 2.1 引物筛选普通刺参和白刺参体壁、呼吸树、消化道混合基因组DNA通过酶切、连接和预扩增之后,由4个HM引物和4个E引物组成的引物对进行选择性扩增,并进行聚丙烯酰胺凝胶电泳、银染、成像和统计。从16对引物组合中筛选出9对组合(表 1),这些引物组合能够稳定地扩增出清晰、多态的谱带,并运用9对引物组合对刺参和白刺参不同组织进行基因组DNA的MSAP分析。
2.2 MSAP带型分析刺参基因组DNA的选扩产物经过6%的聚丙烯酰氨凝胶电泳后,银染成像(图 1),形成3种类型条带,包括不同的甲基化类型和多态性类型。
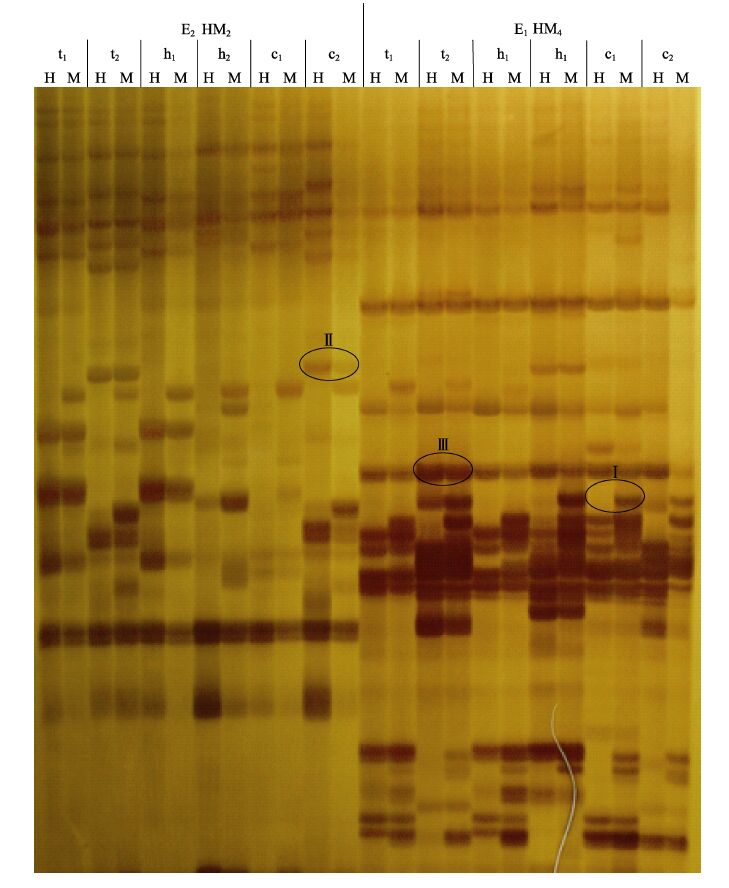
|
图 1 不同引物组合在刺参基因组DNA中的MSAP图谱 Figure 1 DNA methylation profiles in Apostichopus japonicus using different primer pairs Ⅰ型:全甲基化位点;Ⅱ型:半甲基化位点;Ⅲ型:去甲基化位点泳道t1、h1、c1分别代表普通刺参的体壁、呼吸树、消化道;泳道t2、h2、c2分别代表白刺参的体壁、呼吸树、消化道 Ⅰ: fully methylated; Ⅱ: hemi-methylated; Ⅲ: unmethylated Lane t1,h1,and c1 represented body wall,respiratory tree,and digestive tract of A. japonicus,respectively; Lane t2,h2,and c2 represented body wall,respiratory tree,and digestive tract of white A. japonicus,respectively |
用MSAP方法分析普通刺参和白刺参的体壁、呼吸树、消化道3种组织的基因组的甲基化水平,并用SPSS 19.0统计分析软件进行方差分析。扩增条带类型及甲基化水平结果见表 2和表 3。结果显示,普通刺参和白刺参3种组织的总扩增条带数分别为5932和5208,且都以未甲基化位点为主,其中:普通刺参未甲基化位点数为4317,占总扩增条带数的72.78%;白刺参未甲基化位点数为3944,占总扩增条带数的75.73%。而且,3种组织的总扩增位点数变化趋势是一致的,均是体壁最高,其次是呼吸树和消化道。普通刺参中由高到低依次是体壁(2317)、呼吸树(1918)、消化道(1697),白刺参中由高到低依次是体壁(1777)、呼吸树(1733)、消化道(1698)。3种组织的总甲基化率关系如表 4所示,在普通刺参中,体壁(31.07%)>消化道(26.34%)>呼吸树(23.36%),3种组织之间差异显著(P<0.05);在白刺参中,体壁(29.88%)>呼吸树(23.25%)>消化道(19.45%),三者之间存在显著性差异(P<0.05)。刺参和白刺参各组织的全甲基化率之间也存在显著性差异(P<0.05)。
|
|
表 2 普通刺参不同组织中甲基化扩增位点 Table 2 Methylation amplification sites in different tissues of A. japonicus |
|
|
表 3 白刺参不同组织中甲基化扩增位点 Table 3 Methylation amplification sites in different tissues of white A. japonicus |
|
|
表 4 普通刺参和白刺参中3种组织之间甲基化差异 Table 4 Differences of methylation in three tissues of A. japonicus and white A. japonicus |
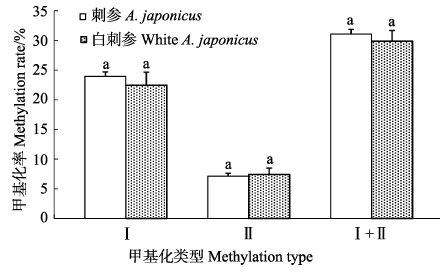
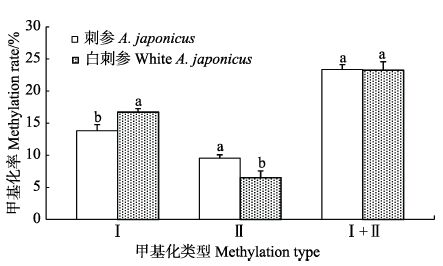
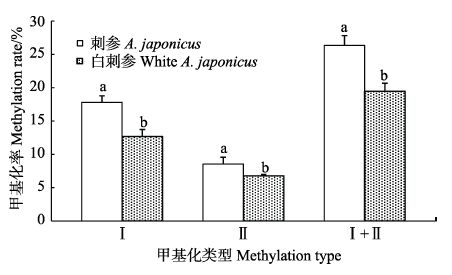
本研究中,普通刺参群体3种组织的甲基化扩增位点数和总甲基化率均高于白刺参群体(表 2和表 3),普通刺参群体和白刺参群体的体壁和呼吸树的总甲基化率之间差异不显著(P>0.05)(图 2和图 3),分别是31.07%、29.88%和23.36%、23.25%,而消化道的总甲基化率群体间差异显著(P<0.05)(图 4);不同组织的甲基化类型也不同,两刺参群体体壁的基因组全甲基化率和半甲基化率差异不显著(P>0.05),普通刺参呼吸树的全甲基化率显著低于白刺参群体(P<0.05),半甲基化率显著高于白刺参群体(P<0.05),而普通刺参消化道的全甲基化率和半甲基化率均显著高于白刺参群体(P<0.05)。
|
图 2 普通刺参和白刺参体壁之间甲基化差异 Figure 2 Differences of methylation in the body wall of A. japonicus and white A. japonicus |
|
图 3 普通刺参和白刺参呼吸树之间甲基化差异 Figure 3 Differences of methylation in the respiratory tree of A. japonicus and white A. japonicus |
|
图 4 普通刺参和白刺参消化道之间甲基化差异 Figure 4 Differences of methylation in the digestive tract of A. japonicus and white A. japonicus 注:不同字母表示差异显著(P<0.05) Note:Different letters denoted significant difference |
近年来,随着MSAP技术的不断革新发展,MSAP在越来越多动植物的研究中得到应用(Waalwijk et al,1978)。DNA甲基化机制和功能的研究多集中在哺乳动物和植物中,无脊椎动物的甲基化研究相对起步较晚。研究表明,无脊椎动物存在不同程度的甲基化(Roberts et al,2012),水产无脊椎动物总甲基化率一般在20%-40%之间,如虾夷扇贝(Patinopecten yessoensis)和栉孔扇贝(Chlamys farreri)甲基化率分别为32.79%和24.13%(于涛等,2010);太平洋牡蛎(Crassostrea gigas)甲基化水平约为26%(Jiang et al,2013);文蛤(Meretrix meretrix)4个品系CCGG序列中有26.7%-30.0%发生甲基化(朱东丽,2011)1);背角无齿蚌(Anodonta woodiana)5个组织的甲基化水平范围在35.5%-56%(曹哲明等,2009)。棘皮动物甲基化研究报道较少,有报道认为棘皮动物的甲基化水平属于中等水平(Regev et al,1998)。棘皮动物门的海星(Echinus esculentus)总甲基化率约为40%(Bird et al,1979);马粪海胆(Glyptocidaris crenularis vivo)甲基化水平约为11.86%-14.57%(王路燕,2013)2)。研究表明,刺参总甲基化率为28.60%-35.77%(郭婷婷等,2013),也有研究表明为27.61%-56.52%(Zhao et al,2015)。本研究得到的普通刺参和白刺参3种组织基因组DNA总甲基化率为23.36%-31.07%,与栉孔扇贝(C. farreri)(于涛等,2010)的类似,处于中等水平。本研究得到刺参的总甲基化率大大低于郭婷婷等(2013)和Zhao等(2015)得到的刺参总甲基化率。在对虾夷扇贝和栉孔扇贝DNA甲基化与生长速度相关性的研究中证明,DNA甲基化与生长速度呈负相关(吴彪等,2012)。对中国明对虾的研究也表明,快速生长的“黄海1号”中国明对虾总体甲基化率比例低于野生组中国明对虾(杜盈等,2013)。本研究采集的普通刺参和白刺参均是海益苗业有限公司人工选育的快速生长的F2代刺参,这可能是导致本研究中刺参总甲基化率较低的主要原因。
1) 朱东丽.2011. 文蛤(Meretrix meretrix)不同壳色花纹品系的分子标记及DNA甲基化分析. 中国海洋大学硕士研究生学位论文,1-53
2) 王路燕.2013. PFOS对黄海胆体内酶活及DNA甲基化的影响研究. 大连海事大学硕士研究生学位论文,1-52
3.2 刺参不同组织基因组DNA甲基化模式具有特异性本研究中,刺参不同组织基因组DNA的甲基化模式具有特异性,3种组织的总甲基化率和全甲基化率大小顺序为:体壁>消化道>呼吸树,而且体壁的半甲基化率与呼吸树和消化道的半甲基化率差异显著(P<0.05),呼吸树和消化道之间差异不显著(P>0.05),这与Zhao等(2015)的研究结果一致。这些结果表明,甲基化在刺参组织分化和发育过程中可能发挥着十分重要的作用(Holiday et al,1975)。动物体不同组织行使着不同的生理功能,而甲基化在功能基因调控中起着一定作用,推测甲基化的组织特异性与此相关。在本研究中,无论普通刺参还是白刺参中体壁组织的总甲基化率都是最高的,分别为31.07%和29.88%,刺参体壁组织富含致密结缔组织,因此其代谢水平和基因表达水平都低于其他组织,这可能是超甲基化修饰的结果(Zhao et al,2015; Takemae et al,2009)。本研究中,普通刺参和白刺参体壁的总甲基化率、全甲基化率和半甲基化率之间差异不显著(P>0.05),而呼吸树和消化道的全甲基化率和半甲基化率在两群体之间具有显著性差异(P<0.05)。呼吸树和消化道分别是刺参非常重要和敏感的器官,极易受到养殖环境中水温、食物等各种因素的刺激和影响,从而影响到基因组DNA甲基化修饰水平的表达,导致该两个组织的甲基化在不同群体中产生差异,这些差异的结果是否与两个刺参群体体色、摄食与生长相关,有待进一步验证。
3.3 白刺参与普通刺参基因组DNA甲基化的比较本研究中普通刺参3种组织的总甲基化率是高于白刺参的。大量研究表明,同一物种不同群体的相同组织基因组DNA甲基化水平也存在着不同。不同品种猪肌肉组织基因组DNA甲基化程度差异显著(杨春,20113)(P<0.05);在对不同品种鸡的研究中发现,鸡同一组织的甲基化水平在不同群体中是不同的,而不同组织甲基化水平的排序在不同的群体也是不一致的(徐青等,2011)。以上研究说明,动物组织基因组甲基化水平会受到群体遗传背景的影响。目前,DNA甲基化也越来越多应用于具有不同体色动物群体间的比较研究,同一物种的不同体色可能与甲基化状态有关。研究表明,不同壳色花纹的文蛤品系之间的甲基化模式存在差异,推测DNA甲基化有可能参与调控文蛤不同壳色花纹的形成(朱东丽,2011)1)。“海大金贝”为中国海洋大学选育的虾夷扇贝新品种,由于类胡萝卜素的积累,使其闭壳肌呈橘红色,吕佳等(2013)通过对扇贝全基因组DNA甲基化进行了研究,发现“海大金贝”和普通虾夷扇贝MSAP图谱有46个位点的甲基化模式存在差异,推测“海大金贝”与普通虾夷扇贝闭壳肌积累类胡萝卜素性状的差异很可能是由于相关基因调控区域甲基化状态的改变引起。目前,在鱼类中已发现多个基因参与动物黑色素的合成,只要影响黑色素生成过程中的任意环节,都会导致白化的发生(马得友,2013)4)。白刺参中黑色素的合成受阻是否受表观遗传影响以及影响的机制有待更进一步的研究。通过对白刺参基因组甲基化的深入研究,还可能获得与刺参体色相关的特异性的甲基化片段,进而获得相关的功能基因,通过对其功能进行分析,可以探索甲基化在调控白刺参体色形成中的作用及机制。
3) 杨春.2011. 猪不同组织和不同品种肌肉组织基因组DNA甲基化分析. 吉林大学博士研究生学位论文,1-107
4) 马得友.2013. 基于高通量测序的刺参白化发生和子代体色分离研究. 中国科学院研究生院(海洋研究所)博士研究生学位论文,1-97
| 于东祥, 孙慧玲, 陈四清, 等. 海参健康养殖技术(第2版). 北京: 海洋出版社, 2005 : 29 -30. | |
| 于涛, 杨爱国, 吴彪, 等. 栉孔扇贝、虾夷扇贝及其杂交子代的MSAP分析. 水产学报 , 2010, 34 (9) : 1335-1342 | |
| 吕佳, 侯睿, 李宁, 等. 应用MSAP技术研究扇贝全基因组DNA甲基化水平. 中国海洋大学学报(自然科学版) , 2013, 43 (10) : 48-53 | |
| 朱华平, 卢迈新, 黄樟翰, 等. 低温对罗非鱼基因组DNA甲基化的影响. 水产学报 , 2013, 37 (10) : 1460-1467 | |
| 杜盈, 何玉英, 李健, 等. 野生和“黄海1号”中国明对虾不同组织基因组DNA的MSAP分析. 中国水产科学 , 2013, 20 (3) : 536-543 | |
| 吴彪, 杨爱国, 刘志鸿, 等. 两种扇贝杂交和自交家系早期生长及甲基化的比较分析. 海洋科学 , 2012, 36 (2) : 1-6 | |
| 战爱斌, 包振民, 陆维, 等. 仿刺参的微卫星标记. 水产学报 , 2006, 30 (2) : 192-196 | |
| 姜群, 于红, 孔令锋, 等. 太平洋牡蛎不同组织DNA甲基化的F-MSAP分析. 中国水产科学 , 2014, 21 (4) : 676-683 | |
| 宫时玉, 蒋曹德, 邓昌彦. DNA甲基化及其生物学功能. 华中农业大学学报(自然科学版) , 2005, 24 (6) : 651-657 | |
| 徐青, 张沅, 孙东晓, 等. 应用MSAP方法检测鸡不同组织基因组的甲基化状态. 遗传 , 2011, 33 (6) : 620-626 | |
| 郭婷婷, 孙国华, 杨建敏, 等. 刺参(Apostichopus japonicus)不同组织基因组甲基化状态MSAP分析. 海洋与湖沼 , 2013, 44 (1) : 77-82 | |
| 曹哲明, 丁炜东, 俞菊华, 等. 草鱼全同胞鱼苗不同个体甲基化位点的差异. 动物学报 , 2007, 53 (6) : 1083-1088 | |
| 曹哲明, 杨健. 背角无齿蚌不同组织的基因组DNA甲基化分析. 生态环境学报 , 2009, 18 (6) : 2011-2016 | |
| 廖玉麟. 中国动物志: 棘皮动物门海参纲. 北京: 科学出版社, 1997 : 1 -2. | |
| Aina R, Sgorbati S, Santagostino A, et al. Specific hypomethylation of DNA is induced by heavy metals in white clover and industrial hemp. Physiologia Plantarum , 2004, 121 (3) : 472-480 DOI:10.1111/ppl.2004.121.issue-3 | |
| Beaujean N, Taylor J, Gardner J, et al. Effect of limited DNA methylation reprogramming in the normal Sheep embryo on somatic cell nuclear transfer. Biol Reprod , 2004, 71 (1) : 185-193 DOI:10.1095/biolreprod.103.026559 | |
| Bird AP, Taggart MH, Smith BA. Methylated and unmethylated DNA compartments in the Sea Urchin Genome. Cell , 1979, 17 (4) : 889-901 DOI:10.1016/0092-8674(79)90329-5 | |
| Brske A, Vockentanz L, Kharazi S, et al. DNA methylation protects hematopoietic stem cell multipotency from myeloerythroid restriction. Nat Genet , 2009, 41 (11) : 1207-1215 DOI:10.1038/ng.463 | |
| Feng SH, Jacobsen SE, Reik W. Epigenetic reprogramming in plant and animal development. Science , 2010, 330 (6004) : 622-627 DOI:10.1126/science.1190614 | |
| Henikoff S, Matzke MA. Exploring and explaining of epigenetic effects. Trends Genetics , 1997, 13 (8) : 293-295 DOI:10.1016/S0168-9525(97)01219-5 | |
| Holiday R, Pugh JE. DNA modification mechanisms and gene activity during development. Science , 1975, 187 (4173) : 226-232 DOI:10.1126/science.1111098 | |
| Jiang Q, Li Q, Yu H, et al. Genetic and epigenetic variation in mass selection populations of Pacific oyster Crassostrea gigas. Genes Genom , 2013, 35 (5) : 641-647 DOI:10.1007/s13258-013-0114-4 | |
| Labra M, Grassi F, Imazio S, et al. Genetic and DNA- methylation changes induced by potassium dichromate in Brassica napus L. Chemosphere , 2004, 54 (8) : 1049-1058 DOI:10.1016/j.chemosphere.2003.10.024 | |
| Rai K, Nadauld LD, Chidester S, et al. Zebra Fish Dnmt1 and Suv39h1 regulate organ-specific terminal differentiation during development. Mol Cell Biol , 2006, 26 (19) : 7077-7085 DOI:10.1128/MCB.00312-06 | |
| Regev A, Lamb MJ, Jablonka E. The role of DNA methylation in invertebrates: developmental regulation or genome defense. Mol Biol Evol , 1998, 15 (7) : 880-891 DOI:10.1093/oxfordjournals.molbev.a025992 | |
| Reyna-Lopez GE, Simpson J, Ruiz-Herrera J. Difference in DNA methylation patterns are detectable during the dimorphic transition of restriction polymorphism. Mol Gen Genet , 1997, 253 (6) : 703-710 DOI:10.1007/s004380050374 | |
| Roberts SB, Gavery MR. Is there a relationship between DNA methylation and phenotypic plasticity in invertebrates. Front Physiol , 2012, 2 : 116 | |
| Sun Y, Hou R, Fu XT, et al. Genome-wide analysis of DNA methylation in five tissues of Zhikong Scallop, Chlamys farreri. PLoS One , 2014, 9 (1) : 1-8 | |
| Takemae N, Nakaya F, Motokawa T. Low oxygen consumption and high body content of catch connective tissue contribute to low metabolic rate of sea cucumbers. Biol Bull , 2009, 216 (1) : 45-54 DOI:10.1086/BBLv216n1p45 | |
| Tsaftaris AS, Kafka M. Mechanism of heterosis in crop plants. J Crop Prod , 1998, 1 (1) : 95-111 | |
| Vandegehuchte MB, Kyndt T, Vanholme B, et al. Occurrence of DNA methylation in Daphnia magna and influence of multigeneration Cd exposure. Environ Int , 2009, 35 (4) : 700-706 DOI:10.1016/j.envint.2009.01.002 | |
| Waalwijk C, Flavell RA. DNA methylation at a CCGG sequence in the large intron of the rabbit beta-globin gene: tissue- specific variations. Nucleic Acids Res , 1978, 5 (12) : 4631-4634 DOI:10.1093/nar/5.12.4631 | |
| Wang WW, Zhao XQ, Pan Y, et al. DNA methylation changes detected by methylation-sensitive amplified polymorphism in two contrasting rice genotypes under salt stress. J Genet Genomics , 2011, 38 (9) : 419-424 DOI:10.1016/j.jgg.2011.07.006 | |
| Xiong LZ, Xu CCCG, Saghai Maroof MA, et al. Patterns of cytosine methylation in an elite rice hybrid and its parental lines, detected by a methylation-sensitive amplification polymorphism technique. Mol Gen Genet , 1999, 261 (3) : 439-446 DOI:10.1007/s004380050986 | |
| Zhang MS, Yan HY, Zhao N, et al. Endosperm-specific hypomethylation, and meiotic inheritance and variation of DNA methylation level and pattern in sorghum (Sorghum bicolor L. ) inter-strain hybrids. Theor Appl Genet , 2007, 115 (2) : 195-207 | |
| Zhao Y, Chen MY, Storey KB, et al. DNA methylation levels analysis in four tissues of sea cucumber (Apostichopus japonicus) based on fluorescence-labeled methylation- sensitive amplified polymorphism (F-MSAP) during aestivation. Comp Biochem Phys B: Biochem Mol Biol , 2015, 181 : 26-32 DOI:10.1016/j.cbpb.2014.11.001 |



