2. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 上海海洋大学水产与生命学院 上海 201306;
4. 中国海洋大学海洋生命学院 青岛 266003
2. National Laboratory for Ocean Science and Technology, Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao 266071 ;
3. College of Fisheries and Life Sciences, Shanghai Ocean University, Shanghai 201306 ;
4. College of Marine Life Sciences, Ocean University of China, Qingdao 266003
半滑舌鳎(Cynoglossus semilaevis)隶属于鲽形目(Pleuronectiformes)、舌鳎科(Cynoglossidae)、舌鳎属(Cynoglossus),主要分布在我国黄、渤海海域,是暖温性近海大型底栖鱼类,具有广温、广盐和适应环境多变的特点,其生长速度快、肉味鲜美、口感爽滑、出肉率高,深受广大消费者喜爱(邓景耀等,1988)。近年来,高密度、集约化的养殖模式以及环境污染等原因,致使半滑舌鳎的腹水、烂鳍、烂尾等细菌性感染问题日益突出,严重制约了半滑舌鳎工厂化养殖产业的发展。因此,开展半滑舌鳎抗病相关基因的分析研究,筛选其抗病相关的分子标记,对于半滑舌鳎抗性品系培育以及该养殖产业的健康可持续发展具有十分重要的意义。
天然抗性相关巨噬细胞蛋白(Nramp)属于膜整合转运蛋白,具有抑制胞内寄生菌侵染、调节巨噬细胞的抗菌活性等作用(Blackwell et al,1995),在物种间保持着高度的保守性(Skamene et al,1991),由于其编码蛋白具有转运质子和二价阳离子的功能,因此,又将其称为溶质转运家族11成员1(Solution carrier family 11 member 1,SLC11A1) (Blackwell et al,1996)。该家族一般具有10−12个典型的跨膜结构,含有1个胞质内转运蛋白特征结构域,以及1−2个糖基化的胞质外环状结构(Bairoch,1993)。Nramp基因最早由Vidal等(1993) 在近交小鼠的研究中发现,该基因可影响宿主对杜氏利什曼原虫、分支杆菌、伤寒沙门氏菌等胞内寄生菌的早期免疫反应。目前,已在人和小鼠等哺乳动物中发现两种天然抗性相关巨噬细胞蛋白即Nramp1和Nramp2(Vidal et al,1993; Kishi,1994; Grunheid et al,1995; Kishi et al,1997)。Nramp1是一个较为保守的基因,主要在网状内皮细胞器官如吞噬细胞、脾脏、肝脏、外周血白细胞中特异表达(Cellier et al,1994; Feng et al,1996),而Nramp2则在绝大多数组织和细胞中均表达(Grunheid et al,1995),在哺乳动物体内对铁的吸收转运和重新利用起到决定性的调控作用(Gunshin et al,1997; Fleming et al,1998)。目前,鱼类中已在虹鳟(Oncorhynchus mykiss)(Dorschner et al,1999)、鲤鱼(Cyprinus carpio)(Saeij et al,1999)、斑点叉尾鮰(Ictalurus punctatus)(Chen et al,2002)、鲈鱼(Lateolabrax japonicus)(Burge et al,2004)、真鲷(Pagrosomus major)(Chen et al,2004)、牙鲆(Paralichthys olivaceus)(Chen et al,2006)、大菱鲆(Scophthalmus maximus)(Chen et al,2007)、草鱼(Ctenopharyngodon idellus)(范玉顶等,2011)等物种中进行了Nramp基因克隆,并对该基因的特征序列及病原菌刺激后的组织和细胞系表达进行了研究,但有关Nramp基因多态性与疾病的关联分析报道较少。本研究对半滑舌鳎Nramp基因进行了克隆、序列比对、组织表达分析,并首次在鱼类中进行Nramp基因多态性与抗病分子标记的筛选,为半滑舌鳎抗性家系的分子标记辅助育种提供了技术支持。
1 材料与方法 1.1 材料健康1龄半滑舌鳎,体重为(145.01±60.02) g,体长为(27.68±5.53) cm,来自山东省海阳市黄海水产有限公司,于实验室暂养7 d无异常后使用。取半滑舌鳎的肝、脾、肾、肠、鳃、血、脑、心、皮肤、肌肉、性腺等组织,迅速放入液氮中,−80℃保存备用。半滑舌鳎感染鳗弧菌家系(1龄)由本实验室2012年于山东省昌邑市三新苗种研究所建立。哈维氏弧菌(Vibrio harveyi)菌种由本实验室保存。
1.2 基因组DNA和总RNA提取及cDNA合成采用常规的酚-氯仿法(Sambrook et al,2001)提取基因组DNA,双蒸水溶解后,利用紫外分光光度计(Biophotometer,Eppendorf)测定DNA浓度,并经1%琼脂糖凝胶电泳检测其完整性,−20℃保存备用;利用总RNA极速抽提试剂盒(上海飞捷生物有限公司)提取各样品总RNA,紫外分光光度计测定RNA浓度,并经1%琼脂糖凝胶电泳检测其完整性;用TaKaRa反转录试剂盒合成cDNA链,−20℃保存备用。
1.3 引物设计及PCR扩增提取半滑舌鳎脾脏中的总RNA,并反转录cDNA(TaKaRa),根据本实验室半滑舌鳎转录组测序得到Nramp转录本信息合成引物Nramp-F/Nramp-R(表 1),以半滑舌鳎脾脏cDNA为模板,经PCR扩增、克隆,测序进行序列验证,根据验证无误后的基因片段序列设计4条特异性引物(表 1),分别进行5´ RACE和3´ RACE反应,获得的片段经过克隆、测序拼接后,得到cDNA全长[具体方法参见SMARTTM RACE cDNA Amplification Kit(Clontech)说明书]。
|
|
表 1 PCR扩增所用引物序列 Table 1 PCR amplification primers used in this study |
实时荧光定量PCR:以β-actin(KF932267) 为内参,用基因特异性引物RT-F和RT-R检测哈维氏弧菌感染前后,鱼的不同组织在不同时间的Nramp表达,实验样本重复数为5(具体方法参见TaKaRa定量试剂盒)。
1.4 目的片段纯化、克隆及测序克隆Nramp基因的PCR产物经1%的琼脂糖凝胶电泳检测,将目的片段切胶回收[具体方法参照Gel Extraction Kit(OMEGA)试剂盒说明书]。将回收产物与pMD-18T载体(TaKaRa)按摩尔数5∶1的比例混合,加入等体积的Solution I于16℃连接,取连接产物10 µl转化至TOP10大肠杆菌(Escherichia coli)感受态细胞,菌液PCR鉴定出阳性克隆后,送上海英潍捷基贸易有限公司测序。
用于筛选Nramp基因SNP的PCR产物,取5 µl经1%的琼脂糖凝胶电泳检测,将条带清晰单一的PCR产物直接送上海英潍捷基贸易有限公司测序。
1.5 序列分析和数据统计利用NCBI网站BLAST工具对测序后拼接结果进行同源性比对;用Signal P(Nielsen et al,1997)分析信号肽序列;利用ScanProsite在线服务器(http://prosite.expasy.org/scanprosite)分析蛋白的二级结构;用DNASTAR 5.0软件分析cDNA序列和开放阅读框;用Clustal W软件(Kyte et al,1982)进行多重序列比对;利用MEGA 4.0(Tamura et al,2007)中的邻位相联法(Neighbor-Joining,NJ)(Saitou et al,1987)构建系统进化树;采用Kyte and Doolittle算法(Kyte et al,1982),通过ProtScale在线工具(http://web.expasy.org/protscale)分析蛋白的亲水性特征;运用PopGene 32分析基因型频率、等位基因频率、Hardy-Weinberg平衡检验等;利用SPSS 17.0和SAS(Version 9.1) 软件对SNP位点与性状关联性进行卡方检验。
2 结果与分析 2.1 半滑舌鳎Nramp基因全长cDNA的克隆利用引物Nramp-F/Nramp-R,以半滑舌鳎脾脏cDNA为模板,PCR扩增得到长度为1085 bp的目的条带,经测序及BLAST比对分析,确定该条带为Nramp基因片段;根据此片段设计合成4条特异性引物进行RACE扩增,分别得到5´ RACE 772 bp和3´ RACE 2068 bp。用软件DNASTAR拼接得到半滑舌鳎Nramp基因全长cDNA序列为3717 bp(GenBank序列号:KP878556) 。半滑舌鳎Nramp全长cDNA由1677 bp的开放阅读框(ORF)、较短的172 bp 5´末端非翻译区(Untranslated Regions,UTR)和一个相对较长的1868 bp的3´ UTR组成。3´末端含有1个典型的加尾信号AATAAA和30 bp的Poly(A)尾;此外,在ORF末端上发现了1个铁反应控制蛋白结合位点(IRE)的特征序列(CNNNNNCAGTG)(Casey et al,1988)(图 1)。

|
图 1 半滑舌鳎Nramp全长cDNA序列及推测出的氨基酸序列 Figure 1 Full-length cDNA and predicted amino-acid sequence of C. semilaevis' Nramp gene 跨膜区(TM)用下划虚线标出,并编号TM 1−10;位于TM6和TM7之间的转运结构域(CTM)用方框和下划虚线标出;加尾信号(aataaa)用阴影和方框标出;位于ORF末端的IRE位点用阴影和下划线标出;N-糖基化位点用双虚线标出;酪蛋白激酶Ⅱ磷酸化位点用方框标出;蛋白激酶C磷酸化位点用下划线标出;酪氨酸激酶磷酸化位点用双下划线标出;N-豆蔻酰化位点用阴影标出 The transmembrane regions(TM)are underlined with broken lines and numbered 1−10. The consensus transport motif(CTM)between TM6 and TM7 is boxed and underlined with broken line. The poly A signal(aataaa)is boxed and shaded. The IRE site located in terminal of ORF is underlined and shaded. The N-glycosylation sites are marked with double broken lines. The casein kinase Ⅱphosphorylation sites are boxed. The predicted protein kinase C phosphorylation sites are underlined with single lines. A tyrosine kinase phosphorylation site is underlined with double lines. The N-myristoylation sites are shown with shade |
根据得到的半滑舌鳎Nramp基因cDNA序列推导其相应的氨基酸序列,结果分析显示,1677 bp的ORF序列编码1个含558个氨基酸的蛋白,预测相对分子量为61.9 kDa,等电点为4.95。利用ScanProsite分析该蛋白的二级结构(图 1),发现该蛋白具有Nramp家族的典型特征:10个跨膜区(TM),1个由20个氨基酸残基组成的、高度保守的胞质内转运结构域(CTM)介于TM6和TM7之间,3个潜在的N-糖基化作用位点(N-X-S/T-X),且均位于TM5和TM6之间,两个蛋白激酶C磷酸化作用位点(S/T-X-R/K),分别位于TM1之前的N端和TM10之后的C端;该蛋白还含有1个位于TM4和TM5之间的酪氨酸激酶磷酸化作用位点(R/K-X-X-X-D/E-X-X-X-Y),13个N-豆蔻酰化作用位点(G-[EDRKHPYFW]-X-X-[STAGCN]-P)以及6个酪蛋白激酶Ⅱ磷酸化作用位点(S/T-X-X-D/E)(图 1)。从蛋白的氨基酸组成上看,Leu、Val、Ile、Ala、Phe、Pro、Met以及Trp等非极性氨基酸占52.6%,表明该蛋白具有较强的疏水性,通过ProScale亲水性分析的结果也进一步验证了这一典型特征(图 2),鱼类Nramp的亲水性分布图与小鼠和人Nramp2几乎一致,半滑舌鳎Nramp与牙鲆、大菱鲆、斑点叉尾鮰等鱼类的Nramp在胞质内外环的间隔大小上也高度保守。
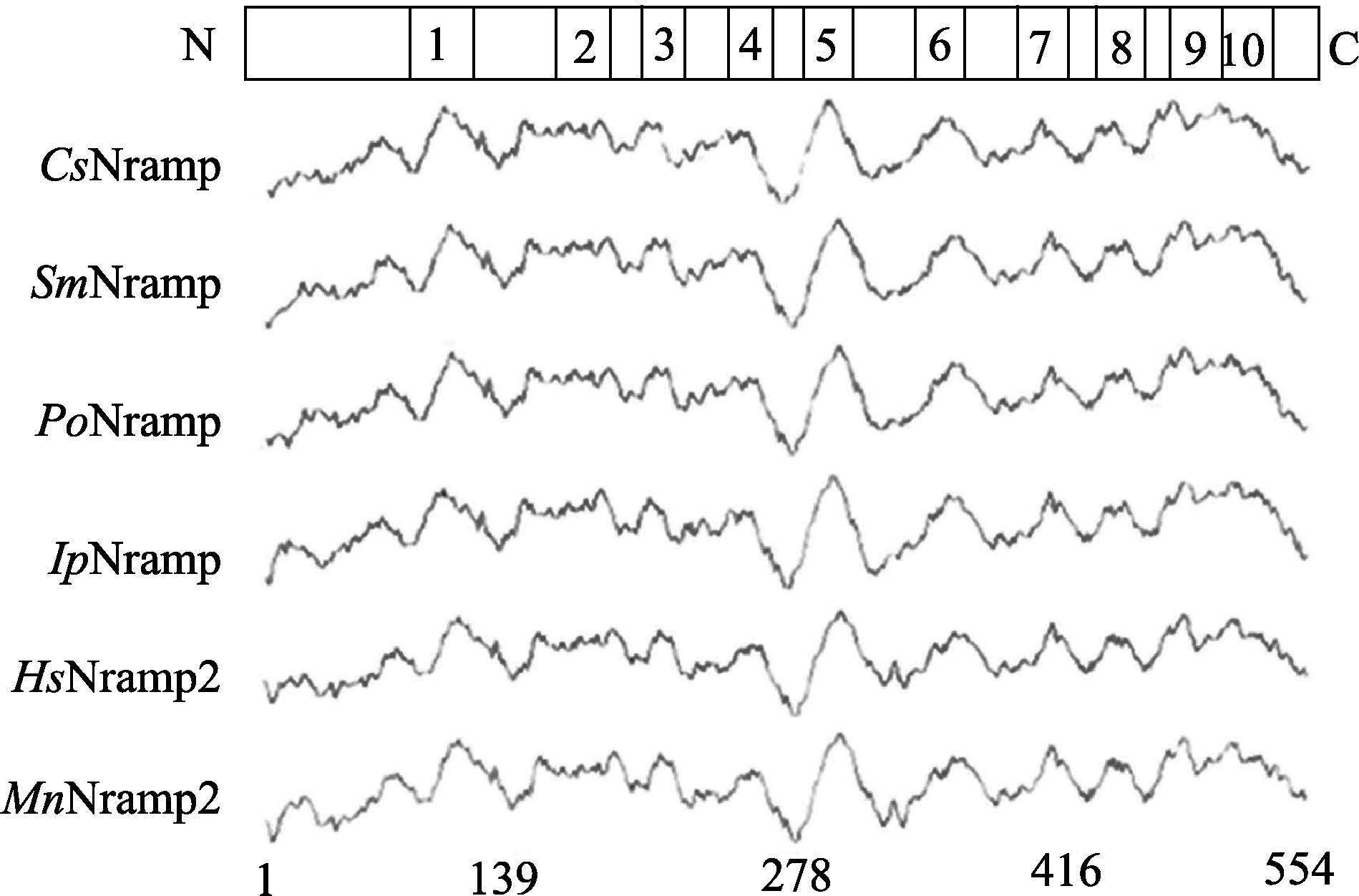
|
图 2 几种脊椎动物Nramp蛋白的亲水性分布 (Kyte和Doolittle算法) Figure 2 Hydropathy profile conservation among seleted vertebrates' Nramp proteins(Kyte and Doolittle algorithm) CsNramp:半滑舌鳎Nramp;SmNramp:大菱鲆Nramp;PoNramp:牙鲆Nramp;IpNramp:斑点叉尾鮰 Nramp;HsNramp2:人Nramp2;MnNramp2:小鼠Nramp2;上面一行数字代表半滑舌鳎Nramp基因的跨膜区;下面一行数字代表6个物种Nramp的氨基酸数目 CsNramp: C. semilaevis Nramp; SmNramp: Scophthalmus maximus Nramp; PoNramp: Paralichthys olivaceus Nramp; IpNramp: Ictalurus punctatus Nramp; HsNramp2: Homo sapiens Nramp2; MnNramp2: Mus musculus Nramp2. Numbers above are the TMs ofCsNramp. Numbers below are the amino acid numbers of six Nramp proteins |
利用Clustal W软件对半滑舌鳎Nramp的氨基酸序列同人、小鼠、牛、绵羊、原鸡及其他鱼类Nramp氨基酸序列进行了比对分析(图 3),发现所比对物种的氨基酸序列在CTM和TM区均相对保守,尤其是TM4区,所有鱼类中Nramp完全保守,和其他物种Nramp也只有1个氨基酸的差异;鱼类的Nramp在TM1、TM2、TM6、TM8区氨基酸的保守性要显著高于TM3、TM5、TM7、TM9、TM10区。在CTM区,除小鼠、人1、绵羊和野猪中有1个氨基酸残基(A)的差异外,在其他的鱼类、两栖类和哺乳类Nramp中高度保守(图 3)。此外,位于TM5和TM6之间的两个N-糖基化作用位点在所比较的物种间保守性较高,其中,靠近TM5区的N-糖基化作用位点在所有鱼类Nramp中高度保守,而接近于TM6区的,除虹鳟Nramp alpha外,在所比较的其他物种间均表现为高度保守。同时本研究还发现,位于TM5和TM6之间,所比较物种的Nramp1均比Nramp2多1个氨基酸残基,这与草鲤鱼和斑点叉尾鮰 Nramp与其他物种Nramp氨基酸序列比对结果是一致的(Chen et al,2002; 范玉顶等,2011)。
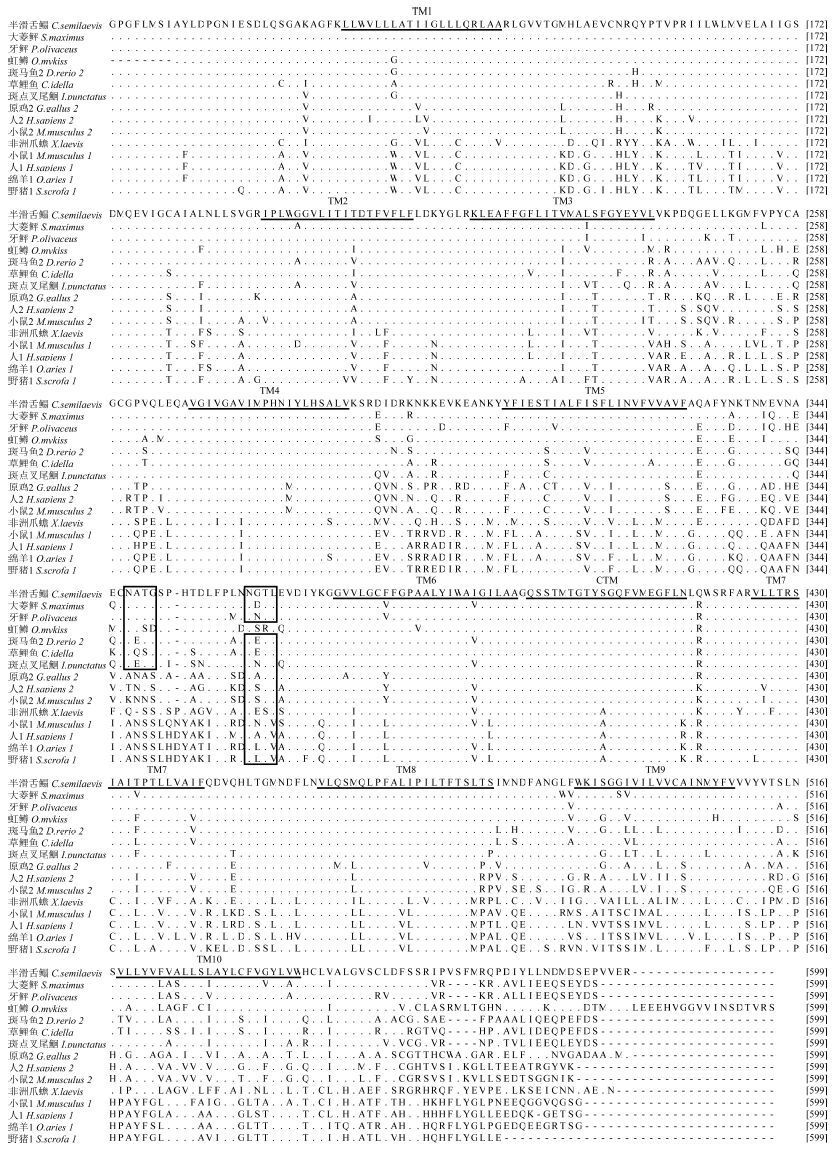
|
图 3 半滑舌鳎和其他物种Nramp氨基酸序列比对分析 Figure 3 Alignment of Nramp's amino acid sequences between C. semilaevis and other vertebrates 半滑舌鳎Nramp的10个TM区及CTM区用下划线标出;保守的N-糖基化位点以方框标出; “∙”表示与半滑舌鳎氨基酸相同的位点;“-”表示此位点为空格 Putative transmembrane regions are underlined and numbered with TM 1−10. The consensus transport motif(CTM)is underlined. Conserved N-glycosylation sites are marked with boxes. Identical sites are indicated by dots(∙),and gaps are shown by dashes(-) |
将半滑舌鳎Nramp的氨基酸序列与其他物种的Nramp氨基酸序列进行了比对分析,并在此基础上构建了系统进化树(图 4)。系统进化树的分析结果表明,半滑舌鳎Nramp和其他鱼类的Nramp聚为一簇,又和鸟类与哺乳类Nramp2聚在一起形成一个分支,而哺乳类Nramp1则单独构成另一个分支,由此可知鱼类的Nramp与哺乳类和鸟类的Nramp2更为类似;与半滑舌鳎Nramp亲缘关系最近的是大菱鲆和牙鲆。
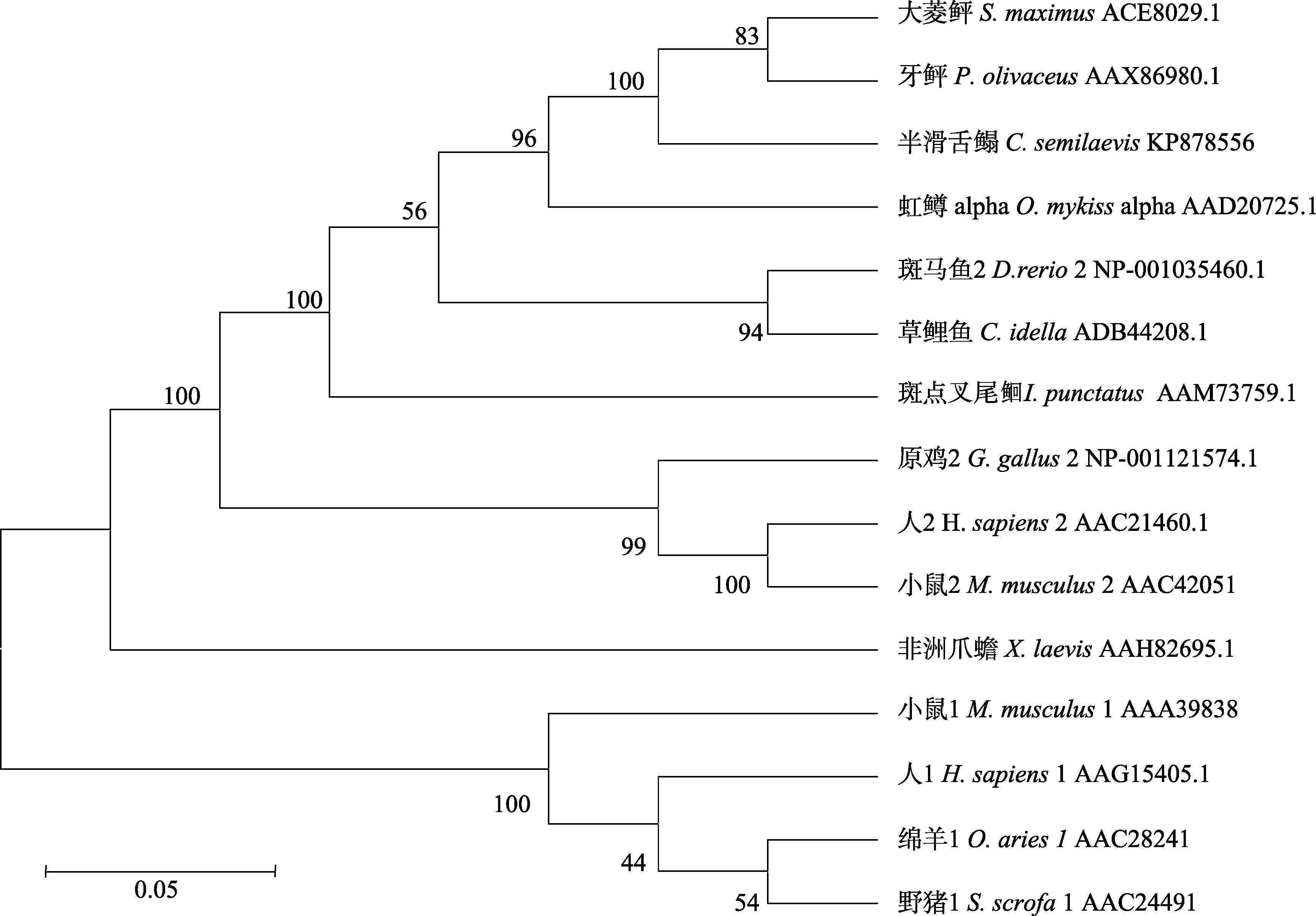
|
图 4 半滑舌鳎和其他物种Nramp氨基酸序列构建的系统发生树(利用Bootstrap法进行1000次评估) Figure 4 Construction of phylogenetic tree based on Nramp amino acids of C. semilaevis and other species (the parameter was evaluated 1000 degree via method of Bootstrap) |
对Nramp基因在半滑舌鳎的肝脏、脾脏、头肾、肠、鳃、血液、脑、心脏、皮肤、肌肉、性腺等11种组织进行实时荧光定量PCR表达分析,发现在所检测的11种组织中Nramp基因表达量差异明显,脾脏和肾脏中的表达量最高,其次是肝脏、皮肤、血液、鳃、肠、心脏和脑,而肌肉和性腺中的表达量最低(图 5)。利用实时荧光定量PCR对哈维氏弧菌感染后半滑舌鳎不同组织Nramp基因表达量进行了分析,结果显示,与PBS对照组相比,感染半滑舌鳎的脾脏、肾脏和肝脏中Nramp基因表达量呈升高趋势,其中,脾脏和肝脏均在感染哈维氏弧菌后24 h表达量最高(图 6、图 7),而在肾脏中则是6 h达到最大值,96 h后表达量基本又回落至对照组的水平(图 8),在鳃中则呈先降低后恢复至正常表达水平的趋势(图 9)。
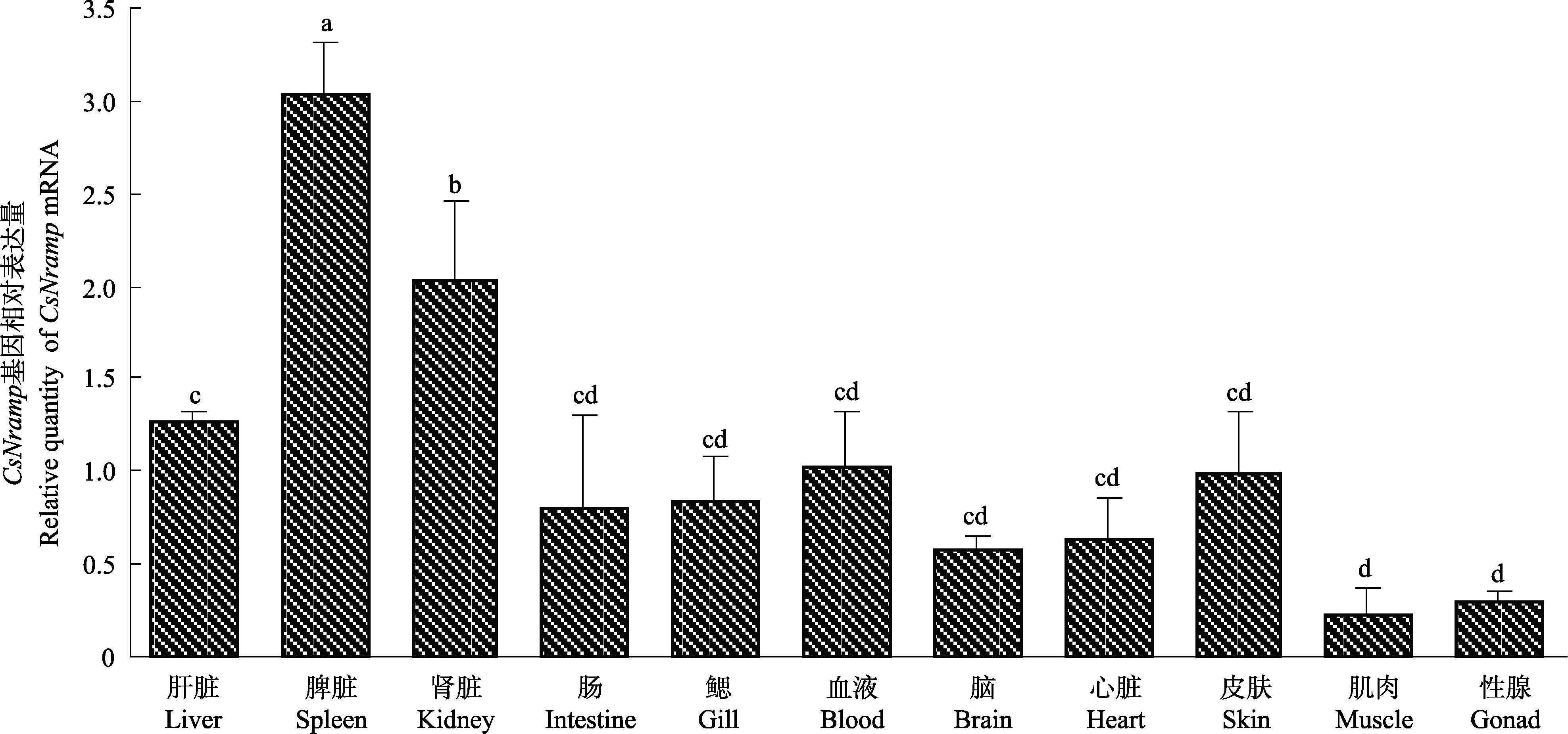
|
图 5 半滑舌鳎Nramp基因各组织相对表达量 Figure 5 Quantitative relative expression of CsNramp in different tissues 各组织相对定量表达分析数据取自5条健康成鱼,以血液中Nramp基因表达量为标准, 用单因素方差分析数据,每个柱子上面的不同字母表示显著差异(P<0.05) Analysis of the relative tissue expression data are from five fish. All results were normalized by the blood Nramp expression levels. Statistical analysis was performed using One-Way ANOVA. Different letters above each bar denote significant differences(P<0.05) |
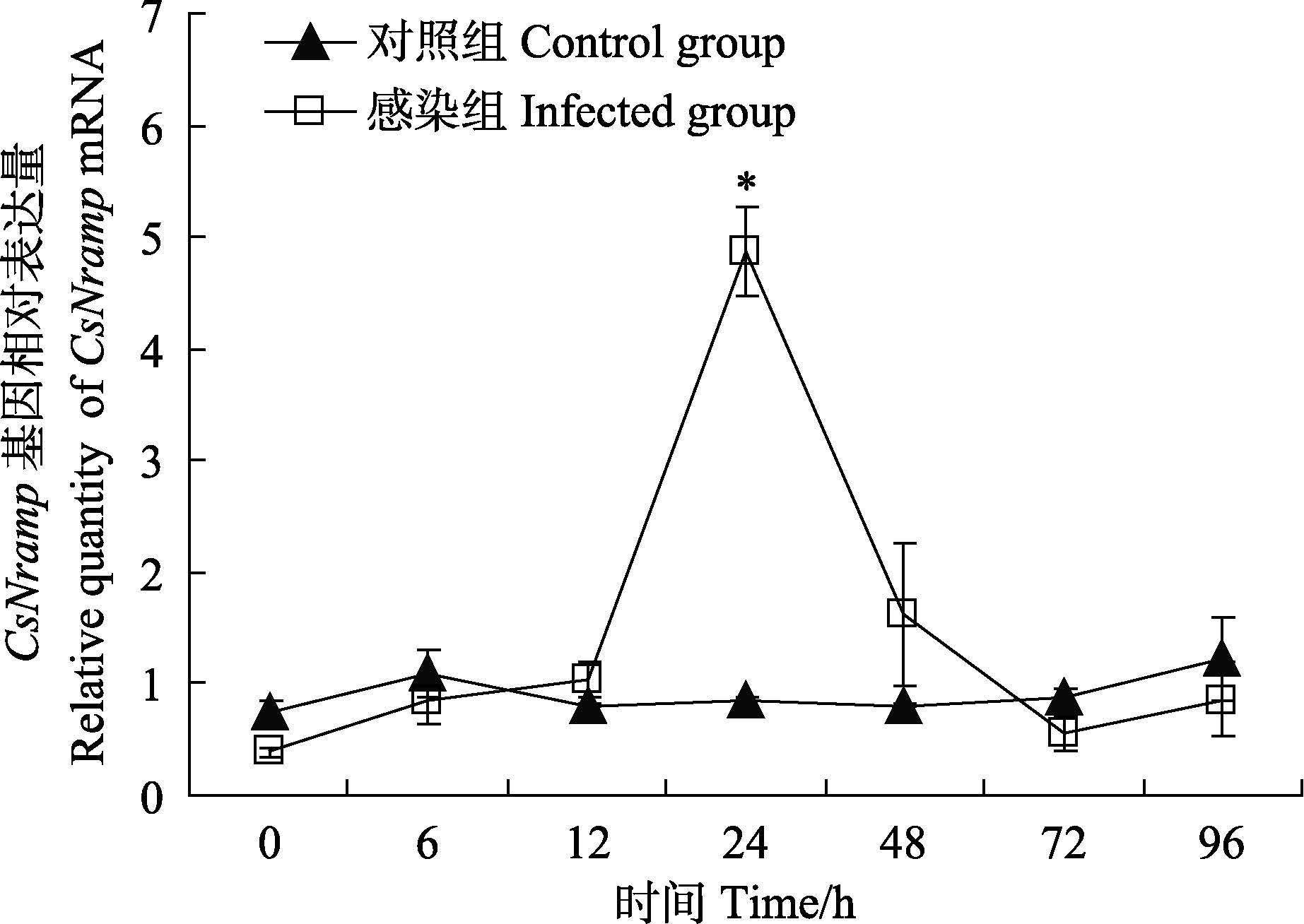
|
图 6 感染哈维氏弧菌后半滑舌鳎肝脏中Nramp 基因相对表达量 Figure 6 Quantitative relative expression of CsNramp in liver after injected with V. harveyi 感染组:腹腔注射哈维氏弧菌,剂量为30 μl/g,滴度为 6.0×105 CFU/ml;对照组:腹腔注射等量的PBS溶液;在感染后0、6、12、24、48、72和96 h共6个时间点,随机选取感染组和对照组各5条半滑舌鳎,解剖鱼体获得肝脏后立即放入液氮中暂存,后转移至−80℃保存,用于总RNA的提取;利用单因素方差分析方法数据,*差异表示显著(P<0.05) ;下同 Injection group: C. semilaevis was injected intraperitoneally with V. harveyi(30 μl/g)or with equal dose of PBS. Five fish's liver tissues were collected at 0,6,12,24,48,72,and 96 h after injection for RNA extraction. Statistical analysis was performed using One-Way ANOVA. * denotes significant difference(P<0.05) . The same as below |
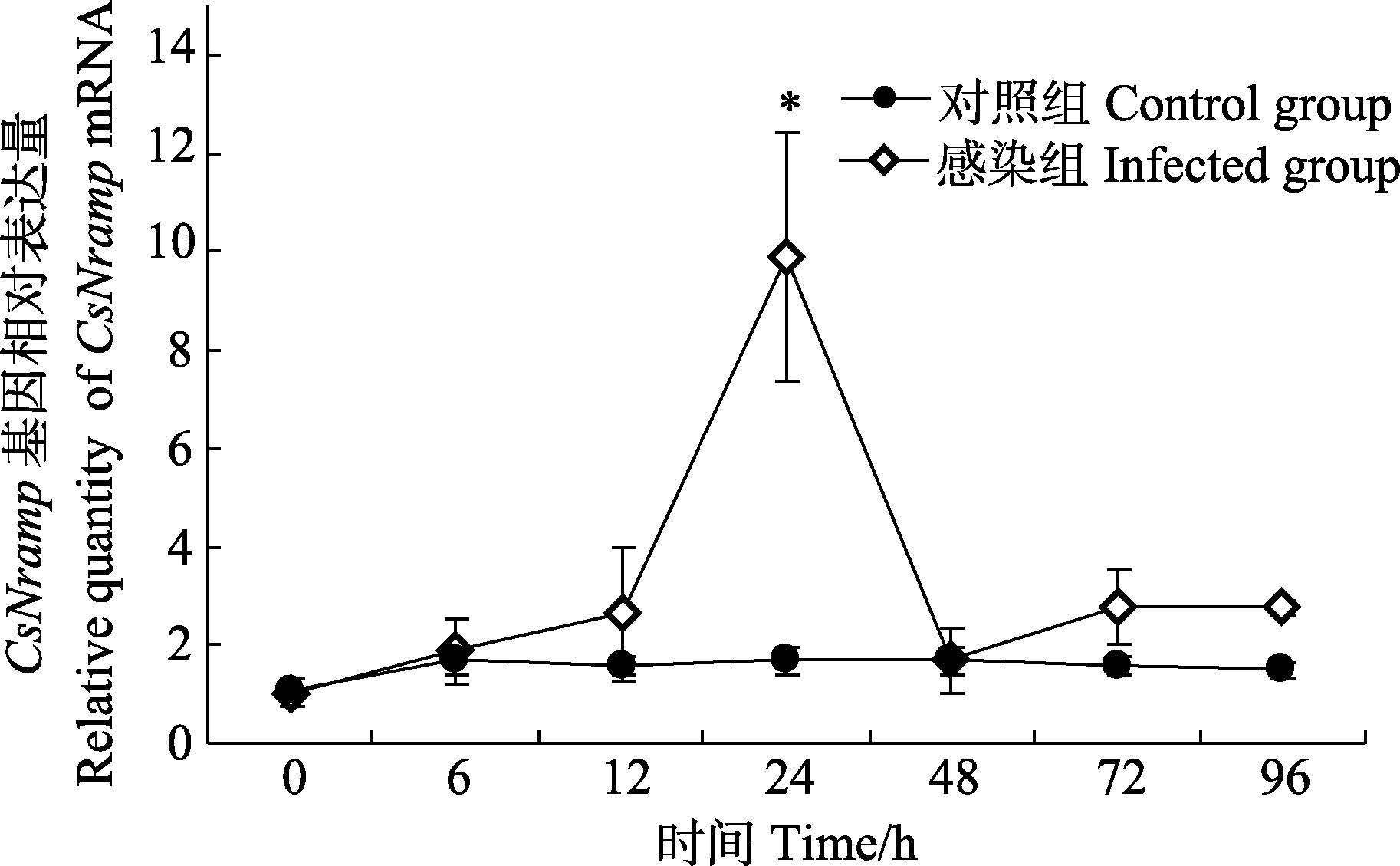
|
图 7 感染哈维氏弧菌后半滑舌鳎脾脏中 Nramp基因相对表达量 Figure 7 Quantitative relative expression of CsNramp in spleen after injected with V. harveyi |
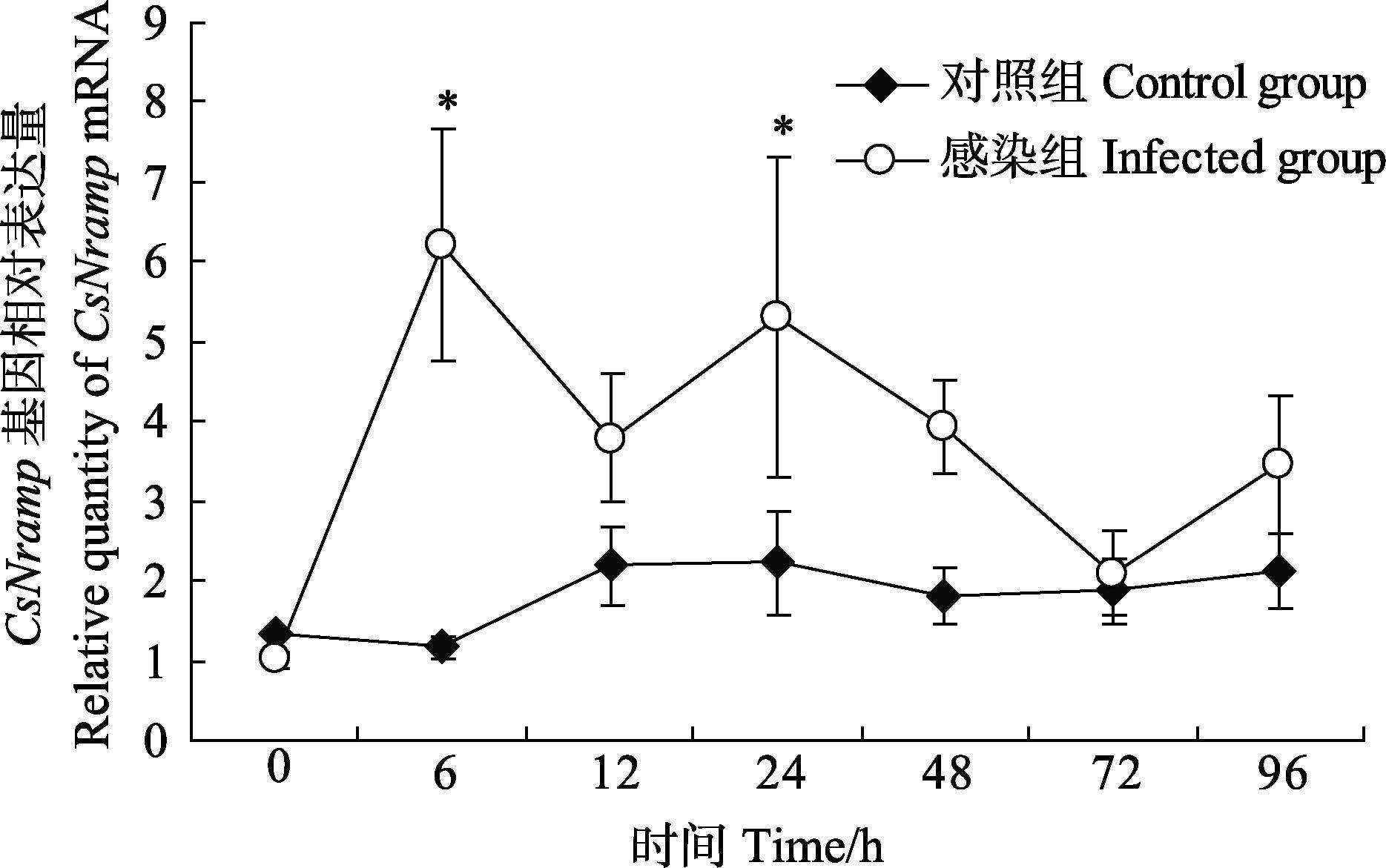
|
图 8 感染哈维氏弧菌后半滑舌鳎肾脏中 Nramp基因相对表达量 Figure 8 Quantitative relative expression of CsNramp in kidney after injected with V. harveyi |
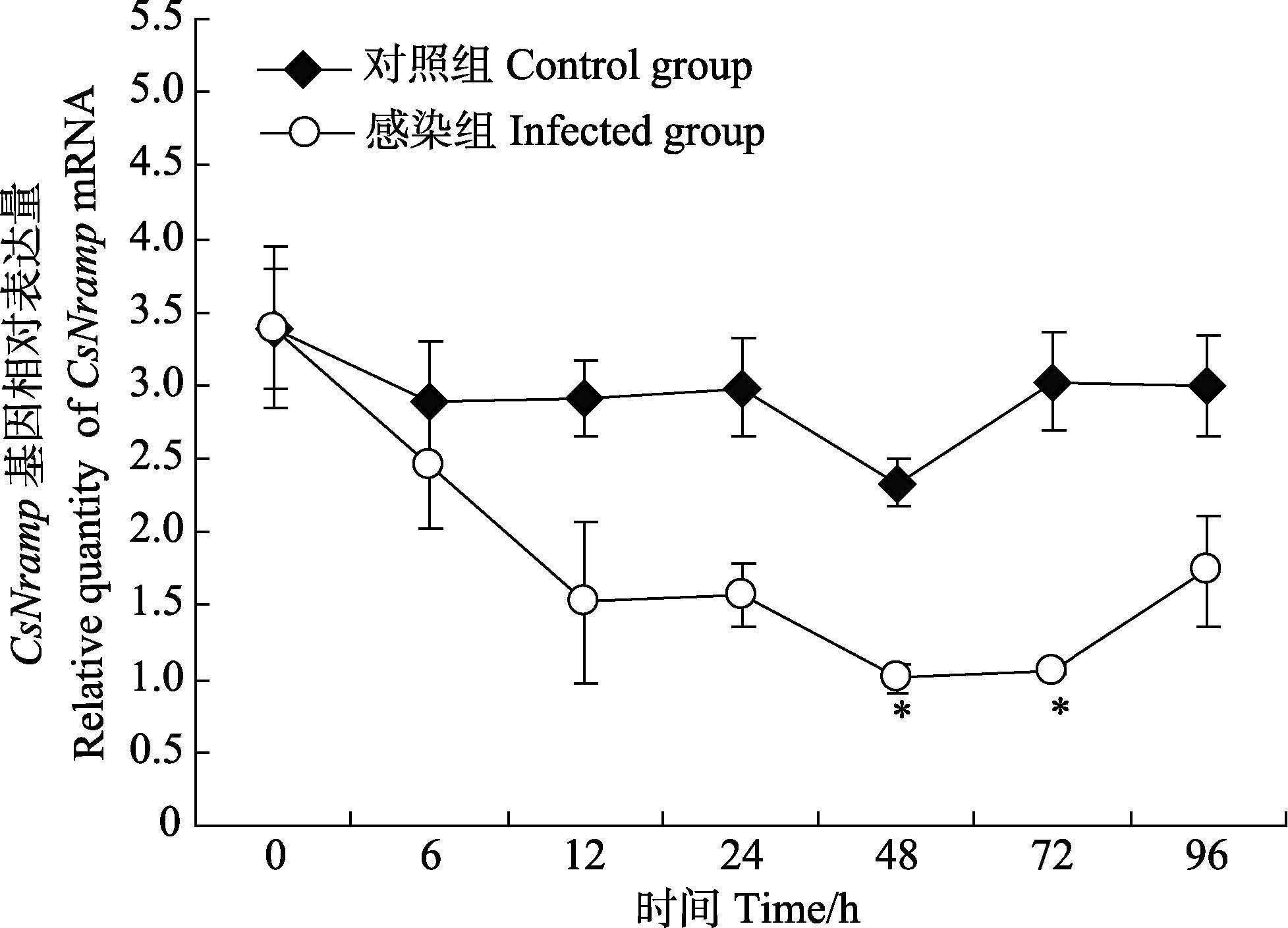
|
图 9 感染哈维氏弧菌后半滑舌鳎鳃中Nramp 基因相对表达量 Figure 9 Quantitative relative expression of CsNramp in gill after injected with V. harveyi |
利用直接测序法检测分析,在1402 bp Nramp序列中,共检测到15个SNP位点,对其中位于第2内含子的3个SNP[SNP-g.3113(T→C)、SNP-g.3125(A→G)和SNP-g.3164(A→T)]位点成功测序分型。对所分型的SNP位点的基因型频率与等位基因频率记录并进行哈德温伯格平衡检验,统计结果见表 2;用软件SPSS 17.0和SAS(Version 9.1) 对其进行性状关联分析,等位基因频率和基因型频率在抗性组及易感组中的卡方检验结果见表 3,其中,SNP-g.3125(A→G)的等位基因频率和基因型频率与半滑舌鳎对于鳗弧菌的抗性显著相关(P<0.01) 。
|
|
表 2 半滑舌鳎Nramp基因内含子2多态性的统计分析 Table 2 Statistical analysis of polymorphism of CsNramp intron 2 |
|
|
表 3 半滑舌鳎Nramp基因SNP与鳗弧菌的关联分析 Table 3 Association analysis of single SNP of CsNramp with V. anguillarum |
本研究克隆得到了半滑舌鳎Nramp基因的全长cDNA。与已报道的脊椎动物Nramp氨基酸序列比对分析结果表明,半滑舌鳎Nramp与其他鱼类Nramp的同源性在83%−91%之间,与其他脊椎动物Nramp2的同源性(74%−78%)要明显高于与Nramp1的同源性(63%−66%);系统进化分析的结果也进一步表明,鱼类的Nramp与其他脊椎动物Nramp2聚在一起,因此,半滑舌鳎Nramp基因与其他脊椎动物的Nramp2基因更为相似,这与在其他鱼类中Nramp基因分析得到的结论是一致的(Chen et al,2002; Chen et al,2004、2006; Dorschner et al,1999; Saeij et al,1999)。
半滑舌鳎Nramp蛋白含有10个TM、1个CTM、6个酪蛋白激酶Ⅱ磷酸化位点、13个N-豆蔻酰化位点、两个蛋白激酶C磷酸化位点、3个N-糖基化位点、1个酪氨酸激酶磷酸化位点等,这与人(Kishi,1994; Kishi et al,1997)、小鼠(Grunheid et al,1995; Govoni et al,1997)、鲤(Saeij et al,1999)、斑点叉尾鮰(Chen et al,2002)、虹鳟(Chen et al,2004)、牙鲆(Chen et al,2006)、大菱鲆(Chen et al,2007)以及草鱼(范玉顶等,2011)序列特征大致相似。此外,本研究发现,在半滑舌鳎Nramp基因ORF末端有1个IRE位点(CNNNNNCAGTG),而草鱼Nramp基因5´ UTR和3´ UTR均发现1个IRE位点(范玉顶等,2011),以及鲤鱼的3´ UTR发现1个IRE位点(Saeij et al,1999)。已有研究表明,Nramp基因5´ UTR和3´ UTR端IRE位点与细胞中铁离子的代谢紧密相关(Klausner et al,1993),在哺乳动物Nramp2基因的研究中,发现该位点与铁离子的转运和吸收密切相关(Forbes et al,2001; Gunshin et al,1997)。据Saeij等(1999) 推测,在鲤鱼Nramp 3´ UTR端发现的IRE位点可能通过与铁调控蛋白结合来调节鲤Nramp的mRNA水平,当铁调控蛋白和Nramp 5´ UTR端IRE结合时,可以组织RNA的翻译,而当与3´ UTR端的IRE位点结合时,则可以保护RNA免受降解。但是,鱼类Nramp基因中的IRE位点是否与哺乳类Nramp2 IRE位点具有相似的作用,以及IRE位于ORF与位于UTR区域是否具有类似的功能,还需进一步的研究和探索。
Nramp2基因在小鼠肝脏、脾脏、肾脏、心脏、肌肉、小肠等组织中广谱表达(Grunheid et al,1995),而Nramp1基因的表达则呈组织特异性,如人的Nramp1可以在肝脏、肾脏和脾脏中检测到表达(Cellier et al,1997),而小鼠的Nramp1基因则主要在脾脏中表达,肝脏中表达量相对较少(Vidal et al,1993)。本研究发现,半滑舌鳎Nramp基因在脾脏、肾脏和肝脏中表达量最高,其次是皮肤、血液、肠、鳃、心脏和脑,而在肌肉和性腺中的表达量最低。半滑舌鳎Nramp基因在器官中这种组成型表达方式与哺乳类Nramp2的表达方式较为相似(Grunheid et al,1995; Forbes et al,2001)。此外,在大菱鲆、草鱼、鲤、鲈以及斑点叉尾鮰中也观察到了Nramp基因在脾脏和肾脏中的表达量较高,这种表达量变化说明了脾脏、肾脏是鱼类主要的免疫器官。
对半滑舌鳎进行哈维氏弧菌感染实验后研究发现,相较于PBS对照组,实验组Nramp基因表达量在脾脏、肾脏和肝脏中明显上调,随着时间推移又恢复至正常表达水平。同样的现象在小鼠(Govoni et al,1997)、猪(Zhang et al,2000)及真鲷(Chen et al,2004)中有过报道,范玉顶等(2011) 利用草鱼呼肠孤病毒感染草鱼肾脏细胞系,结果发现,在感染后Nramp基因表达量明显升高,3 h表达量达到最大,24 h后回落至正常表达水平;Chen等(2007) 利用鳗弧菌感染大菱鲆胚胎细胞系后发现,Nramp基因表达量在感染6−48 h后显著升高,且在12 h达到最大表达量。这些结果表明,Nramp基因与病原菌感染后机体的防御反应密切相关,但半滑舌鳎Nramp基因与哈维氏弧菌感染之间的相互作用机理以及Nramp基因在鱼体感染病原菌后在鱼类免疫系统中所扮演的角色还有待于进一步的研究。
Nramp基因多态性与疾病相关性的研究已在哺乳动物上开展了一些工作。Liu等(2004) 研究发现,一个微卫星位于人SLC11A1基因的5´端,其多态性与结核病易感性以及巨噬细胞调控的疾病有关;Sanchez-Robert等(2005) 对犬Nramp基因的研究表明,TAG-8- 141单倍型与利什曼原虫的易感性有关;Liu等(2003) 在鸡的Nramp1基因上的研究证明,Nramp1基因高度保守区单核苷酸多态性与青年鸡SE疫苗接种及病原感染后的免疫应答相关;Paixiao等(2006) 利用SSCA法分析瘤牛、荷斯坦牛Nramp1基因3´ UTR区的遗传变异,发现不同基因型对布鲁氏菌的抗病性和敏感性差异显著,且不同品种间基因频率差异显著;赵生国等(2013) 研究猪Nramp1基因遗传变异与仔猪腹泻的相关性发现,外显子2的AA基因型个体腹泻评分值显著高于TT基因型个体(P<0.05) ,并极显著高于AT基因型个体(P<0.01) ;内含子6的CC基因型个体腹泻评分值显著高于CT基因型个体(P<0.05) 。分子标记辅助选择育种(Marker-assisted selection,MAS)技术可以定向选育抗病新品种,且已在鱼类上取得了较好的效果(Xu et al,2008)。本研究在半滑舌鳎Nramp部分基因组DNA(1042 bp)中筛选到15个SNP位点,对其中位于第2内含子区的3个SNP[SNP-g.3113(T→C)、SNP-g.3125(A→G)和SNP-g.3164(A→T)]位点进行基因型分型,并对其SNP位点与抗病性的关联进行了分析。结果发现,在同一家系233个个体感染鳗弧菌后,存活个体165个视为抗病个体,死亡个体68个视为易感个体,其中,SNP-g.3125的AG基因型(0.735) 在死亡个体中为优势基因型,而GG基因型(0.546) 在存活个体中为优势基因型,GG基因型个体抗性评分极显著高于AG基因型个体(P<0.01) ,G等位基因抗性评分极显著高于A等位基因(P<0.01) ,可见半滑舌鳎Nramp基因中的SNP-g.3125的等位基因(G)和基因型(GG)与半滑舌鳎对于鳗弧菌抗性呈显著相关性。因此,Nramp基因该SNP位点可作为半滑舌鳎抗病育种的一个潜在的抗性遗传标记位点,为半滑舌鳎抗性品系培育的遗传分子标记提供基础研究资料。
| 邓景耀, 孟田湘, 任胜民, 等. 渤海鱼类种类组成及数量分布. 海洋水产研究 , 1988 (9) : 10-98 | |
| 范玉顶, 徐进, 罗晓松, 等. 草鱼天然抗性相关巨噬蛋白基因全长cDNA的克隆与表达分析. 中国水产科学 , 2011, 18 (1) : 38-47 | |
| 赵生国, 蔡原, 滚双宝, 等. 猪天然抗性相关巨噬细胞蛋白基因(Nramp1)多态性及其与猪仔腹泻相关性分析. 农业生物技术学报 , 2013, 21 (11) : 1351-1357 | |
| Bairoch A. The PROSITE dictionary of sites and patterns in proteins, its current status. Nucleic Acids Res , 1993, 21 (13) : 3097-3103 DOI:10.1093/nar/21.13.3097 | |
| Blackwell JM, Barton CH, White JK, et al. Genetic regulation of leishmanial and mycobacterial infections: the Lsh/Ity/Bcg gene story continues. Immunol Lett , 1995, 43 (1-2) : 99-107 | |
| Blackwell JM. Structure and function of the natural resistance- associated macrophage protein(Nramp1), a candidate protein for infectious and autoimmune disease susceptibility. Mol Med Today , 1996, 2 (5) : 205-211 DOI:10.1016/1357-4310(96)88773-9 | |
| Burge EJ, Gauthier DT, Ottinger CA, et al. Mycobacterium- inducible Nramp in striped bass (Morone saxatilis). Infect Immun , 2004, 72 (3) : 1626-1636 DOI:10.1128/IAI.72.3.1626-1636.2004 | |
| Casey JL, Hentze MW, Koeller DM, et al. Iron-responsive elements: regulatory RNA sequences that control mRNA levels and translation. Science , 1988, 240 (4854) : 924-928 DOI:10.1126/science.2452485 | |
| Cellier M, Govoni G, Vidal S, et al. Human natural resistance- associated macrophage protein: cDNA cloning, chromosomal mapping, genomic organization, and tissue-specific expression. J Exp Med , 1994, 180 (5) : 1741-1752 DOI:10.1084/jem.180.5.1741 | |
| Cellier M, Shustik C, Dalton W, et al. Expression of the human Nramp1 gene in professional primary phagocytes: studies in blood cells and in HL-60 promyelocytic leukemia. J Leukocyte Biol , 1997, 61 (1) : 96-105 | |
| Chen H, Waldbieser GC, Rice CD, et al. Isolation and characterization of channel catfish natural resistance associated macrophage protein gene. Dev Comp Immunol , 2002, 26 (6) : 517-531 DOI:10.1016/S0145-305X(01)00096-9 | |
| Chen SL, Xu MY, Ji XS, et al. Cloning and characterization of natural resistance associated macrophage protein (Nramp) cDNA from red sea bream (Pagrus major). Fish Shellfish Immunol , 2004, 17 (4) : 305-313 DOI:10.1016/j.fsi.2004.04.003 | |
| Chen SL, Wang ZJ, Xu MY, et al. Molecular identification and expression analysis of natural resistance associated macrophage protein (Nramp) cDNA from Japanese flounder (Paralichthys olivaceus). Fish Shellfish Immunol , 2006, 20 (3) : 365-373 DOI:10.1016/j.fsi.2005.05.011 | |
| Chen SL, Zhang YX, Xu JY, et al. Molecular cloning, characterization and expression analysis of natural resistance associated macrophage protein (Nramp) cDNA from turbot (Scophthalmus maximus). Comp Biochem Phys B , 2007, 147 (1) : 29-37 DOI:10.1016/j.cbpb.2006.12.003 | |
| Dorschner MO, Phillips RB. Comparative analysis of two Nramp loci from rainbow trout. DNA Cell Biol , 1999, 18 (7) : 573-583 DOI:10.1089/104454999315123 | |
| Feng J, Li Y, Hashad M, et al. Bovine natural resistance associated macrophage protein 1 (Nramp1) gene. Genome Res , 1996, 6 (10) : 956-964 DOI:10.1101/gr.6.10.956 | |
| Fleming MD, Romano MA, Su MA, et al. Nramp2 is mutated in the anemic Belgrade (b) rat: evidence of a role for Nramp2 in endosomal iron transport. Proc Natl Acad Sci USA , 1998, 95 (3) : 1148-1153 DOI:10.1073/pnas.95.3.1148 | |
| Forbes JR, Gros P. Divalent-metal transport by NRAMP proteins at the interface of host-pathogen interactions. Trends Immunol , 2001, 9 (8) : 397-403 | |
| Govoni G, Gauthier S, Billia F, et al. Cell-specific and inducible Nramp1 gene expression in mouse macrophages in vitro and in vivo. J Leukoc Biol , 1997, 62 (2) : 277-286 | |
| Grunheid S, Cellier M, Vidal S, et al. Identification and characterization of a second mouse Nramp gene. Genomics , 1995, 25 (2) : 514-525 DOI:10.1016/0888-7543(95)80053-O | |
| Gunshin H, Mackenzie B, Berger UV, et al. Cloning and characterization of a mammalian proton-coupled metal-ion transporter. Nature , 1997, 388 (6641) : 482-488 DOI:10.1038/41343 | |
| Liu W, Kaiser MG, Lamont SJ. Natural resistance associated macrophage protein 1 gene polymorphisms and response to vaccine against or challenge with Salmonella enteritidis in young chicks. Poultry Science , 2003, 82 (2) : 259-266 DOI:10.1093/ps/82.2.259 | |
| Liu W, Cao WC, Zhang CY, et al. VDR and Nramp1 gene polymorphisms in susceptibility to pulmonary tuberculosis among the Chinese Han population: a case-control study. Int J Tuberc Lung Dis , 2004, 8 (4) : 428-434 | |
| Nielsen H, Engelbrecht J, Brunak S, et al. Identification of prokaryotic and eukaryotic signal peptides and prediction of their cleavage sites. Protein Eng , 1997, 10 (1) : 1-6 DOI:10.1093/protein/10.1.1 | |
| Kishi F. Isolation and characterization of human Nramp cDNA. Biochem Biophys Res Commun , 1994, 204 (3) : 1074-1080 DOI:10.1006/bbrc.1994.2572 | |
| Kishi F, Tabuchi M. Complete nucleotide sequence of human NRAMP2 cDNA. Mol Immunol , 1997, 34 (12-13) : 839-842 DOI:10.1016/S0161-5890(97)00110-7 | |
| Klausner RD, Rouault TA, Harford JB. Regulating the fate of mRNA: the control of cellular iron metabolism. Cell , 1993, 72 (1) : 19-28 DOI:10.1016/0092-8674(93)90046-S | |
| Kyte J, Doolittle RF. A simple method for displaying the hydropathy of a protein. J Mol Biol , 1982, 157 : 105-132 DOI:10.1016/0022-2836(82)90515-0 | |
| Paixiao TA, Ferreir AC, Borges AM, et al. Frequency of bovine Nramp1(SLC11A1) alleles in Holstein and Zebu breeds. Vet Immunol Immunopathol , 2006, 109 (1/2) : 37-42 | |
| Saeij JPJ, Wiegertjes GF, Stet RJM. Identification and characterization of a fish natural resistance-associated macrophage protein (NRAMP) cDNA. Immunogenetics , 1999, 50 (1) : 60-66 | |
| Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol , 1987, 4 (4) : 406-425 | |
| Sanchez-Robert E, Altet L, Sanchez A, et al. Polymorphism of SLC11A1(Nramp1)gene and canine leishmaniasis in a case-control study. J Heredity , 2005, 96 (7) : 755-758 DOI:10.1093/jhered/esi111 | |
| Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor: Cold Spring Harbor Laboratory Press, 2001 . | |
| Skamene E, Pietrangeli CE. Genetics of the immune response to infectious pathogens. Curr Opin Immunol , 1991, 3 (4) : 511-517 DOI:10.1016/0952-7915(91)90013-Q | |
| Tamura K, Dudley J, Nei M, et al. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4. 0. Mol Biol Evol , 2007, 24 (8) : 1596-1599 DOI:10.1093/molbev/msm092 | |
| Vidal SM, Malo D, Vogan K, et al. Natural resistance to infection with intracellular parasites: isolation of a candidate for Bcg. Cell , 1993, 73 (3) : 469-485 DOI:10.1016/0092-8674(93)90135-D | |
| Xu TJ, Chen SL, Ji XS, et al. MHC polymorphism and disease resistance to Vibrio anguillarum in 12 selective Japanese flounder (Paralichthys olivaceus) families. Fish Shellfish Immunol , 2008, 25 (3) : 213-221 DOI:10.1016/j.fsi.2008.05.007 | |
| Zhang G, Wu H, Ross CR, et al. Cloning of porcine NRAMP1 and its induction by lipopolysaccharide, tumor necrosis factor alpha, and interleukin-1b: role of CD14 and mitogen- activated protein kinases. Infect Immunol , 2000, 68 (3) : 1086-1093 DOI:10.1128/IAI.68.3.1086-1093.2000 |



