2. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 上海海洋大学 上海 201306
2. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071 ;
3. Shanghai Ocean University, Shanghai 201306
对虾白斑综合征病毒(White Spot Syndrome Virus,WSSV)作为线头病毒科(Nimaviridae)、白斑病毒属(Whispovirus)的唯一成员(Vlak et al,2005),由其引起的白斑病已经成为对虾养殖中面临的最主要病害。在1991-1992年间,WSSV首次被报道发现于中国台湾,之后迅速影响至东南亚其他地区(Flegel,1997),而在1994年,在印度等南亚地区病害也呈暴发趋势(Anonymous,1995; Pradeep et al,2008)。WSSV自此开始在全球对虾养殖业中迅速传播,在过去的几十年中,对很多国家的对虾养殖产生很大的影响(Inouye et al,1994; Chou et al,1995; Cai et al,1995; Wongteerasupaya et al,1995; Kasornchandra et al,1998; Mohan et al,1998; Nadala et al,1998; Park et al,1998; Magbanua et al,2000),3-10 d内可达100%的致死率,造成巨大的经济损失(Lightner,1996)。
目前,GenBank上公布有4种不同WSSV病毒株的基因组全序列,分别是中国台湾株(TW,AF440570) (307287 bp)、泰国株(TH,AF369029) (292967 bp)、中国株(CN,AF332093) (305107 bp)和韩国株(KR,JX515788) 。据报道,假定的祖先株约312 kb,最大基因组的WSSV-TH-96-Ⅱ(AY753327) 则起源于泰国株(Pradeep et al,2008)。各种基因组序列之间具有高度的相似度,而其差异主要体现在五个方面:大序列缺失;易于发生基因重组的可变区;同源重复区内重复序列的变化;转座酶基因序列只存在于中国台湾株;单核苷酸突变(童桂香等,2014; Marks et al,2004)。
由于WSSV地理范围、宿主类别和致病力的差异,导致不同的毒株之间具有差异。通过对WSSV基因组两多态性位点分析,ORF14/15和ORF23/24更易于发生缺失和重组。据此,本研究针对中国明对虾养殖不同地区自2014年1月-8月间采集的48份WSSV阳性样本,通过特定引物扩增目的片段,测序分析比较不同地区各分离株ORF14/15和ORF23/24序列缺失情况,探究中国不同地区WSSV分离株ORF14/15和ORF23/24的变异,为WSSV分子流行病研究提供依据。
1 材料与方法 1.1 样本来源在2014年1月-8月WSSV病害暴发期间,赴河北、浙江、山东、江苏、辽宁、福建和广东等地采集对虾样品,然后置于-20℃的冰箱冷冻保存,其来源信息见表 1。
|
|
表 1 样本采集信息 Table 1 Information of sampling |
将保存的样本取出,取约30 mg鳃组织,按照海洋动物组织基因组DNA提取试剂盒(离心柱型)的说明进行DNA提取,最后加30 µl 65℃预热的无菌水溶解,将提取的DNA样本置于-20℃冷冻保存待用。
1.3 PCR检测对WSSV DNA样本的检测采用套式PCR方法,标准按照GB/T 28630.2-2012白斑综合征(WSD)诊断规程第2部分套式PCR检测法。PCR产物通过用1×TAE电泳缓冲液配制的1%的琼脂糖凝胶进行电泳分析。
1.4 WSSV缺失区ORF14/15和ORF23/24扩增将得到的WSSV核酸通过特定的引物进行ORF14/15和ORF23/24目的片段的扩增。实验中25 µl的体系包括:17.3 µl双蒸水,2 µl脱氧核酸混合物,2.5 µl 10×PCR反应缓冲液(含Mg2+),正向和反向引物各1 µl,0.2 µl Ex Taq DNA聚合酶,1 µl待测核酸。目的片段扩增中的PCR条件以及引物序列见表 2(Marks et al,2005; Dieu et al,2010; Tang et al,2013)。
|
|
表 2 PCR 引物与反应条件 Table 2 PCR primers and cycling conditions |
扩增后的PCR产物经1×TBE电泳缓冲液配制的1%琼脂糖凝胶电泳,将目的条带切下,用胶回收试剂盒获得目的DNA,经NanoDrop 2000测定浓度后,通过pMD®18-T Vector进行连接转化,将显示阳性、条带单一的菌液进行测序。测得的序列与NCBI数据库进行比对,其中,将ORF14/15与TH-96-Ⅱ株比对,ORF23/24与中国台湾株(TW)比对。
2 结果与分析 2.1 PCR检测结果对于提取的WSSV核酸,通过在套式PCR中的检测,6份核酸样品在第一轮中呈阳性,42份核酸样品在第二轮中呈阳性。
2.2 ORF14/15扩增结果通过对WSSV样本的扩增,共有21份(2#、3#、4#、5#、6#、7#、13#、23#、24#、27#、30#、31#、32#、33#、37#、40#、42#、44#、45#、46#和47#)样本出现了ORF14/15的检测条带,其能够成功扩增样品的比例为43.75%。其中,13#、23#和24#条带明显比较大,并且在3#、44#和47#一个泳道内均出现了两条大小不一的条带,这可能是由样品中DNA的断裂或片段不完整造成。来源于浙江宁波和湖州、山东即墨、日照和青岛、江苏如东、福建漳浦和广东湛江地区的样品未检出条带(图 1)。
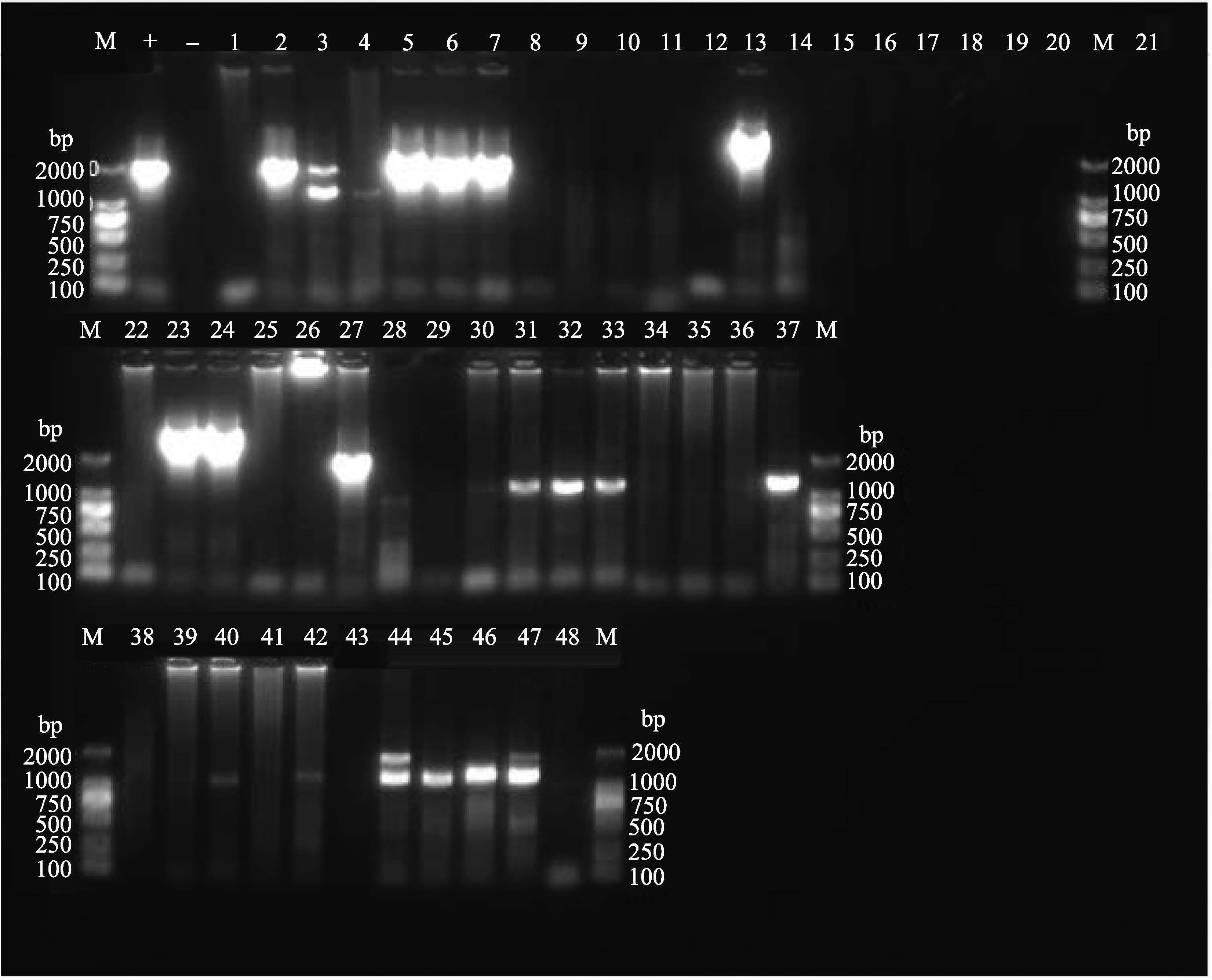
|
图 1 ORF14/15扩增 Figure 1 ORF14/15 amplification M: DNA Marker DL2000; +: 阳性对照; -: 阴性对照; 1-48: 样本编号 M: DNA Marker DL2000; +: positive control; -: negative control; 1-48: number of samples |
在ORF23/24扩增中,有16份(2#、3#、5#、6#、7#、23#、24#、27#、38#、39#、40#、41#、42#、44#和48#)样本可以扩增出条带,能够成功扩增样品的比例为33.33%,所有样本扩增中的条带大小基本没有差异。浙江湖州和宁波、山东日照、即墨和青岛、江苏如东和赣榆、福建漳浦未有ORF23/24扩出(图 2)。
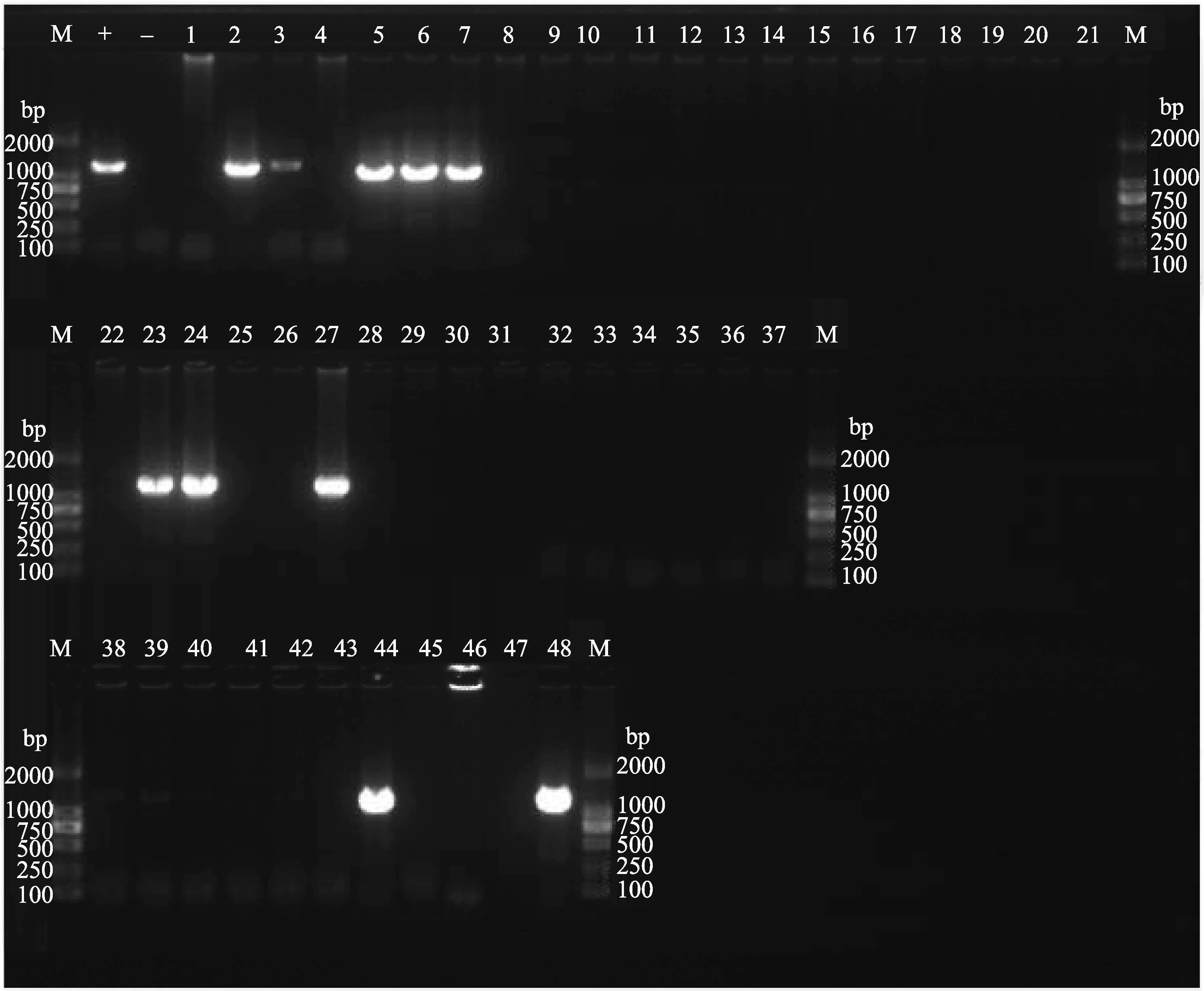
|
图 2 ORF23/24扩增 Figure 2 ORF23/24 amplification M: DNA Marker DL2000; +: 阳性对照; -: 阴性对照; 1-48: 样本编号 M: DNA Marker DL2000; +; positive control; -: negative control; 1-48: number of samples |
在所有样本的ORF14/15扩增中,共有4种大小不一的片段扩增出来,即1260 bp(河北曹妃甸)、1270 bp(浙江温州、广州江门、河北黄骅)、1892 bp(河北曹妃甸、江苏南京)和2662 bp(江苏赣榆、辽宁、福建厦门),同推测有最长序列的TH-96-Ⅱ比对,分别缺失6540 bp、6530 bp、5908 bp和5138 bp(图 4)。而在ORF23/24扩增中,片段大小只有微小差异,分别为1140 bp(河北曹妃甸、黄骅)和1146 bp(辽宁、福建厦门、江苏南京、广州湛江和江门、浙江温州)不等,两种序列中间对应位置相差6个碱基,即GATATC(图 3)。同中国台湾株进行对比,缺失的片段大小分别为12070 bp和12064 bp(图 5)。
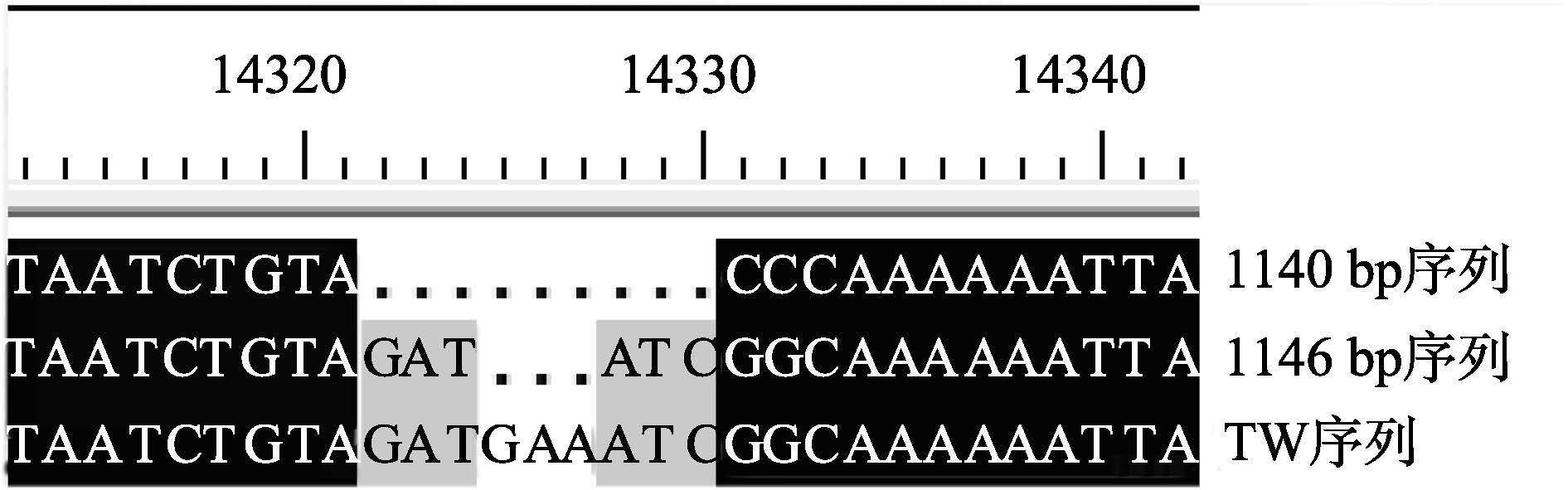
|
图 3 差异位置及碱基序列 Figure 3 Differing sites and nucleotide sequences |
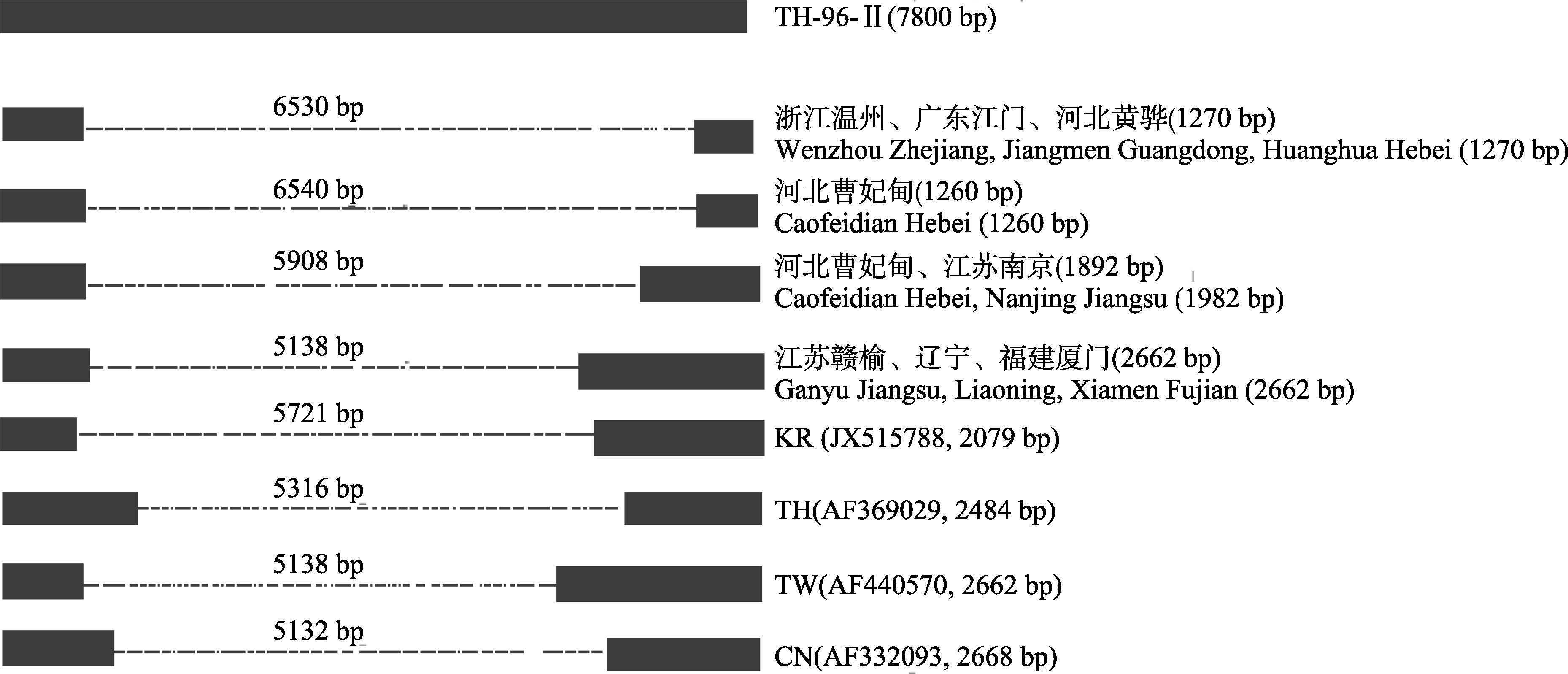
|
图 4 WSSV不同毒株ORF14/15区域的序列比对 Figure 4 Comparison of missing regions of ORF14/15 of different WSSV strains 左右两边的矩形表示用引物扩增出的序列,中间的虚线表示与TW-Ⅱ相比缺失的部分 Rectangles denoted amplified sequences. Dash lines denoted missing sequences compared to TH-96-Ⅱstrain |

|
图 5 WSSV不同毒株ORF23/24区域的序列比对 Figure 5 Comparison of missing regions of ORF23/24 of different WSSV strains 右边粗实线表示引物扩增之外的序列,左右两边的矩形表示用引物扩增出的序列,中间的细虚线表示与中国台湾株(TW,AF440570) 相比缺失的部分,矩形两边的数字表示与TW对应的碱基位置 The right bold lines denoted the region outside the amplified fragments. The right and left rectangles denoted amplified sequences. Dash lines denoted missing sequences compared to the TW strain(AF440570) . Numbers on top of the blocks showed the starting and ending positions corresponding to the TW strain(AF440570) |
比较ORF14/15和ORF23/24扩增结果,可以看出,在河北曹妃甸和黄骅、浙江温州、江苏南京、辽宁的部分样本和广东江门、福建厦门地区样本中,ORF14/15和ORF23/24片段均有扩增,江苏赣榆地区样本中只有ORF14/15的扩增片段。除此之外的浙江宁波和湖州、山东即墨、日照和青岛、江苏如东和福建漳浦地区的所有样本两次扩增均未有相应条带出现,其一致性较高。
已报道亚洲4种毒株,即中国台湾株、中国珠、泰国株、韩国株的ORF14/15片段分别缺失5138 bp、5132 bp、5316 bp和5721 bp。由于TH-96-Ⅱ株(AY753327) ORF14/15片段最为完整,所以通常将ORF14/15与TH-96-Ⅱ株进行比对。在ORF14/15扩增中,可扩增出的片段序列均含有前732个碱基,说明这一段732 bp的序列有较高的保守性,出现缺失差异的位置主要位于全长片段后段部分。河北黄骅、曹妃甸、浙江温州和广东江门地区的毒株出现较大片段的缺失,最大缺失6540 bp。在江苏赣榆、辽宁和福建厦门地区出现的部分缺失为5138 bp的毒株与中国台湾株缺失情况一致。在Tang等(2013) 针对马达加斯加岛、莫桑比亚和沙特阿拉伯地区毒株的实验中发现,序列缺失有5950 bp,同样的缺失情况也出现在Hoa等(2012) 对印度和越南南部地区毒株的调查中。稍微小一点的5892 bp缺失情况发现存在于印度(IN-07,EF468499) 和墨西哥(HQ257380,HQ257383,HQ257381) 的分离株(Tang et al,2013),这与本研究中涉及到江苏南京和河北曹妃甸地区的样本缺失情况相近。在薛晖等(2011) 对2008-2010年兴化等6个地区样本的调查中,报道有4种缺失,即4751 bp、5138bp、5139 bp和5140 bp,后3种相较于中国台湾株仅有1个或两个碱基的差异,但后续的影响可能不仅如此。
在ORF23/24中,共得到两种PCR扩增产物,大小分别为1140 bp和1146 bp。已报道中国台湾株(TW)(AF440570) ORF23/24片段最为完整,因此,通常将ORF23/24与中国台湾株(TW)进行比对。中国台湾株的14322到14330位置的碱基为GATGAAATC,在辽宁、福建厦门、江苏南京、广东湛江和江门、浙江温州地区样本扩增出的1146 bp的序列相较于中国台湾株缺失了GAA 3个碱基,而河北曹妃甸和黄骅地区扩增出的1140 bp序列则缺失了GATGAAATC 9个碱基。二者与中国台湾株相比,分别缺失了12064 bp和12070 bp。目前,已知的泰国株最大的缺失序列为13120 bp,韩国株具有中等程度的缺失,为5654 bp,而中国株缺失最小,仅1169 bp,本研究中的两种情况属于较大程度的片段缺失。童桂香等(2014) 中的广西分离株、Tang等(2013) 中的分离株和两株印度株(EU327499,EU327500) 缺失均为10971 bp,在Pradeep等(2008) 所采集的有关印度地区分离株有8539 bp的片段缺失。总的来说,ORF23/24这段的缺失长度变异较多,在1-13 kb之间不等。
目前为止,Marks等(2005) 以两株最大(约312 kb)和最小(约293 kb)的分离株为实验样本,用RFLP方法呈现出二者之间的差异,同时两分离株之间的毒性差异也已被观测到,即较小的分离株具有更强的毒性,Zwart等(2010) 也证明如此情况。本研究发现,WSSV毒株在可变区ORF14/15和ORF23/24的进化表现出更大程度的缺失,ORF14/15和ORF23/24的缺失没有必然联系,推测两区域的缺失进化是为使病毒基因组更加稳定,从而更好地适应外部环境。有关ORF14/15和ORF23/24变异与缺失的研究,对追溯WSSV毒株世系和发展起到一定帮助,同时为确定“基因组缺失导致更高毒性”是否正确提供了一定的材料依据。
| 童桂香, 黎小正, 韦信贤, 等. 白斑综合征病毒广西株缺失区基因的比较分析. 上海海洋大学学报 , 2004, 23 (1) : 8-13 | |
| 薛晖, 王晓丰, 丁正峰, 等. 白斑综合征病毒江苏分离株变异区和缺失区基因的序列比较. 中国水产科学 , 2011, 18 (5) : 1196-1201 | |
| Anonymous. SEMBV-an emerging viral threat to cultured shrimp in Asia. CP Shrimp New 3 , 1995 : 2-3 | |
| Cai SL, Huang J, Wang CM, et al. Epidemiological studies on the explosive epidemic disease of prawn in 1993-1994. J China Fish , 1995, 19 (2) : 112-117 | |
| Chou HY, Huang CY, Wang CH, et al. Pathogenicity of a baculovirus infection causing white spot syndrome in cultured penaeid shrimp in Taiwan. Dis Aquat Org , 1995, 23 (3) : 165-173 | |
| Dieu BTM, Marks H, Zwart MP, et al. Evaluation of white spot syndrome virus variable DNA loci as molecular markers of virus spread at intermediate spatiotemporal scales. J Gen Virol , 2010, 91 (5) : 1164-1172 DOI:10.1099/vir.0.018028-0 | |
| Flegel TW. Major viral diseases of the black tiger prawn (Penaeus monodon) in Thailand. World J Microbiol Biotechnol , 1997, 13 (4) : 433-442 DOI:10.1023/A:1018580301578 | |
| Hoa TTT, Zwart MP, Phuong NT, et al. Indel-II region deletion sizes in the white spot syndrome virus genome correlate with shrimp disease outbreaks in southern Vietnam. Dis Aquat Org , 2012, 99 (2) : 153-162 DOI:10.3354/dao02463 | |
| Inouye K, Miwa S, Oseko N, et al. Mass mortalities of cultured Kuruma shrimp, Penaeus japonicus in Japan in 1993, electron-microscope evidence of the causative virus. Fish Pathol , 1994, 29 (2) : 149-158 DOI:10.3147/jsfp.29.149 | |
| Kasornchandra J, Boonyaratpalin S, Itami T. Detection of white spot syndrome in cultured penaeid shrimp in Asia, microscopic observation and polymerase chain reaction. Aquaculture , 1998, 164 (1-4) : 243-251 DOI:10.1016/S0044-8486(98)00190-2 | |
| Lightner DV. A handbook of pathology and diagnostic procedures for diseases of penaeid shrimp. Special publication of the World Aquaculture Society, LA, Baton Rouge, 1996 | |
| Magbanua FO, Natividad KT, Migo VP, et al. White spot syndrome virus (WSSV) in cultured Penaeus monodon in the Philippines. Dis Aquat Org , 2000, 42 (1) : 77-82 | |
| Marks H, Goldbach RW, Vlak JM, et al. Genetic variation among isolates of white spot syndrome virus. Arch Virol , 2004, 149 (4) : 674-697 | |
| Marks H, van Duijse JJA, Zuidema D, et al. Fitness and virulence of an ancestral white spot syndrome virus isolate from shrimp. Virus Res , 2005, 110 (1) : 9-20 | |
| Mohan CV, Shankar KM, Kulkarni S, et al. Histopathology of cultured shrimp showing gross signs of yellow head syndrome and white spot syndrome during 1994 Indian epizootics. Dis Aquat Org , 1998, 34 (1) : 9-12 | |
| Nadal ECB, Loh PC. A comparative study of three different isolates of white spot virus. Dis Aquat Org , 1998, 33 (3) : 231-234 | |
| Park JH, Lee YS, Lee S, et al. An infectious viral disease of penaeid shrimp newly found in Korea. Dis Aquat Org , 1998, 34 (1) : 71-75 | |
| Pradeep B, Shekar M Karunasagar I, et al. Characterization of variable genomic regions of Indian white spot syndrome virus. Virology , 2008, 376 (1) : 24-30 DOI:10.1016/j.virol.2008.02.037 | |
| Tang KFJ, Marc LG, Lightner DV. Novel, closely related, white spot syndrome virus (WSSV) genotypes from Madagascar, Mozambique and the Kingdom of Saudi Arabia. Dis Aquat Org , 2013, 106 (1) : 1-6 DOI:10.3354/dao02645 | |
| Vlak JM, Bonami JR, Flegel TW, et al. Nimaviridae. In: Fauquet CM, Mayo MA, Maniloff J, Desselberger U, Ball LA (Eds.), Virus Taxonomy. Eighth Report of the International Committee on Taxonomy of Viruses. Elsevier, Amsterdam, 2005, 187- 192 | |
| Wongteerasupaya C, Vickers JE, Sriurairalana S, et al. A non- occluded, systemic baculovirus that occurs in cells of ectodermal and mesodermal origin and causes high mor?tality in the black tiger prawn, Penaess monodon. Dis Aquat Org , 1995, 21 (2) : 69-77 | |
| Zwart MP, Dieu BTM, Hemerik L, et al. Evolutionary trajectory of white spot syndrome virus (WSSV) genome shrinkage during spread in Asia. PLoS One , 2010, 5 (10) : e13400 DOI:10.1371/journal.pone.0013400 |



