2. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 上海海洋大学水产与生命学院 上海 201306
2. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071 ;
3. College of Fisheries and Life Sciences, Shanghai Ocean University, Shanghai 201306
随着社会经济的快速发展,水产养殖业也呈现出养殖规模和养殖技术的空前提高,尤其在集约化养殖和工厂化养殖方面发展迅速。但在海水经济鱼类的养殖过程中,病毒性疾病的暴发往往会给养殖业带来巨大的危害。淋巴囊肿病毒(Lymphocystis disease virus,LCDV)(徐洪涛等,2000)、肿大细胞病毒属虹彩病毒(Megalocytivirus,Mega)(Chao et al,2004)、赤点石斑鱼神经坏死病毒(Red-spotted grouper nervous necrosis virus,RGNNV)(Lopez-Jimena et al,2011; Choi et al,2013)、传染性造血器官坏死病毒(Infectious haematopoietic necrosis virus,IHNV)(Rudakova et al,2007)、传染性胰脏坏死病毒(Infectious pancreatic necrosis virus,IPNV)(Wallace et al,2008)、病毒性出血败血症病毒(Viral hemorrhagic septicemia virus,VHSV)(Isshiki et al,2001)和传染性鲑鱼贫血症病毒(Infectious salmon anaemia virus,ISAV)(Lyngstad et al,2012; Godoy et al,2013)是养殖鱼类主要的病毒性病原,它们均能引起传染性、暴发性疾病,同时伴随着高死亡率,给养殖企业造成严重的经济损失。大菱鲆红体病虹彩病毒(Turbot reddish body iridovirus,TRBIV)是肿大细胞病毒属虹彩病毒的一个成员,主要感染我国养殖的大菱鲆(史成银等,2005)。因此,建立这些病原的快速、有效、低成本的检测方法,对保障养殖鱼类健康、防治疾病发生具有重要的意义。
多重PCR(Multiplex PCR)技术是目前应用最为广泛的检测技术之一,最早由Chamberlian等(1988) 提出。由于具有高通量、快速和成本低等优势,该检测技术很快被广泛应用在病原检测和临床诊断中(Edwards et al,1994; Elnifro et al,2000)。然而,多重PCR技术也存在引物设计困难、反应相互干扰、扩增效率和准确度差等缺点,其应用范围较为有限。有学者对其进行改进,发明了“扩增子拯救多重PCR”(Amplicon rescue multiplex PCR,Arm-PCR)技术(Han et al,2006),其工作原理是:在多重扩增体系中,设计靶序列特异性的套式PCR引物和通用性的超级引物,通过富集、加标签、拯救、扩增等步骤,实现对多种靶序列的同步、高灵敏、高特异性扩增。目前,该技术在临床病原检测领域已有较多的研究和报道,但在水生动物病原检测领域并不多见(耿伟光等,2013)。
本研究在分析了水产动物LCDV、TRBIV、Mega、RGNNV、IHNV、IPNV、VHSV、ISAV的非结构蛋白(Non-structural protein of ISAV,ISAV-NS)、ISAV的基质蛋白(Matrix protein of ISAV,ISAV-MA)相关基因核苷酸序列的基础上,依据Arm-PCR的原理设计了9套多重PCR引物,优化了反应条件,成功建立了可同步检测上述7种病原的Arm-PCR方法,结合基因芯片技术,实现了多种鱼类病毒性病原的高通量检测,具有较好的应用前景。
1 材料与方法 1.1 材料 1.1.1 实验克隆菌株含有LCDV、TRBIV、Mega、RGNNV、IHNV、IPNV、VHSV、ISAV-NS、ISAV-MA等相关致病基因的T-A克隆质粒和菌株均由本实验室构建,用作检测体系构建的阳性模板。各基因的GenBank检索号见表 1。
|
|
表 1 Arm-PCR 引物序列及目标产物长度 Table 1 Sequences of Arm-PCR primers and products length |
实验中用到的TransStartTM Top Taq DNA Polymerase、High Pure dNTPs购自全式金生物技术有限公司,海洋动物组织基因组DNA提取试剂盒购自天根生化科技有限公司,质粒小量快速提取试剂盒购自博迈德生物技术有限公司,0.5 ml 50 kDa超滤离心管购自德国Millipore公司。
1.2 方法 1.2.1 阳性模板的制备分别将含有LCDV、TRBIV、Mega、RGNNV、IHNV、IPNV、VHSV、ISAV-NS、ISAV-MA等病毒相关致病基因的克隆菌株过夜培养。对扩大培养的菌液提取质粒,用超微量核酸蛋白测定仪(NanoDrop2000,美国)测定核酸的浓度和纯度,保存于-20℃备用。
1.2.2 靶基因的选择与Arm-PCR引物的设计根据GenBank中已公布的LCDV、TRBIV、Mega和RGNNV的衣壳蛋白(CP)、IHNV的核蛋白(N)、IPNV的VP5、VHSV的糖蛋白(G)、ISAV的非结构蛋白(NS)和基质蛋白(MA)等基因序列,使用Primer Premier 5.0分别设计特异性套式PCR引物,用于Arm-PCR第一步PCR的扩增,其中,内引物的5′ 端各有一段通用接头,正向接头的序列为5′-CAG GCC ACG TTT TGT CAT GC-3′,反向接头序列为5′-TTC TTT GCG TTA TGT CTC TG-3′,各引物序列及扩增片段大小见表 1。各引物由生工生物工程(上海)有限公司合成。
1.2.3 Arm-PCR 10 × Primer Mix的准备取合成的各引物,分别稀释至100 μmol/L;然后各取等量,加入新的EP管中,调整每条引物的终浓度至2 μmol/L,分装、保存于-20℃备用。
1.2.4 Arm-PCR反应体系和参数第一步PCR体系:2.5 U/μl Taq DNA聚合酶0.6 μl,10×PCR Buffer(Mg2+ plus)5 μl,2.5 mmol/L dNTPs 5 μl,2 μmol/L 10×Primer Mix 5 μl,模板1 μl,补充ddH2O至50 μl。反应程序:94℃ 5 min;94℃ 15 s,55℃ 15 s,72℃ 15 s 15个循环;94℃ 15 s,70℃ 15 s,6个循环;72℃ 3 min;4℃保存。
产物经超滤管(0.5 ml 50 kDa)离心,收集液作为第二步PCR模板。第二步PCR体系:2.5 U/μl Taq DNA聚合酶0.6 μl,10×PCR Buffer(Mg2+ plus)5 μl,2.5 mmol/L dNTPs 5 μl,10 μmol/L正向通用引物Fs(5x-CAG GCC ACG TTT TGT CAT GC-3x)1 μl,40 μmol/L反向通用引物Rs(5x-Cy3-TTC TTT GCG TTA TGT CTC TG-3x)1 μl,模板(上述收集液)10 μl,ddH2O至50 μl。反应程序:94℃ 5 min;94℃ 30 s,55℃ 30 s,72℃ 30 s,30个循环;72℃ 3 min;4℃保存。
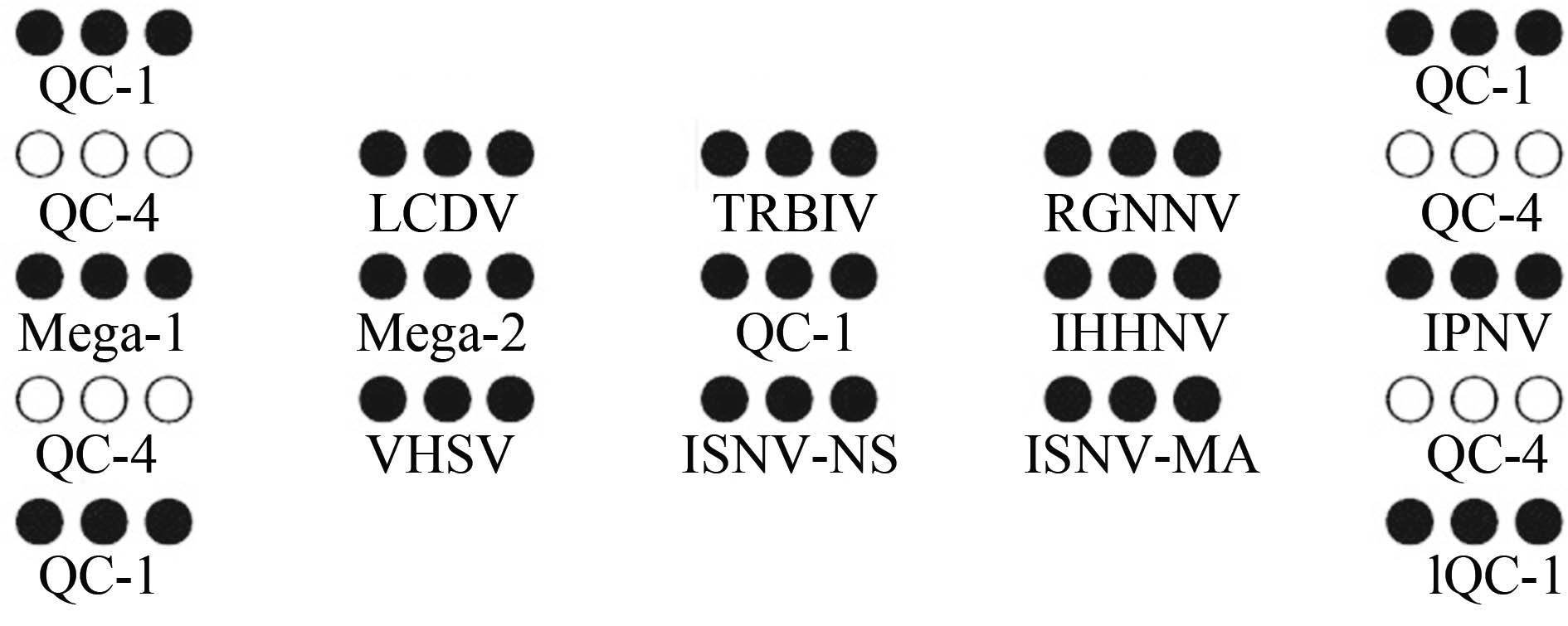
1.2.5 Arm-PCR扩增产物的基因芯片检测由于各病毒的扩增产物大小相近,不能使用常用的琼脂糖凝胶电泳分析扩增结果。故本研究通过已建立的基因芯片技术对上述扩增产物进行杂交检测(王胜强等,2015)。主要方法是:取病毒特异性的且与Arm-PCR扩增产物互补的10条寡核苷酸探针(25-30 mer)点样在醛基修饰玻片上,其中,针对Mega的扩增产物设计两条探针Mega-1和Mega-2;玻片上另设置表面化学质控(QC-1) 和空白对照质控(QC-4) ,制作成基因芯片微阵列(图 1)。将Cy3标记的Arm-PCR扩增产物与制作好的基因芯片微阵列在47℃条件下杂交1.5 h,洗涤后在LuxScan 10K扫描仪上采集荧光信号、判断检测结果。
|
图 1 基因芯片微阵列示意 Figure 1 Schematic diagram of DNA microarray QC-1:表面化学质控;QC-4:空白对照质控 QC-1: surface chemical quality control; QC-4: blank control |
对Arm- PCR第一步PCR中影响扩增结果的Taq酶浓度、Mg2+浓度、dNTPs浓度、引物混合物(Primer Mix)浓度和退火温度5个参数进行调整和优化。其中,Taq酶终浓度为0.045、0.050和0.055 U/μl,Mg2+终浓度为2.0、2.8和3.6 mmol/L,dNTPs终浓度为0.15、0.25和0.35 mmol/L,引物终浓度为0.32、0.36和0.40 μmol/L,退火温度为54℃、56℃和58℃。通过多次实验从而确定最佳参数。
1.2.7 Arm-PCR检测灵敏度的测定对预备的质粒进行拷贝数换算,同时进行10倍梯度稀释。依照1.2.4和1.2.5所述方法,采用1.2.6优化得到的参数进行Arm-PCR反应,测定本方法的检测灵敏度。
1.2.8 检测特异性的验证以每种病毒的质粒为模板,依照1.2.4和1.2.5所述方法,采用1.2.6优化得到的参数进行Arm-PCR反应,测定本研究建立的7种病毒Arm-PCR检测方法的特异性。
1.2.9 应用Arm-PCR方法检测病鱼样品先后收集了19批病鱼样品,包括半滑舌鳎(Cynoglossus semilaevis)、龙胆石斑鱼(Epinephelus lanceolatus)、棕点石斑鱼(E. fuscoguttatus)、卵圆鲳鲹(Trachinotus ovatus)、斑石鲷(Oplegnathus punctatus)等,采用本研究建立的Arm-PCR联合基因芯片检测方法进行相应病毒的检测,同时使用套式PCR和套式RT-PCR方法进行检测,作为对比。检测过程简述如下:取患病样品鱼的鳃、肝、脾、肾、脑和眼等组织,分别依照海洋动物组织基因组DNA提取试剂盒说明书和TRizol法提取组织的总DNA和总RNA。依照全式金的一步法反转录试剂盒说明书将提取的RNA反转录成cDNA,然后对提取的DNA和反转录得到的cDNA进行Arm-PCR扩增。将扩增产物依照1.2.5所述进行杂交、清洗和扫描,然后对扫描的图片提取信号值分析并得出检测结果。
2 结果 2.1 Arm-PCR 第一步PCR参数的优化图 1为用于检测Arm-PCR扩增结果的基因芯片微阵列示意图,Arm-PCR扩增产物与基因芯片微阵列杂交结果见图 2。图 2中表面化学质控QC-1应呈阳性,空白对照质控QC-4应呈阴性。杂交结果显示,当Taq酶终浓度为0.050 U/μl、Mg2+终浓度为2 mmol/L、dNTPs终浓度为0.25 mmol/L、Primer Mix终浓度为0.36 μmol/L、退火温度为56℃时,Arm-PCR各扩增产物的量达到峰值。因此,优化后Arm-PCR第一步PCR体系为:Taq酶(2.5 U/μl)1.0 μl,10×PCR Buffer(含20 mmol/L的Mg2+)5 μl,dNTPs(各2.5 mmol/L)5 μl,10×Primer Mix(2 μmol/L)9 μl,模板1 μl,ddH2O补至50 μl,退火温度为56℃。

|
图 2 Arm-PCR第一步PCR参数的优化 Figure 2 Amplification results under different conditions in the first step of Arm-PCR A:Taq DNA聚合酶浓度;B:Mg2+浓度;C:dNTP浓度;D:Primer Mix浓度;E:退火温度 A1-A3:Taq DNA聚合酶终浓度分别为0.045、0.050和 0.055 U/μl;B1-B3:Mg2+终浓度分别为2.0、2.8和3.6 mmol/L;C1-C3:dNTP的终浓度分别为0.15、0.25和0.35 mmol/L;D1-D3:Primer Mix终浓度分别为0.32、0.36和0.40 μmol/L;E1-E3:退火温度分别为54、56和58℃ A: Concentration of Taq enzyme; B: Concentration of Mg2+; C: Concentration of dNTP; D: Concentration of Primer Mix; E: Annealing temperature A1-A3: The concentrations of Taq enzyme were 0.045,0.050 and 0.055 U/μl respectively. B1-B3: The concentration of Mg2+ was 2.0,2.8 and 3.6 mmol/L respectively. C1-C3: The concentration of dNTP was 0.15,0.25 and 0.35 mmol/L respectively. D1-D3: The concentration of Primer Mix was 0.32,0.36 and 0.40 μmol/L respectively. E1-E3: The annealing temperature was 54,56 and 58℃ respectively |
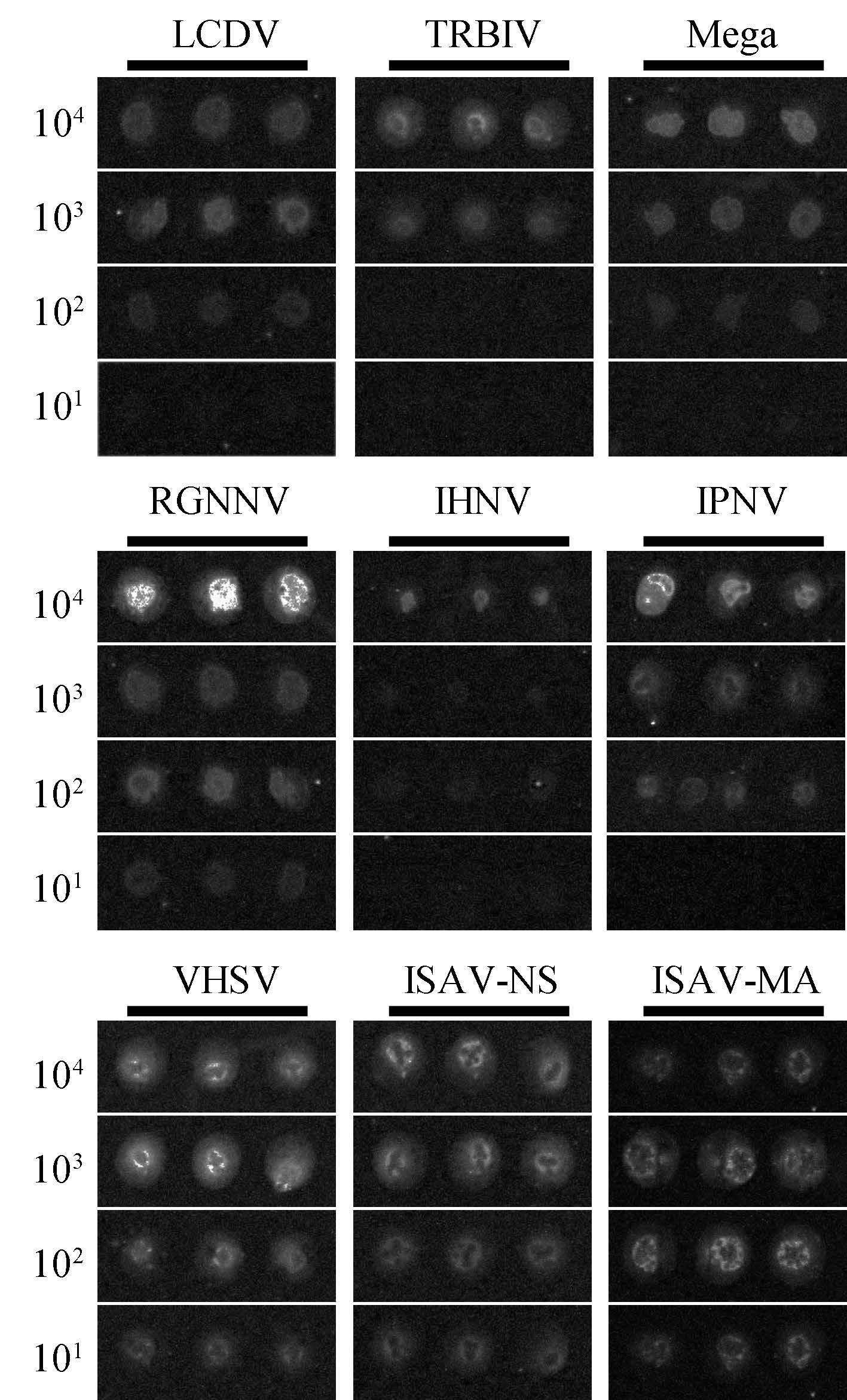
依照1.2.7进行各病毒模板的Arm-PCR扩增、杂交、清洗和扫描,结果如图 3所示。本研究建立的Arm-PCR检测方法,对7种鱼类病毒9个致病基因的检测灵敏度分别为:101 copies/μl(RGNNV、VHSV、ISAV-NS、ISAV-MA)、102 copies/μl(LCDV、Mega、IHNV、IPNV)和103 copies/μl(TRBIV)。
|
图 3 Arm-PCR方法对7种病毒9个基因的 检测灵敏度 Figure 3 The sensitivity test of Arm-PCR for nine genes of seven viruses of fish |
依照1.2.8进行相应的Arm-PCR扩增、杂交、清洗和扫描(图 4)。结果显示,7种病毒9个基因的反应体系中均得到了大量的特异性扩增产物,且杂交信号清晰可见。因此,本研究建立的Arm-PCR检测体系具有良好的扩增特异性。

|
图 4 检测方法的特异性 Figure 4 The specificity test of Arm-PCR |
对19批病鱼样品的检测结果显示,有一批样品(牙鲆,2013-11-15-001) 检出LCDV,两批样品(大菱鲆,2014-03-25-001、2014-12-23-001) 检出TRBIV,两批样品(半滑舌鳎、龙胆石斑鱼精、卵,2014-04-22- 001-009、2014-05-19-001-007) 检出RGNNV,3份样品(棕点石斑鱼、卵圆鲳鲹、斑石鲷,2014-05-25-001、2014- 05-27-001、2015-05-31-001) 检出Megalocytivirus(图 5)。该检测结果与套式PCR和套式RT-PCR检测结果一致(结果未展示)。

|
图 5 Arm-PCR方法检测病鱼样品 Figure 5 Detection of viruses in fish samples using Arm-PCR |
多重PCR技术可以快速、灵敏地同步扩增多个目标片段,已被越来越多地用于病原检测技术研究领域(Han et al,2006; Zou et al,2007; 曾伟伟等,2013),但同时也因其反应体系组成的复杂性而在一定程度上影响其在检测灵敏度的提升。本研究在基因芯片技术的基础上,通过建立和优化扩增子拯救多重PCR(Arm-PCR)的扩增体系,测定反应体系的检测灵敏度以及验证体系扩增的特异性,最终成功建立了灵敏度高、特异性好的Arm-PCR联合基因芯片检测鱼类病毒的方法。
在鱼类病毒的检测领域,多重PCR和基因芯片的联合应用目前并未见报道,但在鱼类、贝类的致病菌检测和分型以及对虾的相关病毒检测当中有相关的报道。Panicker等(2004) 建立的贝类致病菌多重PCR联合基因芯片检测方法在不富集的情况下检测灵敏度为102-103 CFU/ml,通过多重PCR富集之后灵敏度可达单个拷贝每克组织匀浆,本研究建立的鱼类病毒检测方法与Panicker等(2004) 的灵敏度相当。在鱼类致病菌检测领域,González等(2004) 针对海水鱼类5种致病菌中的9个基因所建立的多重PCR联合基因芯片检测方法,检测灵敏度在20 fg纯化基因组DNA以下,而本研究所建立的检测方法的灵敏度相当于在10-3-10-1 fg之间,较González等(2004) 的检测方法高出1-3个数量级。Jeeva等(2014) 所建立的对虾病毒检测方法虽然灵敏度均保持在101 copies/μl左右,但其检测目标病毒的种类仅有对虾白斑病毒和肝胰腺细小病毒两种;刘飞等(2014) 构建的对虾病毒多重检测方法,检测目标病毒的种类可达6种,但该方法的检测灵敏度在102-105 copies/μl之间,较本研究建立的检测方法低1-2个数量级。此外,本研究建立的检测方法采用了基于碱基配对原理的DNA芯片技术,与常规的琼脂糖凝胶电泳分析扩增产物相比,大大提高了检测结果的准确性和可靠性。
本研究建立的Arm-PCR联合基因芯片检测7种鱼类病毒的方法,在预先制备好基因芯片的情况下,经过核酸制备、Arm-PCR扩增、芯片杂交、扫描观察等流程,可以在12 h内完成10个样品7种病毒的高灵敏度、高准确性的同步检测,相对于普通的70个套式PCR反应结合电泳检测过程,更为高效。
总之,本研究建立的可同步检测7种鱼类病毒的Arm-PCR方法具有高通量、高灵敏度、高准确性的优势,能有效提高工作效率,在鱼类病毒的筛查和流行病学调查领域有广泛的应用前景。
| 王胜强, 耿伟光, 李晋, 等. 基因芯片检测鱼类病毒的方法建立与优化. 中国动物检疫 , 2015, 32 (7) : 77-81 | |
| 史成银, 王印庚, 黄倢, 等. 中国大菱鲆虹彩病毒主要衣壳蛋白基因的PCR扩增及序列分析. 中国水产科学 , 2005, 12 (5) : 588-593 | |
| 刘飞, 张宝存, 张晓华, 等. 对虾6种病毒多重PCR检测方法的建立. 渔业科学进展 , 2014, 35 (1) : 60-67 | |
| 耿伟光, 史成银, 李晋, 等. 同步检测海水养殖动物5种病原菌的扩增子拯救多重PCR(Arm-PCR)方法的建立与应用. 农业生物技术学报 , 2013, 21 (9) : 1125-1134 | |
| 徐洪涛, 朴春爱, 姜忠良, 等. 养殖牙鲆淋巴囊肿病病原的研究. 病毒学报 , 2000, 16 (3) : 223-226 | |
| 曾伟伟, 王庆, 王英英, 等. 草鱼呼肠孤病毒三重PCR检测方法的建立及其应用. 中国水产科学 , 2013, 20 (2) : 419-426 | |
| Chamberlain JS, Gibbs RA, Rainer JE, et al. Deletion screening of the Duchenne muscular dystrophy locus via multiplex DNA amplification. Nucleic Acids Res , 1988, 16 (23) : 11141-11156 DOI:10.1093/nar/16.23.11141 | |
| Chao CB, Chen CY, Lai YY, et al. Histological, ultrastructural, and in situ hybridization study on enlarged cells in grouper Epinephelus hybrids infected by grouper iridovirus in Taiwan (TGIV). Dis Aquat Organ , 2004, 58 (2-3) : 127-142 | |
| Choi YR, Kim HJ, Lee JY, et al. Chromatographically-purified capsid proteins of red-spotted grouper nervous necrosis virus expressed in Saccharomyces cerevisiae form virus-like particles. Protein Expres Purif , 2013, 89 (2) : 162-168 DOI:10.1016/j.pep.2013.03.007 | |
| Edwards MC, Gibbs RA. Multiplex PCR: advantages, development, and applications. Genome Res , 1994, 3 (4) : S65-S75 DOI:10.1101/gr.3.4.S65 | |
| Elnifro EM, Ashshi AM, Cooper RJ, et al. Multiplex PCR: and application in diagnostic virology. Clin Microbiol Rev , 2000, 13 (4) : 559-570 DOI:10.1128/CMR.13.4.559-570.2000 | |
| Godoy MG, Kibenge MJ, Suarez R, et al. Infectious salmon anaemia virus (ISAV) in Chilean Atlantic salmon (Salmo salar) aquaculture: emergence of low pathogenic ISAV- HPR0 and re-emergence of virulent ISAV-HPR?: HPR3 and HPR14. Virol J , 2013, 10 (1) : 1-17 DOI:10.1186/1743-422X-10-1 | |
| González SF, Krug MJ, Nielsen ME, et al. Simultaneous detection of marine fish pathogens by using multiplex PCR and a DNA microarray. J Clin Microbiol , 2004, 42 (4) : 1414-1419 DOI:10.1128/JCM.42.4.1414-1419.2004 | |
| Han J, Swan DC, Smith SJ, et al. Simultaneous amplification and identification of 25 human papillomavirus types with templex technology. J Clin Microbiol , 2006, 44 (11) : 4157-4162 DOI:10.1128/JCM.01762-06 | |
| Isshiki T, Nishizawa T, Kobayashi T, et al. An outbreak of VHSV (viral hemorrhagic septicemia virus) infection in farmed Japanese flounder Paralichthys olivaceus in Japan. Dis Aquat Organ , 2001, 47 (2) : 87-99 | |
| Jeeva S, Kim NI, Jang IK, et al. Development of a multiplex PCR system for the simultaneous detection of white spot syndrome virus and hepatopancreatic parvovirus infection. Aquac Res , 2014, 45 (6) : 1073-1083 DOI:10.1111/are.2014.45.issue-6 | |
| Lopez-Jimena B, Alonso MC, Thompson KD, et al. Tissue distribution of Red Spotted Grouper Nervous Necrosis Virus (RGNNV) genome in experimentally infected juvenile European seabass (Dicentrarchus labrax). Vet Microbiol , 2011, 154 (1-2) : 86-95 DOI:10.1016/j.vetmic.2011.06.029 | |
| Lyngstad TM, Kristoffersen AB, Hjortaas MJ, et al. Low virulent infectious salmon anaemia virus (ISAV-HPR0) is prevalent and geographically structured in Norwegian salmon farming. Dis Aquat Organ , 2012, 101 (3) : 197-206 DOI:10.3354/dao02520 | |
| Panicker G, Call DR, Krug MJ, et al. Detection of pathogenic Vibrio spp. in shellfish by using multiplex PCR and DNA microarrays. Appl Environ Microbiol , 2004, 70 (12) : 7436-7444 | |
| Rudakova SL, Kurath G, Bochkova EV. Occurrence and genetic typing of infectious hematopoietic necrosis virus in Kamchatka, Russia. Dis Aquat Organ , 2007, 75 (1) : 1-11 | |
| Wallace IS, Gregory A, Murray AG, et al. Distribution of infectious pancreatic necrosis virus (IPNV) in wild marine fish from Scottish waters with respect to clinically infected aquaculture sites producing Atlantic salmon, Salmo salar L. J Fish Dis , 2008, 31 (3) : 177-186 DOI:10.1111/jfd.2008.31.issue-3 | |
| Zou SM, Han J, Wen LY, et al. Human influenza A virus (H5N1) detection by a novel multiplex PCR typing method. J Clin Microbiol , 2007, 45 (6) : 1889-1892 DOI:10.1128/JCM.02392-06 |



