2. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 青岛瑞滋海珍品发展有限公司 青岛 266409
2. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071 ;
3. Qingdao Ruizi Precious Seafood Development Limited Company, Qingdao 266409
仿刺参(Apostichopus japonicus)属底栖性生物,主要以微生物、微藻、大型藻类碎片、有机碎屑、底泥等为食(于东祥等, 2005; 田传远等, 2008),其中细菌是仿刺参能量的主要来源(廖玉麟, 1997)。仿刺参的消化系统较为简单,肠道是主要的消化器官,能够利用简单的消化系统消化复杂的食物,除了消化系统消化外,肠道中的菌群也起到了重要的作用。食物进入肠道后,大量定植细菌也进入食物内,辅助仿刺参消化复杂的食物,抑制食物中病原菌的繁殖和侵染(Clifford et al,1982)。仿刺参肠道中的复杂细菌主要来源于栖息环境,并与外界环境菌群保持相对稳定的动态平衡。
仿刺参池塘养殖工艺简单、产量和效益可观,是仿刺参养殖主要的方式之一,其中山东省仿刺参池塘养殖面积占23%(李成林等, 2010)。仿刺参池塘养殖过程中可通过投喂益生菌剂调节养殖环境和仿刺参肠道中的菌群结构,实现健康养殖。因此,研究池塘养殖中的仿刺参肠含物及环境中菌群结构,对解析仿刺参肠道消化作用,分析肠道中潜在益生菌具有重要意义。
平板培养法是分析环境细菌数量和种类的传统方法,但受培养基种类、浓度及培养条件的限制,大多数细菌不能在同一种培养基上生长(乔旭东等, 2007)。据报道,目前99%以上的海洋细菌为非可培养细菌(张晓华等, 2007),传统的平板培养法不能客观、真实地反映样品菌群结构。近年来,随着分子生物学技术的迅速发展,相继建立了不依赖于培养法的菌群结构分析方法,其中,PCR-DGGE技术突破了传统方法的局限,且成本低、速度快、操作简单,广泛应用在环境微生物多样性的研究(刘新春等, 2005; 张振冬等, 2008; 马悦欣等, 2003)。本研究利用PCR-DGGE技术分析了池塘养殖仿刺参肠含物和养殖环境的菌群结构,利用生物信息学解析了二者的相关性,以期为仿刺参健康养殖和开发益生菌提供参考。
1 材料和方法 1.1 采样实验在位于青岛市黄岛区琅琊镇的青岛瑞滋海珍品发展有限公司的3个标准化仿刺参养殖池塘中进行,池塘长400 m、宽100 m、水深1.8 m;2014年10月中旬采样,采样时水温为16℃,盐度为32;池塘附着基为瓦片附着基。
底泥样品用采泥器采集池底表层沉积物,每个池塘选3个采样点,样品采集后进行混合,–20℃保存,不同池塘的样品编号为N1、N2、N3。
每个池塘随机取3片上层瓦状附着基,用无菌勺刮取其上的沉积物混合后–20℃保存,不同池塘附着基样品编号为F1、F2、F3。
每个池塘的附着基周围随机采集3头仿刺参,仿刺参规格为(150±10) g,无菌操作下剖取完整肠道,用镊子轻轻挤压肠道,收集肠含物后混合样品,–20℃保存,不同池塘肠含物样品编号为C1、C2、C3。
1.2 基因组DNA提取利用OMEGA公司的Soil DNA Kit试剂盒提取样品的细菌DNA。
1.3 PCR扩增采用细菌16 S rDNA的V3–V5区引物341F-GC(5’-CCTACGGGAGGCAGCAG-3’)和907R(5’-CCGTCAATT CCTTTGAGTTT-3’)。PCR反应采用50 µl反应体系:1×Buffer,引物0.2 μmol/L,dNTP 200 μmol/ml,TaqDNA聚合酶4 U,DNA模板50 ng,灭菌水补充至50 μl。反应条件为:94℃预变性5 min, 前10个循环为94℃ 30 s,65℃-55℃ 30 s,72℃ 1 min(其中每个循环复性温度下降1℃), 后20个循环为94℃ 30 s,55℃ 30 s,72℃ 1 min,72℃延伸10 min,1%琼脂糖凝胶电泳检测。
1.4 变性梯度凝胶电泳(DGGE)用于分离16S rDNA片段的DGGE电泳条件为:6%(w/v)聚丙烯酰胺凝胶(丙烯酰胺:双丙烯酰胺=37.5 : 1),变性剂为尿素,变性梯度浓度范围为30%– 70%,将PCR产物与2×loading Buffer按1 : 1进行混合,每个加样孔的上样量为30 µl,120 V 60℃电泳12 h。使用1% Genefinder对凝胶进行染色,UVP凝胶影像系统拍照。
1.5 DGGE条带的重扩增、克隆及测序在紫外灯下切割各泳道主要的条带,切割后将胶置于30 μl的ddH2O中过夜,12000 r/min离心10 min取上清液。用回收的DNA 1 μl做模板,以不带GC夹的341F/907R进行PCR扩增。对PCR产物纯化后,连接到pEASY-T1载体上,并转化到Top10大肠杆菌感受态细胞中,37℃培养,对检测阳性样品送公司测序。
1.6 数据处理利用Quantity One软件处理与分析DGGE指纹条带,分析不同样品菌群结构的相关性。利用BLAST方法在NCBI数据库中,比较回收条带测序所获得的基因序列的同源性。从中选取与其相似性最高的细菌基因序列,采用Clustalw软件进行多序列匹配排序,用系统发生推断软件包PHYLP 3.67进行聚类分析。采用邻位相连法获得系统发育树,通过自举进行系统进化树的评估,自举数据集为1000次。
2 结果 2.1 16 S rDNA PCR扩增利用细菌16 S rDNA通用引物GC-F341/R907对提取的细菌DNA进行PCR扩增。结果显示,扩增条带亮度和特异性均较好,未出现非特异性扩增条带,通过与DNA Marker对比,片段大小在550 bp左右。
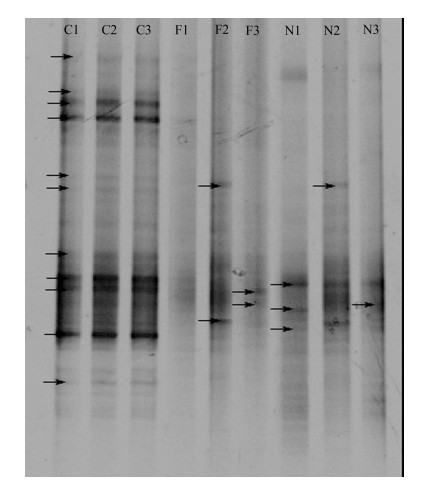
2.2 DGGE指纹图谱分析DGGE电泳结果显示,上述样品DNA扩增产物经聚丙烯酰胺变性梯度凝胶电泳均获得了条带清晰电泳图谱,将DGGE指纹图谱中位于相同位置的条带规定为一个可操作分类学单位(OTU, Operational Taxonomic Unit) (赵兴青等, 2006)。不同样品之间条带的粗细、数目和位置不同,说明仿刺参肠含物与养殖环境中细菌群落结构不同,泳道中条带粗细、数目不一,条带的数目反映出群落的生物种类的多样性,条带信号强弱反映各物种的丰度(罗鹏等, 2009)。由DGGE指纹图谱(图 1)和(图 2)可以看出, C1、C2、C3、F1、F2、F3、N1、N2、N3分别获得29、30、31、17、16、13、24、23、18条可见条带,肠含物、附着基、底泥平均条带数分别为30.00±1.00、15.33±1.70、21.67±2.62。肠含物中细菌种类较多,底泥次之,附着基细菌种类最低,仿刺参肠含物与养殖环境中细菌群落优势条带十分明显,它们分别对应DGGE指纹图谱中的亮条带,肠含物中优势条带明显而且比较多,说明肠道中存在优势度较高的细菌种类。
|
图 1 各样品16SrDNA扩增产物的DGGE电泳结果 Figure 1 The PCR–DGGE electrophoresis results of 16S rDNA from different samples C1、 C2、 C3为肠含物样品;F1、 F2、 F3为附着基样品;N1、 N2、 N3为底泥样品 C1, C2, C3: Gut content; F1, F2, F3: Attachments; N1, N2, N3: Bottom sediment |

|
图 2 仿刺参肠含物与养殖环境样本DGGE指纹图谱 Figure 2 The pattern of PCR–DGGE fingerprint of 16S rDNA from A. japonicus gut content samples and culture environment samples 1、 2、 3为肠含物样品;4、 5、 6为附着基样品;7、 8、 9为底泥样品 1, 2, 3: Gut content; 4, 5, 6: Attachments; 7, 8, 9: Pond sediment |
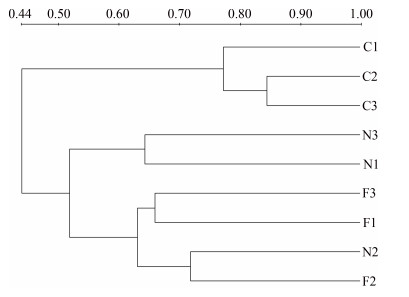
用戴斯系数计算出的各样品相似性矩阵见图 3。图 3显示,各样品按相似程度可以分为4大簇,其中3个肠含物(C1、C2、C3)聚为一簇,相似性在0.725– 0.843之间,平均相似性为0.785。附着基样品(F1、F3)聚为一簇,相似性为0.658,底泥样品(N1、N3)聚为一簇,相似性为0.642,附着基样品F2与底泥样品N2聚为一簇,相似性为0.717。从聚类分析和相似性系数可以看出:3个肠含物(C1、C2、C3)、2个附着基样品(F1、F3)、2个底泥样品(N1、N3)分别具有高度的相似性。其中,附着基F2与F1、F3有差异,底泥样品N2与N1、N3有差异,这种差异反映了它们细菌群落的不同。通过聚类分析还可以看出,附着基与底泥样品在各自聚为一簇的同时,最后聚为一大簇,说明附着基与底泥样品细菌群落结构相似性较高,平均相似性为0.532。
|
图 3 仿刺参肠含物与养殖环境样本DGGE指纹图谱的聚类分析 Figure 3 The clustering analysis of the gut content of the sea cucumber and culture environment samples based on the PCR-DGGE fingerprint |
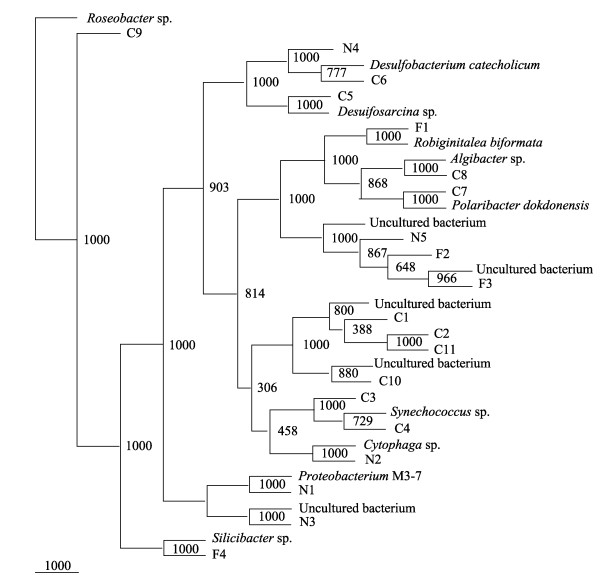
对DGGE指纹图谱的20条主要条带进行切割,切割条带位置如图 1箭头所示。切割条带全部获得阳性克隆,所获得的序列在NCBI中用BLAST进行同源性比较,20个序列所代表的细菌种类比较广泛(图 4),包括以下几个纲(门):蓝藻门(Cyanophyta)、α–变形菌纲、δ–变形菌纲、γ–变形菌纲、黄杆菌纲(Flavobacteria)、鞘脂杆菌纲(Sphingo bacteria),其中8个序列在NCBI比对结果是未可培养的细菌(Uncultured bacterium)。变形细菌门(Proteobacteia)在12个序列中占41.6%,拟杆菌门(Bacteroidetes)占33.3%,说明变形细菌门和拟杆菌门是养殖仿刺参肠含物与环境中主要的细菌类群。12个序列中,肠含物主要以聚球藻属(Synechococcus sp.)、脱硫杆菌属(Desulfobacterium)、脱硫叠球菌属(Desulfosarcina sp.)、极地杆菌属(Polaribacter)、Algibacter sp.为主,附着基样品中主要以Robig-initalea sp.、Silicibacter sp.为主,底泥样品中主要以假单胞菌属(Proteobacterium)、噬胞菌属(Cytophaga)、Desulfosarcina sp.为主。
|
图 4 优势菌16S rDNA序列聚类分析 Figure 4 16S rDNA cluster analysis based on 16S rRNA sequence of predominant bacteria |
仿刺参肠含物与养殖环境样本DGGE指纹图谱戴斯系数见表 1。
|
|
表 1 仿刺参肠含物与养殖环境样本DGGE指纹图谱戴斯系数 Table 1 The similarity index between the gut content of the sea cucumber and culture environment samples based on the PCR-DGGE fingerprint (%) |
PCR扩增片段的大小影响DGGE效果,500 bp以上的片段分辨率降低,200 bp片段分辨率最好,但200 bp在片段分析过程中缺乏足够的序列信息,判断细菌所属类群的片段必须在500 bp以上(Hugenholtz et al,1998)。本研究利用细菌16S rDNA V3–V5区的特异性引物341F/907R,扩增片段大小在550 bp左右,即保证了电泳的分辨率,也保存了细菌的片段信息。
仿刺参能够利用简单的消化系统从藻类和有机碎屑中提取营养成分,合成自身的蛋白质。据报道,在仿刺参肠道中含有大量的消化酶如蛋白酶、淀粉酶、脂肪酶、纤维素酶、褐藻酸酶(王吉桥等, 2007),除了消化酶外,肠道内还具有大量辅助消化的细菌(Naomi et al,1996)。本研究分析了仿刺参肠含物及养殖池塘环境(附着基、底泥)中的菌群结构,结果显示,肠含物、附着基、底泥中细菌群落多样性丰富,肠含物中细菌的种类和丰度最为丰富,推测肠含物中含有大量与消化有关的细菌。有研究表明,海参消化道中细菌数量要比周围环境沉积物中的细菌数量多,这与仿刺参的食性有关(于东祥等, 2005),与本研究结果一致。Barlocher等(1978)研究发现,仿刺参摄入的一些细菌不能被消化,而在肠道中定植下来,通过分解食物或产生一些消化酶来辅助消化或提供营养物质。本研究中,肠含物中的细菌与养殖环境不同,肠道中特有的优势细菌可能与仿刺参苗期环境中细菌在肠道中定植有关,是仿刺参肠道中定植辅助消化的定植菌。一般来自同一仿刺参养殖环境中相同来源样品细菌的多样性具有较高的相似性,聚类分析表明,本研究中来自同一养殖环境的相同样品之间存在着差异,可能与仿刺参养殖环境中的大型藻类有关。
本研究中,PCR-DGGE优势条带测序结果显示,变形菌门是仿刺参养殖系统中最大的细菌菌群,这一结论与其他海洋细菌研究结果一致(Stevens et al,2005)。关晓燕等(2010)应用PCR-DGGE技术对高温季节养殖环境中的菌群进行了分析,其主要的优势菌为假单胞菌属,Uncultured delta bacterium,Ostreococcus sp. Uncultured alpha proteobacterium,其优势细菌的种类与本研究存在差异,可能与采样时间和地点不同有关,研究中未被培养的细菌占有优势,与本研究结果一致。本研究发现,40%的细菌是未被培养的细菌,这些细菌是仿刺参肠含物和养殖环境中细菌群落的重要组成部分,Newberry等(2004)对深海沉积物研究发现,未被培养的细菌在总细菌中占有优势,与本研究结果一致。海水中蓝细菌主要为聚球藻属(Synechococcus)和原绿球藻属(Prochlorococcus),这2个属在海洋食物网中占有重要地位;赵三军等(2005)研究表明,胶州湾聚球菌对海洋初级生产力的贡献为3.7%;Ning等(2000)研究表明,夏季聚球藻属对总初级生产力的贡献比较高,春季较低;罗鹏等(2009)利用DGGE对凡纳滨对虾(Litopenaeus vannamei)海水养殖系统进行调查发现,聚球菌属是凡纳滨对虾海水养殖系统最为常见的属。本研究中,在仿刺参肠含物中发现有大量的聚球藻属细菌,而在养殖环境中没有发现,这可能与聚球藻在仿刺参肠道中的定植有关。仿刺参肠含物中还发现有极地杆菌属(Polaribacter),一些极地微生物多不饱和脂肪酸的含量比较高,能产生蛋白酶(陈吉刚等, 2008),推测这些细菌对仿刺参消化有一定的协助作用,具有潜在开发价值。
孙奕等(1989)发现,弧菌和假单胞菌是仿刺参肠道中的主要细菌类群,冯振飞等(2008)利用纯培养的方式发现,锯缘青蟹(Scylla serrata)养殖池塘底泥中优势菌依次为梭状芽孢杆菌属(Clostridium)、芽孢杆菌属(Bacillus)、假单胞菌属(Pseudomonadaceae)、葡萄球菌属(Staphylococcus)和棒状杆菌属(Corynebacterium)。还有报道,底泥中细菌菌群以芽孢杆菌为主(王亚楠等, 2004;罗兰等,2005)。本研究的DGGE切胶回收测序表明,除了底泥样品中一种优势菌的序列和假单胞菌的序列有较高的相似性外,其他细菌序列和已报道的纯培养所获得的细菌序列不同。因此,纯培养获得的多数细菌并不是优势菌群。本研究利用DGGE技术分析了仿刺参肠含物与养殖环境中的菌群结构,客观反映了细菌群落的多样性,并在仿刺参肠含物与养殖环境中发现大量未被培养的细菌,有些仿刺参肠道细菌很少被关注,可能存在潜在的应用价值。
于东祥, 孙慧玲, 陈四清, 等.
海参健康养殖技术. 北京: 海洋出版社, 2005 : 150 -151.
( 0) 0)
|
|
马悦欣, HolmstromC, WebbJ, 等. 变性梯度凝胶电泳(DGGE)在微生物生态学中的应用.
生态学报 , 2003, 23 (8) : 1561-1569 ( 0) 0)
|
|
王吉桥, 唐黎, 许重, 等. 仿刺参消化道的组织学及其4种消化酶活力的周年变化.
水产科学 , 2007, 26 (9) : 481-484 ( 0) 0)
|
|
田传远, 李琪, 梁英.
刺参健康养殖技术. 青岛: 青岛海洋大学出版社, 2008 : 20 -21.
( 0) 0)
|
|
冯振飞, 王国良, 钱冬, 等. 锯缘青蟹养殖环境中细菌类群及其数量分布.
水产科学 , 2008, 27 (11) : 564-577 ( 0) 0)
|
|
乔旭东, 唐学玺, 肖慧, 等. 渤海湾近岸海域的细菌数量分析.
中国海洋大学学报(自然科学版) , 2007, 37 (2) : 273-276 ( 0) 0)
|
|
刘新春, 吴成强, 张昱, 等. PCR-DGGE法用于活性污泥系统中微生物群落结构变化的解析.
生态学报 , 2005, 25 (4) : 842-847 ( 0) 0)
|
|
关晓燕, 周遵春, 陈仲, 等. 应用PCR-DGGE指纹技术分析高温季节仿刺参养殖水环境中菌群多样性.
海洋湖沼通报 , 2010, 19 (1) : 82-88 ( 0) 0)
|
|
孙奕, 陈騳. 刺参体内外微生物组成及其生理特性的研究.
海洋与湖沼 , 1989, 20 (4) : 300-307 ( 0) 0)
|
|
李成林, 宋爱环, 胡炜, 等. 山东省刺参养殖产业现状分析与可持续发展对策.
渔业科学进展 , 2010, 31 (4) : 127-133 ( 0) 0)
|
|
张振冬, 王淑芬, 曹宇峰. DGGE技术及其在海洋环境微生物多样性研究中的应用.
海洋环境科学 , 2008, 27 (3) : 297-300 ( 0) 0)
|
|
张晓华.
海洋微生物学. 青岛: 中国海洋大学出版社, 2007 : 181 -182.
( 0) 0)
|
|
陈吉刚, 杨秀芳. 极地微生物的工业应用及其与天体生物学研究的联系.
生命的化学 , 2008, 28 (1) : 97-100 ( 0) 0)
|
|
罗兰, 谢丙炎, 袁忠林, 等. 我国土壤中苏云金芽孢杆菌的分离与基因型的鉴定.
应用与环境生物学报 , 2005, 11 (6) : 756-759 ( 0) 0)
|
|
罗鹏, 胡超群, 张吕平, 等. 凡纳滨对虾海水养殖系统内细菌群落的PCR-DGGE分析.
中国水产科学 , 2009, 16 (1) : 32-38 ( 0) 0)
|
|
赵三军, 肖天, 李洪波, 等.胶州湾聚球菌(Synechococcus spp.)
( 0) 0)
|
|
蓝细菌的分布及其对初级生产力的贡献.海洋与湖沼, 2005, 36(6): 534–540
( 0) 0)
|
|
赵兴青, 杨柳燕, 陈灿, 等. PCR-DGGE技术用于湖泊沉积物中微生物群落结构多样性研究.
生态学报 , 2006, 26 (11) : 3610-3616 ( 0) 0)
|
|
廖玉麟.
中国动物志. 北京: 科学出版社, 1997 : 55 -56.
( 0) 0)
|
|
Barlocher FB, Kendrick.Hyphomycetes as intermediaries of energy flow in streams. Recent Advances in Aquatic Mycology, Publisher: Paul Elek, London, 1978, 435–446
( 0) 0)
|
|
Clifford C, Walsh J, Reidy N, et al. Digestive enzymes and subcellular localization of disaccharidases in some echinoderms.
Comp Biochem Physiol B Comp Biochem , 1982, 71 (1) : 105-110 DOI:10.1016/0305-0491(82)90183-3 ( 0) 0)
|
|
Hugenholtz P, Goebel BM, Pace NR. Impact of culture– independent studies on the emerging phylogenetic view of bacterial diversity.
J Bacteriol , 1998, 180 (18) : 4765-4774 ( 0) 0)
|
|
Naomi WR, Fred AR, Erko S. A study of the bacterial flora associated with Holothuria atra.
J Exp Mar Biol Ecol , 1996, 203 (11) : 11-26 ( 0) 0)
|
|
Ning XR, Cloem JE, Cole BE, et al. Spatial and temporal variability of picocyanobacteria Synechococcus spp.In San Francisco Bay.
Limnol Oceanogr , 2000, 45 (3) : 695-702 DOI:10.4319/lo.2000.45.3.0695 ( 0) 0)
|
|
Newberry CJ, Webster G, Cragg BA, et al. Diversity of prokaryotes and methanogenesis in deep subsurface sediments from the Nankai Trough, Ocean Drilling ProgramLeg 190.
Environ Microbiol , 2004, 6 (3) : 274-287 DOI:10.1111/emi.2004.6.issue-3 ( 0) 0)
|
|
Stevens H, Stübner M, Simon M, et al. Phylogeny of Proteobacteria and bacteroidetes from oxic habitats of a tidal flat ecosystem.
FEMS Microbiol Ecol , 2005, 54 (3) : 351-365 DOI:10.1016/j.femsec.2005.04.008 ( 0) 0)
|



