2. 烟台出入境检验检疫局 烟台 264000;
3. 东南大学 南京 210000
2. 2. Yantai Entry-Exit Inspection and Quarantine Bureau, Yantai 264000 ;
3. Southeast University, Nanjing 210000
黄头病(Yellow head disease, YHD)是由黄头病毒(Yellow head virus, YHV)引起的一种对虾疾病,该病与对虾白斑病(White spot syndrome virus, WSSV)、桃拉综合征(Taura syndrome virus, TSV)并称为“影响世界对虾养殖业的三大病害”(OIE, 2015)。黄头病毒属于黄头症候群病毒(Yellow head complex of viruses)基因Ⅰ型,是黄头病的唯一病原(Lightner, 1996),为单正链RNA病毒,毒力较强,对虾被感染后3–5 d内可全军覆灭(OIE, 2015),世界动物卫生组织(International Epizootic Office, OIE)将黄头病列为必须进行疫情上报的疫病,我国农业部也将黄头病列为水生动物二类疫病。OIE水生动物疫病诊断手册(OIE, 2015)认为,在虾生活周期全过程中适用于YHD检测、诊断和确诊的分子生物学方法仅有PCR方法(RT-PCR或巢式RT-PCR)与核酸测序,但这两种方法操作复杂繁琐、测序确证周期较长、成本较高,需要昂贵仪器和熟练的操作人员,检测场所也局限于有条件的实验室中,不能满足现场快速检测的需要。
超分支滚环扩增(Hyper-branched rolling circle amplification, HRCA)技术是Lizardi等(1998)在线性滚环扩增的基础上建立起来的,该方法需要设计特异性的锁式探针(Padlock probe, PLP)(Nilsson et al, 1994)及1对通用引物,通用引物可识别锁式探针并与锁式探针的滚环扩增产物相结合,当DNA聚合酶存在且发挥作用时,实现对锁式探针的HRCA,扩增效率高,在1 h内扩增效率可达到109拷贝(Prins et al, 2008; Zhou et al, 2004),从而实现对检测信号的放大。由于其高敏感性、特异性,HRCA技术已开始应用于单核苷酸多态性研究(Pickering et al, 2002; Cheng et al, 2008)、细胞原位检测(Christian et al, 2001)、DNA芯片检测(Nallur et al, 2001; Prins et al, 2008)和免疫芯片检测(Zhou et al, 2004)等方面。HRCA检测试纸技术是在HRCA检测技术的基础上增加了一个生物素标记的检测探针,从而将HRCA技术与试纸显色技术相结合,从而实现对HRCA检测结果的直观、可视化处理。
本研究旨在依据YHV基因保守核苷酸序列,设计特异的锁式探针及引物,通过优化HRCA条件,构建特异、准确的黄头病毒HRCA检测技术体系,并在此基础上制备YHV HRCA检测试纸,可望为对虾黄头病毒病害的动态检测和控制提供一种新的分子检测技术。
1 材料与方法 1.1 核酸来源YHV及其用于特异性分析的WSSV、TSV、传染性皮下及造血器官坏死病毒(Infectious hypodermal and hematopoietic necrosis virus, IHHNV)均为山东出入境检验检疫局技术中心实验室保存样本。
1.2 主要试剂 1.2.1 商业化试剂本实验所采用的所有引物、探针均由Invitrogen公司合成;脱氧核糖核苷三磷酸(dNTP)、ExTaq DNA聚合酶混液、Triton X-100、T4 DNA连接酶(含10×缓冲液)、one-step RT-PCR试剂盒、DL2000分子标记均购自TaKaRa公司;Bst DNA聚合酶大片段(含10×缓冲液)购自New England Biolabs公司,Qiagen快速凝胶提取试剂盒购于Qiagen公司;辣根过氧化物酶(Horse radish peroxidase, HRP),四甲基联苯胺(Tetramethylbenzidine, TMB)购自上海生工生物工程技术服务有限公司。
1.2.2 实验室配制的试剂变性液:0.4 mol/L NaOH溶液,25 mmol/L乙二胺四乙酸(EDTA)溶液;杂交缓冲液:1×柠檬酸钠缓冲液(SSC),0.1%十二烷基硫酸钠溶液(SDS);洗液:0.5×SSC、0.1% SDS;缓冲液A:磷酸盐缓冲液(PBS)、137 mmol/L NaCl溶液、2.7 mmol/L KCl溶液、1.4 mmol/L KH2PO4溶液、4.3 mmol/L Na2HPO4溶液、体积分数0.3% Triton X-100溶液,pH=7.4;缓冲液B:缓冲液A、1 mol/L尿素溶液、1 g/100 ml硫酸葡聚糖溶液;缓冲液C:100 mmol/L柠檬酸钠,pH=5.0。
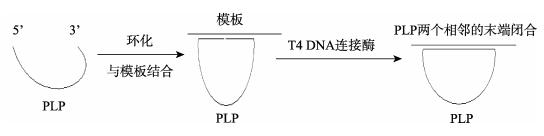
1.3 实验方法 1.3.1 锁式探针和引物的设计根据YHV非结构蛋白N基因序列(GenBank Access No. FJ848674)设计锁式探针两端的特异识别序列(表 1锁式探针大写字母序列),使两端的特异识别序列与单链模板结合后紧密相邻,两端特异性识别序列中间的连接部分为pNAK1质粒中的一段核酸序列(表 1锁式探针小写字母序列),连接部分序列根据特异识别序列进行适当修改。锁式探针与模板的结合见图 1。
|
|
表 1 引物及序列探针 Table 1 Primers and probe sequences |
|
图 1 锁式探针与靶序列的连接(刘青杰等, 2007; Tsui et al, 2010) Figure 1 The connection of PLP and target |
YHV基因RT-PCR特异扩增引物对为上游引物YNS1F和下游引物YNS1R,特异性检测引物对为上游引物YHV-F和下游引物YHV-R,WF1、WF2为经生物标记的检测通用引物,检测探针序列为锁式探针2个特异性末端与模板相结合的衔接区域,各序列详见表 1。
1.3.2 标准品的制备用引物YNS1F/YNS1R对YHV基因组RNA进行RT-PCR扩增,扩增产物经1.5% (m/V)琼脂糖凝胶电泳分离后,目的扩增条带用Qiagen快速凝胶提取试剂盒纯化,回收后连接改造后的pGEM-T载体(肖性龙等, 2011)制备重组质粒,转化大肠杆菌进行克隆增殖。利用T7体外转录试剂盒转录该重组质粒,并获得大量RNA核酸标准物质,测定OD值,计算拷贝数,梯度稀释后作为标准品。
1.3.3 锁式探针连接反应条件的优化最佳探针浓度的确定:分别设置4个浓度梯度的锁式探针即1 μmol/L、100 nmol/L、10 nmol/L、1 nmol/L,连接体系为锁式探针1 μl,T4DNA连接酶1 U/μl,10×T4 DNA连接酶缓冲液1 μl,105拷贝/μl模板RNA 2 μl,加去离子水至反应总体积10 μl。反应条件为:94℃ 5 min后迅速置于冰上,2 min后加T4 DNA连接酶,37℃连接1 h,最后65℃ 10 min终止连接反应。
最佳连接时间的确定:参照上述反应体系和反应条件,在最佳探针浓度条件下,37℃下分别连接10、20、30、40、50、60 min以确定最佳的连接反应时间。
1.3.4 HRCA反应条件的优化HRCA反应体系:dNTPs 0.4 mmol/L,WF1 0.4 μmol/L,WF2 0.4 μmol/L,Tris-HCl (pH=8.8) 20 mmol/L,KCl 10 mmol/L,MgSO4 6.5 mmol/L,(NH4)2SO4 10 mmol/L,0.1% Triton X-100,2 μl连接产物与8 U Bst DNA聚合酶大片段,加dH2O使反应体系总体积为25 μl。61℃分别扩增10、20、30、40、50、60、70、80 min,以确定最佳扩增时间,95℃ 10 min终止反应。反应产物经琼脂糖凝胶电泳检测,若条带呈阶梯状,则表示该样品为YHV阳性,反之为阴性。
1.3.5 检测试纸的制备及检测将3 μl检测探针与17 μl变性液室温下混匀5 min,取5 μl点于硝酸纤维素膜上,室温下自然晾干,每个点样点可重复多样2–3次,每个点设置3个平行对照;UV光源下(波长254 nm,强度60–120 mJ/cm2)照射使DNA固定在膜上;37℃条件下,用脱脂奶粉(5 g/100 ml)封闭1.5 h,备用。
检测试纸按如下流程进行操作:将试纸放入5 ml预热的杂交缓冲液与扩增产物的混合溶液中,60℃温浴2 h;将试纸转移至50 ml预热洗液中,60℃震荡15 min;取出试纸,室温下放入缓冲液A中,加入1 : 2000稀释的HRP-标记链霉亲和素,反应10 min;试纸转移至缓冲液B中,室温下漂洗5 min;试纸转移至缓冲液C中,室温下浸泡5 min;加入TMB显色1–20 min,而后终止反应并观察结果。试纸检测结果也采用Gelpro图像分析软件进行dot值分析。
1.3.6 检测试纸的特异性与灵敏度分析取YHV、TSV、IHHNV和WSSV的核酸,用建立的检测试纸分别对4种病毒进行检测,验证其特异性。取标准品100–105拷贝/μl浓度梯度的核酸分别作为模板,进行连接和扩增反应,产物经试纸检测,显色后采用Gelpro图像分析软件进行dot分析,检测试纸的灵敏度。将该检测试纸灵敏度与常规RT-PCR方法进行比较,常规RT-PCR采用one-step RT-PCR试剂盒(TaKaRa),反应条件为:扩增反应为25 μl体系,包含10 mmol/L引物YHV-F、YHV-R各1 μl,PrimeScript 1 Step Enzyme Mix 1 μl,2×1 step buffer 12.5 μl,dH2O 7.5 μl,YHV病毒基因组RNA 2 μl,反应经50℃ 30 min后,94℃ 1 min,58℃退火30 s,72℃反应2 min,反应30个循环,而后72℃延伸10 min,扩增产物经2%(m/V)琼脂糖凝胶电泳鉴定。
1.3.7 试纸检测方法的实际应用利用建立的YHV超分支滚环试纸对80批次采集的虾样本进行检测(按照表 3中各种虾的明细进行修改),其中,中国对虾样本(Fenneropenaeus chinensis) 30批次、斑节对虾(Penaeus monodon) 16批次、凡纳滨对虾(Litopenaeus vannamei) 4批次,泰国与越南进口斑节对虾、凡纳滨对虾共20批次,澳大利亚进口斑节对虾、墨吉尔虾(Penaeus merguiensis)共10批次。将HRCA试纸的检测结果与常规RT-PCR方法的结果对比,比较2种检测方法的优越性。
|
|
表 3 两种方法的样品检测结果 Table 3 The test results of the 80 YHV samples by two methods |
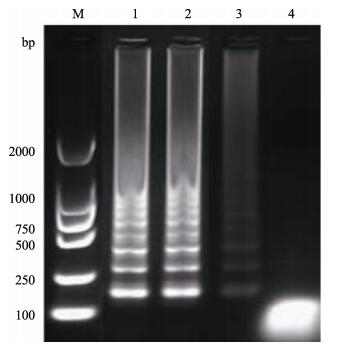
按照1.3.3所述的锁式探针连接反应体系和条件,其中,设置4个磷酸化锁式探针浓度进行连接反应,反应1 h后,以1.3.4中所述体系进行HRCA反应。结果显示,锁式探针浓度≥100 nmol/L时,HRCA反应产物电泳结果条带亮度相近且清晰可见,低于这个浓度扩增带模糊不清,确定该浓度为最佳探针浓度(图 2)。
|
图 2 YHV最优锁式探针浓度的确定 Figure 2 Optimization of the PLP concentration for YHV M: DL2000分子标记Molecular Marker; 1: 10 μmol/L; 2: 100 nmol/L; 3: 1 nmol/L; 4: 10 pmol/L |
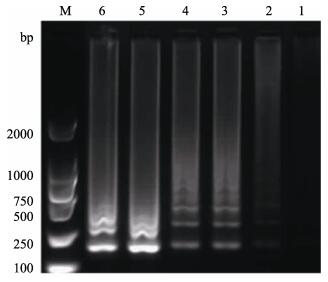
按照1.3.3所述的锁式探针连接反应体系和条件,进行连接反应以确定最优连接时间,锁式探针的浓度为100 nmol/L,并设置6个连接反应时间,反应结束后,以1.3.4中所述体系进行HRCA反应。结果显示,当探针连接时间≥30 min时,HRCA反应产物电泳结果条带亮度相近且清晰可辨,确定最佳连接时间为30 min(图 3)。
|
图 3 锁式探针最适连接时间的确定 Figure 3 Optimization of the PLP connection time for YHV M: DL2000分子标记Molecular Marker; 1–5: 10, 20, 30, 40, 50, and 60 min respectively |
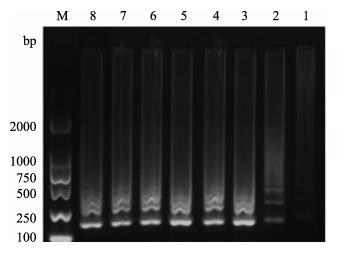
当模板浓度为105copies/μl、锁式探针浓度为100 nmol/L时,锁式探针连接30 min,61℃条件下,在8个不同反应时间进行HRCA,产物经电泳观察,当时间≥30 min时,条带亮度相当且清晰可见(图 4)。因此,确定30 min为HRCA的最优反应时间。
|
图 4 HRCA反应条件的优化 Figure 4 The optimization of reaction conditions of HRCA M: DL2000分子标记Molecular Marker; 1–8: time of the HRCA 10, 20, 30, 40, 50, 60, 70, 80 min respectively |
YHV HRCA试纸特异性检测结果显示,该试纸仅对YHV有检出,并且与其他3种病毒无交叉反应,说明本方法能够保证YHV病毒的特异性检测(图 5)。

|
图 5 YHV HRCA试纸的特异性检测 Figure 5 The specificity test for YHV HRCA test strip 1–4: IHHNV, YHV, TSV, and WSSV respectively; Neg:阴性对照Negative control |
通过对检测试纸斑点的颜色变化和斑点值的分析得出,当样本浓度为10 copies/μl时,试纸仍可检出与阴性对照有明显差异的着色斑点,而RT-PCR方法的结果则无明显扩增带;当样本浓度为1 copy/μl时,试纸虽肉眼可见微弱着色斑,但与阴性对照差异不大,通过核酸样本的斑点扫描结果(表 2),可判定为阴性。因此,本检测方法的灵敏度为10 copies/μl,而常规RT-PCR方法的检测限为1000 copies/μl,HRCA试纸法比常规PCR方法高2个数量级(图 6)。
|
|
表 2 试纸检测YHV梯度核酸样本斑点的结果 Table 2 The dot value of HRCA test strip for YHV |
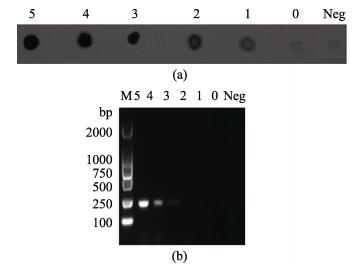
|
图 6 YHV HRCA试纸灵敏度验证 Figure 6 The sensitivity test for the HRCA test strip (a)HRCA检测试纸;(b)常规RT-PCR;M:DL2000;0–5的模板浓度依次为100–105 copies/μl (a) HRCA test strip for YHV; (b) Agarose gel electrophoresis of RT-PCR; M: DL 2000; 0–5: 100–105 copies/μl |
通过对2014年国内与进口共计80批次虾样本的YHV检测,结果显示,50批次国内虾样本中HRCA试纸与RT-PCR检测结果一致,均为YHV阴性;30批次进口虾中2批次呈YHV阳性,阳性样本为越南进口斑节对虾,HRCA试纸检测结果与RT-PCR结果一致,见表 3。但其中1批次YHV RT-PCR阳性样品因扩增条带太弱,不能进行测序确证,另1组YHV阳性RT-PCR结果经测序确证无误。
3 讨论本研究建立了灵敏度高、特异性好的YHV HRCA试纸检测方法,HRCA的特异性是由锁式探针两端的特异、保守核酸序列所决定的,该序列能够特异性地与目的病毒核酸序列相结合;由于HRCA是在滚环扩增的基础上进一步进行双引物多分支扩增,从而大大提高了扩增效率,因此,可以实现短时间内对检测信号的逐级放大,从而提高了检测灵敏度,通过对本实验建立的YHV HRCA试纸灵敏度、特异性的研究,结果显示,该方法灵敏度可以达到10个拷贝,可以满足YHV常规检测的要求。在特异性实验中,本研究选用OIE列出的WSSV、TSV、IHHNV虾类病毒,这3种虾类病毒也是我国国家海域监控的主要虾类病害,本研究结果表明,3种病毒与YHV检测方法均无交叉反应。
对于HRCA方法,锁式探针的设计至关重要,两端特异识别序列决定了反应的特异性,并与假阳性和假阴性的产生密切相关,锁式探针必须与模板正确结合、连接且形成闭合环状结构,当锁式探针不能正常连接时,会出现假阴性;而当锁式探针在无特异模板存在的体系中完成连接,则产生假阳性,所以,锁式探针能否与目的核酸序列正确连接决定了检测结果的成败(孔铖将等, 2011)。目前,已报道的锁式探针的最适长度为140 nt (Antson et al, 2000),而本研究所设计的长度为127 nt (由88 nt的连接序列和39 nt的两端特异识别序列组成),二者长度接近。已有研究表明,HRCA对锁式探针5x末端无严格特异性要求,只需3x末端与目的核酸序列完全互补(Szemes et al, 2005),本研究所设计的锁式探针的5x末端与3x末端DNA熔解温度(Tm)一致,因而探针两端可以同时与模板结合,具有一致性,有利于提高检测的准确率。锁式探针的有效浓度范围一般为4 pmol/L–1 μmol/L (Lizardi et al, 1998; 黄冠军等, 2008; Thomas et al, 1999; Szemes et al, 2005; Tong et al, 2007),本研究确立的锁式探针最适浓度为100 nmol/L,低于这个浓度的锁式探针扩增无明显条带,也有文献报道或呈弥散状(Szemes et al, 2005),这可能是由于本底在Bst DNA聚合酶大片段的作用下扩增而干扰了环化探针扩增的结果,有相关文献(Szemes et al, 2005; Tong et al, 2007; 郝振明等, 2010)采用核酸外切酶Ⅳ对未环化的锁式探针进行消化处理,但本研究经前期条件摸索验证,核酸酶处理前后效果差别不大,且该方法会大大延长反应时间,因此,本研究未采用此方法。
本研究设计的锁式探针是由两端特异的保守序列和中间的链接区域组成,链接区域选自一段pNAK1质粒的核苷酸序列,该链接区域的优点是对多种不同的病毒进行检测时,只需要设计两端的特异性核酸序列,即可以利用1对通用引物,实现对多种疫病的同时检测(刘青杰等, 2007),为后续多病毒检测HRCA检测试纸的建立奠定了基础。
对于整个HRCA的反应,反应温度是由T4 DNA连接酶和Bst DNA聚合酶决定,这2种酶均为恒温反应酶,与以Taq酶为基础的热循环反应方式相比,不需要昂贵的热循环反应装置,从而使得疫病检测更加经济化(彭姣等, 2013)。由于T4 DNA连接酶和Bst DNA聚合酶均为商业化的酶,T4 DNA连接酶有其特定的最佳反应温度,因此,对于疫病检测反应条件的摸索相对比较简单,而Bst DNA聚合酶的反应温度范围较广,其中,61–65℃的温度区间可能影响引物与环化探针的结合(Tsui et al, 2010)。本研究结果表明,Bst DNA聚合酶在3个反应温度梯度(61℃、63℃、65℃)反应产物均无明显差异,因而,选用61℃为最适HRCA反应温度。
HRCA检测试纸是在HRCA反应的基础上,设计检测探针,利用检测探针与锁式探针的互补关系,特异性识别并与目的HRCA产物相结合,这样保证了HRCA试纸检测方法的特异性,而后利用生物素标记的通用引物,对检测结果进行了可视化处理。HRCA检测试纸在保持HRCA方法高灵敏度、高特异性的基础上,实现了检测结果的直接判读,适合水生动物疫病的田间检测。
常规RT-PCR的检测是水生动物疫病YHV检测的金标方法,该方法是针对靶标区域设计特异性引物进行序列扩增,扩增范围内可能存在关键点突变(Wang et al, 2005),因此,在OIE水生动物疫病诊断手册中(OIE, 2015),单纯的RT-PCR反应不能作为疫病确诊的依据,必须与序列测定相结合使用,大大延长了检测时间,并增加了检测成本。与常规RT-PCR检测方法相比,YHV HRCA检测试纸是利用探针与模板的互补关系来保证检测方法的特异性,这一过程类似荧光定量PCR方法的探针与模板的结合过程,具有高度特异性,该特异性可以识别一个碱基的差异,其特异性可以应用于单核苷酸多态性(Single nucleotide polymorphism, SNP)的分析(Prins et al, 2008)。因此,无需进行序列测定,大大缩短了检测周期。
与OIE认可的基于核酸扩增的检测方法–––常规RT-PCR方法和荧光定量RT-PCR方法相比,本研究所建立的YHV HRCA方法及检测试纸无需逆转录过程,反应检测周期短,肉眼直接判读结果,成本低廉,在一定程度上满足了YHV田间检测的需要,为对虾黄头病毒的早期诊断及鉴定提供了一种新的特异、可靠、快速的检测技术。
孔铖将, 史雨红, 黎昊雁, 等.超分支滚环扩增法检测传染性脾肾坏死病毒. 2011, 30(9): 575–579
http://www.cqvip.com/QK/93386X/201109/39335288.html ( 0) 0)
|
|
刘青杰, 陈德清. 滚环DNA扩增技术及其应用.
辐射研究与辐射工艺学报 , 2007, 25 (1) : 5-9 ( 0) 0)
|
|
肖性龙, 余以刚, 翟建新, 等. 含组氨酸纯化标签的假病毒表达载体的构建与应用.
中国生物化学与分子生物学报 , 2011 (7) : 687-692 ( 0) 0)
|
|
郝振明, 赵鑫, 吴孝槐. 超分支滚环扩增技术结合试纸法检测食品中多种转基因组分.
食品科学 , 2010, 31 (6) : 263-266 ( 0) 0)
|
|
黄冠军, 殷幼平, 张仑, 等. 柑桔溃疡病菌滚环扩增检测体系的建立.
微生物学报 , 2008, 8 (3) : 375-379 ( 0) 0)
|
|
彭姣, 王洪强, 李登峰, 等. HRCA技术检测中华鳖虹彩病毒.
微生物学通报 , 2013, 40 (2) : 341-349 ( 0) 0)
|
|
Antson DO, Isaksson A, Landegren U, et al. PCR generated padlock probes detect single nucleotide variation in genomic DNA.
Nucleic Acids Res , 2000, 28 (12) : 596-604 ( 0) 0)
|
|
Cheng YQ, Li ZP, Zhang X, et al. Homogeneous and label-free fluorescence detection of single-nucleotide polymorphism using target-primed branched rolling circle amplification.
Anal Biochem , 2008, 378 (2) : 123-126 DOI:10.1016/j.ab.2008.03.040 ( 0) 0)
|
|
Christian AT, Pattee MS, Attix CM, et al. Detection of DNA point mutations and mRNA expression levels by rolling circle amplification in individual cells.
PNAS , 2001, 98 (25) : 14238-14243 DOI:10.1073/pnas.251383598 ( 0) 0)
|
|
Lightner DV. A Handbook of Shrimp Pathology and Diagnostic Procedures for Diseases of Cultured Penaeid Shrimp. World Aquaculture Society, Baton Rouge, Louisiana, USA, 1996
( 0) 0)
|
|
Lizardi PM, Huang XH, Zhu ZR, et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification.
Nat Genet , 1998, 19 (3) : 225-232 DOI:10.1038/898 ( 0) 0)
|
|
Nallur G, Luo CH, Fang LH, et al. Signal amplification by rolling circle amplification on DNA microarrays.
Nucleic Acids Res , 2001, 29 (23) : e118 DOI:10.1093/nar/29.23.e118 ( 0) 0)
|
|
Nilsson M, Malmgren H, Samiotaki M, et al. Padlock Probes: Circularizing oligonucleotides for localized DNA detection.
Science , 1994, 265 (5181) : 2085-2088 DOI:10.1126/science.7522346 ( 0) 0)
|
|
Office International Des Epizooties. Manual of Diagnostic Tests for Aquatic Animals. [2015-7-20] http://www.oie.int/international-standard-setting/aquatic-manual/access-online/
( 0) 0)
|
|
Pickering J, Bamford A, Godbole V, et al. Integration of DNA ligation and rolling circle amplification for the homogeneous, end-point detection of single nucleotide polymorphisms.
Nucleic Acids Res , 2002, 30 (12) : 205-220 ( 0) 0)
|
|
Prins TW, Dijk JP, Beenen HG, et al. Optimized padlock probe ligation and microarray detection of multiple (non-authorised) GMOs in a single reaction.
BMC Genomics , 2008, 9 (1) : 584 DOI:10.1186/1471-2164-9-584 ( 0) 0)
|
|
Szemes M, Bonants P, Weerdt MD, et al. Diagnostic application of padlock probes-multiplex detection of plant pathogens using universal microarrays.
Nucleic Acids Res , 2005, 33 (8) : e70 DOI:10.1093/nar/gni069 ( 0) 0)
|
|
Thomas DC, Nardone GA, Randall SK, et al. Amplification of padlock probes for DNA diagnostics by cascade rolling circle amplification or the polymerase chain reaction.
Archives Pathology & Laboratory Medicine , 1999, 123 (12) : 1170-1176 ( 0) 0)
|
|
Tong ZS, Kong FR, Wang B, et al. A practical method for subtyping of Streptococcus agalactiae serotype Ⅲ, of human origin, using rolling circle amplification.
J Microbiol Methods , 2007, 70 (1) : 39-44 DOI:10.1016/j.mimet.2007.03.010 ( 0) 0)
|
|
Tsui CK, Wang B, Khadempour L, et al. Rapid identification and detection of pine pathogenic fungi associated with mountain pine beetles by padlock probes.
J Microbiol Methods , 2010, 83 (1) : 26-33 DOI:10.1016/j.mimet.2010.07.016 ( 0) 0)
|
|
Wang B, Potter SJ, Lin YG, et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification.
J Clinical Microbiol , 2005, 43 (5) : 2339-2344 DOI:10.1128/JCM.43.5.2339-2344.2005 ( 0) 0)
|
|
Zhou HP, Bouwman K, Schotanus M, et al. Two-color, rolling-circle amplification on antibody microarrays for sensitive, multiplexed serum-protein measurements.
Genome Biol , 2004, 5 (4) : R28 DOI:10.1186/gb-2004-5-4-r28 ( 0) 0)
|


