2. 上海海洋大学水产与生命学院 上海 201306;
3. 青岛海洋科学与技术国家实验室 海洋生物学与生物技术功能实验室 青岛 266071
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306 ;
3. Marine Biology and Biotechnology Laboratory, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071
水母毒素是目前已知的最毒的海洋生物毒素之一,其毒性是海蛇(Pelamis platuras)毒素的250倍,是河豚毒素的450倍(于华华等,2003)。水母毒素的研究始于20世纪60年代,但因获取困难、稳定性差等因素导致对水母毒素的研究明显滞后于蜂毒、蛇毒、蝎毒。水母毒素成分复杂,既含有能够溶解细胞、具有细胞毒性的多肽、酶类等,又含有一些非蛋白类小分子物质,具有广泛的生物学活性(Martins et al,2009)。磷脂酶A2(Phospholipase A2,PLA2)是水母毒素中含量较为丰富的组分之一,能催化甘油磷脂第2位脂酰键的水解生成溶血磷脂和脂肪酸,参与磷脂的代谢(Glaser et al,1993; Nevalainen et al,2004a、b; Sher et al,2005),具有神经毒性、肌肉毒性、酶活性等多种生物活性。PLA2最先发现于哺乳动物胰液中,随后被发现广泛存在于昆虫、软体动物、蛇和海洋无脊椎动物的毒液中(Nevalainen et al,2004a、b; Razpotnik et al,2010)。根据生物来源、分子量、氨基酸序列同源性、Ca2+是否依赖性以及生理功能等特性的不同,PLA2可以大致分为4类:分泌型PLA2(Secreted phospholipase A2; sPLA2)、胞质型PLA2(Cytoplasmic phospholipase A2; cPLA2)、胞内型PLA2(Intracellular phospholipase A2; iPLA2)和PAF(血小板激活因子)- PLA2(Feng et al,2002)。
海蜇(Rhopilema esculentum)隶属于刺胞动物门、钵水母纲、根口水母目、海蜇属,作为一种大型的可食用经济水母,在我国海洋渔业中占有重要地位。海蜇基础生物学研究深入,人工繁育、增养殖技术成熟,是研究水母毒素组成、结构和功能的理想模式生物。本研究采用转录组454 GS FLX测序和RACE技术,首次解析了海蜇Re-PLA2-1基因的cDNA和基因组结构,分析了其mRNA在海蜇不同发育阶段的表达,这些研究结果将为进一步了解海蜇磷脂酶A2毒素的生物功能奠定基础。
1 材料与方法 1.1 海蜇转录组454 GS FLX测序与EST分析海蜇转录组的构建、454 GS FLX测序与分析见周春娅等(2013)。利用生物信息学方法检索海蜇转录组文库,寻找与已知PLA2基因同源的EST序列。
1.2 Re-PLA2-1 cDNA全长的克隆生物信息学分析发现,海蜇EST(isotig15581)与僧袍芋螺(Conus magus)的PLA2基因具有高度的相似性。根据EST(isotig15581)序列,设计特异性引物Re-PLA2-1 F1和Re-PLA2-1 R1扩增海蜇Re-PLA2-1 cDNA全长(表 1)。3ʹ-RACE使用pBluescript SK(+/-)载体上的通用引物T7与Re-PLA2-1 F1,5ʹ-RACE使用载体通用引物T3与Re-PLA2-1 R1。PCR反应程序为94℃ 5 min;94℃ 30 s,57℃ 30 s,72℃ 30 s,33个循环;72℃ 10 min。PCR产物经琼脂糖电泳检测后胶回收、连接、转化,获得的阳性重组子经菌落PCR验证后送上海鼎安生物科技有限公司测序。
|
|
表 1 引物序列 Table 1 Oligonucleotide primers used in this study |
海蜇于2015年 8月采自青岛沙子口。液氮保存返回实验室后,采用酚-氯仿法提取基因组 DNA:取约100 mg海蜇伞径进行液氮研磨,然后加入400 ml的DNA提取液[其中,Tris-HCl(pH为8.0)100 mmol/L,EDTA(pH为8.0)100 mmol/L,1% SDS 50 ml,20 mg/ml蛋白酶K8 ml],充分混匀,55℃水浴40 min。待裂解完全后,加入等体积的酚-氯仿试剂,静置10min,12000 r/min离心15 min,去除上清液。再加入0.6体积的异丙醇静置7min,12000 r/min离心10 min,除去上清液。然后加入400 ml预冷无水乙醇,沉淀DNA约30 min后,用70%乙醇洗涤沉淀2次,自然干燥后,加入20 ml超纯水溶解。提取的DNA经1.5%琼脂糖电泳进行检测。
根据Re-PLA2-1 cDNA全长设计5对特异性引物进行基因组序列扩增(表 1)。
1.4 Re-PLA2-1 mRNA在不同发育阶段的表达利用Trizol法分别提取海蜇4个不同发育阶段:螅状体(Scyphistoma)、横裂体(Strobila)、碟状体(Ephyra)、水母体(Medusa)的总RNA,然后反转录分别合成cDNA,反应体系及反应条件按说明书要求操作(Invitrogen,美国)。
1.5 Re-PLA2-1基因的生物信息学分析采用实时荧光定量PCR方法,检测Re-PLA2-1在螅状体、横裂体、碟状体和水母体的表达,相关引物见表 1。RT-PCR反应在ABI 7500 Real-time PCR system(Applied Biosystems)上进行。PCR反应体系和流程参照SYBR® Premix Ex TaqTM Ⅱ试剂盒操作说明(TaKaRa)。样品和内参分别设3个重复。反应结束后,采用2-ΔΔCt法和SPSS 18.0统计软件进行数据分析和统计。
用EditSeq和DNAStar软件对DNA序列和推测的氨基酸序列进行生物信息学分析;用Bioedit软件对所获得的测序结果序列进行全长拼接;ORF Finder在线程序(http://www.ncbi.nlm.nih.gov/projects/gorf/)预测开放阅读框,并获得编码的氨基酸序列;用SMART(http://smart.embl-heidelberg.de/)及SingalP 4.1信号肽预测(http://www.cbs.dtu.dk/services/SignalP/)软件对海蜇Re-PLA2-1进行蛋白结构域分析及信号肽预测;用ClustalW(http://www.ebi.ac.uk/clustalw/)进行Re-PLA2-1与其他物种PLA2氨基酸序列的多序列比对。根据多序列比对结果,用Mega 4.1采用邻接法(Neighbor-joining)构建PLA2的系统进化树(Tamura et al,2007)。
2 结果与分析 2.1 Re-PLA2-1基因的全长cDNA分析将3ʹ-RACE和5ʹ-RACE 获得的序列与EST(isotig15581)序列进行拼接,获得海蜇Re-PLA2-1基因的 cDNA全长序列。Re-PLA2-1的cDNA全长序列为824 bp,包括48 bp的5ʹ非编码区(5ʹUTR)、504 bp的开放阅读框(ORF)以及272 bp的3ʹ非翻译区(3ʹUTR),其中,3ʹUTR包括一个多腺苷酸Poly(A)尾和一个多腺苷酸化加尾信号AATAAA(图 1)。Re-PLA2-1为分泌蛋白,信号肽由19个氨基酸组成,酶切后的成熟肽预测的分子量和理论等电点分别为18.93 kDa和7.79。

|
图 1 海蜇Re-PLA2-1基因全长cDNA及推测的氨基酸序列 Figure 1 Full-length Re-PLA2-1 cDNA of R. esculentum and the deduced amino acid sequence 注:下划实线表示信号肽;终止密码子用*标出;方框表示多聚腺苷酸加尾信号;3个内含子插入位点用Y标出 Note: The putative signal peptide was underlined. The asterisk(*)indicated the stop codon and the classical polyadenylation signal was enclosed in a box. The arrow(Y)marked the insertion site of three introns |
BLAST分析发现,预测的海蜇Re-PLA2-1与僧袍芋螺的PLA2氨基酸序列相似性为41%,与刺胞动物门星状海葵(Nematostella vectensis)的PLA2氨基酸序列Nv10-Nv12的相似性在32%-34%之间。多序列比对发现,Ca2+结合位点和组氨酸-天冬氨酸催化位点在所有PLA2 pfam09056成员推测的保守结构域严格保守(图 2)。

|
图 2 海蜇Re-PLA2-1与其他pfam09056家族PLA2s结构域氨基酸多序列比对 Figure 2 Multiple sequence alignment between R. esculentum Re-PLA2-1 and PLA2s from other species 注: 黑色表示相同氨基酸,灰色表示相似氨基酸;*和#分别表示PLA2结构域的Ca2+结合位点和组氨酸-天冬氨酸催化位点 Note: The black shadowing showed identical amino acids and the gray shadowing indicated similar amino acids. The asterisk(*)and pound sign(#)indicatd a calcium-binding site and the His-Asp catalytic active sites of a PLA2 domain respectively |
选择来自不同物种的pfam09056家族的PLA2结构域,应用MEGA 4.1程序,采用邻接法构建系统进化树见图 3。从图 3可以看出,Re-PLA2-1与僧袍芋螺、星状海葵、长牡蛎(Crassostrea gigas)毒素PLA2聚为GⅨ PLA2一支,与GXⅢ、GXⅡ PLA2共聚为pfam-collection,而其他家族共聚为cd-collection。
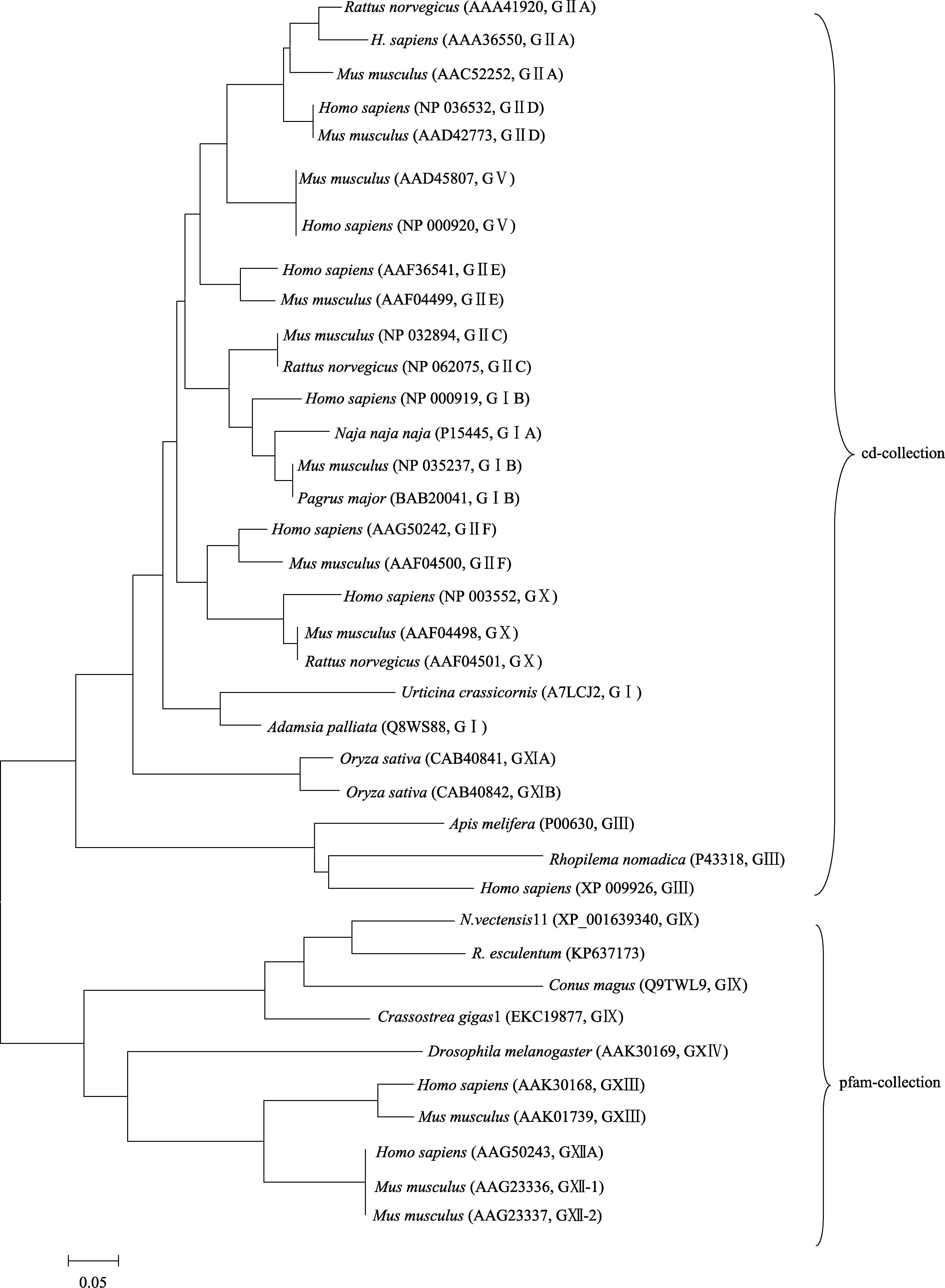
|
图 3 基于邻接法的PLA2 氨基酸序列的系统进化树 Figure 3 Phylogenetic tree of PLA2 based on neighbor-joining method |
利用5对特异性引物扩增获得了5段首尾重叠的Re-PLA2-1基因组片段,将这些片段拼接获得了全长为2671 bp的RePLA2基因组序列。利用NCBI上Splign内含子在线分析工具将Re-PLA2-1的cDNA与基因组序列进行比对分析。结果显示,Re-PLA2-1基因组包含4个外显子和3个内含子(表 2、图 4)。4个外显子大小在39-388 bp之间,3个内含子大小分别为1003 bp、616 bp、494 bp。内含子的A+T含量在62.15%-67.60%之间,而外显子的A+T含量在53.46%-61.08%之间,明显高于外显子。预测的PLA2结构域活性中心“组氨酸-天冬氨酸”(HD)二聚体和Ca2+结合位点天冬氨酸(D)被第2个内含子分隔在第2、3外显子中。所有内含子-外显子边界均符合“AT-CG”剪切规则,按照内含子分型原则,内含子类型分别属于0型和1型(表 2)。
|
|
表 2 Re-PLA2-1内含子/外显子长度、剪切位点及内含子类型 Table 2 Intron/exon lengths,splice junctions,and intron types of Re-PLA2-1 |
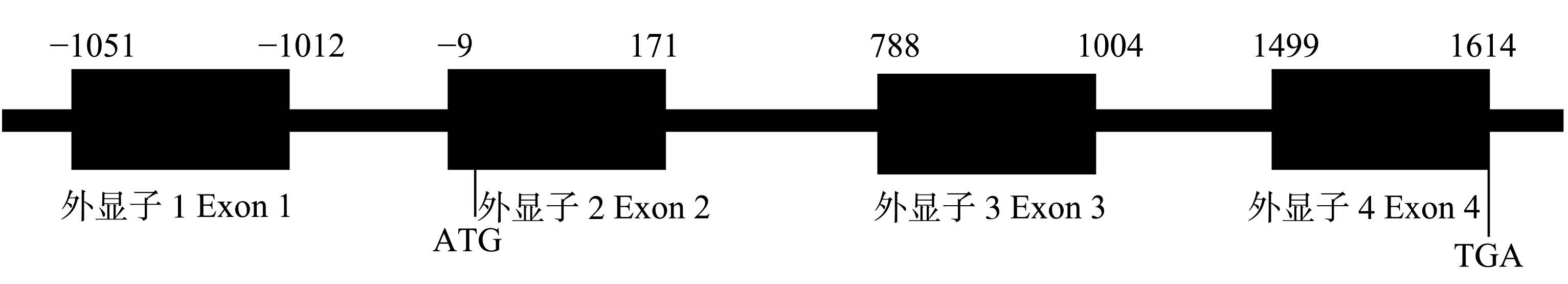
|
图 4 海蜇Re-PLA2-1 基因组结构分布 Figure 4 Genomic organization of R. esculentum Re-PLA2-1 注: 黑色方块代表外显子,黑色粗线代表内含子;数字代表外显子和内含子的位置和大小(bp) |
采用实时荧光定量PCR技术分析了Re-PLA2-1 mRNA在海蜇不同发育阶段的表达见图 5。从图 5可以看出,Re-PLA2-1基因在海蜇的螅状体、横裂体、碟状体和稚水母4个发育阶段均有表达,但存在着明显的表达差异。其中,横裂体阶段表达量最高,碟状体阶段表达量最低,横裂体、螅状体和水母体阶段的表达分别为碟状体阶段表达的12.2、7.6和5.5倍。
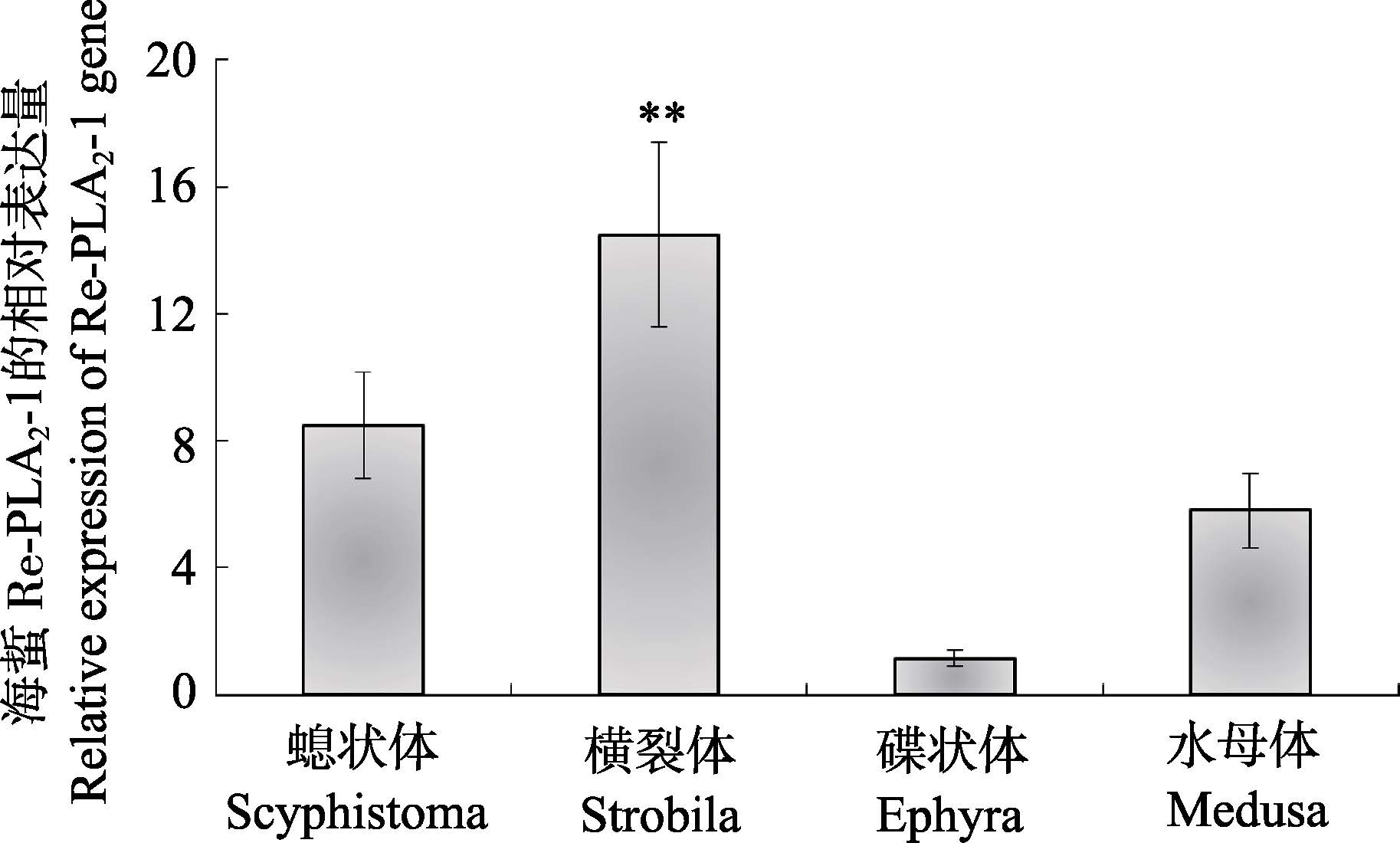
|
图 5 Re-PLA2-1在海蜇不同发育阶段的表达 Figure 5 The expression of Re-PLA2-1 mRNA in different developmental stages ofR. esculentum ** 为差异极显著 ** were considered to be extremely significant differences |
分泌型磷脂酶(sPLA2s)是一类分子量小(14-17 kDa)、由二硫键连接的分泌蛋白,其催化活性依赖于微摩尔的Ca2+存在。根据分子结构的差异,sPLA2s可以分成15个组(Groups,G)和许多亚组(Sub-Groups)(Six et al,2000)。近年来,Nevalainen等(2012)、Punta等(2011)根据保守结构域将sPLA2s分成cd-collection和pfam- collection两大类群。sPLA2s家族在进化上出现较早,从低等微生物到高等脊椎动物均有发现,尽管氨基酸序列差异较大,但Ca2+结合环、催化活性位点在不同家族sPLA2s都严格保守,这说明sPLA2s在不同生物的生命过程中都起关键作用(Nevalainen et al,2012)。
海蜇Re-PLA2-1具有sPLA2s的典型特征:磷脂酶Pfam09056家族严格保守的Ca2+结合位点和催化活性中心“HD”二聚体(图 3)。多序列比对和系统进化分析结果都表明,Re-PLA2-1不但与僧袍芋螺PLA2具有较高的相似性(41%),而且与僧袍芋螺、长牡蛎、水螅等聚类于GⅨ一簇,说明Re-PLA2-1也是pfam09056 GⅨ PLA2家族成员之一。目前,在僧袍芋螺中发现的磷脂酶A2是唯一确定的pfam09056 GIX PLA2s的成员,但其三维结构还未见报道(McIntosh et al,1995; Nevalainen et al,2013)。因此,GⅨ家族磷脂酶Ca2+结合及催化活性中心结构域均基于已知三维结构的紫红链霉菌(Streptomyyce violaceoruber)pfam09056 GIV PLA2s进行预测(Sugiyama et al,2002)。紫红链霉菌PLA2s N端天冬氨酸(Asp)和亮氨酸(Leu)共同参与Ca2+的结合,而刺胞动物PLA2s则衍变为天冬酰胺(A sn)和甘氨酸(Gly),这一变化的具体生物学意义目前还不清楚(Nevalainen et al,2012)。Re- PLA2-1含有11个半胱氨酸(Cys),能形成5对二硫键。在以“HD”二聚体为核心的催化位点具有GⅨPLA2 pfam09056家族成员保守氨基酸序列(ala/ ser-Cys-X-X-His-Asp-X-Cys-Tyr-X-Cys)的特征,其中,Ca2+结合结构域缺失,Ca2+的结合可能也是由N端的Asn和Gly共同介导完成的(Nevalainen et al,2012)
sPLA2在动物中广泛存在,但不同种类的sPLA2基因组结构差异明显。人的GⅡA磷脂酶A2基因组中均含5个外显子4个内含子(Seilhamer et al,1989),而GⅠB磷脂酶的基因组中却含有4个外显子和3个内含子(Jeyaseelan et al,2000)。同时,同一家族的sPLA2在不同物种中其基因组结构也不相同。蜜蜂(Apis mellifera)和蝎子(Anuroctonus phaiodactylus)的GⅢ磷脂酶的基因组均含有4个外显子,人的则有7个外显子,而果蝇(Drosophila melanogaster)的GⅢ磷脂酶A2具有2-6个不等的外显子个数(Valdez-Cruz et al,2007; Xin et al,2009)。海蜇Re-PLA2-1与GⅠB磷脂酶基因组结构相似,均具有4个外显子和3个内含子,说明海蜇Re-PLA2-1可能具有某些与GⅠ磷脂酶相似的基因组进化方式(Jeyaseelan et al,2000)。
PLA2s种类众多,其表达模式和分布随着发育过程发生改变。小鼠(Muroidea sp.)的GⅠB、GⅡA、GⅡD、GⅡE、GⅡF、GⅤ和GⅩ sPLA2s在其胃肠道中均有表达,但在其他组织中分布不同(Eerola et al,2006)。意大利蜂毒PLA2的表达随其日龄和季节发生而变化,如其羽化早期毒囊内表达量很低,羽化后的8-10 d表达量急剧增加,并在随后的成年期保持不变(Owen et al,1990)。本研究发现,Re-PLA2-1在海蜇生活史各时期均有表达,其中,横裂体阶段表达量最高,碟状体表达量最低,2个阶段的表达量相差高达12倍。不同发育阶段Re-PLA2-1的表达差异可能与海蜇在不同发育阶段的摄食方式有关。研究表明,海蜇不同发育阶段具有不同的摄食方式,螅状体依靠触手捕获食物,碟状体依靠缘瓣捕获食物,而水母体则依靠口腕和肩板表面上的吸口完成(刘春洋等,2011)。而利用免疫组化的方法定位地中海水母磷脂酶毒素的分泌部位发现,其磷脂酶毒素是由刺丝囊分泌的(Sher et al,2005),而刺丝囊素作为水母的捕食和防御的重要武器,在其生活史的不同发育阶段均具有重要作用。因此,海蜇Re-PLA2-1尽管在不同发育阶段的表达模式不同,但均在海蜇捕食、防御和食物消化中具有重要作用。
| 于华华, 刘希光, 刘松, 等. 水母毒素的研究现状. 海洋科学 , 2003, 27 (11) : 27-29 | |
| 刘春洋, 王彬, 李轶平, 等. 海蜇不同生长阶段的摄食方式和摄食习性. 水产科学 , 2011, 30 (8) : 491-494 | |
| 周春娅, 朱玲, 潘滢, 等. 海蜇(Rhopilema esculentum)Wnt5基因: cDNA克隆、基因组结构与表达. 海洋与湖沼 , 2013, 44 (4) : 1115-1122 | |
| Eerola LI, Surrel F, Nevalainen TJ, et al. Analysis of expression of secreted phospholipases A2 in mouse tissues at protein and mRNA levels. Biochim Biophys Acta , 2006, 1761 (7) : 745-756 DOI:10.1016/j.bbalip.2006.04.002 | |
| Feng L, Manabe K, Shope JC, et al. A real-time fluorogenic phospholipase A2 assay for biochemical and cellular activity measurements. Chem Biol , 2002, 9 (7) : 795-803 DOI:10.1016/S1074-5521(02)00168-0 | |
| Glaser KB, Mobilio D, Chang JY, et al. Phospholipase A2 enzymes: Regulation and inhibition. Trends Pharmacol Sci , 1993, 14 (3) : 92-98 DOI:10.1016/0165-6147(93)90071-Q | |
| Jeyaseelan K, Armugam A, Donghui M, et al. Structure and phylogeny of the venom group I phospholipase A2 gene. Mol Biol Evol , 2000, 17 (7) : 1010-1021 DOI:10.1093/oxfordjournals.molbev.a026382 | |
| Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ct method. Methods , 2001, 25 (4) : 402-408 DOI:10.1006/meth.2001.1262 | |
| Martins RD, Alves RS, Martins AMC, et al. Purification and characterization of the biological effects of phospholipase A2 from sea anemone Bunodosoma caissarum. Toxicon , 2009, 54 (4) : 413-420 DOI:10.1016/j.toxicon.2009.05.005 | |
| McIntosh JM, Ghomashchi F, Gelb MH, et al. Conodipine-M, a novel phospholipase A2 isolated from the venom of the marine snail Conus magus. J Biol Chem , 1995, 270 (8) : 3518-3526 DOI:10.1074/jbc.270.8.3518 | |
| Nevalainen TJ, Cardoso JCR. Conservation of group XII phospholipase A2 from bacteria to human. Comp Biochem Phys D: Genomics Proteomics , 2012, 7 (4) : 340-350 DOI:10.1016/j.cbd.2012.07.002 | |
| Nevalainen TJ, Peuravuori HJ, Quinn RJ, et al. Phospholipase A2 in cnidaria. Comp Biochem Phys B: Biochem Mol Biol , 2004a, 139 (4) : 731-735 DOI:10.1016/j.cbpc.2004.09.006 | |
| Nevalainen TJ, Quinn RJ, Hooper JNA. Phospholipase A2 in porifera. Comp Biochem Phys B: Biochem Mol Biol , 2004b, 137 (3) : 413-420 DOI:10.1016/j.cbpc.2004.01.010 | |
| Nevalainen TJ, Morgado I, Cardoso JCR. Identification of novel phospholipase A2 group IX members in metazoans. Biochimie , 2013, 95 (8) : 1534-1543 DOI:10.1016/j.biochi.2013.04.003 | |
| Owen MD, Pfaff LA, Reisman RE, et al. Phospholipase A2 in venom extracts from honey bees (Apis mellifera L.) of different ages. Toxicon , 1990, 28 (7) : 813-20 DOI:10.1016/S0041-0101(09)80004-4 | |
| Punta M, Coggill PC, Eberhardt RY, et al. The Pfam protein families database. Nucleic Acids Res , 2011, 40 (D1) : 290-301 | |
| Razpotnik A, Krizaj I, Sribar J, et al. A new phospholipase A2 isolated from the sea anemone Urticina crassicornis - its primary structure and phylogenetic classification. FEBS J , 2010, 277 (12) : 2641-2653 DOI:10.1111/j.1742-4658.2010.07674.x | |
| Seilhamer JJ, Pruzanski W, Vadas P, et al. Cloning and recombinant expression of phospholipase A2 present in rheumatoid arthritic synovial fluid. J Biol Chem , 1989, 264 (10) : 5335-5338 | |
| Sher D, Knebel A, Bsor T, et al. Toxic polypeptides of the hydra—a bioinformatic approach to cnidarian allomones. Toxicon , 2005, 45 (7) : 865-879 DOI:10.1016/j.toxicon.2005.02.004 | |
| Six DA, Dennis EA. The expanding superfamily of phospholipase A2 enzymes: Classification and characterization. Biochim Biophys Acta , 2000, 1488 (1-2) : 1-19 DOI:10.1016/S1388-1981(00)00105-0 | |
| Sugiyama M, Ohtani K, Izuhara M, et al. A novel prokaryotic phospholipase A2: Characterization, gene cloning, and solution structure. J Biol Chem , 2002, 277 (22) : 20051-20058 DOI:10.1074/jbc.M200264200 | |
| Tamura K, Dudley J, Nei M, et al. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4. 0. Mol Biol Evol , 2007, 24 (8) : 1596-1599 DOI:10.1093/molbev/msm092 | |
| Valdez-Cruz NA, Segovia L, Corona M, et al. Sequence analysis and phylogenetic relationship of genes encoding heterodimeric phospholipases A2 from the venom of the scorpion Anuroctonus phaiodactylus. Gene , 2007, 396 (1) : 149-158 DOI:10.1016/j.gene.2007.03.007 | |
| Vandesompele J, Preter KD, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol , 2002, 3 (7) : 1-11 | |
| Xin Y, Choo YM, Hu ZG, et al. Molecular cloning and characterization of a venom phospholipase A2 from the bumblebee Bombus ignitus. Comp Biochem Phys B: Biochem Mol Biol , 2009, 154 (2) : 195-202 DOI:10.1016/j.cbpb.2009.06.003 |



