2. 上海海洋大学水产与生命学院 上海 201306
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
肠道作为微生物大量存在的栖息地,不仅形成了复杂的肠道微生态系统(Björkstén, 2006),而且作为宿主生命活动的有机组成部分,对宿主的生长发育具有重要的影响(Ley et al, 2008; Suez et al, 2014)。肠道菌群对鱼类的健康发挥重要作用(Austin, 2006; 孙云章等, 2008; Round et al, 2009; Wei et al, 2010),例如,肠道微生物可以调节斑马鱼(Danio rerio)消化道基因的表达,促进营养代谢和免疫反应(Rawls et al, 2004)。不同的鱼类具有各自独特的肠道菌群结构,而且与外部环境条件密切相关,生长发育阶段、营养状况、性别和消化系统的复杂性等都会对肠道菌群的形成及多样性产生影响(Kim et al, 2007; Nayak, 2010; Stephens et al, 2016)。鱼类的肠道微生态系统是后天形成的(Yan et al, 2016),因此,了解鱼类肠道菌群的演替和定植过程,对鱼类肠道微生态系统平衡调控具有重要意义。以往对鱼类肠道微生物群落形成及多样性的研究,大多采用传统的梯度稀释平板法,分离培养肠道微生物来研究其结构组成(Griffiths et al, 2001; Huber et al, 2004; Jensen et al, 2002),其缺点是肠道微生物种群中有诸多菌群目前尚无法分离培养。近年来,变性梯度凝胶电泳法(DGGE)和高通量测序技术被应用于研究鱼类肠道微生物群落结构(Dhanasiri et al, 2011; 李金金等, 2013; Giatsis et al, 2015),但DGGE方法在实验结果中只能反应出样品中的优势菌种,无法得到样品中细菌种类的绝对数量,而且难以进行大量样本的测序分析、深入了解肠道菌群的复杂组成,高通量测序技术弥补了这一缺陷(Suenaga, 2012; 夏围围等, 2014)。
牙鲆(Paralichthys olivaceus)作为我国重要的海水养殖经济鱼类,了解其肠道菌群结构变化和功能特性,对牙鲆的健康养殖至关重要。李存玉等(2015)研究了池塘和工厂化2种不同养殖条件下的牙鲆成鱼肠道菌群多样性;Kim等(2013)对养殖条件下的牙鲆与野生牙鲆的肠道微生物进行了比较分析。对于牙鲆仔稚幼鱼肠道微生物的研究仅见Eddy等(2002)和Verner-Jeffreys等(2003),他们采用传统的细菌培养方法,报道了仔稚幼鱼肠道菌群的影响因素。本研究通过MiSeq 16S rRNA高通量测序的方法,分析牙鲆仔稚幼鱼发育阶段肠道菌群多样性及其形成过程,旨在揭示工厂化人工育苗模式下牙鲆仔稚幼鱼肠道菌群结构,为深入研究牙鲆养殖过程中肠道菌群形成机制及调控技术提供理论基础。
1 材料与方法 1.1 苗种来源野生亲鱼经过驯化后,待性腺发育成熟,通过人工授精的方式获取健康的受精卵。在青岛贝宝海洋科技有限公司进行苗种培育,采用牙鲆工厂化人工育苗方法,在5 m×5 m×1 m的方形抹角水泥池中进行。苗种培育的水环境条件:水温为18-20℃,溶氧量为5 mg/L以上,盐度为30,NH4+-N≤0.1 mg/L,换水率为50%-300%。苗种培育的饵料系列为轮虫-卤虫-配合饲料,4-19日龄投喂轮虫(5-8 ind/ml培育水体);17-39日龄投喂卤虫(0.5-1.0 ind/ml培育水体);25日龄时开始投喂配合饲料,随着苗种的生长,逐步增加饲料的投喂量和饲料颗粒粒径(图 1)。

|
图 1 牙鲆仔稚幼鱼饵料系列示意 Figure 1 Feeding pattern during P. olivaceus early life stages, 1 to 120 day-post-hatching s表示取样点 s denoted sampling point |
2015年5-9月进行样品采集,所有样品来自同一批受精卵、同一个育苗池的牙鲆苗种。根据牙鲆摄食饵料的种类和生长发育阶段,分别于1日龄、9日龄、21日龄、45日龄、80日龄和115日龄采取仔稚幼鱼肠道样品(图 1中的黑色菱形标注了取样时间),分别编号为G1、G2、G3、G4、G5、G6。因为G1的样品为初孵仔鱼,无法区分肠道,所以将初孵仔鱼整体保存;G2-G6时期的样品均只取肠道。取样时,提前进行停食处理12 h以上,排空消化道内残留食物。在无菌环境下取样,首先用无菌水将样品表面冲洗干净;然后于无菌培养皿中将肠道迅速取出后,使用无菌水冲洗3次以上,立刻置于液氮中保存;同一阶段样品设置3个生物重复样,每组样品根据鱼的大小分别取50尾×3 (G1)、50尾×3 (G2)、30尾×3 (G3)、5尾×3 (G4)、3尾×3 (G5)和3尾×3 (G6);每次取样的同时测量鱼的全长(表 1)。
|
|
表 1 实验取样信息 Table 1 Information of collected samples of the experiment |
使用QIAamp DNA mini kit (QIAGEN, 德国)提取1.2中肠道样品的DNA,并进行琼脂糖凝胶电泳和DNA浓度检测,以确保所提取DNA质量能够满足后续扩增要求。根据16S rRNA基因序列特点和Miseq平台测序要求,针对V3-V4区域设计特异引物338F:5'-ACTCCTACGGGAGGCAGCA-3',806R:5'-GGAC TACHVGGGTWTCTAAT-3'进行PCR扩增。20 μl PCR反应体系:5×FastPfu Buffer 4 μl,2.5 mmol/L dNTPs 2 μl,Forward Primer (5 μmol/L) 0.8 μl,Reverse Primer (5 μmol/L) 0.8 μl,FastPfu Polymerase 0.4 μl,BSA 0.2 μl,Template DNA 10 ng,补ddH2O至20 μl。反应条件:95℃预变性3 min,95℃变性30 s,55℃退火30 s,72℃延伸45 s,共27个循环,72℃延伸10 min。PCR产物定量和均一化处理后使用MiSeq平台测序分析(上海欧易生物医药科技有限公司)。
1.4 数据处理与分析生物信息分析流程:由Illumina MiSeq测序所得原始数据进行去杂、拼接,对所有的优质序列使用UPARSE clustering方法以相似度97%进行OTU分类。随后使用PyNAST以OTU分类中丰度最大的序列为代表序列与GreenGenes数据库进行比对,以置信度≥0.8进行物种分类注释,进一步进行α多样性和β多样性的分析。
肠道菌群α多样性分析主要包括OTU丰度分析、物种多样性指数分析和物种系统发育分析,用于说明整个发育阶段内肠道菌群的组成和结构变化;肠道菌群β多样性分析主要基于Unweight Unifrac距离和OTU丰度,采取单因素方差分析(One-way ANOVA)和LEfSe分析方法进行分析。所有的统计分析均使用QIIME、R语言软件完成。统计数据均以平均值±标准差(Mean±SD)的形式表示。
2 结果与分析 2.1 基于16S rRNA测序的肠道菌群多样性对牙鲆仔稚幼鱼肠道样品16S rRNA数据分析,18个测序样品共获得489922条有效序列,根据97%的相似度进行OTU分类,获得7462个OTU。将每组的3个重复样品平均或合计后获得6组样品的OTU数据见表 2。其中,G1、G2、G3、G4、G5和G6的OTU的平均值分别为(1298±99)、(768±72)、(400±61)、(754±176)、(1337±527)和(886±107)。通过与数据库16S序列比对,以置信度≥0.8进行物种分类注释,在门(Phylum)分类水平上共有42个门的菌群种类,在属(Genus)分类水平上有972个属的细菌种类(表 2)。其中,G1时期共有38个门490个属,G2时期共有38个门588个属,G3时期的微生物种类最少,只有25个门对应254个属;从G4时期开始微生物种类显著增加,G4中共有32个门378个属,而G5时期增加到36个门498个属,G6时期共有39个门336个属。
|
|
表 2 牙鲆仔稚幼鱼样品16S rRNA测序数据 Table 2 16S rRNA sequencing data summary for samples of P. olivaceus during early life stages |
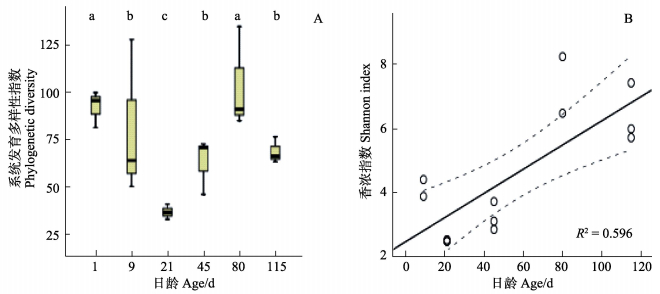
对牙鲆仔稚幼鱼的6组样品的系统发育多样性指数单因素方差分析发现,不同日龄牙鲆肠道的菌群进化多样性指数具有显著差异,这与每组样品对应的菌群种类相一致。初孵仔鱼G1的进化多样性指数与G2、G3样品有明显差异;G1和G5的进化多样性指数最高,G3的最低(P < 0.05)(图 2-A)。另外,对5组摄食后的仔稚幼鱼肠道样品(G2、G3、G4、G5、G6)的香农指数拟合线性回归分析显示,随着日龄的增加,仔稚幼鱼的肠道菌群物种多样性逐渐增加(R2=0.596,P < 0.05)(图 2-B)。
|
图 2 α多样性指数分析 Figure 2 Analysis of bacteria alpha diversity A:系统发育多样性指数(P < 0.05);B:9日龄后样品香农指数(P < 0.05) A: Comparison of phylogenetic diversity (P < 0.05); B: Regression model of Shannon index after 9 d (P < 0.05) |
在门的分类水平上,将每组样品中OTU对应菌种的相对丰度占该样品总OTU≥0.10%的前22个菌门的相对丰度进行比较(图 3),其中的7个优势细菌门类的相对丰度见表 3。由图 3和表 3可知,变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)四者对应OTU的相对丰度在各时期(除G5时期的85%外)均占该组样品总OTU的90%以上。G1期样品为初孵仔鱼,其主要优势菌群种类在门水平上与摄食后的仔稚幼鱼肠道样品基本相同,但在丰度上有较大差别。摄食后,随着仔稚幼鱼生长发育其肠道菌群丰度从门水平上看,变形菌门的相对丰度逐渐降低;厚壁菌门和拟杆菌门的相对丰度逐渐增加;放线菌门的相对丰度基本稳定,只在G3时期有所下降;蓝藻菌门(Cyanobacteria)在各时期的相对丰度均很低,仅在G5时期出现短暂增加。相对丰度前4位的变形菌门、厚壁菌门、拟杆菌门、放线菌门在牙鲆仔稚幼鱼肠道菌群的形成和演替过程中一直处于优势菌群地位。

|
图 3 牙鲆仔稚幼鱼肠道菌群组成(门水平分类) Figure 3 Composition of intestinal bacterial community of P. olivaceus during early life stages at phylum taxonomic level G1:1日龄;G2:9日龄;G3:21日龄;G4:45日龄;G5:80日龄;G6:115日龄 G1: 1 d; G2: 9 d; G3: 21 d; G4: 45 d; G5: 80 d; G6: 115 d |
|
|
表 3 牙鲆仔稚幼鱼肠道优势细菌门类及相对丰度(%) Table 3 Dominant bacterial phyla and the relative abundance for samples of P. olivaceus during early life stages (%) |
在相对丰度最高的前4个菌门中,筛选各时期样品中曾经出现过OTU相对丰度 > 1%并且明确鉴定到属的菌(相似度≥97%)作为优势菌属,进行LEfSe (LDA effect size)分析(Kruskal-Wallis,P < 0.05;Wilcoxon test,P < 0.05;LDA score > 4.0),从而确定组与组之间具有统计学差异的生物标识,即组间差异显著的物种(表 4)。在G1-G6的6组样品中共筛选出29个菌属,其中,15个属于变形菌门,8个属于厚壁菌门中,5个属于拟杆菌门,1个属于放线菌门。
|
|
表 4 相对丰度 > 1%菌属LEfSe分析 Table 4 LEfSe analysis on genus level (the relative abundance > 1%) |
变形菌门各菌属的组成变化:G1时期OTU相对丰度 > 1%的共有11个菌属,其相对丰度水平较低,G2-G6时期OTU相对丰度 > 1%的菌属数量分别为6、3、5、6、2。其中,G1期具有重要贡献值(LDA值 > 4)(P < 0.05)的交替单细胞菌属(Alteromonas)、海单细胞菌属(Marinomonas)和假交替单细胞菌属(Pseudoalteromonas),在G2-G5时期逐渐减少,到G6时期几乎消失,未能作为肠道主要菌群而定植。弧菌属(Vibrio)在G2-G4时期相对丰度显著增高,在G4时期达到60.56%,而到G5、G6时期相对丰度锐减至低水平(0.28%);伯克氏菌属(Burkholderia)、Nautella、弓形杆菌属(Arcobacter)和Aliivibrio在G2-G5时期虽然相对丰度一度较高,但到G6时期其相对丰度很低(几乎检测不到),未在幼鱼期定植为优势菌属;假单胞菌属(Pseudomonas)、萨特氏菌属(Sutterella)、埃希氏杆菌属(Escherichia)、不动杆菌属(Acinetobacter)的相对丰度在各时期均处于较低水平,但一直存在于肠道内;萨特氏菌属在G2-G5阶段相对丰度 < 0.1%,在G6时期增加到1.25%,此时,该菌在牙鲆幼鱼的肠道中可能已成为优势菌属。
拟杆菌门各菌属的组成变化:G1时期OTU相对丰度 > 1%的共有3个菌属,G2-G6时期相对丰度 > 1%的菌属数量分别为0、0、2、2、2。拟杆菌属(Bacteroides)和普氏菌属(Prevotella)从G4时期开始增加,到G5和G6时期到达较高水平,可能已定植成为肠道优势菌群,其贡献率显著(LDA值 > 4)(P < 0.05);Cryomorphaceae某属在G1时期的丰度最高,但在G2-G6时期持续降低直至未检测出;极地杆菌属(Polaribacter)的丰度逐渐增加,在G5时期达到较高丰度,但在G6时期表达极少,尚未固定成为优势菌群。
厚壁菌门各菌属的组成变化:G1时期OTU相对丰度 > 1%的共有2个菌属,且相对丰度均在1%左右,G2-G6时期OTU相对丰度 > 1%的菌属数量分别为0、0、0、2、8。厚壁菌门各菌属在牙鲆摄食活饵料时的G2-G3时期丰度非常低,尤其是G3时期各菌属的相对丰度 < 0.05%。其中,布劳特氏菌属(Blautia)、颤螺菌属(Oscillospira)、毛螺科菌某属和瘤胃球菌属(Ruminococcus)在转换饲料后的G4时期相对丰度增加,到G6时期成为相对丰度最高的前4位优势菌属(相对丰度 > 5%);考拉杆菌属(Phascolarctobacterium)、乳杆菌属(Lactobacillus)在G5时期才开始增加,G6时期相对丰度 > 2%,成为优势菌属。厚壁菌门的8个菌属在G6时期均发展成为优势菌属,其中,布劳特氏菌等6个菌属在G6时期的贡献率显著(LDA值 > 4;P < 0.03)。
3 讨论鱼类的肠道菌群在其生长发育过程中参与重要的生理活动,与鱼类的健康生长密切相关。鱼类仔稚幼鱼阶段是肠道菌群群落形成的关键时期,在该阶段水环境和食物中的微生物开始进入肠道,随着鱼类的生长不断演替最后完成定植(Egert et al, 2005; Austin, 2006; Knapp et al, 2010; Ley et al, 2008)。鲆鲽类作为重要的养殖经济鱼类,其肠道微生物的研究一直备受关注(Thomson et al, 2005; Sugita et al, 2006; Kim et al, 2013)。本研究利用MiSeq 16S rRNA高通量测序技术和生物信息学分析方法,深入认识了工厂化人工育苗条件下牙鲆仔稚幼鱼肠道菌群结构及变化,在门、属水平分类基础上,详细阐述了肠道菌群的结构组成特点以及随生长发育过程的演替变化规律。传统方法分离培养发现,大西洋庸鲽(Hippoglossus hippoglossus)和大菱鲆(Scophthalmus maximus)肠道微生物群落中的弧菌属在仔稚幼鱼摄食后丰度明显增加并成为优势菌属,但随着饵料的改变,其数量迅速减少(Verner-Jeffreys et al, 2003; Jensen et al, 2002; 史秀清等, 2015)。此结果与本研究中弧菌属在仔稚幼鱼肠道中的变化规律相似。但肠道微生物种群中有诸多菌群目前尚无法分离培养,限制了整体菌群结构的分析。本研究在牙鲆仔稚幼鱼肠道菌群中发现了42个菌门下的972个菌属,并分析其优势菌属的变化规律,较全面地反映了肠道菌群结构的多样性变化。
以往研究中发现轮虫和卤虫中含有大量的弧菌(Rawls et al, 2006; Bjornsdottir et al, 2009),在仔鱼开口后,肠道菌群内弧菌属细菌数量迅速增加,取代假交替细胞菌属成为优势菌(Bergh et al, 1994; Eddy et al, 2002; Verner-Jeffreys et al, 2003; 史秀清等, 2015)。Picchietti等(2007)在研究中指出,以卤虫和轮虫为载体,能够有效地帮助益生菌进入仔稚幼鱼肠道内定植。肠道菌落的形成与摄食饲料关系密切,草鱼(Carassius auratus gibelio)的肠道菌群会随着食物的改变而变化(Wu et al, 2012);配合饲料中植物性蛋白会影响金头鲷(Sparus aurata)的肠道菌群结构(Estruch et al, 2015);另外,消化道微生物种间也存在生存竞争关系,有限的肠道环境内微生物种间竞争强度会影响肠道微生物的种类数量(Schryver et al, 2014)。本研究发现在仔鱼期的牙鲆开始摄食轮虫后,肠道的优势菌群结构较单一,变形菌门中的弧菌属最先成为仔鱼期肠道菌群的绝对优势菌;在摄食配合饲料后,变形菌门的相对丰度显著降低,厚壁菌门和拟杆菌门的相对丰度明显增大,在幼鱼阶段替代变形菌门成为肠道菌群的绝对优势菌群,其中,瘤胃球菌属、布劳特氏菌丰度最高。作者认为,在牙鲆仔鱼开始摄食后,作为饵料的轮虫和卤虫所携带的菌群对仔稚幼鱼的肠道菌群结构具有较大影响,从食物转为配合饲料后,肠道菌群中由食物带来的菌群的变化逐渐稳定,同时,牙鲆自身也会有目的的筛选有益菌株,从而使肠道微生态系统达到相对稳定状态。说明摄食饵料的类型与牙鲆仔稚幼鱼肠道菌群结构的变化关系密切。关于饵料对牙鲆肠道菌群结构及变化规律的影响有待深入研究。
本研究中,80日龄后的牙鲆幼鱼肠道菌群趋向稳定,形成了以厚壁菌门、拟杆菌门、变形菌门和放线菌门为主的菌群组成,这与养殖牙鲆成鱼在门水平上的肠道优势菌群结构相似,但是在属水平上其结构却有较大差异(李存玉等, 2015)。牙鲆成鱼肠道中作为优势菌存在的芽孢杆菌属和不动杆菌属,在幼鱼时期肠道中的丰度却较低;而幼鱼肠道的绝对优势菌属在成鱼肠道中不占主要地位。因此,虽然在仔稚幼鱼阶段牙鲆肠道菌群形成了以瘤胃球菌属、布劳特氏菌属和拟杆菌属等为优势菌属的肠道菌群组成,但是随牙鲆幼鱼的生长发育其肠道菌群的结构仍会发生变化。此外,牙鲆的肠道优势菌与其生活环境有关,Kim等(2013)研究发现,野生牙鲆的肠道微生物种类丰富度显著大于养殖牙鲆,且当牙鲆处于较为复杂的环境中,其肠道内的有益菌数量和种类会增加。在池塘养殖条件下,牙鲆肠道的芽孢杆菌属和野生牙鲆肠道的乳酸杆菌属的种类和数量远高于工厂化养殖条件下的牙鲆肠道。
本研究测定的初孵仔鱼的菌群结构多样性丰富,但其肠道菌群与摄食后的仔稚幼鱼肠道菌群结构明显不同。因为该样品是整条初孵仔鱼,此时肠道尚未形成,无法取肠道与后续其他各期的肠道样品进行同步比较。实验结果显示,初孵仔鱼的体内部分微生物是在摄食后的仔稚幼鱼肠道中不存在或逐渐消失的菌群,这些菌群可能来源于母源卵子或受精卵,也可能在受精卵孵化时由水环境中介入,然而这些菌群均无法在仔稚幼鱼肠道内定植。另外一部分菌群在后期仔稚幼鱼发育过程的肠道中一直延续存留下来,成为肠道菌群的组成部分,他们可能具有母源遗传特征。因此,今后应进一步深入开展牙鲆肠道菌群组成与亲本母源的关系研究。
| Austin B. The bacterial microflora of fish, revised. Scientific World Journal , 2006 (6) : 931-945 | |
| Bergh Ø, Naas KE, Harboe T. Shift in the intestinal microflora of atlantic halibut (Hippoglossus hippoglossus) larvae during first feeding. Canadian Journal of Fisheries and Aquatic Sciences , 1994, 51 (8) : 1899-1903 DOI:10.1139/f94-190 | |
| Björkstén B. The gut microbiota: A complex ecosystem. Clinical and Experimental Allergy , 2006, 36 (10) : 1215-1217 DOI:10.1111/cea.2006.36.issue-10 | |
| Bjornsdottir R, Johannsdottir J, Coe J, et al. Survival and quality of halibut larvae (Hippoglossus hippoglossus L.) in intensive farming: Possible impact of the intestinal bacterial community. Aquaculture , 2009, 286 (1-2) : 53-63 DOI:10.1016/j.aquaculture.2008.09.003 | |
| Dhanasiri A, Brunvold L, Brinchmann M, et al. Changes in the intestinal microbiota of wild atlantic cod Gadus morhua L. upon captive rearing. Microbial Ecology , 2011, 61 (1) : 20-30 DOI:10.1007/s00248-010-9673-y | |
| Eddy SD, Jones SH. Microbiology of summer flounder Paralichthys dentatus fingerling production at a marine fish hatchery. Aquaculture , 2002, 211 (1-4) : 9-28 DOI:10.1016/S0044-8486(01)00882-1 | |
| Egert M, Stingl U, Bruun LD. Structure and topology of microbial communities in the major gut compartments of Melolontha melolontha larvae (Coleoptera: Scarabaeidae). Applied and Environmental Microbiology , 2005, 71 (8) : 4556-4566 DOI:10.1128/AEM.71.8.4556-4566.2005 | |
| Estruch G, Collado MC, Peñaranda DS, et al. Impact of fishmeal replacement in diets for gilthead sea bream (Sparus aurata) on the gastrointestinal microbiota determined by pyrosequencing the 16S rRNA gene. PLoS One , 2015, 10 (8) : e0136389 DOI:10.1371/journal.pone.0136389 | |
| Giatsis C, Sipkema D, Smidt H, et al. The impact of rearing environment on the development of gut microbiota in tilapia larvae. Scientific Reports , 2015 (5) : 18206 | |
| Griffiths S, Melville K, Cook M, et al. Profiling of bacterial species associated with haddock larviculture by PCR amplification of 16S rDNA and denaturing gradient gel electrophoresis. Journal of Aquatic Animal Health , 2001, 13 (4) : 355-363 DOI:10.1577/1548-8667(2001)013<0355:POBSAW>2.0.CO;2 | |
| Huber I, Spanggaard B, Appel KF, et al. Phylogenetic analysis and in situ identification of the intestinal microbial community of rainbow trout (Oncorhynchus mykiss, Walbaum). Journal of Applied Microbiology , 2004, 96 : 117-132 DOI:10.1046/j.1365-2672.2003.02109.x | |
| Jensen S, Bergh O, Enger O, et al. Use of PCR-RFLP for genotyping 16S rRNA and characterizing bacteria cultured from halibut fry. Canadian Journal of Microbiology , 2002, 48 (5) : 379-386 DOI:10.1139/w02-033 | |
| Kim DH, Brunt J, Austin B. Microbial diversity of intestinal contents and mucus in rainbow trout (Oncorhynchus mykiss). Journal of Applied Microbiology , 2007, 102 : 1654-1664 DOI:10.1111/jam.2007.102.issue-6 | |
| Kim DH, Kim DY. Microbial diversity in the intestine of olive flounder (Paralichthys olivaceus). Aquaculture , 2013, 414-415 : 103-108 DOI:10.1016/j.aquaculture.2013.06.008 | |
| Knapp BA, Seeber J, Rief A. Bacterial community composition of the gut microbiota of Cylindroiulus fulviceps (Diplopoda) as revealed by molecular fingerprinting and cloning. Folia Microbiologica , 2010, 55 (5) : 489-496 DOI:10.1007/s12223-010-0081-y | |
| Ley RE, Lozupone CA, Hamady M, et al. Worlds within worlds: Evolution of the vertebrate gut microbiota. Nature Reviews Microbiology , 2008, 6 (10) : 776-788 DOI:10.1038/nrmicro1978 | |
| Li CY, Liu XZ, Xu YJ, et al. Comparative analysis of composition, diversity and origin of intestinal bacterial community in pond-and indoor tank-culture Japanese flounder (Paralichthys olivaceus). Journal of Fisheries of China , 2015, 39 (2) : 245-255 [李存玉, 徐永江, 柳学周, 等. 池塘和工厂化养殖牙鲆肠道菌群结构的比较分析. 水产学报 , 2015, 39 (2) : 245-255] | |
| Li JJ, Ni JJ, Li XM, et al. Rrelationship between gastrointestinal bacterial structure and development of silurus soldatovi meridonalls chen. Acta Hydrobiologica Sinica , 2013, 37 (4) : 613-619 [李金金, 倪加加, 李学梅, 等. 南方大口鲇消化道细菌群落结构与其胃肠分化的关系. 水生生物学报 , 2013, 37 (4) : 613-619] | |
| Nayak SK. Role of gastrointestinal microbiota in fish. Aquaculture Research , 2010, 41 (11) : 1553-1573 DOI:10.1111/are.2010.41.issue-11 | |
| Picchietti S, Mazzini M, Taddei AR, et al. Effects of administration of probiotic strains on GALT of larval gilthead seabream: Immunohistochemical and ultrastructural studies. Fish and Shellfish Immunology , 2007, 22 (1-2) : 57-67 DOI:10.1016/j.fsi.2006.03.009 | |
| Rawls J, Samuel B, Gordon J. Gnotobiotic zebrafish reveal evolutionarily conserved responses to the gut microbiota. Proceedings of the National Academy of Sciences , 2004, 101 (13) : 4596-4601 DOI:10.1073/pnas.0400706101 | |
| Rawls JF, Mahowald MA, Ley RE, et al. Reciprocal gut microbiota transplants from zebrafish and mice to germ-free recipients reveal host habitat selection. Cell , 2006, 127 (2) : 423-433 DOI:10.1016/j.cell.2006.08.043 | |
| Round JL, Mazmanian SK. The gut microbiota shapes intestinal immune responses during health and disease. Nature Reviews Immunology , 2009, 9 (5) : 313-323 DOI:10.1038/nri2515 | |
| Schryver PD, Vadstein O. Ecological theory as a foundation to control pathogenic invasion in aquaculture. The ISME Journal , 2014, 8 (12) : 2360-2368 DOI:10.1038/ismej.2014.84 | |
| Shi XQ, Zhang Z, Wang YG, et al. The characteristics of culturable bacterial microflora in the gastrointestinal tract of turbot (Scophthatmus maximus) larvae. Progress in Fishery Sciences , 2015, 36 (4) : 73-82 [史秀清, 张正, 王印庚, 等. 大菱鲆(Scophthalmus maximus)仔稚鱼发育期消化道可培养细菌的菌群特征分析. 渔业科学进展 , 2015, 36 (4) : 73-82] | |
| Stephens WZ, Burns AR, Stagaman K, et al. The composition of the zebrafish intestinal microbial community varies across development. The ISME Journal , 2016, 10 (3) : 644-654 DOI:10.1038/ismej.2015.140 | |
| Suenaga K. Targeted metagenomics: A high-resolution metagenomics approach for specific gene clusters in complex microbial communities. Environmental Microbiology , 2012, 14 (1) : 13-22 DOI:10.1111/j.1462-2920.2011.02438.x | |
| Suez J, Korem T, Zeevi D, et al. Artificial sweeteners induce glucose intolerance by altering the gut microbiota. Nature , 2014, 514 (7521) : 181-186 | |
| Sugita H, Ito Y. Identification of intestinal bacteria from Japanese flounder (Paralichthys olivaceus) and their ability to digest chitin. Letters in Applied Microbiology , 2006, 43 (3) : 336-342 DOI:10.1111/lam.2006.43.issue-3 | |
| Sun YZ, Yang HL. A review: Distribution and manipulation of fish gut microflora. Fisheries Science , 2008, 27 (5) : 257-261 [孙云章, 杨红玲. 浅谈鱼类消化道微生物的分布及调控. 水产科学 , 2008, 27 (5) : 257-261] | |
| Thomson R, Macpherson H, Riaza A, et al. Vibrio splendidus biotype 1 as a cause of mortalities in hatchery-reared larval turbot, Scophthalmus maximus (L.). Journal of Applied Microbiology , 2005, 99 (2) : 243-250 DOI:10.1111/jam.2005.99.issue-2 | |
| Verner-Jeffreys DW, Shields RJ, Bricknell IR, et al. Changes in the gut-associated microflora during the development of Atlantic halibut (Hippoglossus hippoglossus L.) larvae in three British hatcheries. Aquaculture , 2003, 219 : 21-42 DOI:10.1016/S0044-8486(02)00348-4 | |
| Wei H, Dong L, Wang T, et al. Structural shifts of gut microbiota as surrogate endpoints for monitoring host health changes induced by carcinogen exposure. FEMS Microbiology Ecology , 2010, 73 (3) : 577-586 | |
| Wu S, Wang G, Angert ER, et al. Composition, diversity, and origin of the bacterial community in grass carp intestine. PLoS One , 2012, 7 (2) : e30440 DOI:10.1371/journal.pone.0030440 | |
| Xia WW, Jia ZJ. Comparative analysis of soil microbial communities by pyrosequencing and DGGE. Acta Microbiologica Sinica , 2014, 54 (12) : 1489-1499 [夏围围, 贾仲君. 高通量测序和DGGE分析土壤微生物群落的技术评价. 微生物学报 , 2014, 54 (12) : 1489-1499] | |
| Yan QY, Li JJ, Yu YH, et al. Environmental filtering decreases with fish development for the assembly of gut microbiota. Environmental Microbiology , 2016, 18 (12) : 4739-4754 DOI:10.1111/emi.2016.18.issue-12 |



