2. 山东大学海洋学院 威海 264209;
3. 乳山渔政监督管理站 乳山 264500
2. Marine College of Shandong University, Weihai 264209;
3. Fishery Supervision Station, Rushan 264500
星斑川鲽 (Platichthys stellatus) 隶属鲽形目、鲽科、川鲽属 (王波等, 2010),主要分布在33°–73°N和105°W–127°E的太平洋和北冰洋海域以及附近的河流 (Miao et al, 2009)。星斑川鲽鱼体扁平形,体较高,两眼突出,养殖群体中大约有95%–97%的个体两眼位于体左侧,右侧仅占3%–5%(王波, 2010)1)。石鲽 (Kareius bicoloratus) 隶属鲽形目、鲽科、石鲽属,主要分布于我国东海、黄海、渤海以及日本和朝鲜近海 (张少华等, 2004)。石鲽体呈椭圆形,侧扁,两眼位于头的右侧 (刘曼红等, 2009)。星斑川鲽和石鲽营养成分高、肉质鲜美,是我国重要的渔业资源和养殖对象。杂交育种是一种常用的育种方式 (Hashimoto et al, 2012),杂交后代具有亲鱼双方的遗传物质 (Purdom et al, 1974; Šimková et al, 2015),甚至可以表现出比亲鱼还优良的性状 (Garrett, 2005),比如生活能力强、适应范围广、抗逆性强和竞争能力强等。在海洋鱼类杂交实验中,经常研究的是鲆鲽鱼类 (Garrett et al, 2009)。
1) Wang B. Study on the biological characteristics and healthy breeding technology of Platichthys stellatus. Master's Thesis of Chinese Academy of Agricultural Sciences, 2010 [王波.星斑川鲽的生物学及健康养殖技术研究.中国农业科学院硕士研究生学位论文, 2010]
细胞色素b基因 (Cytb) 和细胞色素C氧化酶I (COI) 具有比线粒体rDNA和非编码区更容易排序、进化速率适中、易扩增和测序及富含系统发育信息等优点 (俞建中, 20041); 张迪等, 2012)。基于这些特点,线粒体基因Cytb和COI常常被作为分子标记应用于种群遗传结构和遗传多样性的研究 (Harrison, 1989; Kartavtsev et al, 2008; 彭博等, 2010)。而且同一线粒体引物在邻近种间也可以很好地扩增,因此,在杂交实验中也常常被用来评估杂交子代间的遗传分化程度,以验证杂交子代偏母遗传特性 (肖永双等, 2014b)。鲆鲽鱼类由于形态相似,很难根据一般的形态分类法对其分类,这就给渔业资源保护和管理带来一定困难。由于线粒体基因自然状态下单倍体和母系遗传的特性,相比核基因标记,线粒体基因是一种更好的分子标记。国外学者早已将线粒体基因Cytb用于鲆鲽类的鉴定 (Céspedes et al, 1999; Sanjuan et al, 2002),Cytb基因片段和D-loop区基因片段也曾用于鉴别鲆鲽的杂交亲子代[欧洲川鲽 (Platichthys flesus)×欧鲽 (Pleuronectes platessa)],Cytb鉴定结果与形态鉴定结果一致,D-loop区显示出不同长度的多态性 (Kijewska et al, 2009)。本研究针对星斑川鲽、石鲽以及杂交一代 (星斑川鲽♀×石鲽♂) 线粒体DNA的Cytb和COI基因片段的遗传特性展开研究,可为星斑川鲽和石鲽的线粒体遗传效应的研究提供理论依据,同时为星斑川鲽和石鲽运用杂交育苗手段选育优良品种提供理论支持。
1) Yu JZ. Pilot study of phylogeny and molecular evolution, in Pleuronectiformes of Yellow Sea. Master's Thesis of the Ocean University of China, 2004 [俞建中.黄海鲽形目鱼类系统发生与分子进化的初步研究.中国海洋大学硕士研究生学位论文, 2004]
1 材料与方法 1.1 实验材料将取自山东日照海洋水产资源增殖站的星斑川鲽雌性亲鱼和取自日照近海的野生石鲽雄性亲鱼于2015年4月在烟台蓬莱宗哲养殖公司进行同步促熟培养,人工授精杂交,获得受精卵并进行孵化培养。2015年8月于日照海洋水产资源增殖站采集星斑川鲽24尾 (包含亲本)、石鲽24尾 (包含亲本);于烟台蓬莱宗哲养殖有限公司取30尾平均体长为24 mm的杂交幼苗,包括左眼杂交幼苗15尾和右眼杂交幼苗15尾。所有成鱼于活体状态下取小部分尾鳍后放养。杂交幼苗取其肌肉,在95%的酒精中4℃保存,回实验室后,无菌条件下洗去酒精,并存放于–80℃以备基因组提取。
1.2 基因组DNA提取和PCR扩增取每个样本的尾鳍约30 mg,采用海洋动物组织基因组DNA提取试剂盒 (TIANGEN) 提取,并溶于130 μl TE中,于–20℃保存。水浴时不断摇晃,使样品细胞充分裂解以释放出DNA;加TE溶解前,充分晾干使酒精挥发完全,以免影响后续实验。星斑川鲽中用于扩增线粒体Cytb基因的引物 (张岩等, 2009):上游引物Cytb F (5'-AACCACCGTTGTTATTCAACT-3');下游引物Cytb R (5'-CTCAGAATGACATTTGTCCTCA-3')。用于扩增线粒体COI基因的引物 (肖永双等, 2014a):上游引物COI F (5'-CACAAAGACATTGGCACCCT-3');下游引物COI R (5'-CCTCCTGCAGGGTCAAAGAA-3')。PCR反应条件:94℃预变性4 min;94℃变性30 s,退火温度退火30 s,72℃延伸1 min,共34个循环;最后72℃总延伸10 min。扩增Cytb基因的引物退火温度为53℃,COI的退火温度为55℃。所有的PCR反应体系均为50 μl:DNA模板1.5 μl (15 ng/μl),上下游引物各2 μl (10 μmol/L),2×Reaction Mix 25 μl,ddH2O 18.9 μl,Taq DNA聚合酶0.6 μl (2.5 U/μl)(东盛生物)。PCR反应在TaKaRa PCR Thermal Cycler Dice上进行。取5 μl PCR扩增产物进行1%琼脂糖凝胶电泳检测 (U=5 V/cm)。将含有目的条带的PCR产物送北京美吉桑格生物医药科技有限公司用ABI3730XL自动测序仪进行单向测序,测序反应采用与之相对应的正向引物。
1.3 序列分析测序结果经Dnastar软件包 (DNASTAR, Inc., 美国) 校对后人工截取有效片段,GenBank登录号:日照星斑川鲽KT920092-KT920114,KT920069-KT920091;日照石鲽KT920136-KT920157,KT920115-KT920135;子一代KU097360-KU097415。软件DNA Sequence Polymorphism 5.0(DnaSP 5)(Librado et al, 2005) 计算各个群体单倍型数 (h)、单倍型多样性 (Hd)(Nei, 1987)、核苷酸多样性 (λ) 以及平均核苷酸差异数 (k)(Tajima, 1983);软件MEGA 6.0(Tamura et al, 2013) 计算碱基组成、碱基替换,基于Kimura 2-parameter公式计算种间种内遗传距离,采用 (Neighbor-joining, NJ) 法 (设Interior-branch test次数是1000) 构建系统发育树,进行系统分化分析。
2 实验结果 2.1 mtDNA Cytb序列分析结果 2.1.1 Cytb部分基因序列碱基组成分析将测序结果与GenBank中注册的星斑川鲽和石鲽的mtDNA Cytb基因序列进行比对,确定所得片段序列为目的片段。经Dnastar比对后,截取得到有效片段为356 bp,应用MEGA 6.0中Sequence Date Explore分别计算星斑川鲽、石鲽以及F1代群体的碱基组成频率,结果见表 1。由表 1可见,3种鲽类中 (A+T) 含量均高于 (C+G) 含量,表现出与其他鱼类相似的碱基组成偏向性 (唐文乔等, 2010)。总体来说,3种鲽类的碱基组成差异不大,星斑川鲽、石鲽、杂交F1代的 (A+T) 分别为53.4%、53.5%、53.4%,(C+G) 含量分别为46.6%、46.5%、46.6%,F1代 (C+G) 含量与母本 (C+G) 含量相同。从3个位点分别比较发现,星斑川鲽和杂交F1代没有差异,但二者与石鲽在第2位点和第3位点有轻微差异,从总体分析3个位点,发现第1位点富含T,第2位点富含C,且严重缺少G,第3位点较均一,这表明密码子的碱基使用频率存在明显的偏性,张艳云等 (2011)在乌龟的研究中也发现了类似情况。
|
|
表 1 Cytb基因片段序列的碱基组成 Table 1 Base composition of Cytb partial sequence |
表 2显示,3个鲽类群体共检测到30个变异位点 (8.42%),其中含有29个简约信息位点;星斑川鲽 (♀) 与杂交F1代之间仅有2个变异位点,全部为简约信息位点;石鲽 (♂) 与杂交F1代之间共30个变异位点,其中29个为简约信息位点。可见杂交F1代与母本之间差异性远远小于杂交F1代与父本之间的差异性,这也印证了线粒体母系遗传这一事实。用软件DnaSP检测到3个群体间含有8个单倍型,单倍型多样性为0.792,平均核苷酸差异数为12.679。如表 3所示,3个群体的356个碱基中共有不变位点341个,转换位点13个,颠换位点2个,转换与颠换位点的比值为6.5,转换明显大于颠换,这与Green等 (2012)得出的脊椎动物的数值是一致的。
|
|
表 2 Cytb基因片段平均位点分析 Table 2 The analysis of average sites of Cytb gene sequence |
|
|
表 3 Cytb基因序列碱基替换统计 Table 3 The base substitution of Cytb gene sequence |
从表 4可以看出,星斑川鲽、石鲽和杂交F1代各个群体个体间的序列差异均很小。父本与母本及父本与子代之间序列差异均较大,远远大于母本与子代之间的差异,遗传距离所反映的遗传变异关系与序列差异所反映的一致,即线粒体基因Cytb严格遵守母系遗传。
|
|
表 4 基于Kimura 2-parameter模型3个群体的Cytb序列的遗传距离 Table 4 Genetic distances of Cytb sequences based on the Kimura 2-parameter model of three groups |
将星斑川鲽 (PSTR1-PSTR3)、石鲽 (KBTS1-KBTS3)、其杂交F1代 (左眼PKZY1-PKZY3,右眼PKZY24-PKZY26) 的Cytb基因核苷酸序列随机各取3条,用MEGA 6.0采用NJ法构建分子系统进化树 (图 1)。星斑川鲽KF445179.1和石鲽AB987795.1分别为在GenBank中与星斑川鲽和石鲽Cytb序列 (登录号分别为PSTR4和KBTS4) 相似度最高的序列。由图 1可看出,NJ树形成明显的两大支,石鲽单独聚为一大支,星斑川鲽和杂交F1代聚为一支,而且杂交F1代左右眼并没有分支,而是聚为一小支后与母本星斑川鲽聚为一大支。
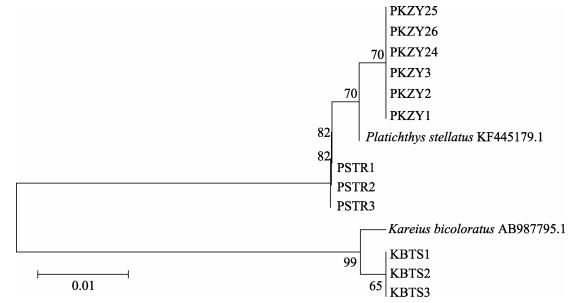
|
图 1 基于Kimura 2-parameter模型建立的邻近关系系统发育树 Figure 1 Neighbor-joining (NJ) phylogeny based on the Kimura 2-parameter model |
对亲子代个体COI基因片段扩增后所获得序列人工拼接后得到557 bp,用软件MEGA 6.0中Sequence Date Explore分别计算星斑川鲽、石鲽以及F1代群体的碱基组成频率,结果见表 5。结果显示,亲子代碱基使用情况差别不大,其中,星斑川鲽和杂交F1代完全一致,每个位点间也一致,再次证明线粒体基因COI具有明显的母系遗传特性。在总碱基频率使用方面,星斑川鲽和杂交F1代 (A+T) 含量为52.4%,(C+G) 含量为47.6%;石鲽 (A+T) 含量为52.1%,(C+G) 含量为48.0%。
|
|
表 5 COI基因片段序列的碱基组成频率 Table 5 Base composition of partial sequence of COI |
在557 bp的COI基因片段中,3个鲽科种群共检测到26个变异位点 (4.8%)(表 6),其中,25个为简约信息位点。母本星斑川鲽和杂交F1代间全部为保守位点,未检测到变异位点,再次有力地说明了线粒体基因母系遗传这一特性;石鲽与杂交F1代间的变异位点和星斑川鲽与石鲽间的变异位点完全一致。如表 7所示,用MEGA 6.0检测,发现亲子代3种鲽科群体间共有12个转换位点,1个颠换位点,转换远远大于颠换;星斑川鲽和杂交F1代间全部为保守位点。
|
|
表 6 COI基因片段平均位点分析 Table 6 The analysis of average sites of COI gene sequence |
|
|
表 7 COI基因序列碱基替换统计 Table 7 The base substitution of COI gene sequence |
基于Kimura 2-parameter模型对3个群体的COI基因序列计算遗传距离,结果如表 8所示。星斑川鲽、石鲽、杂交F1代群体内部的遗传距离,杂交F1代间与母本星斑川鲽的遗传距离,几乎没有差别,杂交F1代与父本石鲽间的遗传距离远远大于子代与母本间的遗传距离。
|
|
表 8 基于Kimura 2-parameter模型3个群体的COI序列的遗传距离 Table 8 Genetic distances of COI sequences based on the Kimura 2-parameter model of three groups |
将星斑川鲽 (PSTRI1-PSTRI3)、石鲽 (KBTSI1-KBTSI3)、杂交F1代 (左眼PKZ1-PKZ3, 右眼PKZ24-PKZ26) 的COI基因核苷酸序列随机各取3条,MEGA6.0采用NJ法构建分子系统进化树 (图 2)。
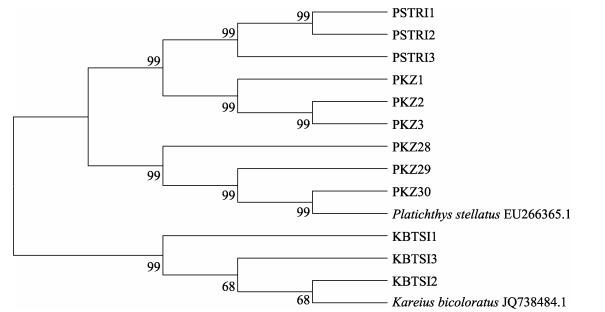
|
图 2 基于Kimura 2-parameter模型建立的邻近关系系统发育树 Figure 2 Neighbor-joining (NJ) phylogeny based on the Kimura 2-parameter model |
星斑川鲽EU266365.1和石鲽JQ738484.1分别为在GenBank中与星斑川鲽和石鲽COI基因序列 (GenBank登录号分别为PSTRI4、KBTSI4) 相似度最高的序列。NJ树显示,3种鲽科鱼分为明显的两支,星斑川鲽和杂交F1代聚为一支,石鲽独自聚为一支,即父本石鲽单独聚成一支,母本与杂交子代聚成一支。杂交F1代与母本星斑川鲽形成的那一大支中,又分为两支:母本星斑川鲽与杂交F1代中外观显示左眼的聚为一支,杂交F1代中外观显示右眼的单独聚为一小支。
3 讨论杂交育苗是鱼类品种改良的重要手段,为了获得具有优良品质的品种,在许多鲆鲽鱼类中进行了杂交实验 (You et al, 2009; 王新成等, 2003; 刘振华等, 2009; 肖永双等, 2014b)。虽然日本学者早已开始研究星斑川鲽和石鲽的杂交育苗,但研究主要集中在星斑川鲽、石鲽和杂交子代的形态学 (Kosaka, 1989)、淡水适应性 (Takeda et al, 2007);遗传方面的研究手段也仅为同工酶 (Park et al, 1991)。运用线粒体基因COI和Cytb探究星斑川鲽、石鲽和杂交子代之间遗传关系的中外报道至今还少有见到。通过线粒体DNA序列变异可以评估物种间的遗传分化程度、遗传距离以及分子系统发育情况 (徐冬冬等, 2010),是一种常用于探究物种间亲缘关系的分子标记手段。本研究通过线粒体基因COI和Cytb在星斑川鲽、石鲽以及杂交F1代间的序列差异、遗传距离、分子系统树等方面,得出杂交F1代线粒体基因具有明显的母系遗传倾向,这也与其他物种的研究结果相一致。基于线粒体基因COI得出的杂交F1代与母本星斑川鲽的遗传距离为0的发现与星斑川鲽和大菱鲆 (星斑川鲽♀×大菱鲆♂) 的杂交实验结果相一致 (肖永双等, 2014b)。周翰林等 (2012)比较了2种杂交石斑鱼子一代与亲本间COI基因序列的差异,表明2种杂交后代在线粒体基因COI序列上都严格遵循母系遗传规律。
NJ树是根据遗传距离建立的分子发育树,其建树思想是先假定一棵无根星状树,再两两比较,将亲缘关系近的聚成聚合分类群,然后再将这聚合分类群看作一个整体与另外的再进行聚合分类,这样重复下去,直至构出完整的NJ树。在本研究中,根据Cytb基因和COI基因建立的NJ树都分为两大支,均为石鲽单独聚为一支。但在基因COI的NJ树中,母本星斑川鲽与杂交F1代又分为明显的两支:其中,外形表现为左眼的子代与星斑川鲽聚为一小支,而后与右眼杂交F1代聚为一支,而在基因Cytb基因的NJ树中没有这种分化。推测线粒体基因COI可能参与鲆鲽杂交F1代的左右眼移位。
鲆鲽左右眼移位的现象早就引起人们的关注,曾有研究调查显示,养殖的星斑川鲽两眼位于体左侧占95%–97%(王波, 2010)1),且有资料显示,在“日本海域100%为左眼型,东北太平洋的北美洲沿岸左右眼型各占50%”(肖志忠等, 2008)。关于左右眼移位发生的内在机制,也有不少研究者做了探究。研究发现,鲆鲽发育过程中有一段变态期,其体尺比例在这一期间发生巨大变化 (Seikai et al, 1986),包含大量的形态上和功能上的变化,也包含眼睛移位这一变化 (Inui et al, 1995)。不少学者认为,鲆鲽发育过程中眼睛移位与一些基因相关,但一直没有得到证明 (Bao et al, 2005; Tagawa et al, 2005; Hildahl et al, 2007)。关于鲆鲽杂交子代中出现左右眼分化的现象,也曾经出现在牙鲆 (♀)×石鲽 (♂) 的子代中,左右眼分别占44%、56%(Wang et al, 2003)。针对这一现象,Chen等 (2009)采用DD-RT-PCR和cDNA-RDA两种方法,针对两个重要的基因——肌酸激酶基因 (CK) 和胰蛋白酶原-2前体基因 (TR2P) 进行了研究,发现右眼的杂交子代中TR2P与父本石鲽相似性高达96%,而和母本牙鲆仅达66%,但并没有证明这2个基因与鲆鲽类的左右眼移位有关。结合本研究和前人的研究来看,可以推测出鲆鲽及鲆鲽杂交子代出现的左右眼移位这一现象不是偶然的,而是在其变态期,通过一系列复杂的的基因调控产生的,并初步得出线粒体基因COI也参与这一复杂过程。
1) Wang B. Study on the biological characteristics and healthy breeding technology of Platichthys stellatus. Master's Thesis of Chinese Academy of Agricultural Sciences, 2010 [王波.星斑川鲽的生物学及健康养殖技术研究.中国农业科学院硕士研究生学位论文, 2010]
4 展望星斑川鲽和石鲽是我国重要的经济鱼类,虽然星斑川鲽的养殖已经规模化、产业化,但养殖的石鲽皆为捕捞的野生幼鱼,养殖成活率较低。为此,星斑川鲽和石鲽杂交为改变石鲽养殖成活率低这一难题起到了积极作用。本杂交实验证明,杂交后代成活率很高,而且在杂交后代中出现了左右眼各半的现象。利用线粒体基因COI建立的NJ树也表明,基因COI与左右眼移位发生有关,为进一步研究鲆鲽左右眼的分化提供了新线索。
| Bao B, Yang G, Liu Z, et al. Isolation of SFRS3 gene and its differential expression during metamorphosis involving eye migration of Japanese flounder Paralichthys olivaceus. Biochimica et Biophysica Acta (BBA)-General Subjects, 2005, 1725(1): 64-70 DOI:10.1016/j.bbagen.2005.04.032 | |
| Céspedes A, García T, Carrera E, et al. Application of polymerase chain reaction-single strand conformational polymorphism (PCR-SSCP) to identification of flatfish species. Central European Journal of Medicine, 1999, 82(4): 903-907 | |
| Chen Y, Zhang Q, Qi J, et al. Screening of eye-position related genes with DD-RT-PCR and RDA in the hybrids between Japanese flounder Paralichthys olivaceus and stone flounder Kareius bicoloratus. Chinese Journal of Oceanology and Limnology, 2009, 27(1): 92-99 DOI:10.1007/s00343-009-0092-1 | |
| Garrett DL. A new intergeneric hybrid flatfish (Pleuronec-tiformes: Pleuronectidae) from Puget Sound and adjacent waters. Copeia, 2005(3): 673-677 | |
| Garrett DL, Pietsch TW, Utter FM, et al. The hybrid sole Inopsetta ischyra (Teleostei: Pleuronectiformes: Pleuronectidae): Hybrid or biological species?. Transactions of the American Fisheries Society, 2009, 136(2): 460-468 | |
| Green MR, Sambrook J. Molecular cloning: A laboratory manual. New York: Cold Spring Harbor Laboratory Press, 2010. | |
| Harrison RG. Animal mitochondrial DNA as a genetic marker in population and evolutionary biology. Trends in Ecology and Evolution, 1989, 4(1): 6-11 DOI:10.1016/0169-5347(89)90006-2 | |
| Hashimoto DT, Senhorini JA, Foresti F, et al. Interspecific fish hybrids in Brazil: Management of genetic resources for sustainable use. Reviews in Aquaculture, 2012, 4(2): 108-118 DOI:10.1111/raq.2012.4.issue-2 | |
| Hildahl J, Galay-Burgos M, Sweeney G, et al. Identification of two isoforms of Atlantic halibut insulin-like growth factor-I receptor genes and quantitative gene expression during metamorphosis. Comparative Biochemistry and Physiology, Part B: Biochemistry and Molecular Biology, 2007, 147(3): 395-401 DOI:10.1016/j.cbpb.2007.02.006 | |
| Inui Y, Yamano K, Miwa S. The role of thyroid hormone in tissue development in metamorphosing flounder. Aquaculture, 1995, 135(1): 87-98 | |
| Kartavtsev YP, Park TJ, Lee JS, et al. Phylogenetic inferences introduced on cytochrome b gene sequences data for six flatfish species (Teleostei, Pleuronectidae) and species synonymy between representatives of genera Pseudop-leuronectes and Hippoglossoides from far eastern seas. Russian Journal of Genetics, 2008, 44(4): 524-531 | |
| Kijewska A, Burzyński A, Wenne R. Molecular identification of European flounder (Platichthys flesus) and its hybrids with European plaice (Pleuronectes platessa). ICES Journal of Marine Science: Journal du Conseil: fsp, 2009, 110 | |
| Kosaka M. Morphological study of hybrid flatfish: Kareius bicoloratus×Platichthys stellatus in Sendai Bay [Japan]. Journal of the Faculty of Marine Science and Technology Tokai University, 1989, 13: 161-166 | |
| Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2005, 25(11): 1451-1452 | |
| Liu MH, Yu HX, Ma YK, et al. Morphological observation of wildlife economical fishes Verasper variegates and Kareius bicoloratus. Journal of Northeast Forestry University, 2009(9): 110-111 [刘曼红, 于洪贤, 马玉堃, 等. 两种野生经济鱼类圆斑星鲽和石鲽的最新形态观测. 东北林业大学学报, 2009(9): 110-111] | |
| Liu ZH, Wang B, Xu ZP, et al. A brief report on cross-breeding of starry flounder and other flounder. Fisheries Science & Technology Information, 2009(6): 267-270 [刘振华, 王波, 徐中平. 星斑川鲽与其他鲆鲽类杂交研究简报. 水产科技情报, 2009(6): 267-270] | |
| Miao GD, Shao CW, Liao XL, et al. Development of polymorphic microsatellite markers from barfin flounder (Verasper moseri) and their cross-species amplification. Conservation Genetics, 2009, 10(3): 701-703 DOI:10.1007/s10592-008-9619-9 | |
| Nei M. Molecular evolutionary genetics. Columbia University Press, 1987 | |
| Park JY, Kijima A. Genetic variability and differentiation within and between the stone flounder (Kareius bicoloratus) and the starry flounder (Platichthys stellatus). Tohoku Journal of Agricultural Research, 1991, 41(3-4): 69-82 | |
| Peng B, Zhang Q, Zhao S, et al. Genetic diversity analysis of Larimichthys polyactis in coastal waters of China based on cytochrome b gene. Guangdong Agricultural Sciences, 2010(2): 131-135 [彭博, 章群, 赵爽, 等. 中国近海小黄鱼遗传变异的细胞色素b序列分析. 广东农业科学, 2010(2): 131-135] | |
| Purdom C, Lincoln R. Gynogenesis in hybrids within the Pleuronectidae. The early life history of fish, Springer, 1974: 537-544 | |
| Sanjuan A, Comesa a AS. Molecular identification of nine commercial flatfish species by polymerase chain reaction-restriction fragment length polymorphism analysis of a segment of the cytochrome b region. Journal of Food Protection, 2002, 65(6): 1016-1023 DOI:10.4315/0362-028X-65.6.1016 | |
| Seikai T, Tanangonan J, Tanaka M. Temperature influence on larval growth and metamorphosis of the Japanese flounder Palalichthys olivaceus in the laboratory. Bulletin of the Japanese Society of Scientific Fisheries, 1986, 52(6): 977-982 DOI:10.2331/suisan.52.977 | |
| Šimková A, Vojtek L, Halačka K, et al. The effect of hybridization on fish physiology, immunity and blood biochemistry: A case study in hybridizing Cyprinus carpio and Carassius gibelio (Cyprinidae). Aquaculture, 2015, 435: 381-389 DOI:10.1016/j.aquaculture.2014.10.021 | |
| Tagawa M, AritakiM. Production of symmetrical flatfish by controlling the timing of thyroid hormone treatment in spotted halibut Verasper variegatus. General and Comparative Endocrinology, 2005, 141(2): 184-189 DOI:10.1016/j.ygcen.2005.01.013 | |
| Tajima F. Evolutionary relationship of DNA sequences in finite populations. Genetics, 1983, 105(2): 437-460 | |
| Takeda Y, Tanaka M. Freshwater adaptation during larval, juvenile and immature periods of starry flounder Platichthys stellatus, stone flounder Kareius bicoloratus and their reciprocal hybrids. Journal of Fish Biology, 2007, 70(5): 1470-1483 DOI:10.1111/j.1095-8649.2007.01424.x | |
| Tamura K, Stecher G, Peterson D, et al. MEGA 6: Molecular evolutionary genetics analysis version 6. 0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729 DOI:10.1093/molbev/mst197 | |
| Tang WJ, Hu XL, Yang JQ. Tang WJ, Hu XL, Yang JQ. Species validities of Coilia brachygnathus and C. nasus taihuensis based on sequence variations of complete mtDNA control regio N. Biodiversity Science, 2010, 15(3): 224-231 [唐文乔, 胡雪莲, 杨金权. 从线粒体控制区全序列变异看短颌鲚和湖鲚的物种有效性. 生物多样性, 2010, 15(3): 224-231] | |
| Wang B, Sun P, Fang HH, et al. Morphological characteristics and parameters mezsurement of starry flounder (platichthys stellatus Pallas). Acta Oceanologica Sinica, 2010(2): 139-147 [王波, 孙萍, 方华华, 等. 星斑川鲽形态特征及相关参数的观测. 海洋学报, 2010(2): 139-147] | |
| Wang XC, You F, Ni GT, et al. Hybridization between Stone flounder Kareius bicoloratus and Olive flounder Paralichthys olivaceus. Marine Sciences, 2003, 27(1): 1-4 [王新成, 尤锋, 倪高田, 等. 石鲽与牙鲆人工杂交的研究. 海洋科学, 2003, 27(1): 1-4] | |
| Wang Z, Wang X, Zhang Q. Interfamilial hybridization between Paralichthys olivaceus and Kareius bicoloratus-Early development and growth characters. Journal of Fishery Sciences of China, 2003, 10(7): 30-37 | |
| Xiao YS, Xiao ZZ, Liu QH, et al. Comparative analysis of genetic variability of mtDNA sequence in Platichthys stellatus, Scophthalmus maximus and their hybrids. Marine Sciences, 2014a, 38(6): 5-9 [肖永双, 肖志忠, 刘清华, 等. 星斑川鲽、大菱鲆及其杂交后代的线粒体DNA序列比较分析. 海洋科学, 2014a, 38(6): 5-9] | |
| Xiao YS, Zhang Y, Gao TX. Study of the relationship between genera Kareius bicoloratus and Platichthys stellatus by mtDNA sequence. Periodical of Ocean University of China, 2014b, 40(6): 69-76 [肖永双, 张岩, 高天翔. 基于线粒体DNA部分片段探讨石鲽与星突江鲽的亲缘关系. 中国海洋大学学报 (自然科学版), 2014b, 40(6): 69-76] | |
| Xiao ZZ, Wang B, Liu ZH. Morphological and anatomical studies on Platichthys stellatus.New Advances in Mariculture Research. Beijing: China Ocean Press, 2008: 260-269. [肖志忠, 王波, 刘振华. 星斑川鲽形态与解剖学研究.海水养殖研究新进展. 北京: 海洋出版社, 2008: 260-269.] | |
| Xu DD, You F, Lou B, et al. Analysis of correlation between pairwise genetic distance of eight flatfishes and hybrid fitness. Journal of Fisheries of China, 2010, 34(2): 178-184 [徐冬冬, 尤锋, 楼宝, 等. 8种鲆鲽鱼种间遗传距离与杂交亲和性的相关性分析. 水产学报, 2010, 34(2): 178-184] | |
| You F, Wang W, Xu DD, et al. Hybrids between olive flounder Paralichthys olivaceus and stone flounder Kareius bicoloratus: Karyotype, allozyme and RAPD analyses. Chinese Journal of Oceanology and Limnology, 2009, 27(2): 135-141 | |
| Zhang D, Lei GC, Gong C, et al. Genetic diversity of Neosalanx taihuensis based on mitochondrial COI sequences. Journal of Lake Sciences, 2012, 24(2): 299-306 DOI:10.18307/2012.0220 [张迪, 雷光春, 龚成, 等. 基于COI基因序列的太湖新银鱼遗传多样性. 湖泊科学, 2012, 24(2): 299-306] | |
| Zhang SH, Zhang XL, Sun AF, et al. Pellet culture technology of stone flounder. Shandong Fisheries, 2004, 21(5): 6-7 [张少华, 张秀丽, 孙爱凤, 等. 石鲽池塘养殖技术. 齐鲁渔业, 2004, 21(5): 6-7] | |
| Zhang Y, Xiao YS, Gao TX, et al. Comparative analysis of mtDNA gene sequences between two species of Pleuronectes. Journal of Fisheries of China, 2009, 33(2): 201-207 [张岩, 肖永双, 高天翔, 等. 两种黄盖鲽线粒体DNA部分片段比较分析. 水产学报, 2009, 33(2): 201-207] | |
| Zhang YY, Bi TT, Song JL, et al. Phylogenetic evolution of hinged turtles in China based on the complete mitochondrial cytochrome b gene sequences. Journal of Biology, 2011, 28(1): 22-26 [张艳云, 毕婷婷, 宋娇莲, 等. 基于线粒体Cytb基因的全长序列探讨闭壳龟类的系统进化. 生物学杂志, 2011, 28(1): 22-26] | |
| Zhou HL, Zhang Y, Qi X, et al. SSR analysis of the heterosis in the two grouper hybrids. Journal of Fisheries of China, 2012, 36(2): 161-168 [周翰林, 张勇, 齐鑫, 等. 两种杂交石斑鱼子一代杂种优势的微卫星标记分析. 水产学报, 2012, 36(2): 161-168] |



