2. 农业部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;
3. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071
海带(Saccharina japonica)是重要的经济褐藻,富含碘、褐藻胶和甘露醇等(刘福利等, 2012),在食品加工、化工、医药等领域都有广泛的应用(李林等, 2001)。自20世纪50年代起,我国就开展了海带遗传育种研究与应用的工作,先后培育出许多新品种(Liu et al, 2012a),在海带养殖的增产、增效中发挥了重要作用。目前,海带仍存在种质退化、混杂、抗逆性差等问题(方宗熙, 1963),影响着海带产业的可持续发展。因此,加强对海带品系间的遗传关系和群体遗传多样性研究意义重大。
分析海带遗传多样性的分子标记方法很多,如RAPD(Random-amplified polymorphic DNA)标记,检测速度快、DNA用量少,但稳定性和重复性不高(夏鹏等, 2003; 周志刚等, 2003);AFLP(Amplified fragment length polymorphism)标记具有较高的稳定性、可用少量的引物短时间内检测大量的位点,但对基因组纯度和反应条件要求较高(Shan et al, 2009; 张全胜等, 2008; 李世国等, 2009);ISSR(Inter-simple sequence repeat)标记实验技术操作简单、快速、高效,但需要时间摸索最适条件,且不能区分显性纯合和杂合基因型(Wang et al, 2005) 以及SSR(Simple sequence repeat)等方法(罗兵等, 2013; 张荣良等, 2016)。RAPD、ISSR和AFLP的标记引物属于通用引物,是非特异性标记。SSR分子标记技术具有以下特点:共显性、特异性高(刘岩等, 2006; 石媛嫄等, 2008; 丛义周等, 20091)),能大大降低非目标生物的干扰;涵盖范围广、揭示多态性高。Gerber等(2000)发现,AFLP标记的多态性远远小于SSR标记的多态性;样本DNA的质量要求低、成本低。Chamberlain等(1998)首次引入多重PCR技术,将SSR的稳定性与多重PCR的高通量特点相结合,大大提高了SSR分型效率,从而降低了遗传分析的时间与成本,另外,新一代测序技术的发展,也降低了微卫星位点的开发成本,开发出大量的微卫星标记(刘福利等, 2013; Song et al, 2013)。据此,本研究采用SSR标记技术对食用海带新品系的遗传多样性进行分析。
1) Cong YZ. Germplasm analysis of Dongfang No.3 hybrid Laminaria. Master's Thesis of Chinese Academy of Agricultural Sciences, 2009 [丛义周.东方3号杂交海带种质分析.中国农业科学院硕士研究生学位论文, 2009]
“海天1号”、“海天2号”和“海天3号”海带是中国水产科学研究院黄海水产研究所与山东荣成寻山集团共同选育的3个食用海带新品系,具有产量高、耐高温、成熟较早等特点。其中,“海天1号”为北方高产海带与南方海带杂交获得,“海天2号”是“奔牛”紫外处理选育获得,“海天3号”是“黄官1号”紫外处理选育所得。为了解4个海带群体遗传多样性和遗传关系,本研究选用SSR分子标记,从20对引物中(Zhang et al, 2015; Liu et al, 2012b; Shi et al, 2007)筛选出8对扩增效果较好的引物,对每个品系的30个样本进行分析评价,为海带品系的遗传关系和群体遗传多样性研究提供科学依据。
1 材料与方法 1.1 实验材料本研究用的“海天1号”、“海天2号”、“海天3号”和“黄官1号”4个海带品系采自山东威海市荣成寻山集团青鱼滩养殖场,每个品系取30个个体(幼孢子体长约10 cm),做好标记后放入自封袋,用冰盒带回实验室。
1.2 实验方法 1.2.1 DNA提取采用天根植物基因组DNA提取试剂盒提取4个海带品系共120个样品的基因组DNA。使用琼脂糖凝胶电泳检测基因组DNA的完整性和纯度,于-20℃冰箱保存备用。
1.2.2 微卫星引物筛选随机取4个样品进行引物筛选,通过预实验筛选出多态性较高的引物进行正式的实验。PCR反应体系为10× Ex Taq Buffer 2.5 μl,dNTP(2.5 mmol/L)2 μl,Mix primer 1 μl,DNA 1 μl,Ex Taq 0.2 μl,H2O 18.3 μl,总体积25 μl。PCR反应程序为95℃ 2 min,95℃ 30 s,56℃ 20 s,72℃ 30 s,25个循环,72℃ 5 min,12℃保温。
1.2.3 数据统计和分析用预实验筛选出的8对特异性、多态性较好的引物,对120个样本进行扩增、上机分型检测。将上机结果原始文件导入Genemarker,选用LIZ500内标准进行数据分析,按位点导出峰图、Excel位点信息表。
根据Excel原始数据,去除非特异性条带,对特异性条带片段大小的相关信息进行整理。记录每对引物所有片段的大小,形成原始数据表征阵。并使用Convert软件将其转换成特定的格式。
用软件Popgen 32对4个海带品系的遗传多样性进行评估,选择的参数包括香农指数(I),多态位点比率(P),等位基因数(Na)和有效等位基因(Ne),Nei's基因多样性指数(H),表观杂合度(Ho)和期望杂合度(He)等。
品系间的遗传分化用Popgen 32计算遗传进化系数(Gst)和基因流(Nm);用Mega 4.1根据遗传距离采用UPGMA(Unweighted pair group method with arithmetic mean)方法构建品系间的聚类分析图。使用Arlequin version 3.11对4个海带品系进行AMOVA (Analysis of molecular variance)分析,计算遗传变异来源及品系间的遗传分化系数。
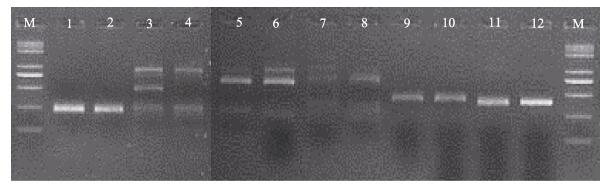
2 结果与分析 2.1 引物筛选随机抽取4个海带样品的基因组DNA,按照SSR-PCR反应体系进行筛选引物,结果见表 1。扩增出2个条带的引物有SSR261、DQ978345、DQ978340、Unigene8756、Unigene15777,扩增出3个条带的引物有CS55、Unigene95068、Unigene17168,其他引物只扩增出1个条带或条带比较弥散。选用扩增出2个或3个条带的这8对引物,对4个品系的120个样本进行毛细管电泳,部分SSR引物筛选的琼脂糖凝胶电泳结果和毛细管电泳结果分别见图 1和图 2,引物信息见表 2。
|
|
表 1 20对引物筛选结果 Table 1 Screening of twenty pair of published primers |
|
图 1 部分SSR引物预实验扩增结果 Figure 1 The results of pre-experiment amplification using partial SSR primers M: DNA Marker; 1, 2: DQ978345; 3, 4: CS55; 5, 6: AW401303; 7, 8: CN468232; 9, 10: DQ98342; 11, 12: DQ978340 |

|
图 2 引物DQ978340在“海天1号”海带的4个个体中的扩增结果 Figure 2 Results of four individual S.japonica "Haitian No.1" amplified by primer DQ978340 |
|
|
表 2 8对海带SSR引物特征 Table 2 Characteristics of eight pairs of SSR primers of S. japonica |
由图 2可以看出,引物DQ978340在“海天1号”的4个个体中的扩增结果显示其具有较好的多态性,扩增的基因型分别为(362、377),(377、377),(368、368),(350、362),多态性等位基因的片段大小分别为350、362、368和377。这些等位基因的峰值较高且无杂峰,具有较高的可信度。该引物与其他7对引物分型效果相似,均能较好地对不同海带品系进行遗传分析。
2.2 引物多态性和扩增结果8对引物的扩增结果见表 3。由表 3可知,8对引物共检测出37个等位基因,观察等位基因数为2-8个,平均为4.6250个;有效等位基因数为1.1353-3.3431个。其中,SSR261观察等位基因、有效等位基因数最多分别为8、3.3431个,CSS55观察等位基因、有效等位基因数最少分别为2、1.1353个。8对SSR引物均具有多态性,Unigene15777、Unigene17168和Unigene8756的多态率最好,达到100%,最低的是DQ978340引物(42.86%),几对引物的平均多态位点百分率是77.86%。
|
|
表 3 不同SSR位点的遗传参数 Table 3 Genetic parameters of different SSR loci |
8对引物共扩增出9个特异性基因,片段大小为156-368 bp (表 4)。“海天1号”海带具有6个特异等位基因,数目最多;“海天2号”和“黄官1号”分别具有2个和1个特异等位基因。由于本研究只选用了8对引物及每个品系的30个样本,不能确切的说这些特异性基因专属于某个品系,可通过选用更多的样品进行验证。
|
|
表 4 特异等位基因统计 Table 4 Unique alleles in four cultivars of S. japonica |
各品系的遗传参数见表 5,4个海带品系的等位基因(Na)为2.6250-3.6250,Nei's基因多样性(H)为0.2943-0.4705,香农指数(I)为0.5072-0.8583,说明这4个海带品系的遗传多样性呈中等程度。其中,“海天1号”和“海天2号”海带的观察杂合度和期望杂合度较高;“黄官1号”海带的最低。“海天1号”海带的Nei's基因多样性(H)、香农指数(I)和多态性位点均最高,“黄官1号”对应的这3个参数最低。
|
|
表 5 4个海带品系的遗传参数 Table 5 Genetic parameters in four cultivars of S. japonica |
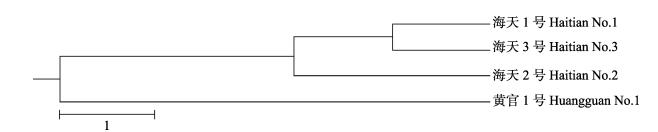
由表 6知,“海天2号”和“海天3号”海带的遗传距离最大为0.9707,“海天1号”和“海天3号”海带的遗传相似系数最低,为0.0297。4个海带品系间的的遗传相似系数和遗传距离分别位于0.8732-0.9707和0.0297-0.1356范围内,说明4个海带品系亲缘关系较近。根据品系间的遗传距离,利用UPGMA法构建了4个海带品系的系统进化树(图 3)。4个海带品系被分为2个大枝,“黄官1号”海带单独构成了1个分枝。另外3个海带品又被分成了2枝,其中,“海天1号”和“海天3号”为1个分枝,“海天2号”为另一分枝。
|
|
表 6 4个海带品系的遗传相似系数(右上角)和遗传距离(左下角) Table 6 Nei's unbiased measures of genetic identity (above diagonal) and genetic distance (below diagonal) among four S. japonica cultivars |
|
图 3 根据遗传距离采用UPGMA法构建的4个海带品系的系统进化树 Figure 3 Dendrogram of four cultivars of S. japonica by UPGMA clustering based on genetic distance |
4个海带品系间的基因分化系数(Gst)值为0.082,基因流(Nm)为2.797。表明4个海带品系的遗传分化程度较低,但有一定的基因交流。AMOVA分析表明,7.13%的遗传变异存在于不同的品系间,而98.94%存在于品系内部(表 7),品系间和品系内的遗传变异存在显著差异(P < 0.05,1000次重复)。
|
|
表 7 4个海带品系的AMOVA分析 Table 7 AMOVA analysis among and within four cultivars of S. japonica |
开发海带微卫星引物的研究已有较多报道,Billot等(1998)用掌状海带(Laminaria digitata)基因组DNA开发出10个具有多态性的微卫星位点,芮凤平(2013)1)从海带转录组(EST-SSR)和基因组DNA中开发出143对具有多态性的引物。本研究从已开发的SSR引物中挑选了20对,其中,只有8对能够进行稳定的PCR扩增并具有清晰的条带。毛细管电泳结果显示,8对引物均具有较高的多态性,其余的12对引物PCR扩增条带弥散,无法进行等位基因的判读或多态性较低。主要原因可能包括海带DNA存在干扰PCR扩增反应的物质(Patwary et al, 1994; Varela-lvarez et al, 2006)和海带微卫星引物区域变异较大,产生基因逃逸(Buchan et al, 2005; Hoffman et al, 2005)。其次,与物种个体间的差异和PCR扩增程序以及毛细管电泳等有关。如毛细管电泳检测结果较灵敏,碱基的插入或缺失都会影响其结果的判定。
1) Rui FP. Development of EST-SSR markers from Saccharina japonica and the study of the genetic diversity. Master's Thesis of Ocean University of China, 2013, 22-23 [芮凤平.海带EST-SSR标记的开发及其遗传多样性研究.中国海洋大学硕士研究生学位论文, 2013, 22-23]
3.2 不同海带品系间的遗传多样性和遗传关系与陆生作物相比,海带育种经过长期的人工干预,连续多代自交或近交,某些重要的经济性状产生分离或丢失,使其呈现较低的遗传多样性。通过分析发现,“海天1号”海带具有较高的遗传多样性,而“黄官1号”海带的遗传多样性相对较低。另外,“黄官1号”海带的杂合度、Nei's基因多样性和香农指数均低于李涛等(2012)的研究结果。作者认为,这主要是因为“黄官1号”海带是经过杂交后,又经历多代自交选育而出,在自交的过程中存在性状分离、基因丢失等现象。理论上,随着时间的积累其杂合度呈下降趋势,具有较低的遗传多样性。
通过对不同海带品系间的基因流、遗传距离以及相似系数的分析发现,3个海带新品系之间的遗传分化呈中等水平,遗传相似系数最高达到0.9707,遗传距离最小为0.0297。UPGMA法构建的系统树结果显示,“海天1号”和“海天3号”海带亲缘性最近,与“黄官1号”海带较远。“海天3号”是“黄官1号”紫外处理后自交获得,理论上与“黄官1号”海带应具有较小的遗传距离和较高的遗传相似性。这说明,紫外处理在一定程度上改变了“海天3号”海带的遗传物质,使其表现出与“黄官1号”遗传物质上存在一定的变异。
AMOVA分析表明,4个海带品系的变异主要来自品系内部,来自品系间的变异仅占总变异的7.94%,说明这4个海带品系的亲缘关系较近。主要是因为在选育过程中,定向选择优良性状如高产、耐高温、高碘、高醇等,使具有目标性状的个体在选育的过程中保留下来,这也导致了遗传多样性降低(李涛等, 2012)。
物种遗传多样性是长期进化的产物,是生物适应、生存、发展和进化的前提,物种的遗传多样性越高或遗传变异越丰富,对环境变化的适应能力就越强(刘坤等, 2013),反之对环境的适应能力就会降低。我国养殖的海带遗传多样性普遍较低,种群基因多样性为0.451,香农指数在0.18-0.55之间(王莹等, 1999),本研究中的3个海带的遗传多样性也在这一范围内。通过分析与评价,对4个海带品系的遗传多样性有了一定的了解,也为将来海带种质创新、杂交亲本选择、保护种群遗传多样性等提供可靠的依据。
| Billot C, Rousvoal S, Estoup A, et al. Isolation and characterization of microsatellite markers in the nuclear genome of the brown algae Laminaria digatata (Phaeophyceae). Molecular Ecology, 1998, 7(12): 1778-1780 DOI:10.1046/j.1365-294x.1998.00516.x | |
| Buchan JC, Archie EA, van Horn RC, et al. Locus effects and sources of error in noninvasive genotyping. Molecular Ecology Notes, 2005, 5(3): 680-683 DOI:10.1111/men.2005.5.issue-3 | |
| Chamberlain JS, Gibbs RA, Rainer JE, et al. Deletion screening of the Duchenne muscular dystrophy locus via multiplex DNA amplification. Nucleic Acids Research, 1988, 16(23): 11141-11156 DOI:10.1093/nar/16.23.11141 | |
| Fang ZX. Piant breeding in Laminaria and the basic principles. Journal of Shandong College of Oceanology, 1963(2): 109-119[方宗熙. 从海带的遗传育种工作谈育种的一般原理. 山东海洋学院学报, 1963(2): 109-119] | |
| Gerber S, Mariette S, Streiff R, et al. Comparison of microsatellites and amplified fragment length polymorphism markers for parentage analysis. Molecular Ecology, 2000, 9(8): 1037-1048 DOI:10.1046/j.1365-294x.2000.00961.x | |
| Hoffman JI, Amos W. Microsatellite genotyping errors: Detection approaches, common sources and consequences for paternal exclusion. Molecular Ecology, 2005, 14(2): 599-612 | |
| Li L, Luo Q, Zhang SH. Analysis on composition of fucoidin in Laminaria japonica Aresch. Journal of Chinese Institute of Food Science and Technonlogy, 2001, 1(1): 46-49[李林, 罗琼, 张声华. 海带中褐藻糖胶的组成分析. 中国食品学报, 2001, 1(1): 46-49] | |
| Li SG, Shan TF, Hou HS, et al. Genetic analyses of principal farmed cultivars of Laminaria japonica in China: AFLP analyses of selfbreeding F1 of nine cultivars. Journal of Fishery Sciences of China, 2009, 16(2): 214-220[李世国, 单体锋, 侯和胜, 等. 9个海带栽培品种自交系后代遗传多样性和亲缘关系的AFLP分析. 中国水产科学, 2009, 16(2): 214-220] | |
| Li T, Liu FL, Wang FJ, et al. The genetic analysis and evaluate of Saccharina Huangguan No. 1. Journal of Qingdao Agricultural University (Natural Science), 2012, 29(3): 212-217[李涛, 刘福利, 王飞久, 等. 黄官1号海带的遗传学分析与评价. 青岛农业大学学报(自然科学版), 2012, 29(3): 212-217] | |
| Li XJ, Liu JL, Cong YZ, et al. Breeding and trial cultivation of Dongfang No. 3, a hybrid of Laminaria gametophyte clones with a more than intraspecific but less than interspecific relationship. Aquaculture, 2008, 280(1-4): 76-80 | |
| Liu FL, Liu K, Wang FJ, et al. EST-SSR marker development and its application in population genetic diversity analysis of Zostera marina. Progress in Fishery Sciences, 2013, 34(4): 91-97[刘福利, 刘坤, 王飞久, 等. 大叶藻EST-SSR标记开发及其在大叶藻群体遗传多样性研究中的应用. 渔业科学进展, 2013, 34(4): 91-97] | |
| Liu FL, Wang FJ, Duan DL. EST-SSR markers derived from Laminaria digitata and its transferable application in Saccharina japonica. Journal of Applied Phycology, 2012a, 24(3): 501-505 DOI:10.1007/s10811-012-9807-3 | |
| Liu FL, Wang FJ, Sun XT, et al. Molecular breeding and its research advances and prospects in Laminaria japonica breeding. Marine Sciences, 2012, 36(9): 128-134[刘福利, 王飞久, 孙修涛, 等. 分子育种及其在海带育种中的研究进展. 海洋科学, 2012, 36(9): 128-134] | |
| Liu FL, Yao JT, Wang XL, et al. Genetic diversity and structure within and between wild and cultivated Saccharina japonica (Laminariales, Phaeophyta) revealed by SSR markers. Aquaculture, 2012b, 358-359: 139-145 DOI:10.1016/j.aquaculture.2012.06.022 | |
| Liu K, Liu FL, Wang FJ, et al. Analysis of genetic diversity and structure of Zostera marina populations in Shandong peninsula. Journal of Shanghai Ocean University, 2013, 22(3): 334-340[刘坤, 刘福利, 王飞久, 等. 山东半岛大叶藻不同地理种群遗传多样性和遗传结构分析. 上海海洋大学学报, 2013, 22(3): 334-340] | |
| Liu Y, Cui JZ, Shen XY, et al. The application of DNA molecular marker technique to heritable breeding of Laminaria. Transactions of Oceanology and Limnology, 2006(1): 75-81[刘岩, 崔建洲, 申雪艳, 等. DNA分子标记技术在海带遗传育种研究中的应用. 海洋湖沼通报, 2006(1): 75-81] | |
| Luo B, Sun HY, Xu GM, et al. Research progress of SSR molecular marker. Journal of Anhui Agriculture Science, 2013, 41(12): 5210-5212,5246 DOI:10.3969/j.issn.0517-6611.2013.12.013[罗兵, 孙海燕, 徐港明, 等. SSR分子标记研究进展. 安徽农业科学, 2013, 41(12): 5210-5212,5246] | |
| Patwary MU, van der Meer JP. Application of RAPD markers in an examination of heterosis in Gelidium vagum (Rhodophyta). Journal of Phycology, 1994, 30(1): 91-97 DOI:10.1111/j.0022-3646.1994.00091.x | |
| Shan TF, Pang SJ. Assessing genetic identity of sporophytic offspring of the brown alga Undaria pinnatifida derived from mono-crossing of gametophyte clones by use of amplified fragment length polymorphism and microsatellite markers. Phycological Research, 2009, 57(1): 36-44 DOI:10.1111/pre.2009.57.issue-1 | |
| Shi Y, Yang G, Liu Y, et al. Development of 18 polymorphic microsatellite DNA markers of Laminaria japonica (Phaeophyceae). Molecular Ecology Notes, 2007, 7(4): 620-622 DOI:10.1111/j.1471-8286.2006.01652.x | |
| Shi YY, Yang GP, Liao MJ, et al. Comparative study on the microsatellite DNA polymorphism of the gametophytes of L.japonica and L. longissima. Periodical of Ocean University of China, 2008, 38(1): 303-308[石媛嫄, 杨官品, 廖梅杰, 等. 海带和长海带配子体无性繁殖系微卫星DNA多态性比较分析. 中国海洋大学学报, 2008, 38(1): 303-308] | |
| Song SL, Lim PE, Phang SM, et al. Microsatellite markers from expressed sequence tags (ESTs) of seaweeds in differentiating various Gracilaria species. Journal of Applied Phycology, 2013, 25(3): 839-846 DOI:10.1007/s10811-012-9943-9 | |
| Varela- lvarez E, Andreakis N, Lago-Lestón A, et al. Genomic DNA isolation from green and brown algae (Caulerpales and Fucales) for microsatellite library construction. Journal of Phycology, 2006, 42(3): 741-745 DOI:10.1111/jpy.2006.42.issue-3 | |
| Wang XL, Liu CL, Li XJ, et al. Assessment of genetic diversities of selected Laminaria (Laminariales, Phaeophyta) gametophytes by inter-simple sequence repeat analysis. Journal of Integrative Plant Biology, 2005, 47(6): 753-758 DOI:10.1111/jipb.2005.47.issue-6 | |
| Wang Y, Dai JX. Comparison of genetic diversity in Laminaria japonica gametophytes. Chinese Biodiversity, 1999, 7(3): 197-201[王莹, 戴继勋. 海带(Laminaria japonica)丝状体遗传多样性的比较研究. 生物多样性, 1999, 7(3): 197-201] | |
| Xia P, Yang YX, Liu SP, et al. Optimization of randomly amplified reaction conditions for gametophyte DNA of Laminaria "901"strain. Marine Sciences, 2003, 27(5): 22-26[夏鹏, 杨迎霞, 刘升平, 等. 海带"901"配子体DNA随机扩增反应条件的优化. 海洋科学, 2003, 27(5): 22-26] | |
| Zhang J, Li W, Qu JQ, et al. Development and characterization of microsatellite markers from an enriched genomic library of Saccharina japonica. Journal of Applied Phycology, 2015, 27(1): 479-487 DOI:10.1007/s10811-014-0301-y | |
| Zhang QS, Shi YY, Cong YZ, et al. AFLP analysis of the gametophyte clones derived from introduced Laminaria (Phaeophyta) and cultured varieties of China. Periodical of Ocean University of China, 2008, 38(3): 429-435[张全胜, 石媛媛, 丛义周, 等. 我国引种海带和栽培品种(系)来源配子体克隆的AFLP分析. 中国海洋大学学报, 2008, 38(3): 429-435] | |
| Zhang RL, Wang WJ, Feng YW, et al. Assessment of genetic variability and microsatellite analysis of Pacific oyster (Crassostrea gigas) after artificial selection of the shell width. Progress in Fishery Sciences, 2016, 37(4): 90-96 DOI:10.11758/yykxjz.20150526004[张荣良, 王卫军, 冯艳微, 等. 长牡蛎(Crassostrea gigas)壳宽快速生长选育群体遗传多样性及遗传结构的微卫星标记分析. 渔业科学进展, 2016, 37(4): 90-96] | |
| Zhou ZG, Shi XZ, Hu YJ, et al. Genetic relationship among brown seaweed Laminaria longissima and various cultivars of L japonica in China revealed by isozyme and RAPD markers.. Journal of Fishery Sciences of China, 2003, 10(6): 474-480[周志刚, 史西志, 胡远皆, 等. 中国不同养殖品系海带与长海带之间的遗传关系. 中国水产科学, 2003, 10(6): 474-480] |



