2. 上海海洋大学水产与生命技术学院 上海 201306
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
肌球蛋白重链(Myosin heavy chain,MYH)的结构分为头区和尾区。常规MYH的N端头区通常与1个调节型肌球蛋白轻链(Regulatory myosin light chain,RLC)和1个必需轻链(Essential myosin light chain,ELC)结合,折叠盘绕形成球状头部作为“马达”功能区;2个MYH的C端棒状区盘绕形成卷曲螺旋,并聚集成束,形成粗肌丝束主体。其中,头部“马达”功能区有ATP结合位点,可结合ATP,将其水解为ADP释放能量,为肌肉收缩提供动力而影响运动、摄食等活动,从而直接影响鱼类生长(Campion et al, 2012)。MYH也广泛存在于非肌细胞中,是细胞骨架的组成成分,参与细胞内的细胞器运动与有丝分裂,细胞外的吞噬、运动、受精和吸收等生理过程,充当非肌细胞生命活动的不同层次的调节者,包括细胞间的信号传递、细胞形状改变等较高级的调节(Sebé-Pedrós et al, 2014)。
哺乳动物肌肉生长只在未成年阶段涉及肌纤维增生和增粗,成体只有增粗而不再增生;而鱼类肌纤维始终存在增生和增粗,鱼类这种生长现象被称为非限制性生长。因MYH基因表达量高低影响肌纤维的增生,被认为与鱼类的非限制性生长有关(Biga et al, 2006)。在鱼类中发现的MYH亚型远远多于哺乳动物,这也被认为是鱼类基因组倍化的可靠证据(Yasmin et al, 2011; Asaduzzaman et al, 2016)。在虹鳟(Oncorhynchus mykiss)、大西洋鲑(Salmo salar)和大菱鲆(Scophthalmus maximus)等鱼类中发现,MYH的表达量与鱼生长速度有显著相关性(Overturf et al, 2001; Dhillon et al, 2008; Churova et al, 2015)。Hevry等(2006) 发现,投喂不同膳食水平68 d后,大西洋鲑肌肉MYH的表达量与肌肉蛋白总量和体重显著相关,MYH的表达量可作为表征鱼类生长的重要标记。
为了解影响翘嘴鳜(Siniperca chuatsi)早期生长的关键基因,本研究从同批孵化、生长存在显著差异的翘嘴鳜的转录组差异表达谱数据中,筛选差异表达MYH基因,克隆获得2个表达量差异显著的MYH全长cDNA,进行序列结构分析,并分析其组织分布及胚胎发育的表达规律;检测这2个基因在孵化出膜后不同阶段的(15、30、60 days post hatching,dph)快长组和慢长组翘嘴鳜白肌中的表达量,探讨其与生长的相关性,为后续翘嘴鳜快长品系的分子选育提供候选基因与位点。
1 材料与方法 1.1 材料实验时间为2014年5月–2015年11月,实验用翘嘴鳜取自广东省清新县宇顺农牧渔业科技服务有限公司养殖基地。转录组测序用翘嘴鳜选择同批孵化、培育30 dph的翘嘴鳜,从中选取生长快和生长慢的个体各12尾,取白肌组织;不同胚胎发育时期的翘嘴鳜源自同一对亲本的受精卵,显微镜下观察胚胎发育时期(刘希良等, 2013),分别取未受精卵、受精卵、囊胚期、神经胚期、体节出现期、尾芽期、肌肉效应期和出膜幼鱼各期样品10枚卵(尾)/组,各3组;随机选取3尾健康成鱼,取眼、鳃、皮肤、白肌、红肌、心肌、胃、肠、肝、脾、肾、性腺、脑及垂体共14个组织作为组织分布样品;取同批15 dph、30 dph和60 dph的快长组和慢长组的翘嘴鳜各5尾,测体重,取白肌组织作为不同生长时期快长组和慢长组样品(平均体重及差异见表 1)。上述样品置于盛有1 ml TRIzol (Invitrogen,美国)的1.5 ml离心管中并匀浆,随即液氮冻存。
|
|
表 1 孵化出膜后各时期翘嘴鳜的平均体重 Table 1 The average body weight of S. chuatsi at different growth stages after hatching (g) |
将上述组织样品按照TRIzol试剂盒说明书提取总RNA,总RNA用DNaseⅠ(TaKaRa,日本)去除基因组DNA后,用Biophotometer (Eppendorf,德国)及琼脂糖凝胶电泳检测RNA浓度及纯度,样品浓度≥400 ng/μl,电泳检测条带清晰,样品–70℃保存备用。cDNA合成,使用Transcriptor First Strand cDNA Synthesis Kit (Roche,瑞士)反转录试剂盒,取1 μg总RNA合成cDNA第1链,–20℃保存备用,具体步骤参考说明书。将快长组和慢长组翘嘴鳜RNA (12个/组)各取3 µl混合,形成快、慢RNA混池,送深圳华大基因公司,经随机打断、反转录、末端修复、加测序接头后,通过Hiseq2000 (Illumina,美国)平台,进行RNA denovo测序。
1.3 转录组表达量分析测序后得到clean reads,用短reads组装软件Trinity对clean reads进行从头组装和过滤后得到高质量的Unigenes。用FPKM (Fragments per kilobase of exon per million fragments mapped)法计算基因表达量(Mortazavi et al, 2008)。通过错误发生率(False discovery rate,FDR)多重检验校正P值,并根据FPKM比较该基因在快长、慢长组中的差异表达倍数。筛选FDR≤0.001且差异倍数不低于2倍(即|log2 ration|≥1) 的基因为差异显著基因。
1.4 翘嘴鳜肌球蛋白重链基因RACE克隆提取健康翘嘴鳜肌肉总RNA,采用SMARTerTM RACE cDNA Amplification Kit (Clontech,美国)合成5'端cDNA第1链和3'端cDNA第1链。根据转录组差异表达谱数据,从转录组表达量差异显著的43个的翘嘴鳜肌球蛋白重链基因中,选出表达量差异最大的2条MYH基因(CL127.Contig1_All和Unigene19394_ All),根据其序列片段,用Primer Premier 5.0软件设计5'、3'RACE引物(5'-GSP/3'-GSP),并与UPM/UPS通用引物进行PCR反应,引物信息见表 2。反应体系:5'/3'端cDNA 2.5 µl,10×UPM 5 µl,5'/3'特异引物(5'/3' GSP-1) 1 µl,2×SeqAmp Buffer 25 µl,SeqAmp DNA酶1 µl,ddH2O补足至50 µl。扩增程序:94℃ 5 min,5个循环:94℃ 30 s,72℃ 4 min;5个循环:94℃ 30 s,70℃ 30 s,72℃ 4 min;20个循环:94℃ 30 s,68℃ 30 s,72℃ 4 min。第2轮巢式PCR以第1轮PCR产物稀释50倍为模板,用通用引物UPS与5'/3'GSP-2进行PCR反应,扩增程序同第1轮PCR。5'和3'RACE扩增产物经凝胶电泳检测目的带并切胶回收,PCR产物克隆至pEASY blunt zero载体(全式金)中,经菌液PCR鉴定后,挑取阳性菌送广州艾基生物技术有限公司测序。测序结果利用DNAMAN 8.0软件将5'RACE片段、3'RACE片段和Unigene片段序列进行拼接。
|
|
表 2 翘嘴鳜肌球蛋白重链基因的克隆及实时荧光定量PCR表达分析使用的引物 Table 2 Primers for cloning and qRT-PCR analysis of S. chuatsi myosin heavy chain |
用BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi)对测序结果进行同源性检索。NCBI ORF Finder (http://www.ncbi.nlm.nih.gov/projects/gorf/)确定其开放阅读框并预测氨基酸序列。用Protparam工具(http://web.expasy.org/protparam/)对推测蛋白进行基本理化参数分析;根据PredictProtein工具(https://www.predictprotein.org/)分析蛋白二级结构及功能;interProScan工具(http://www.ebi.ac.uk/interpro/scan.html)分析结构域;用NLS Mapper (http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi)在线预测核定位序列。用ClustalW软件将翘嘴鳜及其他物种肌球蛋白重链基因的氨基酸头区序列进行多重比对,用MEGA 5.0软件构建M-L(Maximum Likelihood)系统发育树(Bootstraps设为1000次)。
1.6 翘嘴鳜MYH-7s基因表达分析使用合成的cDNA第1链作为实时荧光定量PCR (qRT-PCR)分析的模板,以β-actin为内参基因(Zhou et al, 2010),分别设计目的基因及内参基因的qRT-PCR引物(表 1)。
qRT-PCR采用ABI 7300实时荧光定量PCR仪(Life Technologies,美国),反应体系根据SYBR Premix Ex Taq(Tli RNaseH Plus) (TaKaRa,日本)说明书进行设置:cDNA模板2 µl,上、下游引物(10 μmol/L)各0.4 µl,2×SYBR Premix Ex Taq 10 µl,50×ROX Reference Dye 0.4 µl,ddH2O 6.8 µl。反应程序:95℃ 1 min,40个循环:95℃ 30 s,55℃ 30 s,72℃ 30 s。熔解曲线:95℃ 15 s,55℃ 15 s,95℃ 15 s。采用2–△△CT方法计算目的基因相对表达量。用SPSS 20.0软件进行单因素方差分析(One-way ANOVA),利用Origin6.0对统计结果进行作图。
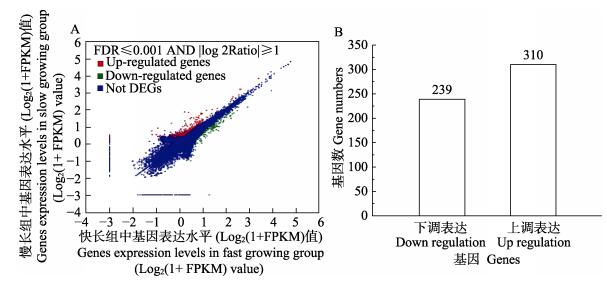
2 结果 2.1 转录组差异表达基因筛选使用Hiseq 2000测序平台分析快长组与慢长组翘嘴鳜的转录组,共获41641条Unigene (平均长度为902 nt),经数字基因表达谱DGE分析,筛选出549个表达量有显著差异Unigene,其中310个上调(图 1)。这549个Unigene中,有43条目的annotation注释为肌球蛋白重链,其中,快长组与慢长组相比,有32个显著上调,11个显著下调。
|
图 1 翘嘴鳜生长快、慢群体中差异表达基因分布
Figure 1 The distribution of differentially expressed genes in fast and slow growing groups of S. chuatsi
A:所有的Unigene表达量分布;B:表达量差异显著基因数统计 FDR:错误发生率;FPKM:每千个碱基的转录每百万映射读取的碎片,表征表达量高低 The expression distribution of all unigenes; B. Genes number of significantly different expression FDR: False discovery rate; FPKM: Fragments per kilobase of exon per million fragments mapped (represent the expression level) |
以5'/3'RACE cDNA为模板分别进行5'/3'端RACE,获得2个MYH基因cDNA全长。NCBI BLAST分析显示,2个基因均与MYH-7基因高度同源,命名为MYH-7a (以Unigene9779_All为模板,GenBank登录号:KX085225)和MYH-7b (以CL2161.Contig6_All为模板,GenBank登录号:KX085226) 亚型。MYH-7a全长为6071 bp,5'UTR为107 bp,3'UTR为143 bp;ORF长为5820 bp (109–5928),编码1939个氨基酸,推测分子量为223 kDa,等电点为5.57。MYH-7b全长为5896 bp,5'UTR为95 bp,3'UTR为146 bp;ORF长为5745 bp (96–5840),编码1914个氨基酸,推测分子量为220 kDa,等电点为5.62。二者核酸相似性为83.71%,氨基酸相似性为89.12%。
核苷酸序列同源性比较显示,MYH-7a与深裂眶锯雀鲷(Stegastes partitus)、尼罗罗非鱼(Oreochromis niloticus)、斑马宫丽鱼(Maylandia zebra)、伯氏朴丽鱼(Haplochromis burtoni)、红丽鱼(Pundamilia nyererei)和红鳍东方鲀(Takifugu rubripes)等的MYH-7的相似性极高,均高于90%。MYH-7b与已报道的翘嘴鳜慢肌MYH (KF601703.1) 相似性最高(97%),与大黄鱼(Larimichthys crocea) MYH-7转录剪接体1和剪接体2、布氏新亮丽鲷(Neolamprologus brichardi)、深裂眶锯雀鲷、斑马宫丽鱼和红丽鱼等的MYH-7的相似性均高于90%。进一步与其他物种的MYH-7a、MYH-7b氨基酸序列推断的头区进行同源比对,结果显示,在头区中都有与“马达”功能相关的关键结构域Loop1和Loop2环。这2个Loop环区上各有1个ATP结合位点,在氨基酸序列起始的175 aa (MYH-7b是176)和403 aa (MYH-7b是404) 处还各有1个ATP结合位点(图 2)。

|
图 2 翘嘴鳜与其他鱼类MYH-7s的头区保守氨基酸位点的比对 Figure 2 Amino acid sequence alignments of the myosin head region of MYH-7s of S. chuatsi and other fish species 灰色区为ATP结合位点;方框为P-loop环;方框上为loop环的保守序列;Sc-s:翘嘴鳜慢肌球蛋白重链亚型1,AHB33632.1;Dr-v:斑马鱼慢肌球蛋白亚型1,AAI63721.1;Sp-MYH7:深裂眶锯雀鲷MYH-7,XP_008303539. 1;On-MYH7:尼罗罗非鱼MYH-7,XP_013131789.1;Mz-MYH7:斑马宫丽鱼MYH-7,XP_012778396.1;Tr-MYH7:红鳍东方鲀MYH-7,XP_011614688.1 The gray regions represented ATP binding sites; the black box represented P-loop; the amino acids sequence above the box represented the conserved sequence of the P-loop; Sc-s: S. chuatsi slow skeletal muscle myosin heavy chain isoform 1, AHB33632.1; Dr-v: D. rerio slow myosin heavy chain 1, AAI63721.1; Sp-MYH7: S.partitus MYH-7, XP_008303539.1; On-MYH7: O. niloticus MYH-7, XP_013131789.1; Mz-MYH7: M. zebra MYH-7, XP_012778396.1; Tr-MYH7: T. rubripes MYH-7, XP_011614688.1 |
根据肌球蛋白重链头区氨基酸序列构建的系统进化树,形成快肌肌球蛋白重链组和慢肌(或心肌)肌球蛋白重链组两大分支。MYH-7a先与红鳍东方鲀的MYH-7聚类,MYH-7b则与鳜鱼、大黄鱼、短鳍花鳉(Poecilia mexicana)的慢肌球蛋白重链聚类,二者同归慢肌球蛋白重链分支(图 3)。

|
图 3 基于翘嘴鳜及其他物种MYH-7s头区序列的系统进化树 Figure 3 Phylogenetic tree based on head domain amino acids of myosin of S. chuatsi and other species 图中氨基酸序列的物种名和相应的登录号分别为(The species and GenBank accession numbers of amino acid sequences were listed as follows):翘嘴鳜慢肌MYH (S. chuasti slow MYH),KF601703.1;大黄鱼MYH-7 (L. crocea MYH-7),KKF11324.1;短鳍花鳉MYH-7 (P. mexicana MYH-7),XP_014864995.1;半滑舌鳎MYH-7 (C. semilaevis MYH-7),XP_008332930.1;墨西哥脂鲤MYH-7 (A. mexicanus MYH-7),XP_015456340.1;网纹鳉MYH-7 (P. reticulata MYH-7),XP_008431929.1;尼罗罗非鱼MYH-7 (O. niloticus MYH-7),XM_013276335.1;红鳍东方鲀MYH-7 (T. rubripes MYH-7),XM_011616386.1;鲤10℃、20℃、30℃适应型MYH (C. carpio 10℃, 20℃, 30℃ acclimated MYH),D89990、D89991、D89992;斑马鱼MYH2、4、7 (D. rerio MYH2, 4, 7),NM_152982、AY921650、XP_009292203.1;人MYH 1–4,6–8,13 (H. sapiens MYH 1–4, 6–8, 13),AF111785、AF111784、NM_002470.3、AF11783、NM_002471、NM_000257、NM_002474、AF111782 |
采用InterPro分析蛋白结构域发现,MYH-7a与MYH-7b主要结构域相似,均可分为头区和尾区。头区均有一种选择性结合多脯氨酸配体的结构域Src同源区3 (Src homology 3, SH3),以及MLC、钙调蛋白或EF手相蛋白超家族结合位点IQ基序(IQ motif)。
Predict Protein分析发现,MYH-7a和MYH-7b有相似的结构组成,均有77.2%的螺旋结构,5.0%的直链,17.8%的loop环(Loop),未发现二硫键形成位点。2个MYH-7s均在各自的178–185 aa处有8个氨基酸(GESGAGKT)为ATP/GTP结合活性位点A [ATP/ GTP-binding site motif A (P-loop)];942–971 aa处30个氨基酸(KKRKLEDECSELKKDIDDLELTLAKV-EKEK)为核定位序列(Nuclear localization sequence,NLS)。此外,MYH-7b还在其632–639 aa处一段8个氨基酸(GGKGKGGG)为核定位序列(图 4)。

|
图 4 2个翘嘴鳜MYH-7s氨基酸序列结构模式 Figure 4 Structure models of the two MYH-7s amino acid sequence of S. chuatsi A: MYH-7a; B: MYH-7b |
进一步用NLS Mapper在线预测与MYH-7a相似性较高的伯氏朴丽鱼(XP_005938260.1)、斑马宫丽鱼(XP_012778396.1)和尼罗罗非鱼(XP_013131789.1)等的MYH-7序列,以及与MYH-7b相似性较高的斑马宫丽鱼(XP_014266369.1)、非洲齿鲤(Nothobranchius furzeri)(XP_015812773.1)和斑马鱼(Danio rerio)(XP_ 009292203.1)等的MYH-7序列,均在其C端对应位置找到NLS序列。
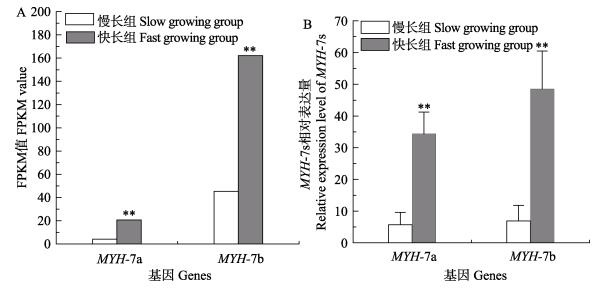
2.4 MYH-7s基因的表达量变化分析 2.4.1 翘嘴鳜MYH-7s的表达验证根据转录组数据计算MYH-7a、MYH-7b的FPKM值,在快长组2个基因的表达量均显著高于慢长组(P < 0.01)(图 5-A)。以用于转录组测序的同批cDNA为模板,qRT-PCR检测验证这2个基因的表达量差异,显示快长组表达量均显著高于慢长组(图 5-B)。qRT-PCR结果与转录组差异表达谱分析结果一致。
|
图 5 翘嘴鳜快长组和慢长组样本的MYH-7s表达量 Figure 5 Expression level of two MYH genes of S. chuatsi in fast and slow growing groups measured by transcriptome sequencing and qRT-PCR A:转录组测序;B:实时荧光定量PCR;**表示差异极显著(P < 0.01),下同 A: Transcriptome sequencing; B: qRT-PCR; **denoted highly significant difference (P < 0.01), the same as below |
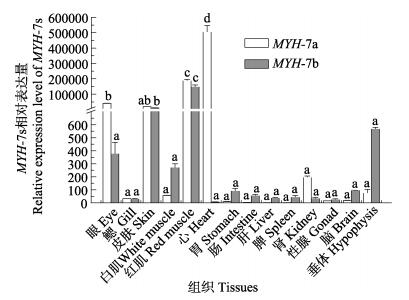
qRT-PCR分析2个MYH基因在翘嘴鳜成鱼14个组织中的表达,结果显示,MYH-7a在心肌中表达量最高,其次是红肌、眼和皮肤,在肝、脾、肠和胃中低表达;MYH-7b在红肌中表达量最高,其次是皮肤、垂体和眼,在鳃、性腺和心肌中低表达(图 6)。
|
图 6 qRT-PCR检测翘嘴鳜不同组织中MYH-7s的表达 Figure 6 qRT-PCR analysis of MYH-7s expression in tissues of adult S. chuatsi 同一基因中不同字母表示差异显著(P < 0.05),相同字母表示差异不显著(P > 0.05),下同 In the same gene expression feature, different letters represented significant difference (P < 0.05), the same letter showed no significant difference (P > 0.05), the same as below |
图 7显示在翘嘴鳜胚胎发育各时期2种MYH-7s的表达情况。MYH-7a在未受精卵中有微量表达,在尾芽期表达量明显上调,并随着孵化出膜,表达量不断增加。MYH-7b在未受精卵中检测到表达,从受精开始表达,随着胚胎发育整体呈上升趋势(差异均不显著)。

|
图 7 MYH-7s在翘嘴鳜各发育时期中的表达水平 Figure 7 The expression levels of MYH-7s in S. chuatsi at different developing stages |
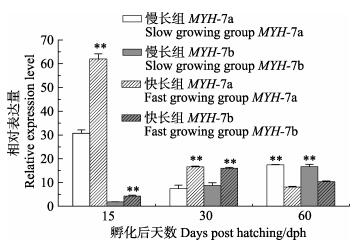
定量检测2个MYH-7s在孵化后15、30和60 dph快长组和慢长组的翘嘴鳜白肌中的表达,2个MYH-7s在15和30 dph快长组白肌中表达量均显著高于慢长组,而到60 dph时,快长组中的表达量均显著低于慢长组(图 8)。
|
图 8 孵化后不同生长时期的翘嘴鳜白肌中MYH-7s的表达 Figure 8 The expression pattern of MYH-7s in white muscle of S. chuatsi during different growth periods |
本研究克隆获得的翘嘴鳜MYH-7a、MYH-7b均具有MYH家族蛋白的典型结构:由头部和尾部组成,头部有ATP结合位点、Loop1和Loop2环区以及头部末端的IQ基序等关键结构。MYH主要由N端球状盘绕的头部和C端α-螺旋型的尾部组成。头区是Myosin动力产生的主要区域,具有ATP酶活性位点,可与ATP结合,水解ATP为ADP,释放能量,供头部迁移、滑动,是肌球蛋白“分子马达”功能的关键结构区,也是MYH家族中的重要保守区(Sebé-Pedrós et al, 2014);头区的2个功能性表面环状结构Loop1和Loop2,Loop1表面环结构靠近ATP结合位点,决定肌纤维收缩速率和动力持续;Loop2则可与肌动蛋白(actin)的非极性末端反应,影响MYH上由actin激活的ATP酶水解活性(Holmes et al, 2002);IQ基序是MYH与MLC以及钙调蛋白结合区,因其保守氨基酸IQxxxRGxxxR(x指任何一种氨基酸残基)的起始2个残基得名;Ca2+与MYH上IQ基序的结合可调节细胞定向运动(如肌细胞收缩),IQ基序也是MYH家族蛋白的重要特征结构之一(Bhler et al, 2002)。此外,在2个MYH-7s还发现SH3结构域和核定位序列等特殊结构。
SH3是一种选择性结合多脯氨酸配体的结构域,在多种生物进程中有重要作用,包括分子内相互作用调节酶活性、调节信号通路中某些成员的局部浓度和亚细胞定位以及介导一些大型多重复合蛋白的组装,在低等的酵母菌到人等物种的数百种蛋白中广泛存在(Mayer, 2001)。含SH3结构的MYH可通过SH3与含有Arp2/3复合体和富脯氨酸区的蛋白结合,通过其上的Arp2/3介导肌动蛋白丝的聚合,对细胞骨架维持和细胞的内吞作用等有重要作用(Okrut et al, 2015)。酵母的2个MYH1亚型:Myo3p和Myo5p的SH3结构域的缺失可导致肌动蛋白细胞骨架的严重缺陷(Evangelista et al, 2000);鸡(Gallus gallus)胸肌中一种MYH的SH3可促进肌ELC的N端延伸与肌动蛋白结合,调节肌纤维运动(Lowey et al, 2007)。SH3是只在MYH-1、2、4、7、15等亚型中发现的特征结构之一(Sebé-Pedrós et al, 2014)。本研究获得的2个翘嘴鳜MYH-7s均有SH3结构,其对细胞骨架维持和肌纤维运动等的功能尚待进一步研究。
核定位序列(NLS)作为蛋白质的一个结构域,与入核载体相互作用,使蛋白能被运进细胞核的特定位置发挥作用。已报道具有NLS的MYH有NM1(myosin ⅠC)、myosinⅥ、myosinⅩⅥB和myosinⅩⅧB (Sobczak et al, 2009)。本研究通过软件预测,在大多数鱼的MYH-7上也发现NLS。Dzijak等(2012) 发现,在NM1的第2个IQ基序中,13 aa的GRRKAAKRK-WAAQ(754–766) 是其必需的NLS,NM1可通过此结构进入细胞核,参与核内转录、染色质移动和染色质重塑等关键核运动。本研究所获得的2个MYH-7s的C端均有核定位序列,推测MYH-7a、MYH-7b蛋白也可能通过NLS入核参与核内运输与转录调控等过程,但具体作用还有待进一步研究验证。
3.2 翘嘴鳜MYH-7s基因的表达分析MYH具有组织和时期表达特异性。研究发现,MYH-7主要在哺乳动物和鱼的胚胎、幼鱼期表达,在成体则主要是心室肌和慢肌中表达;而快肌中受甲状腺激素信号通路的抑制,表达量低( van Rooij et al, 2009; Schiaffino et al, 2015)。Spurgeon等(2008) 检测发现,人的MYH-7主要在心肌和慢肌中表达,在肠、脑、前列腺和睾丸中有一定表达。Ikeda等(2007) 将红鳍东方鲀基因组DNA序列与人DNA序列比较发现,红鳍东方鲀与人同源的MYH-7主要在慢肌、心肌和皮肤中高表达,此外也在眼、胚胎(168 hours post fertilization, hpf)和快肌中有表达。而Akolkar等(2010) 在红鳍东方鲀成鱼快肌中未检测到MYH-7的表达,在外侧浅肌和深层的竖肌与降肌2种慢肌中有表达,在心肌中高表达。本研究中,MYH-7a、MYH-7b在心肌、红肌等部位高表达,与上述MYH-7的组织分布情况相符。
Spurgeon等(2008) 发现,MYH-7在人胎儿期的肝脏和肌肉中均有表达,且肌肉中表达量显著高于肝脏。在大鼠(Rattus norvegicus)和小鼠(Mus musculus)胚胎发育中主要表达胚胎型的MYH-3、神经型的MYH-8和慢肌型的MYH-7 (Schiaffino et al, 2015)。在小鼠胚胎发育中,其MYH-7a在受精后第9天的胚胎期即见表达;鸡的MYH-7则最早出现在神经分化早期(Schiaffino et al, 2015; Nguyen et al, 2015)。用qRT-PCR检测,红鳍东方鲀4–8 dph的胚胎发育期均可见MYH-7表达,幼鱼期的8–16 dph也有表达(Asaduzzaman et al, 2013)。在鲤中,心肌型MYH (也归为MYH-7) 最早在61 hph检测到表达,且随着胚胎发育表达量逐渐升高(Nihei et al, 2006)。本研究中,MYH-7a、MYH-7b在未受精卵即有表达,且随着胚胎发育,表达量呈上升趋势,提示2种MYH-7在翘嘴鳜胚胎发育过程具有重要作用。
MYH基因从早期的慢型逐渐被快型MYH替代,与动物早期生长有很大相关性。在大鼠早期发育中,在出生几天后才能在腿部骨骼肌中检测到成体高表达的快肌型MYH (MYH-1、2和4),而在后续的几周内,快肌型MYH大量表达并迅速替代胚胎型和慢型MYH(MYH-3、7) (Schiaffino et al, 2015)。在小鼠C2C12成肌细胞系,用低血清浓度诱导细胞分化,首先表达MYH-7、MYH-3和MYH-8,3种MYH在第1天即见表达,到2–4 d达到峰值,之后降低;而快肌型的MYH-1和MYH-4在2–4 d才检测到表达,但表达量不断增加(Brown et al, 2012)。鼠机体和细胞中这种MYH由慢型到快型的表达转变受甲状腺激素的调节(Soukup et al, 2015a)。在鱼类中,特别是幼鱼生长阶段,MYH快慢亚型表达量变化可能与肌肉类型在白肌(快肌)、红肌(慢肌)间的转变有关,在斑马鱼、河鲈(Perca fluviatilis)幼鱼中均有发现(McGuigan et al, 2004)。Soukup等(2015b)认为,慢肌球蛋白在鱼胚胎期和幼鱼期表达量较高,而随着鱼类生长,身体其他部位的慢型MYH表达量逐渐降低,被快型MYH取代,而慢型MYH在成体慢肌存在的部位如心脏、红肌表达量持续增加。在孵化后1个月和7个月的鲤躯干肌肉中也能检测到MYH-7表达,但7个月时的表达量低于1个月的(Nihei et al, 2006)。本研究结果显示,MYH-7a、MYH-7b在孵化后30–60 dph的生长时期,在白肌中表达量有明显的降低,与鱼类中肌纤维由慢型向快型转变相符,在对应时期中慢长组MYH-7表达量高于快长组,说明2个MYH-7表达水平的变化与鱼生长进程以及生长速度有极大的相关性。
4 结论本研究从翘嘴鳜快长组和慢长组的转录组差异表达基因中筛选到2个MYH Unigenes (MYH-7a, MYH-7b),进行了cDNA序列克隆、结构分析以及qRT-PCR表达水平分析。这2个基因具有不同的组织分布,在胚胎发育过程早期均有表达且随着发育的推进表达水平明显提高,MYH-7a的表达量明显高于MYH-7b;在不同生长阶段的快长组与慢长组翘嘴鳜白肌中,2个基因的表达量在15 dph和30 dph快长组均显著高于慢长组,而60 dph快长组显著低于慢长组。研究结果显示,这2个基因在翘嘴鳜早期发育过程中发挥着重要的作用,其表达量高低与翘嘴鳜生长快慢相关。
| Akolkar DB, Kinoshita S, Yasmin L, et al. Fibre type-specific expression patterns of myosin heavy chain genes in adult torafugu Takifugu rubripes muscles. Journal of Experimental Biology, 2010, 213(1): 137-145 DOI:10.1242/jeb.030759 | |
| Asaduzzaman M, Ahammad AKS, Asakawa S, et al. 5'-flanking sequences of zebrafish fast myosin heavy chain genes regulate unique expression in the anterior, medial subsection and posterior tail somites of the skeletal muscle. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2016, 191: 1-12 DOI:10.1016/j.cbpb.2015.08.009 | |
| Asaduzzaman MD, Akolkar DB, Kinoshita S, et al. The expression of multiple myosin heavy chain genes during skeletal muscle development of torafugu Takifugu rubripes embryos and larvae. Gene, 2013, 515(1): 144-154 DOI:10.1016/j.gene.2012.10.089 | |
| B hler M, Rhoads A. Calmodulin signaling via the IQ motif. FEBS Letters, 2002, 513(1): 107-113 DOI:10.1016/S0014-5793(01)03239-2 | |
| Biga PR, Goetz FW. Zebrafish and giant danio as models for muscle growth: Determinate vs indeterminate growth as determined by morphometric analysis. American Journal of Physiology-Regulatory, Integrative and Comparative Physiology, 2006, 291(5): R1327-R1337 DOI:10.1152/ajpregu.00905.2005 | |
| Brown DM, Parr T, Brameld JM. Myosin heavy chain mRNA isoforms are expressed in two distinct cohorts during C2C12 myogenesis. Journal of Muscle Research and Cell Motility, 2012, 32(6): 383-390 DOI:10.1007/s10974-011-9267-4 | |
| Campion LA, Choi S, Mistry HL, et al. Myosin heavy chain and parvalbumin expression in swimming and feeding muscles of centrarchid fishes: The molecular basis of the scaling of contractile properties. Comparative Biochemistry and Physiology Part A: Molecular and Integrative Physiology, 2012, 163(2): 223-230 DOI:10.1016/j.cbpa.2012.06.001 | |
| Churova MV, Meshcheryakova OV, Veselov AE, et al. Activity of enzymes involved in the energy and carbohydrate metabolism and the level of some molecular-genetic characteristics in young salmons (Salmo salar L. ) with different age and weight. Russian Journal of Developmental Biology, 2015, 46(5): 254-262 | |
| Dhillon RS, Wang Y, Tufts BL. Using molecular tools to assess muscle growth in fish: Applications for aquaculture and fisheries management. Comparative Biochemistry and Physiology Part C: Toxicology and Pharmacology, 2008, 148(4): 452 | |
| Dzijak R, Yildirim S, Kahle M, et al. Specific nuclear localizing sequence directs two myosin isoforms to the cell nucleus in calmodulin-sensitive manner. PLoS One, 2012, 7(1): e30529 DOI:10.1371/journal.pone.0030529 | |
| van Rooij R, Quiat D, Johnson BA, et al. A family of microRNAs encoded by myosin genes governs myosin expression and muscle performance. Developmental Cell, 2009, 17(5): 662-673 DOI:10.1016/j.devcel.2009.10.013 | |
| Evangelista M, Klebl BM, Tong AHY, et al. A role for myosin-Ⅰ in actin assembly through interactions with Vrp1p, Bee1p, and the Arp2/3 complex. Journal of Cell Biology, 2000, 148(2): 353-362 DOI:10.1083/jcb.148.2.353 | |
| Hevr y EM, Jordal AEO, Hordvik I, et al. Myosin heavy chain mRNA expression correlates higher with muscle protein accretion than growth in Atlantic salmon, Salmo salar. Aquaculture, 2006, 252(2-4): 453-461 DOI:10.1016/j.aquaculture.2005.07.003 | |
| Holmes JM, Whiteley NM, Magnay JL, et al. Comparison of the variable loop regions of myosin heavy chain genes from Antarctic and temperate isopods. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2002, 131(3): 349-359 DOI:10.1016/S1096-4959(01)00509-7 | |
| Ikeda D, Ono Y, Snell P, et al. Divergent evolution of the myosin heavy chain gene family in fish and tetrapods: Evidence from comparative genomic analysis. Physiological Genomics, 2007, 32(1): 1-15 DOI:10.1152/physiolgenomics.00278.2006 | |
| Liu XL, Bin SY, Wang KZ, et al. Artificial propagation and embryonic development observation of mandarin fish. Journal of Guangxi Normal University: Natural Science Edition, 2013, 31(2): 100-106[刘希良, 宾石玉, 王开卓, 等. 翘嘴鳜的人工繁殖与胚胎发育观察. 广西师范大学学报:自然科学版, 2013, 31(2): 100-106] | |
| Lowey S, Saraswat LD, Liu HJ, et al. Evidence for an interaction between the SH3 domain and the N-terminal extension of the essential light chain in class Ⅱ myosins. Journal of Molecular Biology, 2007, 371(4): 902-913 DOI:10.1016/j.jmb.2007.05.080 | |
| Mayer BJ. SH3 domains: Complexity in moderation. Journal of Cell Science, 2001, 114(7): 1253-1263 | |
| McGuigan K, Phillips PC, Postlethwait JH. Evolution of sarcomeric myosin heavy chain genes: Evidence from fish. Molecular Biology and Evolution, 2004, 21(6): 1042-1056 DOI:10.1093/molbev/msh103 | |
| Mortazavi A, Williams BA, McCue K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods, 2008, 5(7): 621-628 DOI:10.1038/nmeth.1226 | |
| Nguyen K, Hall AL, Jones J M. Expression of myosin VIIA in the developing chick inner ear neurons. Gene Expression Patterns, 2015, 19(1): 36-44 | |
| Nihei Y, Kobiyama A, Ikeda D, et al. Molecular cloning and mRNA expression analysis of carp embryonic, slow and cardiac myosin heavy chain isoforms. Journal of Experimental Biology, 2006, 209(1): 188-198 DOI:10.1242/jeb.01978 | |
| Okrut J, Prakash S, Wu Q, et al. Allosteric N-WASP activation by an inter-SH3 domain linker in Nck. Proceedings of the National Academy of Sciences, 2015, 112(47): E6436-E6445 DOI:10.1073/pnas.1510876112 | |
| Overturf K, Hardy RW. Myosin expression levels in trout muscle: A new method for monitoring specific growth rates for rainbow trout Oncorhynchus mykiss (Walbaum) on varied planes of nutrition. Aquaculture Research, 2001, 32(4): 315-322 DOI:10.1046/j.1365-2109.2001.00582.x | |
| Schiaffino S, Rossi AC, Smerdu V, et al. Developmental myosins: Expression patterns and functional significance. Skeletal Muscle, 2015, 5(1): 1-14 DOI:10.1186/s13395-014-0025-3 | |
| Sebé-Pedrós A, Grau-Bové X, Richards TA, et al. Evolution and classification of myosins, a paneukaryotic whole-genome approach. Genome Biology and Evolution, 2014, 6(2): 290-305 DOI:10.1093/gbe/evu013 | |
| Sobczak M, Majewski Ł, Redowicz MJ. Myosins in nucleus. Postepy Biochemii, 2009, 55(2): 239-246 | |
| Soukup T, Diallo M. Proportions of myosin heavy chain mRNAs, protein isoforms and fiber types in the slow and fast skeletal muscles are maintained after alterations of thyroid status in rats. Physiological Research, 2015a, 64(1): 111-118 | |
| Soukup T, Smerdu V. Effect of altered innervation and thyroid hormones on myosin heavy chain expression and fiber type transitions: A mini-review. Histochemistry and Cell Biology, 2015b, 143(2): 123-130 DOI:10.1007/s00418-014-1276-0 | |
| Spurgeon SL, Jones RC, Ramakrishnan R. High throughput gene expression measurement with real time PCR in a microfluidic dynamic array. PLoS One, 2008, 3(2): e1662 DOI:10.1371/journal.pone.0001662 | |
| Yasmin L, Kinoshita S, Asaduzzaman M, et al. A 5x-flanking region of embryonic-type myosin heavy chain gene, MYHM743-2, from torafugu Takifugu rubripes regulates developmental muscle-specific expression. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2011, 6(1): 76-81 DOI:10.1016/j.cbd.2010.05.002 | |
| Zhou RX, Meng T, Meng HB, et al. Selection of reference genes in transcription analysis of gene expression of the mandarin fish, Siniperca chuasti. Zoological Research, 2010, 31(2): 141-146 DOI:10.3724/SP.J.1141.2010.02141 |



