2. 农业部海洋渔业可持续发展重点实验室 山东省渔业资源与生态环境重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071
2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture, Shandong Provincial Key Laboratory of Fishery Resources and Eco-environment, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071
Notch途径是一条进化上十分保守的信号通路,通过受体Notch与配体DSL蛋白的相互作用转导信号,从而调控细胞增殖、分化、凋亡并决定细胞的命运(Lai, 2004; 梁洁等, 2008)。作为Notch信号途径的重要起始蛋白,Notch基因最初在研究果蝇(Drosophila melanogaster)胚胎发育过程中被识别,它的突变导致果蝇翅膀边缘出现缺口,因此被命名为Notch (Mohr, 1919、1922)。随后,果蝇的Notch基因被克隆出来(Allman et al, 2002)。目前,已有Notch的同源蛋白从线虫(Caenorhabditis elegans)、爪蟾(Xenopus laevis)、斑马鱼(Barchydanio rerio var)、鸡(Gallus gallus)、小鼠(Mus musculus)和人(Homo sapiens)等生物中被分离出来(齐润姿等, 2002; Anant et al, 1998)。研究表明,从低等动物到高等动物,Notch基因家族在结构上都具有高度的保守性,均是单链跨膜蛋白,由胞外区、跨膜区和胞内区组成,胞外区均具有多个EGF-like重复结构域、3个NL结构域及1个NOD结构域和1个NODP结构域,而胞内区都具有6个ANK结构域和2个核定位信号(NSL)(李宝园等, 2009; Kortschak et al, 2001; Maine et al, 1995)。在功能上,Notch家族成员也具有高度的相似性,Notch不但参与了骨骼、神经和体节形成等多个发育过程(Zanotti et al, 2010; de Oliveira-Carlos et al, 2013; Artavanis-Tsakonas et al, 1999),而且还调控个体胚胎发育过程中胚层细胞的增殖、分化和凋亡(Coffman et al, 1993; Conboy et al, 1995; Fortini et al, 1993),影响器官形成和形态发生等(郭政等, 2008; 鲁茁壮等, 2004)。
Notch信号途径在高等动物中已经被广泛研究,但在刺胞动物(Cnidaria)中的研究很少,仅在水螅和海葵(Nematostella vectensis)中有相关报道(Bottger et al, 2002; Kasbauer et al, 2007; Marlow et al, 2012)。刺胞动物作为最原始的真后生动物,只有外胚层和内胚层的分化,是其他高等多细胞动物的起点(周春娅等, 2013)。因此,研究刺胞动物Notch信号途径,解析Notch基因的分子结构和不同发育过程的表达特点,将为了解Notch信号途径的起源和功能演化等研究提供重要参考。
海蜇(Rhopilema esculentum)隶属于刺胞动物门、钵水母纲、根口水母目、海蜇属,其人工繁育和增养殖技术成熟,是研究刺胞动物无性繁殖调控途径的理想模式生物。本研究基于转录组454 GS FLX测序结果,利用RACE和RT-PCR技术,克隆了海蜇Notch基因的cDNA全长,并分析了其mRNA在海蜇无性繁殖不同发育阶段的表达差异,这些研究结果将为进一步研究Notch信号通路在海蜇无性繁殖中的调控作用奠定了基础。
1 材料与方法 1.1 实验动物野生性成熟海蜇于2015年9月采于浙江杭州湾,运回山东荣成海蜇养殖场进行人工繁育,以获取海蜇螅状体(Scyphistoma)、横裂体(Strobila)、碟状体(Ephyra)和幼蛰样品。
1.2 海蜇转录组454 GS FLX测序与EST分析海蜇转录组文库构建、454 GS FLX测序见周春娅等(2013)方法。利用生物信息学方法检索海蜇转录组文库,寻找与已知Notch基因同源的EST序列。
1.3 Notch基因cDNA的全长克隆Blast分析表明,海蜇EST(isotig11938)与海葵Notch基因具有高度的相似性,进一步分析发现,这段序列具有Notch基因特有的3个NL结构域、1个NOD结构域和1个NODP结构域。根据EST(isotig11938)序列,设计特异性引物扩增海蜇Notch基因cDNA全长(表 1)。Notch基因的3´末端利用pBluescript SK(+/-)载体上的通用引物T7与特异性引物Notch-F2扩增;Notch基因的5´末端用载体通用引物T3与特异性引物Notch-R2扩增。25 ml的PCR反应体系:2.5 ml 10×PCR Buffer,2 ml dNTP (25 mmol/L),1.5 ml MgCl2 (25 mmol/L),1 ml引物(10 mmol/L),0.2 ml (1U)Taq酶,1 ml cDNA模板,15.8 ml PCR水。扩增产物用琼脂糖凝胶电泳进行分离检测,切胶回收后连接到pMD18-T (TaKaRa)载体,然后转化至大肠杆菌感受态细胞Top10中,通过蓝白斑筛选阳性克隆并送到上海英潍捷基测序。
|
|
表 1 本研究所用引物序列 Table 1 Primer sequences used in the study |
用Bioedit软件对所获得的测序结果序列进行全长拼接;用BLAST(http://www.ncbi.nlm.nih.gov/blast)和蛋白分析系统(http://www.expasy.org/)分析海蜇Notch的cDNA和氨基酸序列。用SMART(http://smart.emblheidelberg.de/)及SingalP 4.1信号肽预测软件(http://www.cbs.dtu.dk/services/SignalP/)对海蜇Notch进行蛋白结构域分析及信号肽预测。利用ClustalX1.83将海蜇Notch与其他物种Notch氨基酸序列进行多序列比对。根据多序列比对结果,用MEGA5.0软件采用邻接法(Neighbor-joining)构建Notch的系统进化树。
1.5 荧光实时定量PCR利用Trizol法分别提取海蜇4个不同发育阶段:螅状体、横裂体、碟状体、水母体(Medusa)的总RNA,每个发育时期15个个体为1组,每组设3个重复。然后分别反转录合成cDNA,反应体系及反应条件按说明书要求操作(Invitrogen)。
PCR反应在ABI 7500 Real-time PCR system (Applied Biosystems)上进行,20 μl反应体系:10 μl SYBR® Premix Ex Taq™Ⅱ,0.4 μl ROX Reference DyeⅡ,0.4 μl特异性引物(Notch-F1和Notch-R1见表 1),2 μl cDNA模板。PCR反应程序如下:95℃预变性30 s,95℃ 5 s、60℃ 34 s,共40个循环。样品和内参分别设3个重复。采用β-actin基因作为实时定量PCR的内参基因。反应结束后的分解步骤(95℃ 15 s,60℃ 1 min,95℃ 15 s)用于绘制熔解曲线,确认Notch基因引物的特异性,采用2-DDCt法进行数据分析。获得的数据采用SPSS 18.0统计软件进行处理,各组间平均数比较采用单因素方差分析(One-way ANOVA),P < 0.05为差异显著,P < 0.01为差异极显著。
2 结果与分析 2.1 海蜇Notch信号通路中Notch基因的克隆和序列分析将3´-RACE和5´-RACE获得的序列与EST(isotig11938)序列进行拼接,获得海蜇Notch基因的cDNA全长序列。海蜇Notch基因的cDNA全长为6768 bp,包括5´非翻译区(5´-UTR) 90 bp,包含1个多腺苷酸Poly(A)尾和AATAAA加尾信号,3´非翻译区(3´-UTR) 612 bp,开放阅读框(Open reading frame, ORF) 6066 bp编码了2021个氨基酸。预测其蛋白分子量和理论等电点分别为218.92 kDa和6.24。
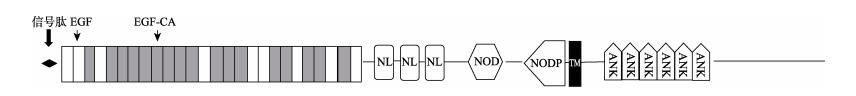
通过SignalP、SMART、PredictProtein 2013和NetNGlyc 1.0 Server等软件分析海蜇Notch的氨基酸序列显示,Notch为分泌蛋白,信号肽由21个氨基酸组成(图 1),酶切后成熟肽的分子量和理论等电点分别为216.55 kDa和6.25。在成熟肽中共有37个结构域(图 2),包括26个表皮生长因子样(EGF-like)重复结构域、3个富含半胱氨酸的Notch/Lin-12(NL)结构域、1个跨膜结构域、1个NOD结构域、1个NODP结构域和6个锚蛋白ANK结构域,还含有25个潜在的N-糖基化位点,其中,NL、NOD和NODP结构域是Notch家族的特有结构域。

|
图 1 海蜇Notch全长cDNA序列及推导的氨基酸序列 Figure 1 Full-length Notch cDNA of R. esculentum and its deduced amino acid sequences 粗体为信号肽;箭头为酶切位点;灰色阴影为Notch结构域EGF-like;双下划线为结构域EGF-CA;下划波浪线为结构域NL;下划点状线为结构域Pfam:NOD;下划为结构域Pfam:NODP;粗体并双下划线为跨膜结构域;下划实线为结构域ANK;方框为多聚腺苷酸加尾信号;*为终止密码子 Black bold indicated signal peptied; Arrow indicated cleavage sites; EGF was printed with grey background; EGF-CA was lined with double underline; NL was underlined with wavy line; Pfam: NODP was underlined with long point line; Pfam: NOD was underlined with dotted line; Transmembrane domain was black bold and underlined with double underline; ANK was under lined with single underline; Poly (A) signals was surrounded by box; The pentagrams indicated stop codon |
|
图 2 预测的海蜇Notch主要结构域 Figure 2 Predicted Notch domains of R.esculentum |
多序列比对结果显示(表 2),在全序列上,海蜇Notch与来自刺胞动物门的海葵(AEW42991) Notch氨基酸全序列相似性为39%,与佛罗里达弓背蚁(Camponotus floridanus)、意大利蜜蜂(Apis mellifera)、蝇蛹金小蜂(Nasonia vitripennis)、囊舌虫(Saccoglossus kowalevskii)等无脊椎动物的氨基酸相似度为35%–37%,而与哺乳动物、两栖动物、鱼类等脊椎动物的氨基酸相似性为34%–38%。NL、NOD和NODP结构域是Notch家族特有的结构,对这3个结构域多序列比对结果显示,海蜇Notch的NL、NOD和NODP结构域与刺胞动物、无脊椎动物、脊椎动物的氨基酸相似性分别为48%、40%–46%、42%–47%;25%、25%–28%、23%–30%;37%、22%–31%、27%–31%(图 3)。
|
|
表 2 海蜇Notch与其他物种的Notch的氨基酸序列一致性 Table 2 Comparison of Notch amino acid identity between R. esculentum Notch and vertebrates/invertebrates |

|
图 3 海蜇与其他物种Notch NOD结构域的氨基酸多序列比对 Figure 3 Multiple sequence alignment NOD domain of Notch of R. esculentum and other species |
对海蜇Notch基因的3个NL结构域进行多序列比对表明,不同物种之间其氨基酸序列保守性很强,且每个NL结构域含有6个半胱氨酸(图 4)。而NOD和NODP 2个结构域的多序列比对表明,海蜇NOD结构域在甘氨酸(Gly)、苯丙氨酸(Phe)、精氨酸(Arg)、赖氨酸(Lys)、苏氨酸(Thr) 5个氨基酸位点保持一致(图 3),NODP结构域在天冬氨酸(Asp)、天冬酰胺(Asn)、2个半胱氨酸(Cys)、苯丙氨酸(Phe)、丙氨酸(Ala)、丙氨酸(Ala) 7个氨基酸位点也保持一致(图 5)。

|
图 4 海蜇与其他物种Notch NL结构域的氨基酸多序列比对 Figure 4 Multiple sequence alignment NL domain of Notch of R. esculentum and other species |

|
图 5 海蜇与其他物种Notch NODP结构域的氨基酸多序列比对 Figure 5 Multiple sequence alignment NODP domain of Notch of R. esculentum and other species 黑色为相同氨基酸;灰色为相似氨基酸;黑色圆点为NL结构域完全一致的半胱氨酸;*为NOD和NODP结构域完全一致氨基酸 Black background indicated the same amino acid; Gray background indicated similar amino acids, the black dots showed the identical cysteine; The pentagrams showed the same amino acid of NOD and NODP |
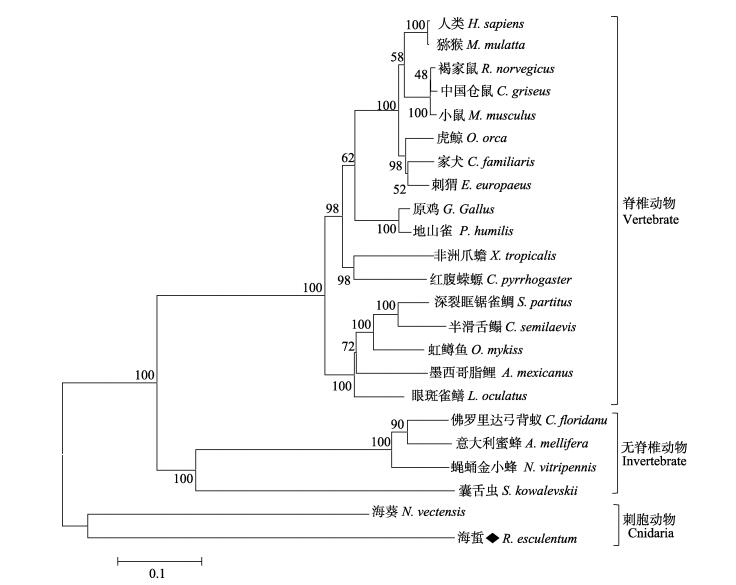
采用邻接法构建Notch的系统进化树(图 6)显示,海蜇与来自刺胞动物门海葵的Notch亲缘关系最近,聚为一支,而佛罗里达弓背蚁、意大利蜜蜂、蝇蛹金小蜂、囊舌虫等非脊椎动物聚为一支,人、原鸡、虹鳟鱼(Oncorhynchus mykiss)等众多脊椎动物又聚为一支。
|
图 6 基于邻接法的Notch氨基酸序列的系统进化树 Figure 6 Phylogenetic tree of Notch based on neighbor-joining method |
利用RT-PCR技术分析了Notch基因在海蜇螅状体、横裂体、蝶状体、水母体4个不同发育时期的表达差异。结果显示,该基因在海蜇无性繁殖4个发育时期均有表达,螅状体阶段Notch的表达量最高,然后是水母体和碟状体(图 7),这3个发育阶段的表达量分别是是横裂体阶段表达量的1.85、1.60、1.33倍,而横裂体阶段Notch的表达量最低。

|
图 7 Notch基因在海蜇不同发育阶段的表达 Figure 7 Expression of Notch mRNA at different developmental stages of R. esculentum |
本研究基于转录组454 GS FLX测序和EST序列分析,利用RACE和RT-PCR技术,解析了海蜇Notch基因结构,发现海蜇Notch基因与其他物种的Notch基因一样都为分泌蛋白,并具有Notch家族特有的NOD、NODP结构域和3个NL结构域,此外,还包括EGF-like结构域、ANK重复结构域和跨膜结
构域(周庆军等, 2004)。Blast分析表明(表 2),不管是全序列还是Notch基因特有结构域相似性分析,海蜇Notch基因与刺胞动物、无脊椎动物和脊椎动物都具有较高的同源性。系统进化树分析也显示,海蜇与来自刺胞动物门海葵的Notch聚为一支,而来自无脊椎动物和脊椎动物的Notch基因分别聚为2个分支,说明Notch基因符合生物进化规律,按照从低等动物到高等动物分别聚类。
Notch广泛存在于各种生物中,是一种单次跨膜蛋白,均由胞外区(NEC)、跨膜区(TM)和胞内区(NICD)3大部分组成(付亚娟等, 2007)。典型的Notch结构域胞外区包括36个的EGF-like结构域、3个NL结构域、NOD和NODP结构域,胞内区主要包括6个ANK结构域以及ANK两侧的2个核定位信号(NSL)(Hori et al, 1997)。研究表明,不同物种之间EGF-like结构域数量为10-36不等,差别明显。在脊椎动物和双翅目动物的Notch基因中EGF-like结构域一般为36个,而在无脊椎动物中,EGF-like结构域数量普遍少于36个,比如蝇蛹金小蜂中有31个(Kortschak et al, 2001),而在本研究中,海蜇Notch的EGF-like结构域数量更少,只有26个,其原因可能是低等动物Notch基因在进化过程中为了适应环境对EGF-like结构域进行了必要的删除,类似的现象也出现在海鞘(Pyrosomella verticilliata) Notch基因的研究中(Hori et al, 1997)。富含Cys的NL(Notch/Lin-12) 结构域是所有Notch基因特有结构域之一。海蜇
Notch基因与其他物种的Notch一样也含有3个NL结构域,每个结构域都由40个左右的氨基酸组成,并含有6个可以形成3对高度保守的二硫键的Cys。多序列比对也发现,从刺胞动物到哺乳动物,NL结构域的氨基酸序列相似性高达48%–83%。这种高度的序列相似性和结构的保守性表明,从低等动物到高等动物,NL结构域的功能相似,都是通过与配体特异性结合、启动Notch蛋白发挥作用(Kortschak et al, 2001; 李文等, 2014)。
无性繁殖是刺胞动物重要的繁殖方式,其调控途径和分子机制的研究已经逐步开展(Trevino et al, 2011)。出芽生殖是水螅的无性繁殖方式之一,Philipp等(2009)研究表明,Wnt信号途径可以启动并调控出芽生殖过程。在水螅的出芽生殖中,Wnt5、Wnt8、Frizzled 2和Dsh基因的表达被局限于水螅出芽部位和触手上,并随着芽体的增长,这些基因的表达量逐步升高。而横裂生殖是海蜇无性繁殖的主要方式,即螅状体经过分节和变态2个紧密联系的发育阶段产生稚水母的过程(周春娅等, 2013)。本实验室的前期研究也表明,Wnt信号途径的几个关键节点基因家族,包括Wnt基因家族、Frizzled基因家族等都参与调控了海蜇的无性繁殖过程。这些基因被局限表达海蜇触手和横裂生殖的分节部位,并且无性繁殖过程中,横裂体阶段的表达量显著高于螅状体、碟状体及其水母体阶段(周春娅等, 2013)。而在本研究中,Notch基因在海蜇无性繁殖过程的表达模式与Wnt等基因正好相反。虽然,Notch基因在海蜇无性繁殖4个发育阶段均有表达,但横裂体表达量最低,而螅状体表达量最高,并随着横裂结束表达量逐步升高,这一结果可能与Notch信号途径在刺胞动物无性繁殖过程中的特殊调控作用相关。Münder等(2010)研究表明,Notch基因的表达是水螅芽体与母体之间形成界限的必需条件,在芽体与母体分离时,Notch通路的主要靶基因-hairy/Enhancer of Split基因在分离区开始表达,进而导致芽体与母体分离。由此可以推断,Wnt信号途径和Notch信号途径在刺胞动物无性繁殖过程中,均发挥重要的调控作用,但其调控机制和模式是不同的,进一步的调控机理值得更深入探讨。
| Allman D, Aster JC, Pear WS. Notch signaling in hematopoiesis and early lymphocyte development. Immunological Reviews, 2002, 187(1): 75-86 DOI:10.1034/j.1600-065X.2002.18707.x | |
| Anant S, Roy S, Raghavan KV. Twist and Notch negatively regulate adult muscle differentiation in Drosophila. Development, 1998, 125(8): 1361-1369 | |
| Artavanis-Tsakonas S, Rand MD, Lake RJ. Notch signaling: Cell fate control and signal integration in development. Science, 1999, 284(5415): 770-776 DOI:10.1126/science.284.5415.770 | |
| Bottger A, Alexandrova O, Cikala M, et al. GFP expression in Hydra: Lessons from the particle gun. Development Genes and Evolution, 2002, 212(6): 302-305 DOI:10.1007/s00427-002-0245-0 | |
| Coffman CR, Skoglund P, Harris WA, et al. Expression of an extracellular deletion of Notch diverts cell fate in Xenopus embryos. Cell, 1993, 73(4): 659-671 DOI:10.1016/0092-8674(93)90247-N | |
| Conboy IM, Conboy MJ, Wagers AT, et al. Rejuvenation of aged progenitor cells by exposure to a young systemic environment. Nature, 2005, 433(7027): 760-764 DOI:10.1038/nature03260 | |
| de Oliveira-Carlos V, Ganz J, Hans S, et al. Notch receptor expression in neurogenic regions of the adult zebrafish brain. PLoS One, 2013, 8(9): 1-21 | |
| Fortini ME, Rebay I, Caron LA, et al. An activated Notch receptor blocks cell-fate commitment in the developing Drosophila eye. Nature, 1993, 365(6446): 555-557 DOI:10.1038/365555a0 | |
| Fu YJ, YE F, Lv WG, et al. Recent researches on the Notch signaling pathway. Journal of Medical Molecular Biology., 2007, 4(5): 447-450 [付亚娟, 叶枫, 吕卫国, 等. Notch信号通路的研究现状. 医学分子生物学杂志, 2007, 4(5): 447-450] | |
| Guo Z, Huang J. Notch signaling pathway and Neuralized protein. Chinese Journal of Biochemistry and Molecular Biology, 2008, 24(11): 987-991 [郭政, 黄瑾. Notch信号通路与Neuralized蛋白. 中国生物化学与分子生物学报, 2008, 24(11): 987-991] | |
| Hori S, Saitoh T, Matsumoto M, et al. Notch homologue from Halocynthia roretzi is preferentially expressed in the central nervous system during ascidian embryogenesis. Development Genes and Evolution, 1997, 207(6): 371-380 DOI:10.1007/s004270050126 | |
| Kasbauer T, Towb P, Alexandrova, Alexandrova O, et al. The Notch signaling pathway in the cnidarian Hydra. Developmental Biology, 2007, 303(1): 376-390 DOI:10.1016/j.ydbio.2006.11.022 | |
| Kortschak DR, Tamme R, Lardelli M. Evolutionary analysis of vertebrate Notch genes. Development Genes and Evolution, 2001, 211(7): 350-354 DOI:10.1007/s004270100159 | |
| Lai EC. Notch signaling: Control of cell communication and cell fate. Development, 2004, 131(5): 965-973 DOI:10.1242/dev.01074 | |
| Li BY, Feng L, Wang YC, et al. Molecular composition, activation pathways and activity of Notch signaling Molecules in cell differentiation. Chinese Journal of Neuro medicine, 2009, 8(6): 633-636 [李宝园, 丰玲, 王雁春, 等. Notch信号转导相关分子的构成、相关机制与调节. 中华神经医学杂志, 2009, 8(6): 633-636] | |
| Li W, Li CS. VEGF-Notch signaling pathway and angiogenesis. Shandong Medical Journal, 2014, 54(13): 83-85 DOI:10.3969/j.issn.1002-266X.2014.13.037 [李文, 李长生. VEGF-Notch信号通路与血管再生的关系. 山东医药, 2014, 54(13): 83-85] | |
| Liang J, Han H. Notch signaling pathway and vascular development. Chinese Journal of Cellular and Molecular Immunology, 2008, 24(12): 1225-1226 DOI:10.3321/j.issn:1007-8738.2008.12.033 [梁洁, 韩骅. Notch信号通路与血管发育. 细胞与分子免疫学杂志, 2008, 24(12): 1225-1226] | |
| Lu ZZ, Wang LS, Wu ZZ. Progress of Notch signaling. Progress in Physiological Sciences, 2004, 35(2): 135-138 [鲁茁壮, 王立生, 吴祖泽. Notch信号通路研究进展. 生理科学进展, 2004, 35(2): 135-138] | |
| Maine EM, Lissemore JL, Starmer WT. A phylogenetic analysis of vertebrate and invertebrate Notch-related genes. Molecular Phylogenetics and Evolution, 1995, 4(2): 139-149 DOI:10.1006/mpev.1995.1014 | |
| Marlow H, Roettinger E, Boekhout M, et al. Functional roles of Notch signaling in the cnidarian Nematostella vectensis. Developmental Biology, 2012, 362(2): 295-308 DOI:10.1016/j.ydbio.2011.11.012 | |
| Mohr OL. Cases of mimic mutations and secondary mutations in the X-chromosome of Drosophila melanogaster. Molecular Genetics and Genomics, 1922, 1(28): 1-22 | |
| Mohr OL. Character change caused by mutation of an entire region of a chromosome in Drosophila. Genetics, 1919, 3(4): 275-282 | |
| Münder S, Kasbauer T, Prexl A, et al. Notch signalling defines critical boundary during budding in Hydra. Developmental Biology, 2010, 344(1): 331-345 DOI:10.1016/j.ydbio.2010.05.517 | |
| Philipp I, Aufschnaiter R, Ozbek S, et al. Wnt/beta-catenin and noncanonical Wnt signaling interact in tissue evagination in the simple eumetazoan Hydra. Proceeding of the National Academy of Science, 2009, 106(11): 4290-4295 DOI:10.1073/pnas.0812847106 | |
| Qi RZ, Cao XT. The structure of the Notch and signal transduction. Chinese Journal of Cancer Biotherapy, 2002, 9(3): 212-214 [齐润姿, 曹雪涛. Notch的结构和信号转导. 中国肿瘤生物治疗杂志, 2002, 9(3): 212-214] | |
| Sun LZ, Hou L. Structure, function and related signaling pathway of Notch. Chinese Journal of Cell Biology, 2010, 32(6): 914-921 [孙丽哲, 侯林. Notch的结构、功能和相关信号通路. 中国细胞生物学学报, 2010, 32(6): 914-921] | |
| Trevino M, Harmon S, Burton PM. Wnt signaling promotes oral fates during regeneration and embryogenesis in the cnidarian Nematostella vectensis. Developmental Biology, 2011, 356(1): 252-254 | |
| Zanotti S, Canalis E. Notch and the Skeleton. Molecular and Cellular Biology, 2010, 30(4): 886-896 DOI:10.1128/MCB.01285-09 | |
| Zhou CY, Zhu L, Pan Y, et al. Wnt5 gene from Rhopilema esculentum: cDNA cloning, genomic organization and mRNA expression. Oceanologia et Limnologia Sinica, 2013, 44(4): 1115-1122 [周春娅, 朱玲, 潘滢, 等. 海蜇(Rhopilema esculentum)Wnt5基因: cDNA克隆、基因组结构与表达. 海洋与湖沼, 2013, 44(4): 1115-1122] | |
| Zhou QJ, Hu RZ, Zou JZ, et al. Notch signal transduction and regulation. Progress in Biochemistry and Biophysics, 2004, 31(3): 198-203 [周庆军, 胡若真, 邵健忠, 等. Notch信号转导与调控. 生物化学与生物物理进展, 2004, 31(3): 198-203] |



