2. 农业部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;
3. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071
脊尾白虾(Exopalaemon carinicauda)是我国近海一种重要的中小型经济虾类,其自身的一些生物学特点使其具备作为甲壳类实验动物的潜力,如成虾个体小,体色透明,生长快速,繁殖力强,环境适应性强等。我国从20世纪90年代开始对脊尾白虾进行了大量的研究,目前,关于脊尾白虾的研究主要集中于养殖技术(王兴强等, 2008; 于天基等, 2014)、病害诊治(Xu et al, 2010)、繁殖生物学(王绪峨, 1987; 梁俊平等, 2012)、微卫星引物筛选(Jia et al, 2013; 朱晓宇等, 20121))、功能基因克隆(王有昆等, 2015)和野生群体遗传多样性分析(马朋等, 2011; 贾舒雯等, 2012)等方面,而关于其近交系的研究尚未见报道。中国水产科学研究院黄海水产研究所海水养殖与生态研究室自2009年开始采用兄妹交配方式培育脊尾白虾近交家系,现已传至第10代,但由于长期的近交繁殖,出现了近交衰退现象,从第8代起,抱卵亲虾死亡率升高,亲虾抱卵量下降,卵成活率降低,传代速度和质量受到影响。因此,了解其遗传背景和遗传纯度是建立脊尾白虾近交系的首要问题。
1) Zhu XY. Isolation and genetic diversity analysis of microsatellite in Exopalaemon carinicaude. Master's Thesis of Zhejiang University, 2010 [朱晓宇.脊尾白虾微卫星分离及其遗传多样性分析.浙江大学硕士研究生学位论文, 2010]
微卫星标记在近交系检测中是一种非常有效的遗传质量控制手段(Tautz et al, 1989)。李凯等(2009)利用14对微卫星引物分析五指山猪3个不同家系F14-F18的遗传规律,结果显示,3个近交群体已各自成为独立家系,且近交程度不完全一致,基因纯合度也不相同。本研究利用33个微卫星基因座对脊尾白虾近交系3个家系进行了遗传学检测,检测它的遗传变异程度及近交程度,评价脊尾白虾近交群体的遗传结构,为开展脊尾白虾近交系遗传质量检测和遗传质量控制提供基础资料,并为其实验动物化提供基础数据支持。
1 材料与方法 1.1 材料 1.1.1 实验动物脊尾白虾近交系样本于2015年12月均取自中国水产科学研究院黄海水产研究所海水养殖与生态研究室建立的近交系,3个家系最初亲本均来自胶州湾,家系之间无明确关系,A系F7、F8、F9各30尾,B系F7、F8各30尾,H系F6 30尾,-80℃保存。
1.1.2 引物本研究涉及的33对微卫星引物均为本实验室开发的脊尾白虾微卫星引物,由生工生物工程(上海)有限公司合成。微卫星引物序列见表 1。
|
|
表 1 脊尾白虾微卫星引物的序列 Table 1 Microsatellite markers of E. carinicauda used in this study |
10×PCR Buffer、dNTP和Taq酶均购自天根公司。
1.1.4 主要仪器和设备PCR扩增仪;电泳仪;分光光度仪。
1.2 方法 1.2.1 DNA的提取参考刘萍等(2000)改进后的酚氯仿抽提法,将提取的DNA溶于灭菌水中,-20℃保存。用琼脂糖凝胶电泳和核酸蛋白质分析仪双重检测DNA的纯度和浓度,然后稀释到50 ng/μl备用。
1.2.2 PCR扩增反应体系20 μl,其中,DNA模板70-100 ng,2.0 μl 10×PCR Buffer, 1.6 μl dNTP (2.5 mmol/L),引物上下游各1 μl,0.2 μl Taq酶(5 U/μl)。PCR反应程序为95℃预变性5 min;95℃变性40 s,47-63℃退火温度40 s (因座位而异),72℃延伸30 s,35个循环;72℃延伸10 min。
1.2.3 电泳结果及记录8%聚丙烯酰氨凝胶500 V电泳2 h,银染显色,照相。每个微卫星座位根据电泳迁移率计算不同扩增片段的分子量,并确定个体基因型。无扩增条带则为无效等位基因,扩增带1条为纯合子,扩增带2条为杂合子。
1.2.4 数据统计与分析用POPGENE 32和Botstein等(1980)的公式计算各家系等位基因数(N)、有效等位基因数(Ne)、期望杂合度(He)、观测杂合度(Ho)、遗传距离(D)和多态信息含量(PIC),计算各家系在33个微卫星座位的平均基因纯合率(个体样本为纯合子的基因位点数与全部基因位点数的比率)并计算各家系间Nei氏标准遗传距离(1972) 进行系统聚类分析。
2 结果 2.1 总群体的遗传变异脊尾白虾近交系3个家系4个世代总群体的遗传多样性见表 2。从表 2可以看出,脊尾白虾近交系群体在33个微卫星基因座上的等位基因数(N)最多为7个(EC02),最少为1个(EC13、EC15、EC31),平均每个基因座2.758个;有效等位基因数(Ne)介于1.000-2.818,平均值为1.611。总群体的杂合度H的范围是0-0.804,其中,4个基因座的H为0,另外有9个在0.570-0.804之间,其余20个在0.008-0.467之间。总群体的平均PIC为0.272。
|
|
表 2 脊尾白虾近交群体的遗传多样性 Table 2 Genetic diversity of inbred E. carinicauda |
此外,3个家系在33个微卫星座位共发现72个等位基因,A、B和H系分别具有40、53、56个等位基因,其中,32个等位基因为各家系所共有。
2.2 家系间的遗传变异根据个体基因型分别计算3个家系的N、H和PIC等指数见表 3。从表 3可以看出,A系中,除EC27、EC28表现为无效等位基因外,其他都表现出多态性,且有5个基因座(EC13、EC15、EC16、EC18、EC31) 表现为单一位点;B系中,EC31表现为无效等位基因,7个基因座(EC11、EC12、EC13、EC15、EC21、EC30、EC32) 表现为单一位点;H系中,EC30表现为无效等位基因,5个基因座(EC13、EC15、EC19、EC31、EC32) 表现为单一位点。在所有表现纯合的基因座中,有2个基因座(EC13、EC15) 在3个家系中都表现纯合,A系特有的纯合基因座有2个(EC16、EC18),B系特有的纯合基因座有4个(EC11、EC12、EC21、EC30),H系特有的纯合基因座只有1个(EC19)。由于这些特有的纯合基因座只在某一近交系中表现单一性,而在其他近交系中仍然表现多态性,故特有纯合基因座可能作为近交系的标志基因座,用来区分不同的近交系。图 1为EC30基因座分别在A系、B系和H系中的电泳图,该位点仅在B系中表现为直带,故可作为B系标志基因座。
|
|
表 3 33个微卫星基因座在脊尾白虾3个近交家系中的遗传变异 Table 3 Genetic variation of 33 microsatellites in three inbred families of E. carinicauda |

|
图 1 EC30分别在A系、B系和H系中的电泳 Figure 1 The electrophoretogram of EC30 on A, B, H inbred lines respectively |
从表 3中还可以看出,A、B和H系的平均N、H和PIC分别为2.258、0.226、0.214,2.469、0.283、0.300和2.219、0.268、0.207,与野生群体(6.697、0.716、0.668) (Wang et al, 2016)相比,已有了大幅度的降低。A、B、H家系中分别有23、19、24个基因座仅拥有2个或3个等位基因,且3个近交系的平均PIC均小于0.5,属中度或低度多态,基因分化系数为0.118。从这些指数的差异来看,3个家系间已经呈现分化的趋势,正向着与本系相关等位基因纯合的方向选育。3个家系在EC13、EC15基因座上表现为纯合,即只有一个等位基因,但在EC03、EC06、EC08、EC17、EC22、EC33基因座上却表现出高度多态性,每个家系均存在2个以上等位基因,并且拥有较高的杂合度(H>0.5)。结果显示,虽然近交的推进使其向着不同家系方向分化,但各自均保持脊尾白虾某些共同的种质特性。
2.3 近交程度基因纯合率是每个个体的纯合座位占总检测座位的比率(牛荣等, 2001)。3个近交家系在全部基因座上的平均基因纯合率见表 4。从表 4可以看出,脊尾白虾近交系A系F7的平均基因纯合率为68.34%,F8为67.11%,稍有下降,但到F9时,平均基因纯合率为77.59%,相比F7、F8有了显著的提高。通过计算得出,A、B、H家系的平均基因纯合率分别为71.01%、57.01%和61.34%,存在一定的差异,即各家系基因纯合的速度不同,A系最快,H系次之,B系最慢,且各家系基因纯合的程度也不相同。近交使基因趋向纯合,因此,平均基因纯合率可以在很大程度上代表一个群体的近交程度,本研究结果显示,脊尾白虾近交系经过人工控制交配已达到了一定的近交程度。
|
|
表 4 脊尾白虾近交系3家系33个微卫星座位平均基因纯合率 Table 4 Mean rate of homozygote at 33 microsatellite loci in 3 lineages of E. carinicauda inbred line (%) |
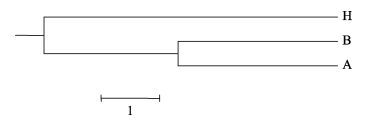
根据等位基因频率计算出脊尾白虾3个近交系间的Nei氏标准遗传距离和遗传相似度见表 5。从表 5可以看出,遗传距离介于0.178-0.352,遗传相似度介于0.653-0.837。A系和B系的遗传距离最近(0.178),A系和H系的遗传距离最远(0.352)。利用UPGMA法构建的聚类见图 2。从图 2可以看出,A系和B系先聚为一支,再与H系聚在一起。推测可能与近交系最初亲本之间亲缘关系远近有关,也可能跟建系过程有关。
|
|
表 5 3个脊尾白虾近交系间的遗传距离(D,对角线下)及遗传相似性(I,对角线上) Table 5 Nei's genetic distance (D, below diagonal) and genetic identity (I, above diagonal)among E. carinicauda inbred lines |
|
图 2 基于D遗传距离的UPGMA聚类 Figure 2 UPGMA clustering tree based on D genetic distance |
通常用来检验近交系遗传多样性的指标有N、H、PIC、基因分化系数(Gst)和平均基因纯合率等。从各家系的等位基因组成可以评价其遗传背景。33个微卫星座位在脊尾白虾3个家系中共检测到72个等位基因,等位基因数在1-7个不等,平均每个位点2.180个,而这33个位点在江苏野生群体上检测到的等位基因数范围为3-11个,平均每个位点等位基因数为6.69个(Wang et al, 2016),由此可见,近交系的等位基因数远远少于野生群体。但这一结果与李霞等(2004)对剑尾鱼(Xiphophorus hellerii) RW-H系F8的结果(平均等位基因数2.600个)相似。等位基因最多的位点为EC02,共发现7个等位基因;最少的位点仅有1个等位基因(EC13和EC15),即在3个家系所有个体中均为同一等位基因。该结果说明,近交使大量等位基因丢失。3个家系都拥有的等位基因有32个,占所有等位基因的44.40%,另外,40个等位基因则在各家系间表现出不同程度的差别,说明在近交过程中,脊尾白虾各近交家系发生了严重分化,各家系均已成为独立遗传群体。
杂合度又称基因多样度,是度量群体遗传变异的最适参数之一。利用33个微卫星座位测得脊尾白虾3个近交家系的总群体平均杂合度为0.294,各家系内平均杂合度为0.259,该值远远低于脊尾白虾野生群体的平均杂合度(0.689),略高于近交至F7时的广西巴马小型猪近交群体(0.277) (叶香尘, 2006)1)。脊尾白虾近交群体由于长期的近交和人工选育产生近交分化,群体中不断淘汰不利基因,纯化有利基因,使群体基因型趋于一致和纯合,而导致了遗传基因多样性的低水平。
1) Ye XC. Genetic analysis of 21 microsatellite loci in 5 lineages of Guangxi Bama mini-pig inbred line. Master's Thesis of Guangxi University, 2006, 31-55 [叶香尘.应用21个微卫星座位对广西巴马小型猪五个近交系群体的遗传学分析.广西大学硕士研究生学位论文, 2006, 31-55]
PIC是衡量位点多态性的指标。当PIC>0.5时,该座位为高度多态座位,0.25<PIC<0.5时为中度多态性座位,PIC<0.25时为低度多态座位(战爱斌等, 2006)。本研究所用的33个微卫星位点用于野生群体的检验时,有27个表现为高度多态,5个为中度多态,1为个低度多态。用于近交系群体的检验时,A系、B系和H系分别有16、11和17个低度多态位点,且其平均PIC分别为0.214、0.300、0.207,均小于0.5,属中度或低度多态。本研究的总群体PIC为0.272,远低于野生群体的PIC (0.668) 和近交至F7时小型猪近交群体(叶香尘, 2006)1),却与近交至F3时的广西巴马小型猪近交群体(0.278) (牛荣, 2004)接近。表明长期近交和人工选育已经产生了基因分离,即脊尾白虾已分化成多个家系,且在该过程中大量等位基因丢失,PIC降低。
基因分化系数(Gst)是衡量群体间在多个座位上遗传分化的指标,为总群体平均杂合度(Ht)和各亚群内平均杂合度(Hs)的函数,即:Gst=1-Hs/Ht。本研究中3个家系总群体平均杂合度为0.294,各家系内平均杂合度为0.259,基因分化系数为0.118。各家系内平均杂合度比较低,表明大多数为纯合位点,但并未达到近交系动物的水平,即群体内平均杂合度为0,故仍然存在进一步分化的可能。遗传分化系数是衡量基因分化相对量的一个较好的指标,脊尾白虾近交系3家系的基因分化系数为0.118,表明群体间的遗传变异占总群体遗传变异的11.80%,表明脊尾白虾近交系3个家系间的差异已经比较大,均已成为独立品系。
平均基因纯合率在很大程度上代表一个群体的近交程度。本研究近交系H系F6的平均基因纯合率为61.34%,与三疣梭子蟹(Portunus trituberculatus) 2个近交系F4的平均基因纯合率分别55.87%和56.69% (韩智科等, 2011)2)高出5%左右。A系F9的平均基因纯合率为77.59%,与版纳小耳猪807近交系F10的平均基因纯合率(75%) (牛荣等, 2001)值大小相近。通过以上数据推测,在长期的近交过程中,基因不断被淘汰,最后趋向纯合,导致平均基因纯合率提高。
2) Han ZK. The genetic variation analysis of Inbred Portunus trituberculatus. Master's Thesis of Shanghai Ocean Univeristy, 2011, 30-53 [韩智科.三疣梭子蟹近交家系遗传变异分析.上海海洋大学硕士研究生学位论文, 2011, 30-53]
3.2 近交系的标志位点近交系动物中微卫星长度变化发生率很低,微卫星位点的等位基因可能是某一近交系动物的恒定参数(Tascon et al, 2000)。可以用来鉴别不同的近交系动物。刘宇飞等(2007)用20个微卫星位点检测剑尾鱼RW-H近交系F15遗传结构,发现有17个位点表现为纯合,可作为RW-H系的标志基因位点。本研究得出脊尾白虾近交A系特有的纯合基因座有2个(EC16、EC18),B系特有的纯合基因座有4个(EC11、EC12、EC21、EC30),H系特有的纯合基因座只有1个(EC19),可分别作为各自近交系的潜在特异标志基因用于遗传监测。
| Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314 | |
| Jia SW, Liu P, Li J, et al. Isolation and characterization of polymorphic microsatellite loci in the ridgetail white prawn Exopalaemon carinicauda. Genetics and Molecular Rese arch, 2013, 12(3): 2816-2820 DOI:10.4238/2013.August.8.1 | |
| Jia SW, Liu P, Li J, et al. Microsatellite analysis of genetic diversity in 3 wild populations of Exopalaemon carinicauda. Journal of Fisheries of China, 2012, 36(12): 1819-1825 [贾舒雯, 刘萍, 李健, 等. 脊尾白虾3个野生群体遗传多样性的微卫星分析. 水产学报, 2012, 36(12): 1819-1825] | |
| Li K, Feng ST, Mou YL, et al. Study on genetic regulation of microsatellite loci gene of three inbred families of Wuzhishan Miniature pig. Scientia Agriculture Sinica, 2009, 42(5): 1751-1760 [李凯, 冯书堂, 牟玉莲, 等. 五指山猪3个近交家系内微卫星等位基因的遗传变化. 中国农业科学, 2009, 42(5): 1751-1760] | |
| Li X, Bai JJ, Wu SQ, et al. Genetic structure analysis of Xiphophorus helleri RW-H inbred strain. Journal of Fisheries of China, 2004, 28(4): 365-370 [李霞, 白俊杰, 吴淑勤, 等. 剑尾鱼RW-H近交系的遗传结构分析. 水产学报, 2004, 28(4): 365-370] | |
| Liang JP, Li J, Liu P, et al. Research progress of biological characteristics and artificial breeding of ridgetail white prawn, Exopalaemon carinicauda. Chinese Agricultural Science Bulletin, 2012, 28(17): 109-116 DOI:10.11924/j.issn.1000-6850.2011-3631 [梁俊平, 李健, 刘萍, 等. 脊尾白虾生物学特性与人工繁育的研究进展. 中国农学通报, 2012, 28(17): 109-116] | |
| Liu P, Xu HS, Kong J, et al. RAPD analysis of wild stock of penaeid shrimp (Penaeus chinensis)in the China's coastal waters of Huanghai and Bohai seas. Acta Oceanologica Sinica, 2000, 22(5): 89-93 [刘萍, 徐怀恕, 孔杰, 等. 中国对虾黄, 渤海沿岸地理群的RAPD分析. 海洋学报, 2000, 22(5): 89-93] | |
| Liu YF, Bai JJ, Li KB, et al. Genetic structure of 20 microsatellite loci in RW-H swordtail fish (Xiphopphorus helleri) inbred strain. Journal of Fishery Sciences of China, 2007, 14(4): 602-607 [刘宇飞, 白俊杰, 李凯彬, 等. 剑尾鱼RW-H近交系20个微卫星位点的遗传结构. 中国水产科学, 2007, 14(4): 602-607] | |
| Ma P, Liu P, Li J, et al. The genetic diversity and phylogenetic analysis of COI gene in mitochondrial DNA of three populations of Exopalaemon carinicauda. Progress in Fishery Sciences, 2011, 32(6): 50-56 [马朋, 刘萍, 李健, 等. 脊尾白虾3个野生群体线粒体COI基因的遗传多样性及其系统发育分析. 渔业科学进展, 2011, 32(6): 50-56] | |
| Niu R, Huang ZB, Wei H, et al. Genetic analysis of 12 microsatellite loci in Xishuangbanna miniature pig inbred. Acta Laboratorium Animalis Scientia Sinica, 2004, 11(1): 42-45 [牛荣, 黄中波, 魏泓, 等. 用12对微卫星引物对版纳小耳猪近交系的遗传学分析. 中国实验动物学报, 2004, 11(1): 42-45] | |
| Niu R, Zeng YZ, Shang HT, et al. Genetic analysis of 35 microsatellite loci in 5 lineages of Xishuangbanna miniature pig inbred line. Acta Genetic Sinica, 2001, 28(6): 518-526 [牛荣, 曾养志, 商海涛, 等. 版纳小耳猪近交系5家系35个微卫星座位的遗传分析. 遗传学报, 2001, 28(6): 518-526] | |
| Tascon CD, Littlejohn RP, Almeida AR, et al. Genetic variation within the Merino sheep breed: Analysis of closely related populations using microsatellites. Animal Genetics, 2000, 31: 243-251 DOI:10.1046/j.1365-2052.2000.00636.x | |
| Tautz D. Hypervariabflity of simple sequences as a general source for polymorphic DNA markers. Nucleic Acids Research, 1989, 17(16): 6463-6471 DOI:10.1093/nar/17.16.6463 | |
| Wang XE. Preliminary observation of Exopalaemon carinicauda breeding biology. Chinese Journal of Zoology, 1987, 22(1): 7-10 [王绪峨. 脊尾白虾繁殖生物学的初步观察. 动物学杂志, 1987, 22(1): 7-10] | |
| Wang XQ, Cao M, Yan BF, et al. The aquaculture technology research of Exopalaemon carinicauda. Fisheries Science and Technology Information, 2008, 35(6): 265-269 [王兴强, 曹梅, 阎斌伦, 等. 脊尾白虾养殖技术研究. 水产科技情报, 2008, 35(6): 265-269] | |
| Wang YK, Liu P, Duan YF, et al. The cloning and expression of Alpha2-Macrogloblin gene of Exopalaemon carinicauda. Progress in Fishery Sciences, 2015, 36(2): 63-71 DOI:10.11758/yykxjz.20150208 [王有昆, 刘萍, 段亚飞, 等. 脊尾白虾(Exopalaemon carinicauda) a2-巨球蛋白cDNA全长的克隆和表达分析. 渔业科学进展, 2015, 36(2): 63-71] | |
| Wang RF, Li J, Li JT, et al. Isolation and characterization of microsatellite markers in the ridgetail white prawn Exopalaemon carinicauda. Conservation Genetics Reso urces, 2016, 8(1): 43-81 DOI:10.1007/s12686-016-0522-2 | |
| Xu WJ, Xie JJ, Shi H, et al. Hematodinium infections in cultured ridgetail white prawns, Exopalaemon carinicauda, in eastern China. Aquaculture, 2010, 300(1-4): 25-31 DOI:10.1016/j.aquaculture.2009.12.024 | |
| Yu TJ, Li J, Li JT, et al. The growth and reproduction characteristic of ridgetail white prawn (Exopalaemon carinicauda) cultured in ponds. Chinese Fishery Quality and Standards, 2014, 4(2): 16-25 [于天基, 李健, 李吉涛, 等. 池养脊尾白虾的生长与繁殖特性. 中国渔业质量与标准, 2014, 4(2): 16-25] | |
| Zhan AB, Bao ZM, Lu Wei, et al. Microsatellite markers of Apostchopus japonicas. Journal of Fisheries of China, 2006, 30(2): 192-196 [战爱斌, 包振民, 陆维, 等. 仿刺参的微卫星标记. 水产学报, 2006, 30(2): 192-196] |




