2. 江苏省洪泽湖渔业管理委员会办公室 淮安 223002
2. Hongze Lake Fisheries Administration Committee Office of Jiangsu Province, Huai'an 223002
河蚬(Corbicula fluminea)属软体动物门、瓣鳃纲、真瓣鳃目、蚬科、蚬属,是我国常见的小型双壳经济贝类,在淡水、咸淡水的江河、湖泊、池塘中广泛分布,是底栖动物群落中的优势种群(杞桑等, 1987; 蔡炜等, 2010; 李德亮等, 2013)。河蚬在水生态系统的物质循环和能量流动中具有重要作用,也是构建生物浮床净化富营养化水体常用的底栖生物种类(Li et al, 2010)。同时,河蚬营养丰富、味道鲜美,是很多国家居民的美食,国际市场对河蚬需求量也在逐渐增加。近年来,河蚬养殖业悄然兴起,已成为淡水经济贝类的养殖热点之一。由于养殖所需苗种主要来自于采捕野生苗,导致对河蚬资源的捕捞强度逐渐加大,再加之水环境污染,使得其天然资源量呈现急剧下降。因此,保护和恢复河蚬野生种质资源成为亟待解决的问题。
洪泽湖是中国第四大淡水湖泊,位于江苏省西部淮河干流中下游,是具有防洪、灌溉、调水、水产、水运、水电等综合利用功能的平原型浅水湖(王兆群等, 2010)。历史上洪泽湖河蚬资源量较为丰富,分布遍及全湖,是洪泽湖重要的出口创汇水产品之一。在20世纪80年代初期,洪泽湖年产河蚬32万t,到90年代初减少到21.29万t。近年来,资源量下降到5万t,分布区域也大大缩小(袁永浒等, 1994; 严维辉等,2007; 毕婷婷等, 2014),这较大地制约了河蚬产业的可持续发展。目前,有关洪泽湖河蚬的研究主要集中在资源量调查及其分布、种群年龄结构、营养价值等方面(袁永浒等, 1994; 严维辉等, 2007; 毕婷婷等, 2014; 张超文等, 2012; 刘晶晶等, 2014),而对于其种质资源遗传多样性的研究较少(丁怀宇等, 2011; 李大命等, 2015)。在本研究中,以洪泽湖河蚬2种表型群体(黑色和黄色)为研究对象,采用形态学分析和分子标记技术(COⅠ基因)研究河蚬2种群体的形态及遗传多样性,以期为洪泽湖河蚬种质资源保护提供科学依据。
1 材料与方法 1.1 样本来源及形态学测量本研究于2015年3月在洪泽湖临淮水域采集河蚬样品,采用工具为划耙,耙齿间距为8 mm,耙宽为70 cm。根据河蚬外壳颜色差异,将洪泽湖河蚬分为黑色群体和黄色群体(图 1)。随机选择部分河蚬样品,其中,黑色河蚬28个,黄色河蚬30个。用电子天平称量河蚬湿重(Wet weight, WW),精确到0.01 g。称量前,用滤纸将河蚬体表的水分吸干。用电子游标卡尺测量河蚬壳长(Shell length, SL)、壳宽(Shell width, SW)和壳高(Shell height, SH),精确到0.01 mm。

|
图 1 河蚬外壳差异 Figure 1 Distinct difference of external shell color of C. fluminea |
取河蚬的肌肉组织,用无水乙醇固定,-20℃保存备用。基因组DNA的提取方法为酚-氯仿抽提法(Sambrook et al, 1995)。DNA经75% (体积分数)乙醇洗涤并在室温干燥,最后用100 μl TE溶解,置于-20℃保存备用。
COⅠ基因扩增引物分别为F(5'-GGTCAACAAATCATAAAGATATTGG-3')、R(5'-TAAACTTCAGGGTGACCAAAAAATCA-3') (Folmer et al, 1994)。PCR反应体积为50 μl,其中,包括25 μl PCR mix,上、下游引物各2 μl (10 μmol/L),模板2 μl,其余部分用超纯水补足。PCR反应条件:94℃预变性4 min;94℃变性40 s,50℃退火40 s,72℃延伸50 s,30个循环;72℃延伸10 min;最后保持在4℃。
取PCR扩增产物进行1.5%的琼脂糖凝胶电泳检测,采用DNA凝胶回收试剂盒对目的片段进行回收纯化,然后送上海生工生物技术有限公司进行双向测序。
1.3 数据处理和分析采用SPSS 16.0软件对2种河蚬群体的形态学参数进行t检验,当P < 0.05时为显著性差异。采用Bioedit 7.5软件对COⅠ基因序列进行编辑、排序和比对,并辅以人工校对。利用MEGA 4.0软件计算序列的碱基组成、多态性位点及转换/颠换比值(Tamura et al, 2007)。利用邻接法(Neighbor joining)构建COⅠ单倍型分子系统进化树,经1000次重复抽样检测其置信度。运用DnaSP 5.0软件计算群体的遗传多样性参数,包括单倍型数、单倍型多样性指数、核苷酸多样性指数和平均核苷酸差异数(Librado et al, 2007)。使用Arlequin 3.1软件中的分子方差分析(AMOVA)方法估算遗传变异在群体间和群体内的分布,并计算群体间遗传分化指数Fst值(F-statistics, Fst)及其P值(Excoffier et al, 2005)。
2 结果 2.1 河蚬形态学特征洪泽湖黑色和黄色河蚬群体的形态学参数见表 1。由表 1可以看出,黑色河蚬群体的重量、壳长、壳宽、壳高、壳长/壳宽、壳高/壳宽均大于黄色河蚬群体,而壳长/壳高则小于黄色群体。统计分析结果显示,除壳长/壳高之外,2个群体的形态学指标均存在极显著性差异(P < 0.01)。
|
|
表 1 河蚬2个表型群体形态学参数 Table 1 Morphological parameters of two different phenotypical population of C. fluminea |
黑色和黄色河蚬群体的COⅠ基因序列碱基组成基本一致,COⅠ基因为614 bp,碱基序列中A、T、G和C的平均含量分别为22.5%、42.3%、21.2%和14.0%,均表现出碱基A+T含量(64.8%)大于G+C含量(35.2%)。2个群体的COⅠ基因序列中均检出53个多态性位点,转换/颠换比分别为2.71和2.74,没有插入、缺失位点。
2.3 种群遗传多样性参数黑色河蚬群体检出7个单倍型(Hb1–Hb7),黄色种群检出5个单倍型(Hy1–Hy5)。单倍型Hb1、Hy5及Hb5、Hy2是2个群体的共享单倍型。黑色群体中单倍型Hb1的数量最多(10个),占比35.7%;黄色群体中单倍型Hy2的数量最多(16个),占比53.3%。2个河蚬群体的遗传多样性见表 2。从表 2可以看出,共检出洪泽湖河蚬单倍型10个,单倍型多样性指数为0.726,核苷酸多样性指数为0.04021,平均核苷酸差异数为24.687。由表 2可以得出,洪泽湖河蚬群体的遗传多样性较高,其中,黑色群体的遗传多样性高于黄色群体。
|
|
表 2 河蚬群体的遗传多样性参数 Table 2 Genetic diversity parameters of two populations of C. fluminea |
洪泽湖河蚬2个群体的方差分析(AMOVA)结果显示(表 3),遗传变异固定指数Fst=0.2174,且统计结果为极显著(P < 0.01),表明2个群体之间存在显著的遗传分化。在整个遗传变异中,2个群体之间的遗传变异占21.74%,而大部分变异(78.26%)发生在群体内部。
|
|
表 3 洪泽湖河蚬群体COⅠ基因的AMOVA分析 Table 3 AMOVA analysis of all mtDNA COⅠ gene sequences from two C. fluminea populations |
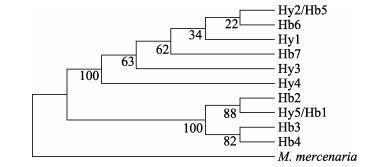
分析比较了10个单倍型之间的Kimura-2-parameter遗传距离,其值在0.002-0.091之间,平均遗传距离为0.048 (表 4)。根据遗传距离大小,可以将10个单倍型分为2个类群,其中,单倍型Hb1/Hy5、Hb2、Hb3和Hb4为一个类群,而单倍型Hy1、Hy2/Hb5、Hy3、Hy4、Hb6和Hb7为另一个类群。
|
|
表 4 河蚬10个COⅠ单倍型之间的遗传距离 Table 4 Genetic distances among 10 COⅠ gene haplotypes of C. fluminea |
以中国圆蛤(Mercenaria mercenaria)为外群,采用邻接法(NJ)和最大简约法(MP)构建COⅠ基因单倍型系统发生树(图 2和图 3)。结果显示,10个单倍型聚为2个明显的分支,其中,单倍型Hy1、Hy2/Hb5、Hy3、Hy4、Hb6和Hb7聚为一支,单倍型Hb1/Hy5、Hb2、Hb3和Hb4聚为另一支。2个分支系统树的置信度统计值较高,说明COⅠ单倍型系统进化树具有较高的可信度。
|
图 2 河蚬COⅠ基因10个单倍型NJ系统发生树 Figure 2 Phylogenetic tree of 10 COⅠ haplotypes of C. fluminea |

|
图 3 河蚬COⅠ基因10个单倍型MP系统发生树 Figure 3 Maximum parsimony tree of 10 COⅠ haplotypes of C. fluminea |
本研究中洪泽湖黑色河蚬和黄色河蚬群体的mtDNA COⅠ基因序列中的4种碱基组成平均值为T(42.3%)、A(22.5%)、C(14.0%)和G(21.2%),其中,A+T的平均含量为64.8%,远高于G+C的含量(35.2%),显示出明显的碱基组成偏倚性,与Park等(2003)研究的亚洲8个国家和地区的河蚬mtDNA COⅠ基因序列组成相似,其中,A+T的含量平均值为64.5%。在其他双壳贝类中mtDNA COⅠ基因序列组成也有类似规律,比如缢蛏(Sinonovacula constricta)的COⅠ基因序列中A+T的含量为66.2% (牛东红等, 2008),褶纹冠蚌(Cristaria plicata)的A+T含量为60.1% (贾名静等, 2009),三角帆蚌(Hyriopsis cumingii) 10个群体的COⅠ基因序列中A+T的含量都明显高于G+C的含量(李家乐等, 2008),说明COⅠ基因序列中A+T含量较高在双壳贝类中普遍存在,也符合动物线粒体基因组成不均一的特征。
3.2 河蚬遗传多样性及群体结构分析遗传多样性即基因多样性是生物多样性的核心。生物的遗传多样性高低与其适应能力、生存能力和进化潜力密切相关。单倍型多样性(Hd)和核苷酸多样性(Pi)是衡量生物遗传多样性的2个重要指标。本研究中,基于线粒体COⅠ基因序列研究了洪泽湖河蚬黑色和黄色群体的遗传多样性。结果显示,黑色和黄色群体的单倍型多样性和核苷酸多样性指数分别为0.794和0.04274、0.607和0.02825 (表 2)。根据线粒体DNA序列的遗传变异分析,可以将不同核苷酸多样性和单倍型多样性间的组合分成4种类型[低Pi( < 0.005) 与低Hd( < 0.5);低Pi( < 0.005) 与高Hd( > 0.5);高Pi( > 0.005) 与低Hd( < 0.5);高Pi( > 0.005) 与高Hd( > 0.5)] (Grant et al, 1998)。本研究表明,2个河蚬群体的Pi和Hd均较高,属于第4种类型,表明洪泽湖河蚬群体的遗传多样性较高。丁怀宇等(2011)采用微卫星分子标记对洪泽湖4个河蚬群体遗传特征进行了研究,结果显示,4个群体的平均期望杂合度均大于0.752,显示出较高的遗传多样性水平。李大命等(2015)研究表明,洪泽湖河蚬的单倍型多样性指数和核苷酸多样性指数分别为0.870和0.045,均与本研究结果一致。整体来看,一方面,洪泽湖河蚬种质资源具有较高的遗传多样性,意味着河蚬群体有较强的环境适应能力及进化潜力,为河蚬良种选育及资源可持续发展和利用提供了保证;另一方面,洪泽湖河蚬天然资源量急剧下降,目前已无法形成捕捞群体。因此,必须加强洪泽湖河蚬种质资源的保护,通过建立种质资源保护区、增殖放流、控制捕捞强度及延长禁渔期等措施避免河蚬资源进一步衰减,以保护其种质资源的多样性及完整性。
根据Balloux等(2002)提出的标准,Fst值在0-0.05、0.05-0.15、0.15-0.25分别表明群体间的低、中、高遗传分化水平。在本研究中,方差分析结果显示,2个群体的遗传变异主要发生群体内,占78.26%,而种群间遗传变异占21.74%。群体间遗传分化系数Fst = 0.2174,且统计结果为极显著(P < 0.01),表明洪泽湖河蚬黑色和黄色河蚬群体之间存在较高的遗传分化。因此,应将洪泽湖河蚬黑色和黄色群体作为独立单元分别进行管理和保护。
3.3 河蚬单倍型遗传分化分析在本研究中,共检测出10个单倍型,NJ系统进化树显示,COⅠ单倍型分为2个明显分支,且2个分支内单倍型之间的遗传距离较小(0.002-0.005),而2个分支间单倍型之间的遗传距离较大(0.081-0.091) (表 4)。根据Hebert等(2003)提出的物种鉴定最小种间距离为2%,且种间遗传距离大于种内的遗传距离大约10倍的标准,可以初步认定,洪泽湖河蚬存在2个种类的COⅠ单倍型,二者之间有明显的遗传差异。同样,基于COⅠ基因序列,对德国、法国、荷兰及中国多个水体河蚬群体的遗传多样性进行研究,也发现了河蚬群体COⅠ单倍型呈现出明显遗传分化(Renard et al, 2000; Pfenninger et al, 2002; Wang et al, 2014)。由此可以推测,河蚬群体内部COⅠ单倍型出现遗传分化是一个普遍存在的现象,可能是由于河蚬种源不同或环境差异所致(Baker et al, 2003; Sousar et al, 2007)。
一般来说,双壳贝类具有较强的表型可塑性以适应不同的环境和生态条件(Baker et al, 2003)。根据河蚬壳表面颜色差异,可将河蚬分为不同的群体类型。在太湖和洪泽湖中均发现河蚬有黑色和黄色2个群体。通过调查发现,太湖河蚬黑色和黄色群体的分布区域不同,且黑蚬高产区的底泥硫化物含量较高(曹文明等, 2000)。而洪泽湖河蚬黑色和黄色群体的分布区域一致,导致其颜色差异的环境因子还有待进一步研究。另外,统计分析结果显示,洪泽湖河蚬黑色和黄色群体的形态学特征有显著差异,而湖南大通湖河蚬白色和紫色群体的形态学参数无显著性差异(Wang et al, 2014)。研究还发现,根据颜色划分河蚬类群与其遗传特征不具有对应性。本研究中黑色和黄色河蚬群体拥有2个共享单倍型(Hb1、Hy5及Hb5、Hy2),却分布在2个不同的分支上(图 2),且群体内单倍型之间的遗传距离大于群体间的遗传距离(表 4)。在大通湖河蚬2个群体(白色和紫色)中,基于mtDNA COⅠ基因序列分析得到4种单倍型(DT-1、DT-7、DT-19和DT-31),单倍型DT-7与另外3种单倍型有明显遗传差异,其中,单倍型DT-31仅在白色群体中出现,其他3种单倍型在2个群体中都有出现(Wang et al, 2014)。基于形态学和COⅠ基因序列对葡萄牙2个地理分布河蚬群体进行比较分析,结果显示,2个河蚬群体的形态学参数具有显著性差异,而COⅠ基因序列却高度相似,Sousar等(2007)认为,应将2个地理分布群体归为同一河蚬种类。因此,只有将形态学和遗传学特征结合起来,才能准确认识不同表型河蚬群体之间的相互关系。
| Baker AM, Bartlett C, Bunn SE, et al. Cryptic species and morphological plasticity in long-lived bivalves (Unionoida: Hyriidae) from inland Australia. Molecular Ecology, 2003, 12(10): 2707-2717 DOI:10.1046/j.1365-294X.2003.01941.x | |
| Balloux F, Lugon-Moulin N. The estimation of population differentiation with microsatellite markers. Molecular Ecology, 2002, 11(2): 155-165 DOI:10.1046/j.0962-1083.2001.01436.x | |
| Bi TT, Hou G, Zhang SY, et al. Study on age structure, growth and mortality of Corbicula fluminea in Hongze Lake, China, through shell length-frequency data analysis. Acta Hydrobiologica Sinica, 2014, 38(4): 797-800 DOI:10.7541/2014.111 [毕婷婷, 侯刚, 张胜宇, 等. 基于壳长频率分析的洪泽湖河蚬渔获物年龄结构、生长与死亡参数估计. 水生生物学报, 2014, 38(4): 797-800] | |
| Cai W, Cai YJ, Gong ZJ, et al. Temporal and spatial patterns of Corbicula fluminea in Lake Taihu. Journal of Lake Sciences, 2010, 22(5): 714-722 [蔡炜, 蔡永久, 龚志军, 等. 太湖河蚬时空格局. 湖泊科学, 2010, 22(5): 714-722] | |
| Cao WM, Zhou G, Sheng JM, et al. The present situation and evolution of the Corbicula fluminea resources in Tai Lake. Journal of Nanjing Forestry University (Natural Science Edition), 2000, 24(S1): 125-128 [曹文明, 周刚, 盛建明, 等. 太湖河蚬资源现状及演变. 南京林业大学学报(自然科学版), 2000, 24(S1): 125-128] | |
| Ding HY, Jiang HC, Feng JB, et al. Isolation of microsatellite loci and population genetic structure analysis of wild Corbicula fluminea in Hongze Lake. Journal of Fisheries of China, 2011, 35(11): 1624-1632 [丁怀宇, 姜虎成, 冯建彬, 等. 河蚬微卫星引物筛选及洪泽湖野生群体遗传结构分析. 水产学报, 2011, 35(11): 1624-1632] | |
| Excoffier L, Laval G, Schneiders S. Arlequin Ver. 3.0: An integrated software package for population genetics data analysis. Evolution Bioinformation Online, 2005, 1(1): 47-50 | |
| Folmer O, Black M, Hoeh W, et al. DNA primers for amplification of mitochondrial cytochrome coxidase subunit Ⅰ from diverse metazoan invertebrates. Molecular Marine Biology and Biotechnology, 1994, 3(5): 294-299 | |
| Grant WS, Bowen BW. Shallow population histories in deep evolutionary lineages of marine fishes: Insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 1998, 89(5): 415-426 DOI:10.1093/jhered/89.5.415 | |
| Hebert PD, Ratnasingham S, Dewaard JR. Barcoding animal life: Cytochrome c oxidase subunit 1 divergences among closely related species. Proceedings of the Royal Society B: Biology Sciences, 2003, 270(S1): 96-99 | |
| Jia MJ, Li JL, Niu DH, et al. Sequence variation of COI gene in ten populations of Cristaria plicata from the middle and lower Yangtze River. Chinese Journal of Zoology, 2009, 44(1): 1-8 [贾名静, 李家乐, 牛东红, 等. 长江中下游褶纹冠蚌10个群体COI基因序列变异分析. 动物学杂志, 2009, 44(1): 1-8] | |
| Li DM, Zhang TQ, Tang SK, et al. Genetic diversity of wild Corbicula fluminea population in the Hongze Lake analyzed by mitochondrial DNA COI gene sequence. Progress in Fishery Sciences, 2015, 36(5): 81-86 DOI:10.11758/yykxjz.20150511 [李大命, 张彤晴, 唐晟凯, 等. 洪泽湖野生河蚬(Corbicula fluminea)线粒体COI基因序列的遗传多样性分析. 渔业科学进展, 2015, 36(5): 81-86] | |
| Li DL, Zhang T, Xiao TY, et al. Standing crops and temporal-spatial distribution of Corbicula fluminea (Müller, 1774) in Lake Datong, Hunan Province. Journal of Lake Sciences, 2013, 25(5): 743-748 DOI:10.18307/2013.0517 [李德亮, 张婷, 肖调义, 等. 湖南省大通湖河蚬(Corbicula fluminea)现存量及其时空分布. 湖泊科学, 2013, 25(5): 743-748] | |
| Li JL, Wang JJ, Wang GL, et al. Sequence analysis of mitochondrial cytochrome c oxidase sununit (COI) gene of Hyriopsis cumingii from the five freshwater Lakes of China. Acta Hydrobiologica Sinica, 2008, 32(5): 779-782 [李家乐, 王建军, 汪桂玲, 等. 我国五大淡水湖三角帆蚌群体mtDNA COI基因片段变异分析. 水生生物学报, 2008, 32(5): 779-782] | |
| Li XN, Song HL, Li W, et al. An integrated ecological floating-bed employing plant, freshwater clam and biofilm carrier for purification of eutrophic water. Ecological Engineering, 2010, 36(4): 382-390 DOI:10.1016/j.ecoleng.2009.11.004 | |
| Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinfomatics, 2009, 25(11): 1451-1452 DOI:10.1093/bioinformatics/btp187 | |
| Liu JJ, Xu YJ, Han YP, et al. Analysis and evaluation on the nutritional components of wild Corbicula fluminea from Hongze Lake. Science and Technology of Food Industry, 2014, 35(15): 360-364 [刘晶晶, 徐云婕, 韩曜平, 等. 洪泽湖野生河蚬营养盐成分的分析与评价. 食品工业科技, 2014, 35(15): 360-364] | |
| Niu DH, Li JL, Shen HD, et al. Sequence variability of mitochondrial DNA--COI gene fragment and population genetic structure of six Sinonovacula constricta populations. Acta Oceanologica Sinica, 2008, 30(3): 109-116 [牛东红, 李家乐, 沈和定, 等. 缢蛏六群体线粒mtDNA-COI基因序列变异及群体遗传结构分析. 海洋学报, 2008, 30(3): 109-116] | |
| Park JK, Kim W. Two Corbicula (Corbiculidae: Bivalvia) mitochondrial lineages are widely distributed in Asian freshwater environment. Mology Phylogenetics and Evolution, 2003, 29(3): 529-539 DOI:10.1016/S1055-7903(03)00138-6 | |
| Pfenninger M, Reinhardt F, Streit B. Evidence for cryptic hybridization between different evolutionary lineages of the invasive clam genus Corbicula (Veneroida, Bivalvia). Journal of Evolutionary Biology, 2002, 15(5): 818-829 DOI:10.1046/j.1420-9101.2002.00440.x | |
| Qi S, Lin MX. Studies on ecological aspects of the population of Corbicula fluminea (Müller) (Mollusca) in the pearl river, Guangzhou. Acta Ecologica Sinica, 1987, 7(2): 161-169 [杞桑, 林美心. 珠江广州段河蚬种群的若干生态学研究. 生态学报, 1987, 7(2): 161-169] | |
| Renard E, Bachmann V, Cariou ML, et al. Morphological and molecular differentiation of invasive freshwater species of the genus Corbicula (Bivalvia, Corbiculidea) suggest the presence of three taxa in French rivers. Molecular Ecology, 2000, 9(12): 2009-2016 DOI:10.1046/j.1365-294X.2000.01104.x | |
| Sambrook J, Fritsch EF, Maniatis T. Molecular cloning: A laboratory manual. Beijing: Science Press, 1995: 27-29. | |
| Sousar R, Freire R, Rufino M, et al. Genetic and shell morphological variability of the invasive bivalve Corbicula fluminea (Müller, 1774) in two Portuguese estuaries. Estuarine Coastal and Shelf Science, 2007, 74(1-2): 166-174 DOI:10.1016/j.ecss.2007.04.011 | |
| Tamura K, Dudley J, Nei M, et al. MEGA 4: Molecular evolutionary genetic analysis (MEGA) software version 4.0. Molecular Biology and Evolution, 2007, 24(8): 1596-1599 DOI:10.1093/molbev/msm092 | |
| Wang GP, Zhang T, Zhang J, et al. Morphological and molecular differentiation of genus Corbicula suggests that two species are sympatrically distributed in Datong Lake in the Central Yangtze River Basin. Zoological Studies, 2014, 53(1): 1-8 DOI:10.1186/1810-522X-53-1 | |
| Wang ZQ, Zhang NH, Zhang Y, et al. Eutrophication assessment of water quality in Hongze Lake. Environmental Monitoring and Forewarning, 2010, 2(6): 31-35 [王兆群, 张宁红, 张咏, 等. 洪泽湖水质富营养化评价. 环境监测与预警, 2010, 2(6): 31-35] | |
| Yan WH, Pan YC, Hao C, et al. Investigation reports of benthos in Hongze Lake. Reservoir Fisheries, 2007, 27(3): 65-66 [严维辉, 潘元潮, 郝忱, 等. 洪泽湖底栖生物调查报告. 水利渔业, 2007, 27(3): 65-66] | |
| Yuan YH, Wang XY, Chen AL, et al. Investigation of resources of Margarya and Corbicular in Hongze Lake. Aquaculture, 1994(6): 15-16 [袁永浒, 王兴元, 陈安来, 等. 洪泽湖螺蚬资源调查报告. 水产养殖, 1994(6): 15-16] | |
| Zhang CW, Zhang TL, Zhu TB, et al. Community structure of macrozoobenthos and its relationship with enviromental factors in Lake Hongze. Journal of Hydroecology, 2012, 33(3): 27-33 [张超文, 张堂林, 朱挺兵, 等. 洪泽湖大型底栖动物群落结构及其与环境因子的关系. 水生态学杂志, 2012, 33(3): 27-33] |



