2. 海洋生态与环境科学重点实验室 中国科学院海洋研究所 青岛 266071
2. Key Laboratory of Marine Ecology and Environmental Sciences, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071
再生是动物应对环境不适的一种适应能力,且对动物存活具有重要作用,可以认为是细胞、组织或器官的重新生长过程(Rychel et al, 2009; Sánchez Alvarado et al, 2006)。目前,国内外有关无脊椎动物再生方面的研究多集中于腔肠动物门的水螅(Hydra attenuate) (Schmidt et al, 1986),扁虫动物门涡虫类扁虫(Schmidtea mediterranea)、环节动物门寡毛类蚯蚓(Enchytraeus japonensis)(Agata et al, 1999; Alvarado et al, 2002; Norman et al, 2001; Yoshida-Noro et al, 2010),棘皮动物门海盘车(Asterina Burtoni; Coscinasterias tenuispina; Oxycomanthus japonicus)和海参(Holothuria glaberrima)等(Achituv et al, 1991; Alves et al, 2002; García-Arrarás et al, 1998; Rubilar et al, 2005; Shibata et al, 2010)。
刺参(Apostichopus japonicus)是我国一种经济价值极高的海珍品。组织器官损伤后,刺参可以在短时间内完成重建。“吐脏”是刺参的一种特殊行为,遇外界刺激或遇敌后,其将肠道、呼吸树、生殖腺等内脏器官排出体外,3周左右再生出完整的、功能完善的内脏器官(孙丽娜, 2013)1)。
1) Sun LN. Histocytological events and analysis of key genes during intestine regeneration in sea cucumber Apostichopus japonicus (Selenka). Doctoral Dissertation of University of Chinese Academy of Sciences, Institute of Oceanology, Chinese Academy of Sciences, 2013, 13-31[孙丽娜.仿刺参Apostichopus japonicus (Selenka)消化道再生的组织细胞特征与关键基因分析.中国科学院研究生院(海洋研究所)博士研究生学位论文, 2013, 13-31]
目前,海参(Echinodermata, Holothuroidea)肠道再生的研究主要集中在肠壁组织层的起源与变化(Mashanov et al, 2001; Rojascartagena et al, 2007),细胞起源、迁移与增殖等细胞学事件的发生(San Miguel-Ruiz et al, 2007)。研究发现,与海参肠道再生特异性相关的基因主要有细胞骨架基因(如Actin)、和发育密切相关的基因(如Wnt和Hox)(Sun et al, 2013b)、对抗炎症反应和形态发生有重要作用的血清淀粉样蛋白A (Serum Amyloid A Protein, SAA)同源物(Santiago et al, 2000)、神经重塑有关的室管膜蛋白基因(Suarez-Castillo et al, 2004)。近年来,人们借助EST库的构建、芯片技术和高通量测序等技术,构建了EST文库(Ortiz-Pineda et al, 2009; Zheng et al, 2006)、再生转录组(Sun et al, 2013a)、基因表达谱(Sun et al, 2011),以期在更高通量上对这一多基因调控过程的机制进行探索。
HMG(High-mobility group box protein)是一种多功能细胞因子,也是一类核酸内非组蛋白类蛋白,能与特异性的受体结合,参与组织损伤修复、细胞迁移、再生等重要生命进程,并应答炎症反应等,已被证实在再生行为中发挥作用(Kuehl et al, 1979; De Mori et al, 2007; Straino et al, 2008; Limana et al, 2005),而在低等动物再生中的调控作用尚不清楚。在刺参再生转录组与表达谱研究中发现,HMG参与了刺参肠道再生过程(Sun et al, 2013a),并测定了该基因在刺参肠道再生的第3、7、14、21天的mRNA水平表达情况:再生期间HMG的表达量呈先增高后降低的趋势,再生的第7天,HMG表达量达到最大值,为对照组的4.5倍,后逐渐降低,至第21天表达量基本恢复到正常水平,推测其在肠道再生过程中使免疫系统识别损伤信号,启动修复再生功能(孙丽娜, 2013)1)。而刺参肠道再生过程中的HMG蛋白水平的表达情况还未见报道,再生早期阶段的mRNA水平的表达情况也未知。
1) Sun LN. Histocytological events and analysis of key genes during intestine regeneration in sea cucumber Apostichopus japonicus (Selenka).Doctoral Dissertation of University of Chinese Academy of Sciences, Institute of Oceanology, Chinese Academy of Sciences, 2013, 93-102 [孙丽娜.仿刺参Apostichopus japonicus (Selenka)消化道再生的组织细胞特征与关键基因分析.中国科学院研究生院(海洋研究所)博士研究生学位论文, 2013, 93-102]
本研究采用实时荧光定量PCR (qRT-PCR)技术测定HMG在刺参肠道再生第0.5、1、2、6小时及第1、3、7天mRNA水平的表达情况,并通过免疫荧光实验测定了HMG蛋白在刺参肠道再生关键时期的空间表达情况,以期更深入的探明HMG对刺参肠道再生的调控作用。
1 材料与方法 1.1 样品采集实验用体重为100–120 g的刺参于2015年11月采集于青岛。实验前将刺参在实验室水池中暂养7 d,暂养水温为15–17℃,每日换水1/2,投饵1次。暂养结束后,向刺参腹腔注射0.35 mol/L KCl溶液1–2 ml,诱导其吐脏,吐脏后的刺参养于水池中,水温为15–17℃,每日换水1/2,不投饵料。
分别在刺参吐脏后的第0.5、1、2、6小时,第1、3、7天解剖刺参,取出再生的肠道,每次取样15只刺参。将肠道组织放入冻存管中,经液氮速冻后保存于-80℃超低温冰箱,用于后续qRT-PCR实验。用同样的方法取15只未刺激吐脏刺参的正常肠道作为对照组。
同时,在吐脏后第2、6小时和第1、3、7天解剖刺参,取出再生的肠道,每次取样10只刺参。肠道置于4%多聚甲醛固定液中浸泡过夜,分别经过25%、50%、75%、100%酒精梯度脱水后,石蜡包埋,4℃冰箱暂时保存,用于后续免疫组化实验。以同样的方法取15只未刺激吐脏刺参的正常肠道作为对照组。
1.2 RNA提取及基因表达定量RNA提取实验采用TaKaRa MiniBEST Universal RNA Extraction kit (TaKaRa, 日本)试剂盒,根据说明书中的要求进行。RNA的完整性和浓度分别通过1%凝胶电泳与Nanodrop 1000 (Thermo Scientific)仪器测定。qRT-PCR实验以提取的RNA为模板,用Prime-ScriptTM RT reagent kit with gDNA eraser试剂盒(TaKaRa)反转出第一链cDNA作为后续qRT-PCR实验的模板。每组15只刺参肠道样品,5只肠道混为1个样本,每组共计3个样品。NADH脱氢酶为本研究的内参基因。实验所需引物NADH-F,NADH-R,HMG-F,HMG-R的序列(表 1)参考孙丽娜(2013)1)。实验体系及PCR程序参考SYBR PrimeScripTM RT-PCR Kit Ⅱ试剂盒(TaKaRa)说明书,设置见表 2。qRT-PCR程序:95℃预变性5 s;95℃变性10 s,60℃复性20 s,72℃延伸30 s,共40个循环;添加溶解曲线,以检测扩增产物是否单一。每个反应设置4次平行并记录好对应的Ct值,用于后续基因表达量分析。
|
|
表 1 qRT-PCR引物序列(孙丽娜, 2013)1) Table 1 Primers sequences for qRT-PCR (Sun, 2013)1) |
1) Sun LN. Histocytological events and analysis of key genes during intestine regeneration in sea cucumber Apostichopus japonicus (Selenka).Doctoral Dissertation of University of Chinese Academy of Sciences, Institute of Oceanology, Chinese Academy of Sciences, 2013, 93-102 [孙丽娜.仿刺参Apostichopus japonicus (Selenka)消化道再生的组织细胞特征与关键基因分析.中国科学院研究生院(海洋研究所)博士研究生学位论文, 2013, 93-102]
|
|
表 2 qRT-PCR体系 Table 2 Reagents for qRT-PCR |
实验前以5个稀释点(10倍稀释)的cDNA为模板,绘制HMG基因的标准反应曲线并计算PCR反应的效率(The PCR efficiency, E)和相关系数(Correlation coefficient, R2)。采用2-ΔΔCT的方法对HMG基因的相对表达量进行计算,对照组与实验组之间的差异采用SPSS 18.0进行单因素方差分析(One-way ANOVA),P < 0.05为显著差异。
1.3 免疫组化将包埋好的刺参肠道标本进行切片处理,切好的蜡片贴附于多聚赖氨酸处理过的载玻片上,60℃烘5 h使石蜡融化。免疫组化具体实验步骤:将石蜡切片置于二甲苯中脱蜡5 min,重复2次;石蜡切片分别经过100%乙醇1 min、95%乙醇1 min、75%乙醇1 min、50%乙醇1 min、25%乙醇1 min、蒸馏水2 min、PBS 2 min×2次;将切片置于柠檬酸钠抗原修复液中,微波炉中火加热6 min至微沸,重复4次;将切片置于1%牛血清蛋白中,室温轻摇封闭30 min;一抗以1: 1000稀释于封闭液中,在切片上滴加稀释好的一抗孵育液,于湿盒中37℃孵育1 h,实验用抗体为Anti-HMG rabbit polyclonal antibody (BBI Life Science);用PBS充分清洗3次,每次10 min,用滤纸擦去标本外的PBS;荧光二抗以1: 2000稀释于PBS中,在切片上滴加稀释好的二抗孵育液,于湿盒中37℃避光孵育45 min,实验用抗体为Alexa Fluor 594-conjugated AffiniPure Goat Anti-Rabbit IgG (H+L);用PBS充分清洗3次,每次10 min,用滤纸擦去标本外的PBS;用含抗荧光淬灭剂的封片液封片,在荧光显微镜下立即观察。
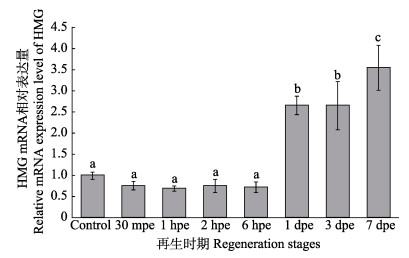
2 结果 2.1 刺参肠道再生初始阶段HMG mRNA的表达利用qRT-PCR技术分析测定了HMG mRNA在刺参肠道再生初始阶段的表达情况(图 1)。qRT-PCR最后的溶解曲线为单峰,证明扩增的PCR产物具有特异性。
|
图 1 刺参肠道HMG基因相对表达量 Figure 1 Relative expression levels of intestine HMG of A. japonicus Control:未经处理的肠道组织;30 mpe、1 hpe、2 hpe、6 hpe、1 dpe、3 dpe和7 dpe分别为刺激排脏后再生30 min、1 h、2 h、6 h、1 d、3 d、7 d的肠道组织;不同字母表示差异显著(P < 0.05),纵坐标值为平均值±标准差(n=3) Control: Non-eviscerated sea cucumbers; 30 mpe, 1 hpe, 2 hpe, 6 hpe, 1 dpe, 3 dpe, and 7 dpe were 30 minutes, 1 hour, 2 hours, 6 hours, 1 day, 3 days, and 7 days post-evisceration, respectively. Different letters indicated significant differences (P < 0.05). The values of ordinate were shown as Mean±SD (n=3) |
qRT-PCR结果显示,在刺参肠道再生的较早期阶段(吐脏后第30分钟、第1、2、6小时),HMG mRNA的相对表达量与正常刺参肠道相比并无显著变化;从再生第1天开始,该基因的表达量显著升高,再生第7天达到最大值(图 1)。
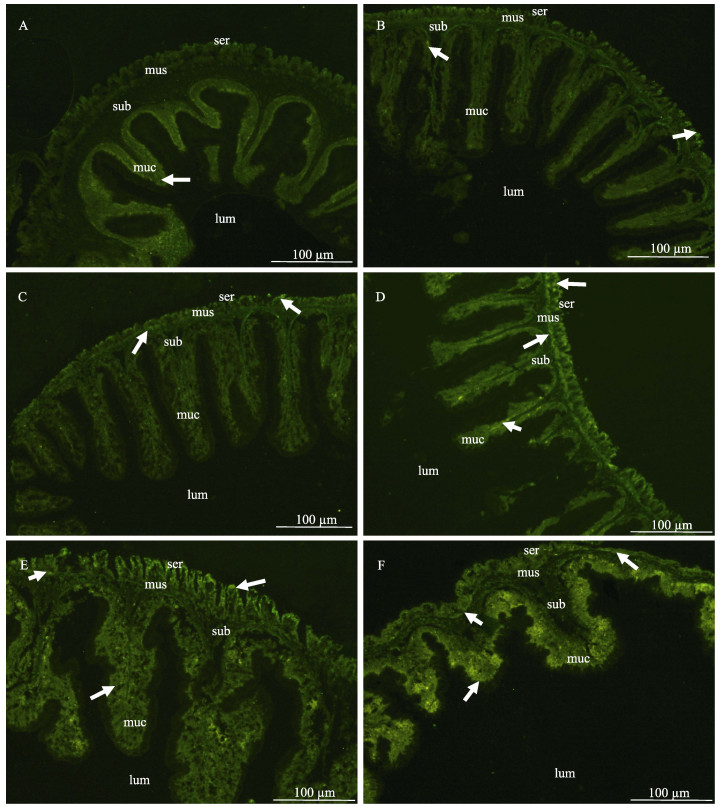
2.2 刺参肠道再生各阶段HMG蛋白表达情况利用免疫荧光技术,用多克隆抗体分别与再生各阶段的刺参肠道组织切片中的相应蛋白进行免疫反应,分析HMG蛋白在刺参肠道再生各阶段的空间表达情况,免疫荧光显色结果见图 2(绿色荧光为杂交信号)。对照组刺参肠道各层(从外向内分别为:浆膜层、肌肉层、黏膜下层、黏膜层)分化明显,黏膜下层为最厚层,肌肉层较薄,HMG蛋白荧光信号较弱,主要集中于黏膜下层。再生过程中,肠道组织结构发生巨大变化,肠壁各层组织明显变薄且较为杂乱,分化不明显。再生第2、6小时,HMG蛋白荧光信号弱,仅在浆膜层和黏膜层有少量分布。再生第1–7天,HMG蛋白荧光信号逐渐增强,再生第1天,HMG荧光主要分布在浆膜层和肌肉层,再生第3、7天,荧光信号强,各层均有分布。
|
图 2 再生过程中刺参HMG在肠道组织的表达分布 Figure 2 Expression of A. japonicus HMG protein at different regeneration stages A:对照组;B:再生第2小时;C:再生第6小时;D:再生第1天;E:再生第3天;F:再生第7天;箭头表示荧光信号 A: Control; B: 2 hours post-evisceration (hpe); C: 6 hpe; D: 1 day post-evisceration (dpe); E: 3 dpe, F: 7 dpe; Arrows indicated fluorescence signals; ser:浆膜层Serosa; mus:肌肉层Muscle; sub:黏膜下层Submucosa; muc:黏膜层Mucosa; lum:肠腔Intestinal lumen |
HMG是一种多功能的细胞因子,对高等动物胚胎发育、血管发生、血管再生、神经突生长、干细胞迁移、肿瘤发生等多种生物学进程都有重要作用(Degryse et al, 2001; Kim et al, 2006; Palumbo et al, 2004)。HMG已被证实在许多再生行为中发挥重要作用,如大鼠(Rattus norvegicus)肝脏再生(Kuehl et al, 1979)和骨骼肌肌肉再生(De Mori et al, 2007)、成年人皮肤再生(Straino et al, 2008)以及心肌再生(Limana et al, 2005)等,而HMG对低等动物再生的调控作用尚未知。
HMG mRNA表达量分析显示,HMG在刺参正常的肠道中表达,表明其基因产物是一种维持正常生命活动的转录因子。再生第30分钟,第1、2、6小时,HMG mRNA的表达量与正常刺参肠道相比并无显著变化;免疫荧光结果显示,肠道再生第2、6小时与对照组HMG蛋白的表达情况并无明显变化,这与HMG mRNA表达情况一致。结合刺参HMG mRNA水平和蛋白的表达情况来看,在肠道再生第1天以前,HMG可能并不发挥调控作用。再生的第1、3、7天,该基因的mRNA表达量显著高于正常生理状态下,于第7天达到最大值,这与孙丽娜等(2013)1)测定的HMG在刺参肠道再生过程中的mRNA表达情况相一致;HMG蛋白荧光信号也逐渐增强,浆膜层、肌肉层、黏膜下层、黏膜层均有荧光信号分布,到第7天达到荧光亮度的最大值,表明HMG蛋白在这一阶段大量积累。
1) Sun LN. Histocytological events and analysis of key genes during intestine regeneration in sea cucumber Apostichopus japonicus (Selenka). Doctoral Dissertation of University of Chinese Academy of Sciences, Institute of Oceanology, Chinese Academy of Sciences, 2013, 93-102 [孙丽娜.仿刺参Apostichopus japonicus (Selenka)消化道再生的组织细胞特征与关键基因分析.中国科学院研究生院(海洋研究所)博士学位论文, 2013, 93-102]
在动物再生的过程中,细胞外基质(Extracellular matrix, ECM)成分往往会发生剧烈变化,ECM降解与重建可以调控细胞活性和细胞迁移与分化(Brown et al, 2014)。高等动物HMG与再生相关研究表明,HMG在组织受损修复的过程中可以通过激活糖基化终产物受体来调控ECM中胶原的合成与降解(Zhang et al, 2012)。被破坏的或者凋亡的细胞中,HMG产物可以“被动释放”至细胞外基质中;ECM中的HMG蛋白成为被先天免疫系统识别的“损伤标记物”,当其含量过多时证实组织受损伤,启动修复功能(Ulloa et al, 2006)。从本研究中可以看出,从刺参肠道再生第1天开始,HMG表达量相比对照组增多,很有可能作为“损伤标记物”被先天免疫系统识别,启动肠道再生修复的功能。
研究发现,在组织修复再生的过程中,HMG可以激活干细胞,通过其蛋白浓度效应诱导干细胞迁移通过内皮屏障,进入健康正常的组织中分化成普通的细胞(Palumbo et al, 2004)。刺参肠道再生第0-2天为肠系膜伤口愈合阶段,第2-5天为原基形成阶段,一般认为,原基的形成是刺参肠道再生真正的起点。在刺参肠道再生早期,新生组织的细胞来源于细胞去分化和细胞迁移,原基细胞主要来源于肠系膜的间充质干细胞(Yang et al, 2015)。HMG从肠道再生第1天开始表达量逐渐升高,因此推测,HMG可能对再生早期原基形成过程中的干细胞迁移起作用。Zhang等(2012)研究发现,在小鼠(Mus musculus)上皮组织伤口愈合过程中,HMG在多种促炎因子的作用下释放到细胞外基质中,在伤口愈合结束后又反过来诱导受损部位的细胞分泌后续免疫反应的相关调控因子及巨噬细胞免疫蛋白,HMG可能在伤口愈合阶段(第0-2天)过后也参与了细胞免疫过程。
结合HMG在其他物种组织修复再生中的作用与其在刺参肠道再生过程中的表达变化来看,推测HMG可能使免疫系统识别损伤信号,启动修复再生功能,调控ECM成分的降解与生成,诱导干细胞迁移、分化,并在肠道再生后期参与细胞免疫。
| Achituv Y, Sher E. Sexual reproduction and fission in the sea star Asterina burtoni from the Mediterranean coast of Israel. Bulletin of Marine Science, 1991, 48(3): 670-678 | |
| Agata K, Watanabe K. Molecular and cellular aspects of planarian regeneration. Seminars in Cell and Developmental Biology, 1999, 10(4): 377-383 DOI:10.1006/scdb.1999.0324 | |
| Alvarado AS. The Schmidtea mediterranea database as a molecular resource for studying platyhelminthes, stem cells and regeneration. Development, 2002, 129(24): 5659-5665 DOI:10.1242/dev.00167 | |
| Alves L, Pereira A, Ventura C. Sexual and asexual reproduction of Coscinasterias tenuispina (Echinodermata: Asteroidea) from Rio de Janeiro, Brazil. Marine Biology, 2002, 140(1): 95-101 DOI:10.1007/s002270100663 | |
| Brown BN, Badylak SF. Extracellular matrix as an inductive scaffold for functional tissue reconstruction. Translational Research, 2014, 163(4): 268-285 DOI:10.1016/j.trsl.2013.11.003 | |
| De Mori R, Straino S, Di Carlo A, et al. Multiple effects of high mobility group box protein 1 in skeletal muscle regeneration. Arteriosclerosis, Thrombosis, and Vascular Biology, 2007, 27(11): 2377-2383 DOI:10.1161/ATVBAHA.107.153429 | |
| Degryse B, Bonaldi T, Scaffidi P, et al. The high mobility group (HMG) boxes of the nuclear protein HMG1 induce chemotaxis and cytoskeleton reorganization in rat smooth muscle cells. Journal of Cell Biology, 2001, 152(6): 1197-1206 DOI:10.1083/jcb.152.6.1197 | |
| García-Arrarás JE, Estrada-Rodgers L, Santiago R, et al. Cellular mechanisms of intestine regeneration in the sea cucumber, Holothuria glaberrima Selenka (Holothuroidea: Echinodermata). Journal of Experimental Zoology, 1998, 281(4): 288-304 DOI:10.1002/(ISSN)1097-010X | |
| Kim JB, Choi JS, Yu YM, et al. HMGB1, a novel cytokine-like mediator linking acute neuronal death and delayed neuroinflammation in the postischemic brain. Journal of Neuroscience, 2006, 26(24): 6413-6421 DOI:10.1523/JNEUROSCI.3815-05.2006 | |
| Kuehl L. Synthesis of high mobility group proteins in regenerating rat liver. Journal of Biological Chemistry, 1979, 254(15): 7276-7281 | |
| Limana F, Germani A, Zacheo A, et al. Exogenous high-mobility group box 1 protein induces myocardial regeneration after infarction via enhanced cardiac C-kit+ cell proliferation and differentiation. Circulation Research, 2005, 97(8): e73-e83 DOI:10.1161/01.RES.0000186276.06104.04 | |
| Mashanov VS, Dolmatov IY. Regeneration of digestive tract in the pentactulae of the far-eastern holothurian Eupentacta fraudatrix (Holothuroidea, Dendrochirota). Invertebrate Reproduction & Development, 2001, 39(2): 143-151 | |
| Norman MD, Finn J. Revision of the Octopus horridus species-group, including erection of a new subgenus and description of two member species from the Great Barrier Reef, Australia. Invertebrate Systematics, 2001, 15(1): 13-35 DOI:10.1071/IT99018 | |
| Ortiz-Pineda PA, Ramírez-Gómez F, Pérez-Ortiz J, et al. Gene expression profiling of intestinal regeneration in the sea cucumber. BMC Genomics, 2009, 11061106(10): 262 | |
| Palumbo R, Bianchi ME. High mobility group box 1 protein, a cue for stem cell recruitment. Biochemical Pharmacology, 2004, 68(6): 1165-1170 DOI:10.1016/j.bcp.2004.03.048 | |
| Rojascartagena C, Ortizpineda PA, Ramirezgomez F, et al. Distinct profiles of expressed sequence tags during intestinal regeneration in the sea cucumber Holothuria glaberrima. Physiological Genomics, 2007, 31(2): 203-215 DOI:10.1152/physiolgenomics.00228.2006 | |
| Rubilar T, de Ward CTP, de Vivar MED. Sexual and asexual reproduction of Allostichaster capensis (Echinodermata: Asteroidea) in Golfo Nuevo. Marine Biology, 2005, 146(6): 1083-1090 DOI:10.1007/s00227-004-1530-4 | |
| Rychel AL, Swalla BJ. Regeneration in hemichordates and echinoderms. Stem Cells in Marine Organisms, 2009: 245-265 | |
| Sánchez Alvarado A, Tsonis PA. Bridging the regeneration gap: Genetic insights from diverse animal models. Nature Reviews Genetics, 2006, 7(11): 873-884 DOI:10.1038/nrg1923 | |
| Santiago P, Roig-López JL, Santiago C, et al. Serum amyloid A protein in an echinoderm: Its primary structure and expression during intestinal regeneration in the sea cucumber Holothuria glaberrima. Journal of Experimental Zoology, 2000, 288(4): 335-344 DOI:10.1002/(ISSN)1097-010X | |
| San Miguel-Ruiz JE, García-Arrarás JE. Common cellular events occur during wound healing and organ regeneration in the sea cucumber Holothuria glaberrima. BMC Developmental Biology, 2007, 11061106(7): 115 | |
| Schmidt T, David CN. Gland cells in Hydra: Cell cycle kinetics and development. Journal of Cell Science, 1986, 85(1): 197-215 | |
| Shibata TF, Oji T, Akasaka K, et al. Staging of regeneration process of an arm of the feather star Oxycomanthus japonicus focusing on the oral-aboral boundary. Developmental Dynamics, 2010, 239(11): 2947-2961 DOI:10.1002/dvdy.v239:11 | |
| Straino S, Di Carlo A, Mangoni A, et al. High-mobility group box 1 protein in human and murine skin: Involvement in wound healing. Journal of Investigative Dermatology, 2008, 128(6): 1545-1553 DOI:10.1038/sj.jid.5701212 | |
| Suárez-Castillo EC, Medina-Ortiz WE, Roig-López JL, et al. Ependymin, a gene involved in regeneration and neuroplasticity in vertebrates, is overexpressed during regeneration in the echinoderm Holothuria glaberrima. Gene, 2004, 334: 133-143 DOI:10.1016/j.gene.2004.03.023 | |
| Sun L, Chen M, Yang H, et al. Large scale gene expression profiling during intestine and body wall regeneration in the sea cucumber Apostichopus japonicus. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2011, 6(2): 195-205 DOI:10.1016/j.cbd.2011.03.002 | |
| Sun L, Yang H, Chen M, et al. RNA-Seq reveals dynamic changes of gene expression in key stages of intestine regeneration in the sea cucumber Apostichopus japonicas. PLoS One, 2013a, 8(8): e69441 DOI:10.1371/journal.pone.0069441 | |
| Sun LN, Yang HS, Chen MY, et al. Cloning and expression analysis of Wnt6 and Hox6 during intestinal regeneration in the sea cucumber Apostichopus japonicus. Genetics Molecular Research, 2013b, 12(4): 5321-34 DOI:10.4238/2013.November.7.7 | |
| Ulloa L, Messmer D. High-mobility group box 1 (HMGB1) protein: Friend and foe. Cytokine & Growth Factor Reviews, 2006, 17(3): 189-201 | |
| Yang H, Hamel JF, Mercier A. The sea cucumber Apostichopus japonicus: History, biology and aquaculture. Amsterdam: Academic Press, 2015. | |
| Yoshida-Noro C, Tochinai S. Stem cell system in asexual and sexual reproduction of Enchytraeus japonensis (Oligochaeta, Annelida). Development, Growth and Differentiation, 2010, 52(1): 43-55 | |
| Zhang Q, O'Hearn S, Kavalukas SL, et al. Role of high mobility group box 1 (HMGB1) in wound healing. Journal of Surgical Research, 2012, 176(1): 343-347 DOI:10.1016/j.jss.2011.06.069 | |
| Zheng FX, Sun XQ, Fang BH, et al. Comparative analysis of genes expressed in regenerating intestine and non-eviscerated intestine of Apostichopus japonicus Selenka (Aspidochirotida: Stichopodidae) and cloning of ependymin gene. Hydrobiologia, 2006, 571(1): 109-122 DOI:10.1007/s10750-006-0231-z |



