2. 上海海洋大学水产与生命学院 上海 201306
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
魁蚶(Scapharca broughtonii),俗称大毛蛤、赤贝、血贝,属埋栖型冷水性贝类,多栖息于底质为泥质粉砂和砂质粉砂的海区(张玉玺等, 1991)1)。在我国黄海北部资源量丰富,主要分布于山东、辽宁等沿海,是我国北方重要的经济贝类之一。因魁蚶肉质鲜美、营养及药用价值高,在中国及韩国市场深受欢迎。但捕捞过度、环境恶化等因素导致魁蚶自然资源量不断衰减、遗传多样性水平降低。深入开展魁蚶种质资源调查,查明不同群体间的遗传结构,是进行种质改良的前提和基础。本课题组已经开展了一些有关魁蚶种质资源调查等方面的研究,运用多变量形态度量学分析方法、RAPD和SSR标记技术,对韩国和我国沿海4个魁蚶地理群体的遗传变异和遗传多样性情况进行了研究(梁超等, 2010; 吴彪等, 2012),结果显示,4个群体遗传多样性丰富,有较大的育种潜力;对4个魁蚶地理群体样本的核糖体RNA的2个内转录间隔区域(ITS-1和ITS-2)进行扩增,发现各群体之间存在一定的遗传差异,综合考虑不同群体相对优良的生物学特征,可开展选择或杂交培育良种的研究(周丽青等, 2012)。另外,An等(2005)用10个多态微卫星位点对魁蚶进行了遗传多样性分析,等位基因的数量从11到23不等,群体遗传学分析显示,10个位点之间不存在连锁不平衡的现象;孙楠等(2015)利用微卫星标记对魁蚶混交家系进行了鉴定。但利用线粒体基因和核糖体基因进行魁蚶种质资源分析的相关研究报道较少。
1) Zhang YX, Zhou XJ, Jiang HB, et al. Study on the habits and modes of life in the natural environment of Scapharca broughtonii (Shrenck) in Shandong offshore. Transactions of the Chinese Society of Malacology, 1991 [张玉玺, 周学家, 姜海滨, 等.山东近海魁蚶生态习性的调查.中国动物学会、中国海洋湖沼学会贝类学分会第四次代表大会暨第五次学术讨论会论文集, 1991]
线粒体基因组具有突变率高、分子量小、无组织特异性的特点,在分子系统发育中应用广泛。COⅠ(线粒体细胞色素C氧化酶亚基Ⅰ, coxidase Ⅰ)基因通用性比较高,可操作性强(侯丫等, 2016),可以提供丰富的遗传信息,已成功应用于遗传分析和系统进化研究中(Chen et al, 2009; Shen et al, 2009; Zheng et al, 2009)。12S rRNA基因具有高度的保守性,在贝类的分子系统学研究中发挥着重要的作用(刘春芳, 20141); 冯艳微, 20122))。18S rRNA和28S rRNA在生物体内含量较大,且在进化过程中具有保守性,因此,适合不同层次系统发育的研究,是研究生命起源和早期生物进化、高级分类阶元系统发育关系的主要分子标记,已广泛应用于动植物的种质鉴定和系统发育研究。薛东秀等(2012)从GenBank下载翼形亚纲11个总科80个种类的28S部分序列,进行系统发育关系研究,取得较好结果。
1) Liu CF. Phylogenetic analysis of Arcoidea and genetic diversity of different populations of Tegillarca granosa along Chinese coasts. Master's Thesis of Chinese Academy of Sciences (Institute of Oceanology), 2014, 32-54 [刘春芳.中国近海蚶总科(Arcoidea)贝类的系统发育及泥蚶(Tegillarca granosa)不同群体遗传多样性研究.中国科学院研究生院(海洋研究所)硕士研究生学位论文, 2014, 32-54]
2) Feng YW. DNA barcoding and molecular phylogeny of the Pteriina and Arcoida. Doctoral Dissertation of Ocean University of, 96-101 [冯艳微.珍珠贝亚目和蚶目DNA条形码与系统发生学研究.中国海洋大学博士研究生学位论文, 2012, 96-101]
本研究对魁蚶5个地理群体进行COⅠ、12S、18S和28S序列变异分析及系统进化分析,旨在明确魁蚶群体遗传多样性水平和不同地理群体的系统进化关系,为魁蚶资源保护及种质改良提供基础资料。
1 材料与方法 1.1 实验材料实验所用的5个地理群体的魁蚶分别取自山东青岛胶南(J)、山东荣成(R)、山东长岛(C)、山东蓬莱(P)、韩国统营(H),样品均为近海野生个体,低温运至实验室迅速解剖,取闭壳肌,置于-80℃超低温冰箱保存。
1.2 基因组DNA提取和PCR扩增每个群体随机取15个样品,采用常规酚-氯仿法提取基因组DNA,实验用DNA模板稀释至100 ng/μl。运用PCR扩增魁蚶COⅠ、12S、18S和28S基因,所用引物序列见表 1。PCR反应体系为30 μl:3 μl PCR 10×buffer,3 μl高纯度的dNTP (2.5 mmol/L),10 μmol/L的正反方向引物各1 μl,0.5 μl Easy Taq (5 U/μl),1 μl DNA(80 ng/μl),20.5 μl灭菌水。PCR反应程序:94℃预变性5 min;35个循环(94℃变性20 s, 52℃退火45 s, 72℃延伸1 min);最后,72℃延伸10 min。得到清晰、单一的目的片段,所得PCR产物用1%琼脂糖凝胶电泳检测(1×TAE, 130 V, 30 min),然后利用凝胶成像系统进行观察拍照。切取目的条带、胶回收,将目的片段与T载体连接,转化感受态细胞,进行单克隆,用M13通用引物进行双向测序。其中,8个个体没有扩增出COⅠ条带,3个个体没有扩增出12S条带。
|
|
表 1 COⅠ、12S、18S和28S引物序列 Table 1 Primer sequences of COⅠ, 12S, 18S, and 28S |
先用Chromas检查所得序列的测序质量,并在NCBI数据库中进行Blast比对,验证其同源性。运用BioEdit和ClustalX 1.8软件进行序列比对,利用DnaSp软件分析魁蚶群体单倍型数(Number of haplotype, H)、平均核苷酸差异(Average number of nucleotide difference, K)及核苷酸多样性指数(Nucleotide diversity, Pi)等遗传多样性参数,运用MEGA 5.0软件对序列的碱基组成(Nucleotide composition)、多态位点数(Number of polymorphic sites, S)、变异位点(Variable sites)进行分析,采用NJ (Neighbor-joining)法构建魁蚶5个地理群体的系统发育树。
2 结果与分析 2.1 COⅠ、12S、18S和28S序列的碱基组成及变异分析测序后获得的COⅠ、12S、18S和28S基因片段长短不一,经序列比对去除序列两端部分碱基后,分别得到67条长度均为776 bp的COⅠ序列、72条长度为443 bp的12S序列、75条长度均为909 bp的18S基因片段、75条长度均为894 bp的28S基因片段。魁蚶5个群体的COⅠ、12S、18S和28S基因部分片段的碱基组成见表 2、表 3。5个群体的18S基因部分序列碱基组成比例相对稳定,COⅠ基因部分序列A+T含量要比12S、18S和28S的A+T含量高,而C的含量最少,28S基因部分序列中,G的含量相对较高。
|
|
表 2 魁蚶不同群体COⅠ和12S基因部分序列的碱基组成 Table 2 Base compositions of partial sequences of COⅠ and 12S gene of five populations of S. broughtii |
|
|
表 3 魁蚶不同群体18S和28S基因部分序列的碱基组成 Table 3 Base compositions of partial sequences of 18S and 28S gene of five populations of S. brouhtonii |
5个群体间的COⅠ、12S、18S和28S的核苷酸遗传多样性参数见表 4。从5个群体67个个体的COⅠ序列中共检测到466个多态位点、32种单倍型、单倍型多样性指数为0.925,说明5个群体的COⅠ基因单倍型很丰富。核苷酸多样性指数为0.086,其中,韩国群体的多态位点(S)和平均核苷酸差异数(K)高于其他群体。12S序列共检测到292个多态位点、43种单倍型,其中,单倍型3有7个个体,分别是荣成(R)群体3个、长岛(C)群体7个、蓬莱(P)群体7个。18S序列中共检测到909个多态位点、74种单倍型,单倍型多样性指数为1.000,说明5个群体的18S单倍型比较丰富,核苷酸多样性指数也很丰富(0.717)。基于18S序列,胶南群体的多态位点最多,核苷酸多样性指数也最丰富,蓬莱群体的多态性最低。从5个群体75个个体的28S序列中共检测到27个多态位点、18种单倍型,其中,单倍型8有26个个体,分别是长岛(C)群体7个,韩国(H)群体5个,蓬莱(P)群体2个,胶南(J)群体5个,荣成(R)群体12个,单倍型多样性指数为0.778,核苷酸多样性指数为0.289。其中,蓬莱群体的多态性是最高的,其次是荣成群体,韩国群体的多态性最低。
|
|
表 4 魁蚶不同群体COⅠ、12S、18S和28S基因片段的遗传多样性指数 Table 4 The genetic diversity indices of partial COⅠ, 12S, 18S, and 28S in different population of S. broughonii |
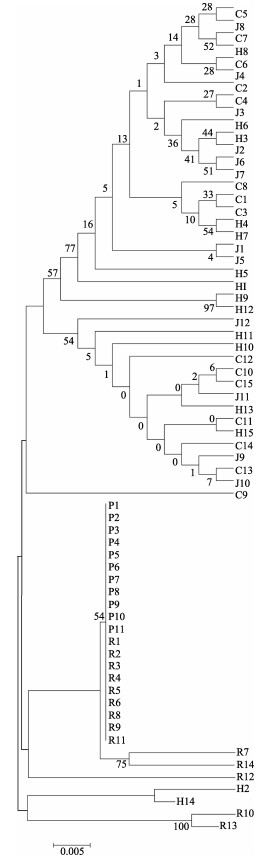
用MEGA 5.0软件计算5个群体内和群体间的遗传距离(表 5和表 6),构建5个群体个体间基于COⅠ、12S、18S和28S的NJ分子系统树。COⅠ和12S分析结果显示,群体内遗传距离大多小于群体间遗传距离,且韩国群体与其他4个群体的遗传距离都较大,其中,12S序列,韩国群体与其他4个群体的遗传距离较远。18S序列的分析结果显示,5个群体间的遗传距离很小,群体内遗传距离甚至高于群体间遗传距离。韩国群体与其他4个群体间的遗传距离相对较大。在28S序列中,群体间的遗传距离均较大,胶南群体和蓬莱群体间的遗传距离最远。COⅠ的NJ系统进化树见图 1。虽然韩国群体遗传距离与其他4个群体较大,但聚类分析显示,韩国群体并没有形成单独一支。
|
|
表 5 基于COⅠ和12S序列的5个魁蚶群体间和群体内的遗传距离 Table 5 Genetic distances between and within five different populations of S. broughtonii |
|
|
表 6 基于18S和28S序列的5个魁蚶群体间和群体内的遗传距离 Table 6 Genetic distances between and within five different populations of S. broughtonii |
|
图 1 基于魁蚶5个群体COⅠ部分序列建立的NJ系统发育关系 Figure 1 Phylogenetic relationships among five groups of S. broughtonii based on the partial sequences of COⅠ gene |
遗传多样性是指生物所携带遗传信息的总和,包括遗传变异程度及其分布格局,即群体的遗传结构,是物种适应多变的环境条件、维持长期生存和进化的遗传基础,是评价群体资源状况的一个重要依据。某个种群的遗传变异越丰富,其对生存环境的适应能力就越强,进化的潜力也越大(张文静等, 2003; 马波等, 2009)。本研究中,基于COⅠ和12S基因的单倍型非常丰富,分别是32种和43种,尤其是韩国群体,多态位点和平均核苷酸差异数都很多。基于18S序列,5个群体都表现出丰富的遗传多样性,共有74种单倍型,胶南群体多态位点有488个,平均核苷酸差异指数是69.724,核酸多样性指数是0.080,相比于其他群体,此群体的变异位点最多,多样性也最丰富。基于28S序列,75个个体共有18种单倍型,蓬莱群体多样性最丰富。周丽青等(2012)对山东蓬莱、山东黄岛、江苏前三岛及韩国统营4个地理群体魁蚶样本的核糖体RNA两个内转录间隔区域(ITS-1和ITS-2)进行扩增,序列比对结果显示,ITS区序列变异相对较高,4个群体的ITS区序列均表现出丰富的遗传多样性,这与本研究结果一致。
遗传距离是起源于共同祖先的相同基因进化趋异的一种测度(张爱兵等, 2002)。在此假定下,遗传距离通常指由基因频率的函数表示群体间的差异(Nei, 1987),根据其大小可以确定群体间亲缘关系的远近。群体间的遗传距离越大,则基因型差异越大,表明所比较群体的亲缘关系越远,遗传相似性越小;反之亦然(田吉腾等, 2013)。本研究中,魁蚶5个群体间的遗传距离在0.001-3.229之间,韩国群体与其他4个群体的遗传距离相对较大。这与吴彪等(2012)、田吉腾等(2013)的结果较一致,均表现出韩国群体与中国群体遗传距离相差较大。
3.2 基于COⅠ、12S、18S和28S序列特征进行群体鉴定和遗传多样性分析的可行性DNA碱基序列分析可以准确地检测出碱基替换、插入和缺失等变异信息,利用这些变异数据来研究个体或者群体的遗传结构,不仅可以获得丰富的遗传变异资料,而且还可以准确地揭秘种群间进化关系,甚至更准确地鉴别不同的个体(周丽青等, 2012)。线粒体COⅠ基因是DNA条形码技术中常用的分子标记(冯艳微, 20111); 孟玮等, 2010; 胡玉婷等, 2016),它蕴含着丰富的遗传进化信息,可以用来进行物种鉴定和隐存种的发现(陈双雅等, 2012; 张馨月等, 2014),通过分析物种的种间和种内遗传距离,来判断亲缘关系的远近。18S和28S基因都是高度保守的,适合研究较高阶元的系统发生(Hwang et al, 1999),也是近年来在海洋贝类分类中研究利用较多的序列之一(Carapelli et al, 2000)。吴文健等(2010)对9个群体的石磺科贝类进行外部形态特征差异分析和内部结构比较,在初步分类基础上利用18S rRNA基因部分序列对9个群体进行系统发育分析,探讨我国大陆沿海石磺科属种间的亲缘关系;孟学平等(2011)利用18S来探讨双壳纲贝类的系统发生关系,发现18S序列有插入∕缺失序列,存在长度多态性;Giribet等(2005)使用18S rDNA、28S rDNA、COⅠ基因以及形态学数据证实了蚶总科的单系性。
1) Feng YW. DNA barcoding and molecular phylogeny of the Pteriina and Arcoida. Doctoral Dissertation of Ocean University of China, 2012, 96–101 [冯艳微.珍珠贝亚目和蚶目DNA条形码与系统发生学研究.中国海洋大学博士研究生学位论文, 2012, 96-101]
本研究表明,基于COⅠ和12S基因,5个群体均表现出较高的遗传多样性,相比于其他4个群体,韩国群体的遗传多样性较丰富。18S和28S序列的遗传信息并不完全一致,序列变异频率也存在差异。因此,为进一步提高遗传多样性评估的准确性,选择进化过程中变异比较大的基因序列,能够更加全面地分析物种的进化和遗传多样性。
| An HS, Park JY. Ten new highly polymorphic microsatellite loci in the blood dam Scapharca broughtonii. Molecular Ecology Notes, 2005, 5(4): 896-898 DOI:10.1111/men.2005.5.issue-4 | |
| Carapelli A, Frati F, Nardi F, et al. Molecular phylogeny of the apterygotan insects based on nuclear and mitochondrial genes. Pedobiologia, 2000, 44(3-4): 361-373 DOI:10.1078/S0031-4056(04)70055-4 | |
| Chen AH, Li ZX, Feng GN. Phylogenetic relationships of the genus Meretrix (Mollusca: Veneridae) based on mitochondrial COⅠ gene sequences. Zoology Research, 2009, 30(3): 233-239 DOI:10.3724/SP.J.1141.2009.03233 | |
| Chen SY, Wang JH, Chen WL, et al. Application of 16S rRNA and COⅠ gene sequence analysis on grouper species identification. Biotechnology Bulletin, 2012, 11061106(10): 124-130 [陈双雅, 王嘉鹤, 陈伟玲, 等. 16S rRNA基因和COⅠ基因序列分析在石斑鱼物种鉴定中的应用. 生物技术通报, 2012, 11061106(10): 124-130] | |
| Giribet G, Wheeler W. On bivalve phylogeny: A high-level analysis of the Bivalvia (Mollusca) based on combined morphology and DNA sequence data. Invertebrate Biology, 2005, 121(4): 271-324 DOI:10.1111/ivb.2002.121.issue-4 | |
| Hou Y, Tian JT, Liu ZH, et al. Genetic diversity of different populations of Scapharca subcrenata based on mitochondrial COⅠ gene. Marine Sciences, 2016, 40(1): 1-9 DOI:10.11759/hykx20140429002 [侯丫, 田吉腾, 刘志鸿, 等. 基于线粒体COⅠ基因的毛蚶群体遗传多样性. 海洋科学, 2016, 40(1): 1-9] | |
| Hu YT, Jiang H, Pan TS, et al. Genetic differentiation of Monopterus albus populations from Anhui Province in Yangtze River basin based on mitochondrial COⅠ barcode sequence. Journal of Northeast Agricultural University, 2016, 47(2): 74-80 [胡玉婷, 江河, 潘庭双, 等. 基于线粒体COⅠ序列探讨安徽长江流域黄鳝群体遗传分化. 东北农业大学学报, 2016, 47(2): 74-80] | |
| Hwang UW, Kim W. General properties and phylogenetic utilities of nuclear ribosomal DNA and mitochondrial DNA commonly used in molecular systematics. Korean Journal of Parasitology, 1999, 37(4): 215-228 DOI:10.3347/kjp.1999.37.4.215 | |
| Liang C, Yang AG, Liu ZH, et al. RAPD analysis of genetic variations in four geographical populations of Scapharea broughtonii. Progress in Fishery Sciences, 2010, 31(1): 59-64 [梁超, 杨爱国, 刘志鸿, 等. 魁蚶4个地理群体遗传结构的RAPD分析. 渔业科学进展, 2010, 31(1): 59-64] | |
| Ma B, Jiang ZF, Huo TB. Analysis of genetic variation and geographic divergence of Thymallus tugarinae by microsatellite. Journal of Fishery Sciences of China, 2009, 16(5): 678-688 [马波, 姜作发, 霍堂斌. 下游黑龙江茴鱼种群遗传变异及地理分化的微卫星分析. 中国水产科学, 2009, 16(5): 678-688] | |
| Matsumoto M. Phylogenetic analysis of the subclass Pteriomorphia (Bivalvia) from mtDNA COⅠ sequences. Molecular Phylogenetics & Evolution, 2003, 27(3): 429-440 | |
| Meng W, Yang TY, Hai S, et al. Study of DNA barcoding based on the mitochondrial COⅠ gene sequences in salmo Trutta fraio. Chinese Journal of Fisheries, 2010, 23(1): 6-10 [孟玮, 杨天燕, 海萨, 等. 基于线粒体COⅠ基因序列的亚东鲑DNA条形码研究. 水产学杂志, 2010, 23(1): 6-10] | |
| Meng XP, Shen X, Cheng HL, et al. 18S rRNA gene variation and phylogenetic analysis among 6 Orders of Bivalvia Class. Acta Ecologica Sinica, 2011, 31(5): 1393-1403 [孟学平, 申欣, 程汉良, 等. 双壳纲贝类18S rRNA基因序列变异及系统发生. 生态学报, 2011, 31(5): 1393-1403] | |
| Nei M. Molecular evolutionary genetics. Columbia University Press, 1987: 599-599. | |
| Okusu A, Schwabe E, Eernisse DJ, et al. Towards a phylogeny of chitons (Mollusca, Polyplacophora) based on combined analysis of five molecular loci. Organisms Diversity & Evolution, 2003, 3(4): 281-302 | |
| Shen YB, Li JL, Feng BB. Genetic analysis of cultured and wild populations of Mytilus coruscus based on mitochondrial DNA. Zoology Research, 2009, 30(3): 240-246 DOI:10.3724/SP.J.1141.2009.03240 | |
| Simon C, Frati F, Beckenbach A, et al. Evolution, weighting and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers. Annals of the Entomological Society of America, 1994, 87(6): 651-701 DOI:10.1093/aesa/87.6.651 | |
| Sun N, Li Q, Yu H, et al. Parentage determination of Scapharca broughtonii based on microsatellite analysis. Periodical of Ocean University of China, 2015, 46(9): 42-48 [孙楠, 李琪, 于红, 等. 利用微卫星标记的魁蚶混交家系鉴定. 中国海洋大学学报(自然科学版):, 2015, 46(9): 42-48] | |
| Tian JT, Liu ZH, Yang AG, et al. Microsatellite analysis of genetic diversity in four geographic populations of Scapharca broughtonii. Progress in Fishery Sciences, 2013, 34(6): 59-67 DOI:10.11758/yykxjz.20130609 [田吉腾, 刘志鸿, 杨爱国, 等. 魁蚶4个地理群体遗传多样性微卫星分析. 渔业科学进展, 2013, 34(6): 59-67] | |
| Wu B, Liang C, Yang AG, et al. Genetic variation in different populations of Scapharca broughtonii Schrenck inferred from microsatellite data. Oceanologia et Limnologia Sinica, 2012, 43(4): 863-869 DOI:10.11693/hyhz201204026026 [吴彪, 梁超, 杨爱国, 等. 基于SSR标记的魁蚶(Scapharca broughtonii)不同群体遗传结构的分析. 海洋与湖沼, 2012, 43(4): 863-869] | |
| Wu WJ, Shen B, Chen C, et al. Preliminary classification and phylogenetic relationship among Onchidiidae in China inferred from 18S rRNA partial sequence. Zoological Research, 2010, 11061106(4): 381-386 [吴文健, 沈斌, 陈诚, 等. 基于18S rRNA的中国大陆沿海石磺科贝类分类的初步分析. 动物学研究, 2010, 11061106(4): 381-386] | |
| Xue DX, Wang HY, Zhang T, et al. Phylogenetic analysis of the subclass Pteriomorphia (Bivavia) based on partial 28S rRNA sequence. Oceanologia et Limnologia Sinica, 2012, 43(2): 348-356 DOI:10.11693/hyhz201202024024 [薛东秀, 王海艳, 张涛, 等. 基于28S rRNA基因片段的翼形亚纲(Bivalvia: Pteriomorphia)系统发育的初步研究. 海洋与湖沼, 2012, 43(2): 348-356] | |
| Zhang AB, Wang ZJ, Tan SJ, et al. The important concept of molecular ecology-genetic distance and its measuring. Acta Ecologica Sinica, 2002, 22(6): 943-949 [张爱兵, 王正军, 谭声江, 等. 分子生态学重要概念-遗传距离及其测度的理论研究概况. 生态学报, 2002, 22(6): 943-949] | |
| Zhang WJ, Yu YH, Shen YF. Advances on genetic analysis of microsatellite DNA in protozoology. Acta Hydrobiologica Sinica, 2003, 27(2): 185-190 [张文静, 余育和, 沈韫芬. 微卫星DNA遗传分析在原生动物学中的研究进展. 水生生物学报, 2003, 27(2): 185-190] | |
| Zhang XY, Liu Y, Zhang XM, et al. Species identification of some commercial fishes in southwest Atlantic based on COⅠ barcoding. Acta Hydrobiologica Sinica, 2014, 38(6): 1161-1167 DOI:10.7541/2014.168 [张馨月, 刘岩, 张秀梅, 等. 基于COⅠ基因的西南大西洋部分经济鱼类DNA条形码鉴定. 水生生物学报, 2014, 38(6): 1161-1167] | |
| Zheng WJ, Zhu SH, Shen XQ, et al. Genetic differentiation of Tegillarca granosa based on mitochondrial COⅠ gene sequences. Zoology Research, 2009, 30(1): 17-23 DOI:10.3724/SP.J.1141.2009.01017 | |
| Zhou LQ, Yang AG, Wang QY, et al. ITS sequence variation and phylogenetic analysis on four geographical populations of blood clam Scapharca broughtonii. Progress in Fishery Sciences, 2012, 33(5): 78-84 [周丽青, 杨爱国, 王清印, 等. 魁蚶4个地理群体ITS序列变异及系统发生分析. 渔业科学进展, 2012, 33(5): 78-84] |



