2. 河北农业大学海洋学院 秦皇岛 066100;
3. 潍坊市海洋与渔业局 潍坊 261000
2. Ocean College, Agriculture University of Hebei, Qinhuangdao 066100;
3. Weifang Municipal Ocean and Fishery Administration, Weifang 261000
牙鲆(Paralichthys olivaceus)是渤海重要土著经济鱼类,其个体硕大、肉质细嫩鲜美,深受消费者喜爱。但由于掠夺式的捕捞和严重的近海污染等原因,牙鲆野生资源日渐枯竭。为了应对资源衰退,我国自20世纪80年代开始在渤海海域进行牙鲆的增殖放流活动(金显仕等, 2014)。秦皇岛近海增殖放流区位于河北省东北部近海,每年有数百万的牙鲆苗种被投放到该海域,是我国牙鲆增殖放流的主要区域之一。增殖放流活动在促进渔民增产增收和修复渔业种群资源方面起到了积极的作用,但大规模放流人工培育群体所引起的生态和遗传风险同样不能忽视(Araki et al, 2010; González-Wangüemert et al, 2012; Taranger et al, 2015)。
遗传多样性是生物多样性的重要组成部分,它是衡量一个种群种质资源质量的重要标准,通过衡量生物所携带遗传信息的变异程度来反映遗传多样性的高低,由于DNA是遗传信息的载体,所以DNA的变化直接反应了物种遗传变异的程度(刘海金等, 2008; 姚韩韩, 20111))。微卫星DNA分子标记,又称简单序列重复(Simple sequence repeat, SSR),在基因组中数量众多且分布均匀,具有多态性丰富、杂合度高和检测方便等优点,已经广泛应用于群体遗传多样性的研究(Wang et al, 2013)。至今,利用微卫星DNA分子标记进行牙鲆遗传多样性及遗传结构分析报道较多,主要集中在自然群体、养殖群体和繁育后代群体,如马晓冰等(2012)和刘永新等(2013)分别用18对和72对微卫星分子标记对90尾和74尾野生牙鲆群体进行了遗传多样性分析;刘海金等(2008)和邵长伟等(2009)分别利用5对和16对微卫星分子标记对3个和5个牙鲆养殖群体进行了遗传多样性分析;徐田军等(2009)利用10对微卫星分子标记对由3个牙鲆基础群体组合交配建立的4个选择性繁育后代群体进行遗传多样性分析。但关于增殖放流对牙鲆回捕群体遗传多样性影响的研究还未见报道。本研究利用12对微卫星分子标记对秦皇岛近海增殖放流区内的牙鲆回捕群体的遗传多样性进行分析,并与放流用牙鲆亲本和放流前群体的遗传多样性进行对比,从分子遗传学角度对牙鲆增殖放流可能引起的遗传风险进行评估,以期为我国牙鲆种质资源保护和增殖放流工作提供理论基础。
1) Yao HH. Studies on molecular genetic basis of wild and breeding populations in Tegillarca granosa. Master's Thesis of Shanghai Ocean University, 2011, 70–90 [姚韩韩.泥蚶野生群体与选育群体的分子遗传基础研究.上海海洋大学硕士研究生学位论文, 2011, 70-90]
1 材料与方法 1.1 样品采集和DNA提取共3个群体:2015年河北北戴河中心实验站放流用野生牙鲆亲鱼(以下简称亲本M)、增殖放流前牙鲆幼鱼(以下简称放流前群体P)及2015年禁渔期结束后从秦皇岛近海增殖放流区回捕的牙鲆幼鱼(以下简称回捕群体C)。亲本群体及放流前群体各采集样本30尾。其中,亲本M体重为(3675.22±1572.45) g,体长为(81.66±12.38) cm;放流前群体P体重为(1.59±1.06) g,体长为(5.94±0.97) cm。回捕群体C分别从河北山海关、北戴河和昌黎码头收集,每处采集样本30尾,该群体体重为(55.84±48.65) g,体长为(12.49±6.82) cm。剪取胸鳍平铺于采样纸上,37℃烘干后保存。使用海洋动物基因组DNA提取试剂盒(TIANGEN, DP324)提取基因组DNA。提取完成后,采用琼脂糖凝胶电泳和紫外分光光度计分别检测DNA的质量和浓度。
1.2 微卫星分析从马晓冰等(2012)和刘永新等(2013)发表的72对牙鲆微卫星标记中选取12对多态性高的标记用于本研究。各标记的名称、引物序列、退火温度等见表 1,引物由上海生工生物工程技术有限公司合成。PCR反应体系为20 μl:dH2O 7.4 μl,2×Es Taq MasterMix 10 μl,上下游引物(10 pmol/μl)各0.3 μl,DNA模板2 μl。PCR反应程序为:94℃预变性5 min;94℃变性30 s,54℃复性35 s,72℃延伸40 s,共35个循环;最终72℃延伸3 min。PCR扩增在PE9700型PCR仪上进行。将甲酰胺与分子量内标按100:1的体积比混匀后,取15 μl加入上样板中,再加入1 μl稀释10倍的PCR产物。然后使用3730XL测序仪进行毛细管电泳,利用Genemarker中的Fragment (Plant)片段分析软件对测序仪得到的原始数据进行分析,将各泳道内分子量内标的位置与各样品峰值的位置进行比较分析,得到片段大小。
|
|
表 1 12对微卫星标记的名称、重复序列、引物序列、退火温度及GenBank登录号 Table 1 Loci, repeat motifs, primer sequences, annealing temperatures and GenBank accession numbers of 12 microsatellite markers |
利用Genepop 4.0软件分析分型结果,统计各位点等位基因数(Na)、观测杂合度(Ho)、期望杂合度(He)和多态信息含量(PIC),并分析群体的Hardy-Weinberg遗传平衡状态。计算群体间各群体的基因分化系数(Gst)、基因流、遗传相似性系数(I对角线下方)和Nei氏遗传距离。使用MEGA 5.0软件,采用UPGMA方法根据3个群体的遗传距离进行聚类。
2 结果 2.1 遗传多样性分析12对微卫星标记的遗传多样性参数Na、Ho、He、PIC值列于表 2。从表 2可以看出,12对微卫星标记在3个群体中的PCR扩增得到的平均Na为11.917–22.167个,平均Ho为0.800–0.836,平均He为0.814–0.845,平均PIC为0.775–0.818。其中,回捕群体C的平均Na最多,为22.167,放流前群体P的平均Na最少,为11.917。平均Ho最大的是回捕群体C (0.836),最小的是放流前群体P (0.800)。平均He最大的是亲本M(0.845),最小的是放流前群体P (0.814)。哈迪–温伯格平衡检验结果显示,在36个群体–位点组合中,有23个群体–位点组合显著偏离哈迪–温伯格平衡状态(P < 0.05)。在3个群体中偏离哈迪–温伯格平衡相对最少的是放流前群体P,仅在微卫星标记Poli1396TUF、8-D2和Poli1024TUF中显著偏离(P < 0.05)。偏离哈迪–温伯格平衡相对最多的是回捕群体C。
|
|
表 2 牙鲆不同群体的哈迪–温伯格平衡(HWE)检验及遗传多样性 Table 2 The genetic diversity and HWE test of different populations of P. olivaceus |
3个群体各位点的基因分化系数Gst值为0.005– 0.043,位点HLJYP90的基因分化系数Gst值最大(0.043),位点Poli1408TUF的基因分化系数Gst值最小(0.005)。3个群体各位点的基因流Nm值为5.515–45.983(表 3)。位点Poli1408TUF的Nm值最大,为45.983,位点HLJYP90的Nm值最小,为5.515。3个群体相互间的遗传相似系数和Nei氏遗传距离分别为0.848–0.941和0.061–0.164(表 4)。根据各群体之间的遗传距离,对3个群体进行聚类(图 1),3个群体主要分为两类,放流前群体P和回捕群体C遗传距离较小,首先聚类,然后再与亲本M聚类。
|
|
表 3 牙鲆3个群体各位点的基因分化系数(Gst)和基因流(Nm) Table 3 The coefficient of gene differentiation (Gst) and gene flow (Nm) among three P. olivaceus populations |
|
|
表 4 牙鲆3个群体遗传相似性系数(I对角线下方)和Nei氏遗传距离(D对角线上方) Table 4 The genetic identity (under the diagonal) and Nei's unbiased genetic distances (above the diagonal) of three P. olivaceus populations |
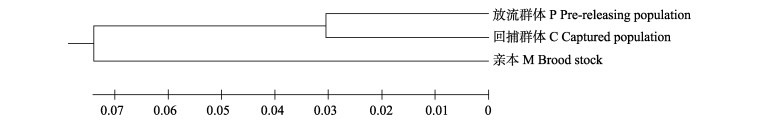
|
图 1 根据Nei氏遗传距离用UPGMA法构建的牙鲆3个群体的进化树 Figure 1 UPGMA dendrogram for three P. olivaceus populations based on Neixs unbiased genetic distances |
牙鲆是黄渤海重要的增殖放流鱼类,北戴河中心实验站自2009年开始在秦皇岛近海增殖放流区进行牙鲆增殖放流活动,至今累计放流不同规格牙鲆苗种1300万尾。随着放流数量的增多,放流群体对固有群体遗传多样性的影响值得关注。鉴于此,本研究以秦皇岛近海增殖放流区域捕捞的牙鲆幼鱼为研究对象,分析回捕群体的遗传多样性,并与放流用牙鲆亲鱼和放流前群体的遗传多样性参数作对比分析。
多态信息含量(PIC)为衡量标记遗传信息含量高低的主要参数,当PIC > 0.5时,表明该遗传标记具有高度的可提供遗传信息性,即高度多态;当0.25 < PIC < 0.5时,表明该遗传标记能够较为合理地提供遗传信息,为中度多态;而当PIC < 0.25时,表明该遗传标记可提供的遗传信息较差,为低度多态(McDonald et al, 2003)。本研究中,除标记Poli1408TUF在放流前群体中的PIC值为0.482外,各标记的PIC范围为0.565– 0.944,表明所选用的微卫星标记具有高度多态性,可以用于分析牙鲆群体的遗传多样性。各标记在3个群体中的平均Na范围为11.917–22.167,其中,回捕群体的Na最高(22.167),说明这3个群体的遗传信息含量都比较丰富,且回捕群体的多态性水平要高于其他群体。马晓冰等(2012)和刘永新等(2014)分别于2009年和2012年对秦皇岛附近海域牙鲆自然群体的等位基因数进行研究,结果分别为8.9和6.789,均低于本研究结果(17.667)。这说明,作为国家级牙鲆良种场,我站每年春季从秦皇岛海域捕捞野生牙鲆对放流用牙鲆亲本群体进行补充更换,确保了放流牙鲆亲本群体遗传多样性的丰富度。
杂合度包括观测杂合度(Ho)和期望杂合度(He),是反映微卫星标记在群体中遗传变异程度的最优参数,是描述遗传多样性的最适参数,杂合度越高,表明群体内遗传多样性就越高,遗传变异程度就越大,反之则群体内遗传变异程度就小(Nei et al, 1975)。本研究He在3个群体中的范围为0.814–0.845,亲本群体最高(0.845),放流前群体最低(0.814)(表 2)。放流前群体He低于亲本群体的主要原因是并非所有的亲鱼都参与繁殖活动。之前的研究结果表明,牙鲆亲本对后代的贡献率严重失衡(Hara et al, 2006; 陈睿毅等, 2013),存在有效亲本群体比实际群体小的问题,且后代仅来自少数几尾亲本。生产上为了克服这个问题,常用的方法是增加繁殖亲本的数量。此外,生殖细胞移植技术在水产动物方面的发展应用为解决这个问题提供了新的方法(Lacerda et al, 2013; Silva et al, 2016)。回捕群体C的He值为0.838,远高于邵长伟等(2009)研究中的养殖群体,略低于野生群体(马晓冰等, 2012),说明秦皇岛近海增殖放流区的人工放流活动未引起明显的遗传风险。
基因分化系数(Gst)是反映群体间遗传分化程度的重要参数。一般认为,Gst值在0–0.05之间时,群体的遗传分化较弱,为轻度遗传分化;Gst值在0.05–0.15之间时,遗传分化水平中等;Gst值大于0.15时,遗传分化较大(Balloux et al, 2002)。本研究中,牙鲆3个群体不同位点的遗传分化系数Gst值为0.005–0.043,3个群体之间表现为轻度分化水平,遗传分化均为较弱。不同群体之间遗传分化系数平均值为0.019,说明3个群体的变异中约有1.9%是由不同群体之间的基因变异引起的,而98.1%的变异来源于群体内。检验结果表明,亲本群体M和回捕群体C微卫星位点偏离哈迪–温伯格平衡较多,其中,亲本群体M多数表现出显著的杂合子缺失,回捕群体C多数表现为显著的杂合子过剩。基因流是指一些个体从一个群体迁移到另一个群体时把某些基因带到新的群体,从而产生的基因流动(曲若竹等, 2004)。基因流与基因分化系数呈负相关,即基因流越大,群体间相似性越大,遗传分化越小(曲若竹等, 2004; 刘海金等, 2008)。本研究各位点基因流较大(Nm > 5),说明3个群体间遗传分化较小,足以抵抗遗传漂变,与本研究中基因分化系数相关结果相互支持。
遗传多样性和遗传结构是种群繁衍生息的重要原因,遗传多样性降低可能导致种群生长缓慢、个体变小、性成熟提早、抗病力下降等不良影响(黎中宝等, 2009; 李树华, 20141))。本研究中,亲本群体M与回捕群体C较高的遗传多样性水平说明秦皇岛附近海域牙鲆资源在增殖放流活动的实施下,其遗传多样性得到了有效的保护,进而说明,保持放流亲本群体较高的遗传多样性,对有效提高资源量修复水平有促进作用,对渔业资源及生态平衡保护有重要意义。
1) Li SH. Genetic and enhancement effects of released parent grass carp in the middle Yangtze River. Master's Thesis of Huazhong Agriculture University, 2014, 27–44 [李树华.长江中游草鱼亲本放流的增殖效果及遗传多样性影响评估.华中农业大学硕士研究生学位论文, 2014, 27–44]
| Araki H, Schmid C. Is hatchery stocking a help or harm?: Evidence, limitations and future directions in ecological and genetic surveys. Aquaculture, 2010, 308(S1): 2-11 | |
| Balloux F, Lugon-Moulin N. The estimation of population differentiation with microsatellite markers. Molecular Ecology, 2002, 11(2): 155-165 DOI:10.1046/j.0962-1083.2001.01436.x | |
| Chen RY, Wang GX, Liu HJ, et al. Difference in parental contribution to reproduction in Japanese flounder (Paralichthys olivaceus). Journal of Fishery Science of China, 2013, 20(4): 698-705 [陈睿毅, 王桂兴, 刘海金, 等. 牙鲆亲本对子代贡献率的实验研究. 中国水产科学, 2013, 20(4): 698-705] | |
| González-Wangüemert M, Fernández TV, Pérez-Ruzafa A, et al. Genetic considerations on the introduction of farmed fish in marine protected areas: The case of study of white seabream restocking in the Gulf of Castellammare (Southern Tyrrhe-nian Sea). Journal of Sea Research, 2012, 68(1-2): 41-48 | |
| Hara M, Sekino M. Efficient detection of parentage in a cultured Japanese flounder Paralichthys olivaceus using microsatellite DNA marker. Aquaculture, 2003, 217(1-4): 107-114 DOI:10.1016/S0044-8486(02)00069-8 | |
| Jin XS, Qiu SY, Liu XZ, et al. The foundation and prospect of fisheries resources of Huanghai and Bohai Sea. Beijing: Science Press, 2014. [金显仕, 邱盛尧, 柳学周, 等. 黄渤海渔业资源增殖基础与前景. 北京: 科学出版社, 2014.] | |
| Lacerda S, Costa GMJ, Campos-Junior PHA, et al. Germ cell transplantation as a potential biotechnological approach to fish reproduction. Fish Physiology and Biochemistry, 2013, 39(1): 3-11 DOI:10.1007/s10695-012-9606-4 | |
| Li ZB, Fang X, Chen J, et al. Loss of the genetic diversity in cultivated populations of Pseudosciaena crocea by AFLP. Oceanologia et Limnologia Sinica, 2009, 40(4): 446-450 DOI:10.11693/hyhz200904009009 [黎中宝, 方秀, 陈锦, 等. 大黄鱼(Pseudosciaena crocea)养殖群体遗传多样性的降低. 海洋与湖沼, 2009, 40(4): 446-450] | |
| Liu HJ, Zhu XC, Sun XW, et al. Analysis of genetic structure among 5 cultured stocks of Japanese flounder Paralichthys olivaceus. Journal of Fishery Science of China, 2008, 15(1): 30-37 [刘海金, 朱晓琛, 孙效文, 等. 牙鲆5个养殖群体的遗传多样性分析. 中国水产科学, 2008, 15(1): 30-37] | |
| Liu YX, Zhu YM, Liu YJ, et al. Analysis of genetic diversity in Bohai natural population of Paralichthys olivaceus. Journal of Fisheries of China, 2013, 37(11): 1609-1617 [刘永新, 朱以美, 刘英杰, 等. 牙鲆渤海自然群体的遗传多样性分析. 水产学报, 2013, 37(11): 1609-1617] | |
| Ma XB, Wang GX, Liu HJ, et al. Genetic diversity analysis of the offshore Japanese flounder (Paralichthys olivaceus) population within Qinhuangdao, China. Journal of Fishery Science of China, 2012, 19(6): 963-969 [马晓冰, 王桂兴, 刘海金, 等. 秦皇岛海域野生牙鲆群体遗传多样性分析. 中国水产科学, 2012, 19(6): 963-969] | |
| McDonald GM, Wiley J. Space, time and life. Seattle: University of Washington Press, 2003: 109-120. | |
| Nei M, Maruyama T, Chakraborty R. The bottleneck effect and genetic variability in populations. Evolution, 1975, 29(1): 1-10 | |
| Qu RZ, Hou L, Lv HL, et al. The gene flow of population genetic structure. Hereditas, 2004, 26(3): 377-382 [曲若竹, 侯林, 吕红丽, 等. 群体遗传结构中的基因流. 遗传, 2004, 26(3): 377-382] | |
| Shao CW, Liao XL, Tian YS, et al. Microsatellite marker analysis of genetic structures of three populations of cultured Japanese flounder Paralichthys olivaceus. Progress in Fishery Sciences, 2009, 30(1): 41-46 [邵长伟, 廖小林, 田永胜, 等. 牙鲆3个养殖群体遗传结构的微卫星分析. 渔业科学进展, 2009, 30(1): 41-46] | |
| Silva MA, Costa GMJ, Lacerda S, et al. Successful xenogeneic germ cell transplantation from Jundia catfish (Rhamdia quelen) into adult Nile tilapia (Oreochromis niloticus) testes. General and Comparative Endocrinology, 2016, 230: 48-56 | |
| Taranger GL, Karlsen Ø, Bannister RJ, et al. Risk assessment of the environmental impact of Norwegian Atlantic salmon farming. ICES Journal of Marine Science: Journal du Conseil, 2015, 72(3): 997-1021 DOI:10.1093/icesjms/fsu132 | |
| Wang YH, Fu DK, Xia JJ. The genetic diversity of the noble scallop (Chlamys nobilis, Reeve 1852) in China assessed using five microsatellite markers. Marine Genomics, 2013, 9(1): 63-67 | |
| Xu TJ, Chen SL, Tian YS. Genetic structure analysis among four selective offspring stocks of Japanese flounder Paralichthys olivaceus by microsatellite markers. Progress in Fishery Sciences, 2009, 30(4): 57-63 [徐田军, 陈松林, 田永胜. 牙鲆4个选择性繁育后代群体遗传结构的微卫星分析. 渔业科学进展, 2009, 30(4): 57-63] |



