2. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 上海海洋大学水产与生命学院 上海 201306;
4. 大连海洋大学水产与生命学院 大连 116023
2. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071;
3. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306;
4. College of Fisheries and Life Science, Dalian Ocean University, Dalian 116023
微卫星DNA(Simple sequence repeat, SSR),是由基本单元为1~6个核苷酸所构成的简单重复序列,是目前应用较为广泛的分子标记之一(OxConnell et al, 1997),其稳定性和可重复性高,特异性和共显性强,现已应用于生物遗传多样性分析、全基因组相关分析、分子标记辅助育种等诸多方面(刘志毅等, 2001; Alam et al, 2005; Cavagnaro et al, 2010; Zhu et al, 2012; 王日芳等, 2017)。海洋生物的微卫星筛选及多态性分析、利用微卫星构建连锁图谱和抗病相关标记的连锁分析等工作已陆续开展(赵莹莹等, 2006a、b; 李春艳等, 2009; Hou et al, 2011)。
虾夷扇贝(Patinopecten yessoensis)自20世纪80年代引入中国,经过几十年的培育与发展,现已成为我国重要的海洋养殖物种。目前,已有采用分子标记开展虾夷扇贝种群遗传研究的报道,如同工酶电泳技术(高悦勉等, 2003、2004)、AFLP(鲍相渤等, 2009; 丁君等, 2010)、SSR(Sato et al, 2005; 赵莹莹等, 2006b; 常亚青等, 2007; 李春艳等, 2009; Hou et al, 2011),而对虾夷扇贝基因组水平上的微卫星及其分布特征还知之甚少。本研究采用第二代高通量测序技术对虾夷扇贝进行简化基因组测序(Restriction-site associated DNA sequencing, RAD-seq),通过对基因组中不同类型微卫星的统计与分析,了解其微卫星分布频率和数量,从基因组水平阐明虾夷扇贝基因组SSR特征及组成,对进一步利用微卫星开展遗传标记辅助育种和种群遗传学研究具有重要的参考价值。
1 材料与方法 1.1 材料虾夷扇贝于2015年7月采自我国北黄海獐子岛虾夷扇贝底播养殖海域,冰冻保存运送至实验室,解剖处理,取虾夷扇贝闭壳肌组织,存于–20℃备用。
1.2 基因组DNA提取采用海洋动物组织基因组DNA提取试剂盒(离心柱型) (天根,北京)进行基因组DNA的提取,琼脂糖凝胶电泳初步检测提取的DNA质量,采用核酸蛋白分析仪检测DNA浓度与纯度,选取质量较好的DNA样品保存于–20℃备用。
1.3 文库构建及测序将所获取的虾夷扇贝DNA样品送至北京诺禾致源生物信息科技有限公司进行测序。DNA样品通过随机酶切打断,经末端修复、加A尾、加测序接头、纯化、PCR扩增等步骤完成整个RAD文库的制备,构建好的文库通过Illumina(测序仪)进行双末端测序,测序个数乘以测序序列长度即为原始数据值。对原始数据进行过滤:去除带接头(Adapter)的reads pair;去除单端测序read中含有的N(未读碱基)含量超过该条read长度比例的10%的reads pair;去除单端测序read中含有的低质量(Q≤5)碱基数超过该条read长度比例的50%的reads pair。对所得数据样本含有的酶识别位点reads用cd-hit-est软件(Li et al, 2006)进行聚类,并保证每类中reads支持数在10~400之间。根据聚类结果,用Velvetopt软件(Zerbino et al, 2008)进行组装,得到最后的组装序列,并对125 bp以下的contig进行过滤。
1.4 分析方法使用SR search软件(Rozen et al, 2000)对过滤所得的contig进行简单重复序列的检测,检测标准如下:(1) SSR重复的最小长度为2;(2) SSR重复的最大长度为6;(3) SSR序列的最小长度为12;(4) SSR上下游序列长度为100 bp;(5) 2个SSR的最小距离为12 bp。使用Primer 3软件(Rozen et al, 2000)对鉴定出的含有SSR重复基元的序列片段进行引物设计:引物的长度范围为20~28,最适长度为24;引物退火温度范围为60~65℃,最适退火温度为63℃,且一对引物退火温度差值不超过1℃。
SSR分布密度(D)计算公式为:
| $ D = N/L $ |
式中,D为不同重复微卫星分布密度(ind./Mb);L为虾夷扇贝基因组重叠群(contig)总长(Mb);N为各重复类型微卫星数量(ind.)。
SSR不同长度的分布频率(F)计算公式为:
| $ F = N/T $ |
式中,F为不同长度微卫星的分布频率;N为各长度微卫星数量(ind.);T为微卫星总数量(ind.)。
2 结果与分析 2.1 简化基因组测序及拼接测序结果共产生原始数据2.872 G,经过滤后共获得有效数据2.847 G (表 1)。Illumina HiSeq产生的序列的碱基质量用Qphred表示,Phred值为20和30,碱基正确识别率分别为99%和99.9%。本测序中Phred数值大于20和30的碱基占所有碱基的百分比分别为96.77%和92.66%,RAD-Tag捕获率为93.07%,数据量符合预先设定,表明建库成功,测序质量合格。
|
|
表 1 虾夷扇贝简化基因组测序主要数据 Table 1 Mean data of reduced-representation genome sequencing in P. yessoensis |
通过聚类组装获得序列总长度为92, 551, 435 bp,过滤掉小于125 bp的contig后获得259, 535个contig,其中,N50 (将序列从大到小排列,当长度达到组装总长度一半时,contig的长度)的长度为419 bp,组装基因组的平均覆盖深度为14.55×,其中,4×覆盖度在86.64%以上,GC含量为35.63%,拼接结果正常,拼接序列质量较高。
2.2 基因组微卫星的数量及分布密度通过对虾夷扇贝基因组的序列进行微卫星查找,在虾夷扇贝的高通量测序数据库中进一步搜索含有2~6重复单元的SSR,从总长为92, 551, 435 bp的259, 535个contig中筛选出3618个潜在微卫星位点,经引物设计共获得3460个微卫星引物,利用微卫星密度计算公式可得,平均26, 748.97 bp出现1个SSR (表 2)。由表 2可以看出,虾夷扇贝微卫星各重复类型之间SSR数量均相差较大,各类型SSR分布密度差异明显。所获得的3460条微卫星序列中,分布最多的重复类型为三核苷酸重复(45.87%);其次是二核苷酸重复(37.05%)、四核苷酸重复(14.48%)、五核苷酸重复(2.02%);分布最少的是六核苷酸重复(0.58%)。
|
|
表 2 虾夷扇贝不同重复类型微卫星所占比例及分布密度 Table 2 The proportion and density of different repeat types in SSR database of P. yessoensis |
统计微卫星各重复类型中不同重复单元的数量显示,二核苷酸重复有12种重复单元,三核苷酸重复59种,四核苷酸重复100种,五核苷酸重复47种,六核苷酸重复20种。统计微卫星各重复类型中不同重复单元的百分比(表 3),在二核苷酸重复微卫星中,AT/TA重复单元所占比例最高(79.17%),共计1015个;其次是TG/GT重复单元103个(8.03%)。在三核苷酸重复的微卫星中,ATA重复单元所占比例最高(11.41%),共计181个;AAT、TAT、ATT、TAA这4种重复单元紧随其后,分别有155个(9.77%)、150个(9.45%)、137个(8.36%)和120个(7.56%)。四核苷酸重复中每种重复单元所占比例相对均匀,其中,数量最多的为CAAA重复单元,共计48个,所占比例为9.58%。五核苷酸重复中,数量最多的重复单元为AAACC,共计11个,占比15.71%。六核苷酸重复共计20个,每一种重复单元仅有1个。
|
|
表 3 虾夷扇贝SSR重复单元的数量及百分比 Table 3 The numbers and percentage of SSR repeat motifs in P. yessoensis |
虾夷扇贝基因组微卫星长度范围为12~36 bp,平均长度为14.96 bp。微卫星长度分布频率计算结果显示,微卫星长度越长其分布频率越低,其中,12~16 bp的微卫星占绝对优势,比例高达79.35% (图 1)。
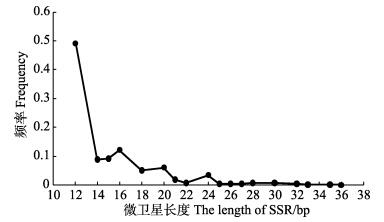
|
图 1 虾夷扇贝微卫星长度分布及不同长度微卫星频率 Figure 1 The length distribution and frequency of P. yessoensis SSR |
针对虾夷扇贝基因组微卫星数量进行分析发现,当同一重复类型的微卫星随着重复数增加其数量相对减少(表 4纵向),而相同重复数的微卫星随着重复单元长度的增加其数量也呈下降趋势(表 4横向),且随着不同重复单元长度的增加其重复数范围逐渐减小(表 4)。如,二核苷酸重复单元按照重复数多少可分成11种,其中,重复数为6的二核苷酸重复单元数量有695,随着重复数增加其数量逐渐减少,当重复数增加至16时该重复单元的数量降至7;当重复单元长度增加至六核苷酸时,该重复类型数量仅有20种。可见,随着重复单元长度的增加,微卫星数量和重复数范围均呈下降趋势,推测虾夷扇贝基因组中长度较短的微卫星变异速率比长度较长的微卫星变异速率快。
|
|
表 4 虾夷扇贝SSR不同重复数及不同重复类型的数量 Table 4 The number of SSR with different repeat types and different copy number in P. yessoensis |
本研究基于虾夷扇贝基因组获得有效SSR数目为3460个,其中出现频率最高的为三核苷酸重复(45.87%),主要以ATA(11.41%)和AAT(9.77%)重复单元为主,然后依次是TAT、ATT、TAA重复单元;而二核苷酸重复中,则是以AT最多,其次为TA。Hou等(2011)采用454测序技术对虾夷扇贝转录组进行测序,在其转录组中搜索到的2748个SSR中,三核苷酸重复的频率最高(39.4%),其次是二核苷酸重复(21.1%)、四核苷酸重复(15.5%)、五核苷酸重复(14.6%)和六核苷酸重复(9.4%)。可见,无论是在转录组还是基因组水平上,虾夷扇贝微卫星三核苷酸重复类型的出现频率均最高。
通过二核苷酸、三核苷酸和四核苷酸3种重复类型中各重复单元对比研究,在各种长度的重复中,A/T都较G/C含量高很多,这与多数生物基因组微卫星具有的特点相一致,如中国对虾(Fenneropenaeus chinensis)微卫星(高焕等, 2004),红鳍东方鲀(Sargassum fusiforme)微卫星(崔建洲等, 2006),三疣梭子蟹(Portunus trituberculatus)微卫星(宋来鹏等, 2008)。生物基因组微卫星富含A/T的可能原因是微卫星序列A/T含量高,则Tm值降低,DNA链容易解开,通过DNA复制滑动机制和重组机制,产生富含AT重复类型的机率更高。此外,Schlotterer等(1992)认为,基因组DNA因CpG甲基化的胞嘧啶C很容易经过脱氨基作用转变成胸腺嘧啶T,使其具有较高的突变可能性,并且少量的GC又是维持DNA热力学稳定性所必需的,一定程度上可解释生物体基因组微卫星中G/C含量如此之少的原因(黄杰等, 2015)。还有一种可能是,统计结果中GC重复过少也可能与GC重复的测序工作比较困难有关(高焕等, 2004)。具体原因,现今仍无定论,尚有待进一步的研究探讨。
对虾夷扇贝基因组不同重复类型微卫星的长度变异情况进行分析,发现这些微卫星的数量与重复长度成反比,微卫星的数量随着重复长度的增加而降低。Samadi等(1998)的模拟分析研究认为,重复长度越长,经受的选择压力越大,则它们的重复数就越少,数量越低。此外,在中国对虾(高焕等, 2004)、红鳍东方鲀(崔建洲等, 2006)、枸杞(Lycium barbarum) (党少飞等, 2016)等不同种类的动植物基因组微卫星的研究中也得到了相同的结论。这意味着虾夷扇贝基因组中重复长度较短的微卫星比重复长度较长的微卫星变异速率快,为虾夷扇贝进一步的多态性微卫星标记筛选工作提供选择方向和依据。
3.2 SSR开发与筛选方法对SSR特征分析的影响目前,比较成熟的微卫星开发与筛选技术有磁珠富集法、构建cDNA文库筛选EST-SSR法以及基于二代测序的转录组测序和简化基因组测序法。不同方法获得微卫星标记数量不同,所体现出的分布特征也不尽相同。传统的磁珠富集法筛选微卫星标记,获得率较低,每次仅能获得百余个微卫星标记,因其筛选探针多用二核苷酸重复的微卫星序列,在富集过程中,磁珠经过多个洗涤过程将一些因重复数目较少而附着不牢固的微卫星序列洗去,仅能获得较长序列的微卫星标记,因此,不能完全反映其微卫星的分布情况(赵莹莹等, 2006a; 李春艳等, 2009)。李云峰等(2010)通过构建虾夷扇贝cDNA文库获得74个EST-SSR,由于EST序列仅能反映转录加工后的mRNA信息,除去了许多内含子和其他序列,因此,并不能反映基因组DNA的真实信息。随着测序技术的发展,获取效率高、技术难度低且花费成本与传统方法相近的二代测序被广泛应用到微卫星引物开发中,基于转录组或基因组进行全部或部分高通量测序的方法可以获得大量的核酸序列信息,微卫星标记的开发也就水到渠成,而且二代测序一次性开发标记数据量大,可以比较完整地反应物种的微卫星分布特征。
3.3 与其他贝类微卫星分布特征比较由虾夷扇贝基因组微卫星特征分析得知,其三碱基重复类型微卫星数量最多,在已开展基因组微卫星开发的贝类中(表 5),栉孔扇贝(Chlamys farreri) (Zhang et al, 2008)和三角帆蚌(Hyriopsis cumingii)(Bai et al, 2009)微卫星数量最多的同样为三碱基重复类型,而在马氏珠母贝(Pinctada martensii)(王忠良等, 2015)、里氏拟石磺(Paraoncidium reevesii)(吴欣等, 2015)、菲律宾蛤仔(Ruditapes philippinarum)(闫路路等, 2015)和文蛤(Meretrix meretrix)(Wang et al, 2011)中,却呈现出单核苷酸重复数量最多,二核苷酸、三核苷酸和四核苷酸重复数量依次递减的趋势。而长牡蛎(Crassostrea gigas)(张琳琳等, 2011)的EST-SSR则呈现六核苷酸重复数目最多(851),单核苷酸次之(805),二核苷酸、三核苷酸和四核苷酸重复依次递减的趋势。由此可见,在已有贝类微卫星研究中,各重复类型的微卫星分布并未呈现较明显的规律,产生这样的结果,一方面可能是微卫星开发手段不同而导致结果不同,另一方面,也可能是不同物种其微卫星组成存在较大差异,这些都有待进一步研究。
|
|
表 5 不同贝类SSR分布特征 Table 5 The characteristics of SSR distribution in different molluscs |
| Alam MS, Islam MS. Population genetic structure of Catla catla (Hamilton) revealed by microsatellite DNA markers. Aquaculture, 2005, 246(1-4): 151-160 DOI:10.1016/j.aquaculture.2005.02.012 | |
| Bai Z, Yin Y, Hu S, et al. Identification of genes involved in immune response, microsatellite, and SNP markers from expressed sequence tags generated from hemocytes of freshwater pearl mussel (Hyriopsis cumingii). Marine Biotechnology, 2009, 11(4): 520-530 DOI:10.1007/s10126-008-9163-0 | |
| Bao XB, Dong Y, He CB, et al. Studies of Japanese scallop (Mizuhopecten yessoensis) germplasm by using AFLP markers. Biotechnology Bulletin, 2009(4): 126-129 [鲍相渤, 董颖, 赫崇波, 等. 基于AFLP技术对中国虾夷扇贝群体种质资源的研究. 生物技术通报, 2009(4): 126-129] | |
| Cavagnaro PF, Senalik DA, Yang L, et al. Genome-wide characterization of simple sequence repeats in cucumber (Cucumis sativus L.). BMC Genomics, 2010, 11(1): 569 DOI:10.1186/1471-2164-11-569 | |
| Chang YQ, Chen XX, Ding J, et al. Genetic diversity in five scallop populations of the Japanese scallop (Patinopecten yessoensis). Acta Ecologica Sinica, 2007, 27(3): 1145-1152 [常亚青, 陈晓霞, 丁君, 等. 虾夷扇贝(Patinopecten yessoensis) 5个群体的遗传多样性. 生态学报, 2007, 27(3): 1145-1152] | |
| Cui JZ, Shen XY, Yang GP, et al. Extraction, purification and chemical composition studies on fucodian from Sargassum fusiforme. Periodical of Ocean University of China (Natural Science Edition), 2006, 36(2): 249-254 [崔建洲, 申雪艳, 杨官品, 等. 红鳍东方鲀基因组微卫星特征分析. 中国海洋大学学报(自然科学版), 2006, 36(2): 249-254] | |
| Dang SF, Wang ZL, Zhang DF, et al. Characteristic analysis of microsatellites of Lycium barbarum. Journal of Northwest Forestry University, 2016, 31(1): 97-102 [党少飞, 王占林, 张得芳, 等. 枸杞基因组微卫星特征分析. 西北林学院学报, 2016, 31(1): 97-102] | |
| Ding J, Wang T, Chang YQ, et al. AFLP analysis on genetic diversity and differentiation of different populations of Patinopecten yessoensis. Journal of Yantai University (Natural Science and Engineering Edition), 2010, 23(1): 47-53 [丁君, 王婷, 常亚青, 等. 基于AFLP技术的不同群体虾夷扇贝遗传结构及多样性研究. 烟台大学学报(自然科学与工程版), 2010, 23(1): 47-53] | |
| Gao H, Liu P, Meng XH, et al. Analysis of microsatellite sequences in Chinese shrimp Fenneropenaeus chinensis genome. Oceanologia et Limnologia Sinica, 2004, 35(5): 424-431 [高焕, 刘萍, 孟宪红, 等. 中国对虾(Fenneropenaeus chinensis)基因组微卫星特征分析. 海洋与湖沼, 2004, 35(5): 424-431] | |
| Gao YM, Li GX, Zhao YL. Genetic structure of Japanese scallop Patinopecten yessoensis hatched population in Dalian Sea areas. Journal of Dalian Fisheries University, 2004, 19(2): 142-145 [高悦勉, 李国喜, 赵银丽. 大连沿海虾夷扇贝养殖群体遗传结构的研究. 大连水产学院学报, 2004, 19(2): 142-145] | |
| Gao YM, Li GX. Biochemical genetic analysis of isozymes from Patinopecten yessoensis. Journal of Dalian Fisheries University, 2003, 18(4): 269-272 [高悦勉, 李国喜. 虾夷扇贝同工酶的生化遗传分析. 大连水产学院学报, 2003, 18(4): 269-272] | |
| Hou R, Bao ZM, Wang S, et al. Transcriptome sequencing and de novo analysis for Yesso scallop (Patinopecten yessoensis) using 454 GS FLX. PLoS One, 2011, 6(6): e21560 DOI:10.1371/journal.pone.0021560 | |
| Huang J, Zhou Y, Liu YZ, et al. Characteristics of microsatellites in Arborophila rufipectus genome sequences using 454 GS FLX. Sichuan Journal of Zoology, 2015, 34(1): 8-14 [黄杰, 周瑜, 刘与之, 等. 基于454 GS FLX高通量测序的四川山鹧鸪基因组微卫星特征分析. 四川动物, 2015, 34(1): 8-14] | |
| Li CY, Ding J, Chang YQ, et al. Isolation of microsatellite markers in Japanese scallop and its application in genetic structure analysis. Journal of Fishery Sciences of China, 2009, 16(1): 39-46 [李春艳, 丁君, 常亚青, 等. 虾夷扇贝微卫星标记的分离及其养殖群体的遗传结构分析. 中国水产科学, 2009, 16(1): 39-46] | |
| Li WZ, Godzik A. Cd-hit: A fast program for clustering and comparing large sets of protein or nucleotide sequences. Bioinformatics, 2006, 22(13): 1658-1659 DOI:10.1093/bioinformatics/btl158 | |
| Li YF, Liu WD, Gao XG, et al. Construction of cDNA libraries from mantle and kidney of Japanese scallop (Mizuhopecten yessoensis) and ESTs analysis. Journal of Fishery Sciences of China, 2010, 17(3): 578-585 [李云峰, 刘卫东, 高祥刚, 等. 虾夷扇贝外套膜和肾脏组织cDNA文库构建以及EST的初步分析. 中国水产科学, 2010, 17(3): 578-585] | |
| Liu ZY, Xiang JH. Application of the microsatellite technique for genetic analysis in marine animals. Marine Sciences, 2001, 25(6): 11-13 [刘志毅, 相建海. 微卫星DNA分子标记在海洋动物遗传分析中的应用. 海洋科学, 2001, 25(6): 11-13] | |
| O'Connell M, Wright JM. Microsatellite DNA in fishes. Reviews in Fish Biology and Fisheries, 1997, 7(3): 331-363 DOI:10.1023/A:1018443912945 | |
| Rozen S, Skaletsky H. Primer3 on the WWW for general users and for biologist programmers. Methods in Molecular Biology, 2000, 132(3): 365-386 | |
| Samadi S, Artiguebielle E, Estoup A, et al. Density and variability of dinucleotide microsatellites in the parthenogenetic polyploid snail Melanoides tuberculata. Molecular Ecology, 1998, 7(9): 1233-1236 DOI:10.1046/j.1365-294x.1998.00405.x | |
| Sato M, Kawamata K, Zaslavskaya N, et al. Development of microsatellite markers for Japanese scallop (Mizuhopecten yessoensis) and their application to a population genetic study. Marine Biotechnology, 2005, 7(6): 713-728 DOI:10.1007/s10126-004-0127-8 | |
| Schlotterer C, Tautz D. Slippage synthesis of simple sequence DNA. Nucleic Acids Research, 1992, 20(2): 211-215 DOI:10.1093/nar/20.2.211 | |
| Song LP, Liu P, Li J, et al. Analysis of microsatellite sequences in genome of crab, Portunus trituberculatus. Journal of Fishery Sciences of China, 2008, 15(5): 738-744 [宋来鹏, 刘萍, 李健, 等. 三疣梭子蟹基因组微卫星特征分析. 中国水产科学, 2008, 15(5): 738-744] | |
| Wang HX, Huan P, Lu X, et al. Mining of EST-SSR markers in clam Meretrix meretrix larvae from 454 shotgun transcriptome. Genes and Genetic Systems, 2011, 86(3): 197-205 DOI:10.1266/ggs.86.197 | |
| Wang RF, Li J, Li JT, et al. Genetic analysis of 33 microsatellite loci in 3 families of Exopalaemon carinicauda inbred line. Progress in Fisheries Science, 2017, 38(4): 78-86 [王日芳, 李健, 李吉涛, 等. 脊尾白虾(Exopalaemon carinicauda)近交系3个家系33个微卫星座位的遗传分析. 渔业科学进展, 2017, 38(4): 78-86] | |
| Wang ZL, Ding J, Xu YH, et al. Polymorphism of EST-SSRs from Pinctada martensii based on transcriptome datasets. Oceanologia et Limnologia Sinica, 2015, 46(3): 687-693 DOI:10.11693/hyhz20141200361 [王忠良, 丁燏, 许尤厚, 等. 基于转录组数据的马氏珠母贝EST-SSR位点的信息分析及其多态性检测. 海洋与湖沼, 2015, 46(3): 687-693] | |
| Wu X, Shen HD, Wang DF, et al. Development of microsatellite markers for Paraoncidium reevesii based on transcriptome sequencing Illumina HiseqTM 2000. Marine Sciences, 2015, 39(11): 20-25 DOI:10.11759/hykx20150803002 [吴欣, 沈和定, 王冬凤, 等. 基于Illumina HiseqTM 2000高通量转录组测序的里氏拟石磺SSR标记开发. 海洋科学, 2015, 39(11): 20-25] | |
| Yan LL, Qin YJ, Yan XW, et al. Development of microsatellite markers in Ruditapes philippinarum using next-generation sequencing. Acta Ecologica Sinica, 2015, 35(5): 1573-1580 [闫路路, 秦艳杰, 闫喜武, 等. 基于转录组平台的蛤仔微卫星标记筛选. 生态学报, 2015, 35(5): 1573-1580] | |
| Zerbino DR, Birney E. Velvet: Algorithms for de novo short read assembly using de Bruijn graphs. Genome Research, 2008, 18(5): 821-829 DOI:10.1101/gr.074492.107 | |
| Zhang LL, Bao ZM, Wang S, et al. FISH mapping and identification of Zhikong scallop (Chlamys farreri) chromosomes. Marine Biotechnology, 2008, 10(2): 151-157 DOI:10.1007/s10126-007-9045-x | |
| Zhang LL, Li L, Zhang GF. Bioinformatics data mining of EST-tandem repeats of the Pacific oyster (Crassostrea gigas). Marine Sciences, 2011, 35(4): 9-14 [张琳琳, 李莉, 张国范. 长牡蛎(Crassostrea gigas)EST串联重复序列的组成和分布. 海洋科学, 2011, 35(4): 9-14] | |
| Zhao YY, Zhu XC, Sun XW, et al. Microsatellite enrichment by magnetic beads in Japanese scallop, Patinopecten yessoensis. Journal of Fishery Sciences of China, 2006a, 13(5): 749-755 [赵莹莹, 朱晓琛, 孙效文, 等. 磁珠富集法筛选虾夷扇贝微卫星序列. 中国水产科学, 2006a, 13(5): 749-755] | |
| Zhao YY, Zhu XC, Sun XW, et al. Polymorphic microsatellite loci of the Japanese scallop Patinopecten yessoensis. Acta Zoologica Sinica, 2006b, 52(1): 229-233 [赵莹莹, 朱晓琛, 孙效文, 等. 虾夷扇贝的多态性微卫星座位. 动物学报, 2006b, 52(1): 229-233] | |
| Zhu H, Senalik D, McCown BH, et al. Mining and validation of pyrosequenced simple sequence repeats (SSRs) from American cranberry (Vaccinium macrocarpon Ait.). Theoretical and Applied Genetics, 2012, 124(1): 87-96 DOI:10.1007/s00122-011-1689-2 |



