曼氏无针乌贼(Sepiella japonica)属软体动物门(Mollusca)、头足纲(Cephalopoda)、十腕目(Decapoda)、乌贼科(Sepiidae)、无针乌贼属(Sepiella),和大黄鱼(Pseudosciaena crocea)、小黄鱼(P. polyactis)、带鱼(Trichiurus haumela)并称为我国东海“四大海产”,是我国重要的海洋渔业资源。曼氏无针乌贼地理分布范围较广,朝鲜西南海岸、日本濑户内海沿岸、俄罗斯远东海和我国沿海及其邻近海域都有分布(常抗美等, 2008)。其肉味鲜美,蛋白质含量高,营养丰富,具有很高的经济和药用价值。以曼氏无针乌贼及其性腺为原材料的产品主要有“墨鱼干”、“乌鱼蛋”等(董正之, 1988)。
由于过度捕捞、生境破坏等原因,资源受到严重损害,20世纪中后期开始,曼氏无针乌贼资源已无法形成渔汛,自然海域已很难见到野生的曼氏无针乌贼。因此,为了保护曼氏无针乌贼资源,恢复资源量,人工培育乌贼种苗进行增殖放流的工作已经陆续开展。从21世纪初起,本团队对曼氏无针乌贼做了大量研究工作,如繁殖习性及产卵场修复(吴常文等, 2010)、胚胎发育与人工育苗技术(常抗美等, 2009)等,并已解决人工繁育和养殖难题,在2006~2016年期间,本团队每年均在曼氏无针乌贼的主要栖息地浙江东极等海域投放百万头(颗)幼乌贼和乌贼卵。在放流海域已可见曼氏无针乌贼资源恢复迹象(董智勇等, 2010; 史会来等, 2015),但放流曼氏无针乌贼主要以卵的形式放流,所以传统的物理、化学等标记方法不完全适用,因此,至今仍不能有效评估增殖放流效果。
微卫星(Microsatellite)又称简单重复序列(Simple sequence repeat, SSR),是真核生物基因组中的高度重复序列,是由几个核苷酸组成的串联重复序列,其长度一般较短(何平, 1998)。在众多分子标记中,微卫星DNA(Microsatellite DNA)标记位点数目巨大且随机分布于整个基因组中,呈共显性遗传,易于检测且重复稳定,在亲代和子代之间遵从孟德尔遗传,因而成为水产养殖业中遗传多样性和亲缘关系研究的重要分子标记(陈睿毅等, 2013)。目前,微卫星标记已经应用于头足纲中的欧洲横纹乌贼(Sepia officinalis)和真蛸(Octopus vulgaris)的遗传多样性研究,及欧洲乌贼(Loligo vulgaris)的遗传结构研究(Pérez-Losada et al, 2002; Moreira et al, 2011; Garoia et al, 2004)。微卫星标记对样本的非损伤性对于一些濒危珍稀物种而言更是不二选择,国内外已有将微卫星标记应用于渔业资源增殖放流效果评价的研究(Sekino et al, 2003; 成为为等, 2014)。近年来,利用磁珠富集法和FIASCO法构建微卫星富集文库法,开发出多对曼氏无针乌贼微卫星标记,并利用开发标记对群体多态性进行了分析(Wu et al, 2010; 吴璋等, 2011; Guo et al, 2013; 张川等, 2014),之后鲜见关于曼氏无针乌贼微卫星标记开发的报道。
为进一步开发可用的微卫星标记资源,本研究利用曼氏无针乌贼高通量转录组数据,获得127575条Unigene序列。利用计算机软件进行微卫星位点的搜索,并分析微卫星序列的特征、分布和组成等信息,为后续筛选大量的微卫星遗传标记提供基础数据。
1 材料与方法 1.1 实验材料及数据来源曼氏无针乌贼视腺转录组数据(未发表)来自本实验室,组装并去冗余后得到127575个Unigene序列,总长度103104058 bp。
1.2 简单重复序列(SSR)的分析搜索为检测曼氏无针乌贼转录组中SSR位点,利用Perl语言操作平台下的MISA软件(MISA- MIcroSAtellite identification tool, MISA)(Lu et al, 2013)搜索Unigene中的SSR位点,搜索参数分别设置为:二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸的最少重复次数分别为6、5、5、4、4。复合SSR 2个位点间最大间隔碱基数为(Maximal number of bases interrupting two SSRs in a compound microsatellite) 100。
1.3 数据分析将Perl语言操作平台下的MISA软件生成的结果MISA文件和STATISTICS文件导入Excel,利用高级筛选功能对结果进行细化和汇总分析,分析SSR类型、数目、序列长度、出现频率和分布情况。用SSR出现频率及分布的平均距离、重复类型、基元组成等,分析曼氏无针乌贼转录组SSR的分布特征,其中,
SSR出现频率=搜索到的SSR数量/总Unigene序列数量;
SSR发生频率=含SSR的Unigene数/Unigene总数;
SSR分布的平均距离=总Unigene长度/搜索到的SSR数量。
利用SPSS软件计算微卫星出现频率与序列长度的皮尔逊(Pearson)相关系数。显著性检验的水平定义为P≤0.05,差异显著;P≤0.01,差异极显著。
2 结果与分析 2.1 转录组序列中SSR的数量和分布特点对127575条Unigene序列进行SSR检测,共得到62124个SSR(完整型SSR 36813个),分布在50626条Unigene序列当中。SSR发生频率(含SSR的Unigene数/Unigene总数)为39.68%,SSR出现频率(检出SSR位点数/Unigene总数) 48.70%,平均每16.6 kb含有1个SSR位点。其中,25548条Unigene含有1个以上SSR位点,复合型SSR数目为22001个。经对曼氏无针乌贼转录组中不同碱基完整型SSR进行分析,部分SSR信息见表 1。
|
|
表 1 曼氏无针乌贼转录组中微卫星数据库的部分结果 Table 1 SSR database of S. japonica transcriptome |
在62124个曼氏无针乌贼微卫星的所有碱基重复类型中,二碱基重复SSR含量最多,约占总数的50.27%,其次为三碱基重复SSR(26.13%)、四碱基重复SSR(22.37%),五碱基和六碱基重复SSR所占比例最少(表 2)。研究发现,曼氏无针乌贼微卫星中共有122种重复基元(motif)。其中,二、三、四、五和六碱基重复基元中在各自重复类型出现频率最多的分别是(AT/AT)n、(AAT/ATT)n、(AAAG/CTTT)n、(AAAAG/ CTTTT)n和(AAAAAG/CTTTTT)n。它们在各自重复基元类型中的比例分别是46.41%、26.58%、77.87%、32.67%和20.61%。
|
|
表 2 曼氏无针乌贼转录中不同微卫星重复基元(motif)出现的频率 Table 2 Occurrence frequency of different microsatellites motifs of S. japonica transcriptome |
在曼氏无针乌贼微卫星序列122种碱基重复模式中,二、三、四、五及六碱基重复基元分别有4、10、28、45和35种。从每种重复基元在总SSR(62124)所占的比例来看,所占比例最高的是(AT/AT)n (23.33%),其次为(AAAG/CTTT)n (17.42%)、(AC/GT)n (16.30%)、(AG/CT)n (10.61%)、(AAT/ATT)n (6.94%)和(AAG/ CTT)n (6.46%)等。不同类型重复基元SSRs占总SSR的比例分布见图 1。
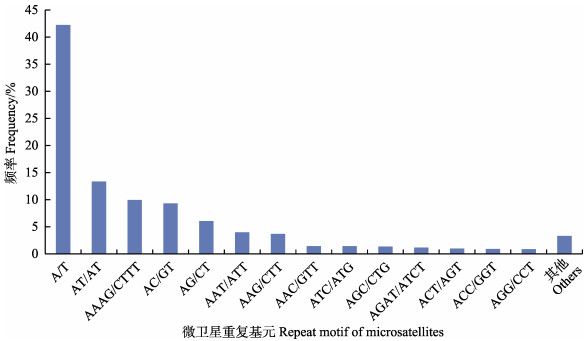
|
图 1 基于重复基元(motif)类型的微卫星分布(考虑到碱基互补作用) Figure 1 Microsatellites distribution on different repeat motifs (considering sequence complementary) “其他”表示频率小于0.50%的重复基元类型 "Other motifs" denoted the repeat motifs with frequency below 0.50% |
曼氏无针乌贼转录组中所发现的36813个完整型微卫星中,微卫星长度存在极显著变异,片段长度从12~147 bp不等,平均长度为25.87 bp。曼氏无针乌贼微卫星序列中,主要是重复长度大于20 bp的长重复序列,长度在20 bp以上的有18774条,达到微卫星(完整型)总数的51%;而重复长度小于20 bp的短重复序列仅占微卫星(完整型)总数的49%,其中,长度在12~15 bp的数量略多,长度在16~20 bp之间的次之(图 2)。此外,片段长度在20 bp以上的低级重复基元(二、三核苷酸重复基元)占20 bp以上SSR(完整型)总数的36.78%,共计13539条。本研究利用SPSS软件进行Pearson相关性分析表明,曼氏无针乌贼微卫星的出现频率和微卫星的长度呈极显著负相关(P<0.01),相关系数为–0.594。
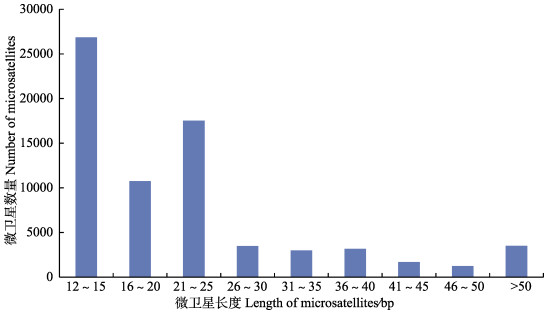
|
图 2 曼氏无针乌贼转录组中微卫星的长度分布 Figure 2 Length distribution of microsatellites in S. japonica transcriptome |
曼氏无针乌贼转录组中微卫星数量与片段长度的分布关系如图 3所示。从图 3可以看出,曼氏无针乌贼SSR数量随着重复次数的增加呈明显下降趋势。二碱基重复随重复次数的增加,SSR数量开始直线下降,当重复次数从10次增加到11次时,曼氏无针乌贼SSR数量陡然增加,之后继续下降,速率趋于平缓。三碱基重复SSR数量则一直随重复次数增加而减少。当二碱基重复次数达到11次、三碱基重复次数达到9次、其他碱基重复次数达到12次时,SSR的下降速率降低趋于平缓,下降速率较为稳定。
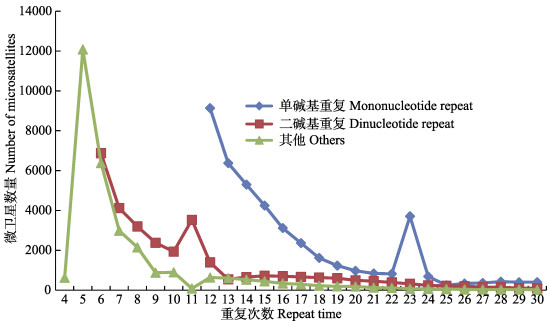
|
图 3 曼氏无针乌贼转录组中SSR数量随重复次数的变化 Figure 3 Variation of repeat times on number of microsatellites in S. japonica transcriptome “其他”表示四、五、六碱基重复SSR的总和 "Others" denoted the total of Tetra-, Penta-, and Hex- nucleotides repeats |
本研究基于对曼氏无针乌贼转录组进行测序所得数据,使用MISA软件对127575个Unigene进行微卫星特征分析,共发现62124个微卫星位点,分布在50626条Unigene序列中。SSR的发生频率(含SSR的Unigene数/Unigene总数)为39.68%,平均间隔16.6 kb出现1个SSR序列。不同物种间微卫星的出现频率不同且差异较大,曼氏无针乌贼视腺组织转录组中微卫星位点出现频率达到48.70%,远大于茶树花(Camellia sinensis)(16.66%)、野三七(Panax vietnamensis var. fuscidiscus)(16.82%)、油松(Pinus tabulaeformis)(2.23%)(王丽鸳等, 2014; 李翠婷等, 2014; 安俊等, 2016)等植物;沟眶象(Eucryp- torrhynchus chinensis) (10.36%)、粘虫(Mythimna separate) (1.93%)、小胸鳖甲(Microdera punctipennis) (7.94%)等昆虫动物(武政梅等, 2016; 胡艳华等, 2015; 库尔班·吐松等, 2017)和中华鳖(Pelodiscus sinensis)(22.3%)、诸氏鲻虾虎鱼(Mugilogobius chulae)(12.97%)、黄姑鱼(Nibea albiflora Richardson) (39.30%)等水生生物(Wang et al, 2013; 蔡磊等, 2015; 龚诗琦等, 2016)。与其他软体动物,如马氏珠母贝(Pinctada martensii)(13.34%)、缢蛏(Sinonovacula constricta)(8.89%)、虾夷扇贝(Mizuhopecten yessoensis) (3.69%)(王忠良等, 2015; 刘博等, 2012; 李云峰等, 2010)等相比,本研究SSR出现频率也较高,说明曼氏无针乌贼视腺组织转录组SSR数量非常丰富。分析其中的原因,一方面,搜索SSR位点时,不同的搜索软件、搜索参数的设置和序列数据量大小等都可能对研究结果产生影响。不同物种的物种特异性可能是最根本的原因(黄海燕等, 2013)。
本研究发现,曼氏无针乌贼微卫星序列的重复类型中,二碱基重复类型的数量最高,约占总数的50.27%;三碱基重复类型的数量次之,约占总数的26.13%。这与草鱼(Ctenopharyngodon idellus)、红鳍东方鲀(Takifugu rubripes)等以二碱基重复为主的水生生物相同(李偲等, 2011; 崔建洲等, 2006)。也有水生生物,如翘嘴鳜(Siniperca chuatsi)、缢蛏等以三碱基重复为主(袁文成等, 2015; 刘博等, 2012)。造成以上差异的原因除了物种特异性的差异之外,与搜索SSR软件的不同和搜索SSR参数的设置也有很大关系(Wei et al, 2011)。
SSR分子标记多态性的高低是判断其潜在利用价值的重要依据,而其长度大小是决定标记多态性高低的关键(李珊等, 2010)。当SSR长度≥20 bp时,多态性较高;当12<SSR长度<20 bp时,SSR呈中度多态性;当SSR长度<12 bp时,SSR呈低多态性(Temnykh et al, 2001)。本研究在SSR筛选设置参数时,已将长度在12 bp以下的SSR序列筛除,发现曼氏无针乌贼转录组中完整型SSR长度在12~147个碱基之间,平均长度为25.87个碱基。在36813个完整型SSR中,片段长度在12~20 bp之间的SSR有18039条(占SSR总数的49%),具有中等多态性;而片段长度≥20 bp的SSR有18774条(占SSR总数的51%),此类SSR具高度多态性。此外,研究发现,片段长度在20 bp以上的低级重复基元(二、三核苷酸重复基元)占20 bp以上SSR总数的36.78%,比例较大,共计13539条。由此可见,曼氏无针乌贼转录组来源的SSR具有较高的多态性潜能,在开发曼氏无针乌贼微卫星分子标记研究方面将具有较高的利用价值(杨华等, 2011; Dreisigacker et al, 2004)。利用SPSS软件进行Pearson相关性分析,发现曼氏无针乌贼微卫星出现频率和其长度呈极显著负相关(P<0.01),相关系数为–0.594。模拟分析研究得出,微卫星重复单位长度与选择压力相关,经受的选择压力越小,重复次数越多;经受的压力越大,重复次数越少(Samadi et al, 1998)。郭文久(2004)分析了29个真核生物基因组微卫星真实统计数据,计算发现,二者相关系数r=–0.304007852,概率值P=1.31967×10‒28,小于0.01,验证了Samadi等(1998)的结果。发现重复motif长度越长,重复次数越少,说明不仅在曼氏无针乌贼基因组微卫星序列中,微卫星序列会随着重复motif长度的增加,重复次数显著减少,在大部分真核生物基因组微卫星序列中也是如此。
4 结论本研究利用高通量测序技术首次对曼氏无针乌贼视腺组织转录组进行测序,对Unigene序列进行SSR检索,共查找到62124个SSR (36813个完整型SSR)位点,分布在50626条Unigene序列中。SSR重复类型主要以二碱基重复为主,其次为三碱基和四碱基重复。曼氏无针乌贼SSR序列中,主要为重复片段长度大于20 bp的长序列微卫星,占微卫星总数(完整型)的51%,呈现较高的多态性。近年来,由于二代测序技术的迅速发展,使得大批量的测序成为可能,推动基因组学和生物信息学研究的不断深入,而借助高通量测序技术有关转录组微卫星方面的研究也逐渐兴起。微卫星分子标记技术的应用越来越广泛,但关于曼氏无针乌贼转录组微卫星的研究仍很少。本研究通过对曼氏无针乌贼视腺组织转录组数据进行微卫星位点的搜索,并分析SSR特征,研究表明,利用转录组数据发掘SSR是高效可行的。研究结果为开发高多态性微卫星引物来进行曼氏无针乌贼亲缘鉴定、放流效果评估、种群遗传结构、种群遗传多样性等研究奠定了基础。
| An J, Zheng CX, Yao Y, et al. Characterization of microsatellites of ovule transcriptome in Pinus tabulaeformis. Journal of Beijing Forestry University, 2016, 38(5): 77-83 [安俊, 郑彩霞, 姚阳, 等. 油松胚珠转录组微卫星特征分析. 北京林业大学学报, 2016, 38(5): 77-83] | |
| Cai L, Yu LJ, Chen XQ, et al. A preliminary screening and characteristic analysis of microsatellite markers from transcriptome sequences in Mugilogobius chulae. Biotechnology Bulletin, 2015, 31(9): 146-151 [蔡磊, 余露军, 陈小曲, 等. 诸氏鲻虾虎鱼转录组序列中微卫星标记的初步筛选及特征分析. 生物技术通报, 2015, 31(9): 146-151] | |
| Chang KM, Wu CW, Lv ZM, et al. Comparison in biochemistry of tissues of wild and cultured Sepiella maindroni. Oceanologia et Limnologia Sinica, 2008, 39(2): 145-151 [常抗美, 吴常文, 吕振明, 等. 曼氏无针乌贼(Sepiella maindroni)野生及养殖群体的生化特征及其形成机制的研究. 海洋与湖沼, 2008, 39(2): 145-151] | |
| Chang KM, Wu CW, Lv ZM, et al. Study on embryo development and artificial breeding of Sepiella maindroni. Journal of Zhejiang Ocean University (Natural Science), 2009, 28(3): 257-263 [常抗美, 吴常文, 吕振明, 等. 曼氏无针乌贼胚胎发育与人工育苗技术的研究. 浙江海洋学院学报(自然科学版), 2009, 28(3): 257-263] | |
| Chen RY, Wang GX, Liu HJ, et al. Difference in parental contribution to reproduction in Japanese flounder (Paralichthys olivaceus). Journal of Fishery Sciences of China, 2013, 20(4): 698-705 [陈睿毅, 王桂兴, 刘海金, 等. 牙鲆亲本对子代贡献率的实验研究. 中国水产科学, 2013, 20(4): 698-705] | |
| Cheng WW, Wang DQ, Wei QW, et al. Effect of restocking enhancement of Chinese sucker in the middle and upper reaches of Yangtze River based on microsatellite loci. Journal of Fishery Sciences of China, 2014, 21(3): 574-580 [成为为, 汪登强, 危起伟, 等. 基于微卫星标记对长江中上游胭脂鱼增殖放流效果的评估. 中国水产科学, 2014, 21(3): 574-580] | |
| Cui JZ, Shen XY, Yang GP, et al. The analysis of simple sequence repeats in Takifugu rubripes genome. Periodical of Ocean University of China, 2006, 36(2): 249-254 [崔建洲, 申雪艳, 杨官品, 等. 红鳍东方鲀基因组微卫星特征分析. 中国海洋大学学报, 2006, 36(2): 249-254] | |
| Dong ZY, Wu CW, Ye DF. A fluorescent method for marking the cuttlefish, Sepiella maindroni de Rochebrune. Journal of Zhejiang Ocean University (Natural Science), 2010, 29(2): 120-128 [董智勇, 吴常文, 叶德锋. 曼氏无针乌贼荧光标记技术初步研究. 浙江海洋学院学报(自然科学版), 2010, 29(2): 120-128] | |
| Dong ZZ. Fauna sinica, Mollusca, Cephalopoda. Beijing: Science Press, 1988: 181-182. [董正之. 中国动物志软体动物门头足纲. 北京: 科学出版社, 1988: 181-182.] | |
| Dreisigacker S, Zhang P, Warburton ML, et al. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments. Crop Science, 2004, 44(2): 381-388 DOI:10.2135/cropsci2004.3810 | |
| Garoia F, Guarniero I, Ramsak A, et al. Microsatellite DNA variation reveals high gene flow and panmictic populations in the Adriatic shared stocks of the European squid and cuttlefish (Cephalopoda). Heredity, 2004, 93(2): 166-174 | |
| Gong SQ, Wang ZY, Xiao SJ, et al. Development and verification of SSR based on transcriptome of yellow drum, Nibea albiflora. Journal of Jimei University (Natural Science), 2016, 21(4): 241-246 [龚诗琦, 王志勇, 肖世俊, 等. 黄姑鱼转录组SSR的开发与验证. 集美大学学报(自然科学版), 2016, 21(4): 241-246] | |
| Guo BY, Qi PZ, Zhu AY, et al. Isolation and characterization of new polymorphic microsatellite markers from the cuttlefish Sepiella maindroni (Cephalopoda; Sepiidae). Genetics and Molecular Research, 2013, 12(3): 2376-2379 DOI:10.4238/2013.July.11.1 | |
| Guo WJ. Primary research on the microsatellite distribution and function in genomes and the relevant computational methodology. Doctoral Dissertation of Sichuan Agricutural University, 2004, 79-83[郭文久. 微卫星在基因组上的分布与功能及其计算方法初步研究. 四川农业大学博士研究生学位论文, 2004, 79-83] | |
| He P. Abundance, polymorphism and applications of microsatellite in eukaryote. Hereditas, 1998, 20(4): 42-47 [何平. 真核生物中的微卫星及其应用. 遗传, 1998, 20(4): 42-47] | |
| Hu YH, Li M, Zhang HF, et al. The information analysis of SSR loci in the Mythimna separate (Walker) transcriptome. Journal of Shanxi Agricultural University (Natural Science Edition), 2015, 35(5): 484-489 [胡艳华, 李敏, 张虎芳, 等. 粘虫转录组中SSR位点的信息分析. 山西农业大学学报(自然科学版), 2015, 35(5): 484-489] | |
| Huang HY, Du HY, Wuyun TN, et al. Development of SSR molecular markers based on transcriptome sequencing of Eucommia ulmoides. Scientia Silvae Sinicae, 2013, 49(5): 176-181 DOI:10.11707/j.1001-7488.20130523 [黄海燕, 杜红岩, 乌云塔娜, 等. 基于杜仲转录组序列的SSR分子标记的开发. 林业科学, 2013, 49(5): 176-181] | |
| Kuerban TS, Chen Z, Liu XN, et al. Bioinformatic analysis of the microsatellites from the transcriptome of desert beetle Microdera punctipennis. Genomics and Applied Biology, 2017(3): 865-873 [库尔班·吐松, 陈真, 刘小宁, 等. 荒漠昆虫小胸鳖甲转录组微卫星的生物信息学分析. 基因组学与应用生物学, 2017(3): 865-873] | |
| Li C, Liu H, Huang R, et al. Identification of typeⅠmicrosatellite markers and their polymorphism in grass carp (Ctenopharyngodon idellus). Acta Hydrobiologica Sinica, 2011, 35(4): 681-687 [李偲, 刘航, 黄容, 等. 草鱼Ⅰ型微卫星标记的发掘及其多态性检测. 水生生物学报, 2011, 35(4): 681-687] | |
| Li CT, Zhang GH, Ma CH, et al. Analysis on SSR loci information in transcriptome of Panax vienamensis var. fuscidiscus and its polymorphism. Chinese Traditional and Herbal Drugs, 2014, 45(10): 1468-1472 DOI:10.7501/j.issn.0253-2670.2014.10.021 [李翠婷, 张广辉, 马春花, 等. 野三七转录组中SSR位点信息分析及其多态性研究. 中草药, 2014, 45(10): 1468-1472] | |
| Li S, Zhou TH, Zhao GF, et al. Data mining for simple sequence repeats in expressed sequence tags from Saruma henryi. Chinese Traditional and Herbal Drugs, 2010, 41(3): 464-468 [李珊, 周天华, 赵桂仿, 等. 马蹄香表达序列标签资源的SSR信息分析. 中草药, 2010, 41(3): 464-468] | |
| Li YF, Liu WD, Gao XG, et al. Construction of cDNA libraries from mantle and kidney of Japanese scallop(Mizuhopecten yessoensis) and ESTs analysis. Journal of Fishery Sciences of China, 2010, 17(3): 578-585 [李云峰, 刘卫东, 高祥刚, 等. 虾夷扇贝外套膜和肾脏组织cDNA文库构建以及EST的初步分析. 中国水产科学, 2010, 17(3): 578-585] | |
| Liu B, Shao YQ, Teng SS, et al. Characterization, development and utilization of EST-derived microsatellites in Sinonovacula constricta. Oceanologia et Limnologia Sinica, 2012, 43(1): 132-137 [刘博, 邵艳卿, 滕爽爽, 等. 缢蛏(Sinonovacula constricta)EST-SSR分布特征及引物开发利用. 海洋与湖沼, 2012, 43(1): 132-137] | |
| Lu X, Wang H, Liu B, et al. Three EST-SSR markers associated with QTL for the growth of the clam Meretrix meretrix revealed by selective genotyping. Marine Biotechnology, 2013, 15(1): 16-25 DOI:10.1007/s10126-012-9453-4 | |
| Moreira AA, Tomás ARG, Hilsdorf AWS. Evidence for genetic differentiation of Octopus vulgaris (Mollusca, Cephalopoda) fishery populations from the southern coast of Brazil as revealed by microsatellites. Journal of Experimental Marine Biology and Ecology, 2011, 407(1): 34-40 DOI:10.1016/j.jembe.2011.06.029 | |
| Pérez-Losada M, Guerra A, Carvalho GR, et al. Extensive population subdivision of the cuttlefish Sepia officinalis (Mollusca:Cephalopoda) around the Iberian Peninsula indicated by microsatellite DNA variation. Heredity, 2002, 89(6): 417-424 DOI:10.1038/sj.hdy.6800160 | |
| Samadi S, Artiguebielle E, Estoup A, et al. Density and variability of dinucleotide microsatellites in the parthenogenetic polyploid snail Melanoides tuberculata. Molecular Ecology, 1998, 7(9): 1233-1236 DOI:10.1046/j.1365-294x.1998.00405.x | |
| Sekino M, Saitoh K, Yamada T, et al. Microsatellite-based pedigree tracing in a Japanese flounder Paralichthys olivaceus hatchery strain:Implications for hatchery management related to stock enhancement program. Aquaculture, 2003, 221(1-4): 255-263 DOI:10.1016/S0044-8486(02)00667-1 | |
| Shi HL, Zhang T, Ping HL, et al. Study on the technology of large scale artificial breeding and enhancement of Sepiella maindroni. Scientific Fish Farming, 2015(12): 44-46 [史会来, 张涛, 平洪领, 等. 曼氏无针乌贼规模化人工繁育及增殖放流技术探讨. 科学养鱼, 2015(12): 44-46] | |
| Temnykh S, DeClerck G, Lukashova A, et al. Computational and experimental analysis of microsatellites in rice (Oryza sativa L. ): Frequency, length variation, transposon associations, and genetic marker potential. Genome Research, 2001, 11(8): 1441-1452 | |
| Wang LY, Wei K, Zhang CC, et al. Characterization of microsatellites in tea (Camellia sinensis) floral transcriptome. Acta Agronomica Sinica, 2014, 40(1): 80-85 [王丽鸳, 韦康, 张成才, 等. 茶树花转录组微卫星分布特征. 作物学报, 2014, 40(1): 80-85] | |
| Wang W, Li C, Ge C, et al. De-novo characterization of the soft-shelled turtle Pelodiscus sinensis transcriptome using Illumina RNA-Seq technology. Journal of Zhejiang University Science B, 2013, 14(1): 58-67 DOI:10.1631/jzus.B1200219 | |
| Wang ZL, Ding Y, Xu YH, et al. Polymorphism of EST-SSRs from Pinctada martensii based on transcriptome datasets. Oceanologia et Limnologia Sinica, 2015, 46(3): 687-693 DOI:10.11693/hyhz20141200361 [王忠良, 丁燏, 许尤厚, 等. 基于转录组数据的马氏珠母贝EST-SSR位点的信息分析及其多态性检测. 海洋与湖沼, 2015, 46(3): 687-693] | |
| Wei WL, Qi XQ, Wang LH, et al. Characterization of the sesame (Sesamum indicum L. ) global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers. BMC Genomics, 2011, 12: 451 | |
| Wu CW, Chi CF, He GY, et al. Isolation via enrichment and characterization of ten polymorphic microsatellite loci in the cuttlefish, Sepiella maindroni de Rochebruns. Acta Oceanologica Sinica, 2010, 29(6): 121-124 DOI:10.1007/s13131-010-0083-2 | |
| Wu CW, Dong ZY, Chi CF, et al. Reproductive and spawning habits of Sepiella maindroni off Zhejiang, China. Oceanologia et Limnologia Sinica, 2010, 41(1): 39-46 DOI:10.11693/hyhz201001005005 [吴常文, 董智勇, 迟长凤, 等. 曼氏无针乌贼(Sepiella maindroni)繁殖习性及其产卵场修复的研究. 海洋与湖沼, 2010, 41(1): 39-46] | |
| Wu Z, Zhang XJ, Jiang XM, et al. Isolation of GT microsatellite loci from Sepiella maindroni. Biotechnology Bulletin, 2011(3): 120-124 [吴璋, 张晓菊, 蒋霞敏, 等. 曼氏无针乌贼GT微卫星位点的筛选. 生物技术通报, 2011(3): 120-124] | |
| Wu ZM, Gao P, Wen JB. Characteristic analysis of microsatellite in Eucryptorrhynchus chinensis transcriptome. Journal of Environmental Entomology, 2016, 38(5): 979-983 [武政梅, 高朋, 温俊宝. 沟眶象转录组微卫星特征分析. 环境昆虫学报, 2016, 38(5): 979-983] | |
| Yang H, Chen Q, Wei ZL, et al. Analysis on SSR information in Camellia sinensis transcriptome. Journal of Anhui Agricultural University, 2011, 38(6): 882-886 [杨华, 陈琪, 韦朝领, 等. 茶树转录组中SSR位点的信息分析. 安徽农业大学学报, 2011, 38(6): 882-886] | |
| Yuan WC, Huang HZ, Li WL, et al. Analysis of EST-SSRs information in Siniperca chuatsi transcriptome and detection of polymorphism. Oceanologia et Limnologia Sinica, 2015, 46(2): 403-409 DOI:10.11693/hyhz20140900248 [袁文成, 黄鹤忠, 李文龙, 等. 翘嘴鳜(Siniperca chuatsi)转录组EST-SSR位点的信息分析及其多态性检测. 海洋与湖沼, 2015, 46(2): 403-409] | |
| Zhang C, Guo BY, Wu CW, et al. Construction and identification of DNA libraries enriched for microsatellite repeat sequences of Sepiella maindroni. Journal of Zhejiang Ocean University (Natural Science), 2014, 33(6): 483-487 [张川, 郭宝英, 吴常文, 等. 曼氏无针乌贼微卫星DNA富集文库构建与鉴定. 浙江海洋学院学报(自然科学版), 2014, 33(6): 483-487] |



