2. 青岛海洋科学与技术国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 农业部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071
2. Laboratory for Marine Fisheries Science and Food Production Process, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071;
3. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071
MicroRNAs(miRNA)是一类长约18~25个核苷酸的非编码小RNA分子,广泛分布于真核生物中(Guarnieri et al, 2008),主要在转录后水平参与基因表达的调控,在细胞增殖、分化、代谢、凋亡等一系列生理过程中发挥着重要的作用(Ott et al, 2016; Li et al, 2016; Shigeyasu et al, 2017; Han et al, 2013; Yan et al, 2012; Nothnick et al, 2010; Passon et al, 2012)。已有研究表明,miRNA与性腺发育密切相关(Grossman et al, 2016),其在原始生殖细胞分化(Yang et al, 2016; Wu et al, 2011; Huszar et al, 2013)、卵泡发育和精子发生过程中发挥着重要的调控作用(Modzelewski et al, 2015; Kaneda et al, 2009; Kim et al, 2013)。
miRNA的合成过程受到多个基因的共同调控,其中,Drosha和Exportin 5基因发挥着至关重要的作用(Kim et al, 2016; Kuehbacher et al, 2007)。首先,在细胞核中miRNA基因的初级转录产物(pri-miRNA)由RNA酶ⅢDrosha剪切成前体miRNA (pre-miRNA) (Denli et al, 2004);随后,pre-miRNA被转运蛋白Exportin 5转运到细胞质中进行后续处理(Romero-Cordoba et al, 2014)。已有研究报道,Drosha和Exportin 5基因能够通过调节性腺miRNA的合成,进而调控性腺的发育(Yang et al, 2016; Wu et al, 2012; Song et al, 2007)。Yang等(2016)发现,Drosha能够调控miRNA的合成,从而影响果蝇(Drosophila)原始生殖细胞的分化和卵巢的发育;Wu等(2012)发现,敲除雄性小鼠(Mus musculus)Drosha基因,能够造成生殖细胞miRNAs严重缺失,精子发生异常,最终导致雄性不育;Song等(2007)发现,Drosha和Exportin 5基因在海胆(Strongylocentrotus purpuratus)卵巢中均有较高表达。目前,Drosha和Exportin 5基因在性腺发育调控方面的研究主要集中在模式生物中,在三疣梭子蟹(Portunus trituberculatus)中尚未见相关报道。
三疣梭子蟹广泛分布于中国、日本、韩国等海域,是我国重要的捕捞对象和海水养殖品种(管卫兵等, 2009b)。无论对于三疣梭子蟹野生资源的合理利用,还是养殖业的可持续发展,研究其繁殖生物学都是十分必要的基础性工作。但目前,有关三疣梭子蟹繁殖生物学的研究还相对匮乏。本研究利用RACE技术,首次克隆了三疣梭子蟹miRNA合成通路重要基因Drosha和Exportin 5,通过qRT-PCR分析了其在不同组织以及性腺发育不同时期的表达水平,为进一步探究三疣梭子蟹等甲壳类性腺发育调控机制提供参考资料。
1 材料与方法 1.1 材料实验用健康三疣梭子蟹均取自中国水产科学研究院黄海水产研究所的实验基地山东昌邑海丰水产有限公司,在塑料箱(560 mm×360 mm×280 mm)中暂养7 d,持续充氧,控制水温为20±2℃,pH=8.2,盐度为33,氨氮含量为0.078~0.127 mg/L,亚硝酸盐含量为0.074~0.132 mg/L,每天换水1/3,并清理箱底排泄物,早晚各投喂适量蛤蜊肉。
SMART RACE cDNA Amplification Kit购于Clontech公司,Trizol Reagent购于Invitrogen公司;E.Z.N.A-Gel Extraction Kit购于Omega公司;LA Taq酶,pMD18-T载体,DH5α感受态细胞,PrimeScript RT Reagent Kit和SYBR-Premix Ex Taq Ⅱ均购于TaKaRa公司;实验中所有引物合成和测序反应均在青岛擎科梓熙生物技术有限公司进行。
1.2 三疣梭子蟹性腺不同发育时期组织样品的采集从2015年7月~2016年3月,每月于实验基地连续取样,在性腺不同发育时期,每时期各取5只。参考相关文献(管卫兵等, 2009a; 吴旭干等, 2007),根据精巢和卵巢的外部特征进行粗略分期,取不同个体相同部位的精巢和卵巢样品,分别固定、切片、HE染色后,根据精巢和卵巢组织学特征,确定精巢和卵巢组织的具体分期。
1.3 总RNA提取及RACE一链cDNA的合成Trizol试剂提取三疣梭子蟹各组织总RNA,通过核酸定量仪和1.0%的琼脂糖凝胶检测其质量。选取质量好的总RNA样品,用SMART RACE Amplification Kit合成3ʹ和5ʹ RACE的一链cDNA。
1.4 三疣梭子蟹Drosha和Exportin 5基因全长克隆根据三疣梭子蟹性腺转录组数据库中Drosha和Exportin 5基因的序列片段,用Primer Premier 5.0设计其3ʹ和5ʹ RACE引物(表 1),3ʹ和5ʹ末端使用Advantage 2 PCR Kit进行扩增,运用巢氏PCR方法,第1轮反应使用外侧引物DS-3ʹ-F、DS-5ʹ-F、E-3ʹ-F和E-5ʹ-F,分别与UPM配对,反应程序:94℃ 5 min;94℃ 30 s,68℃ 3 s,72℃ 1 min,30个循环。第2轮反应以第1轮扩增产物为模板,使用内侧引物DS-3ʹ-S、DS-5ʹ-S、E-3ʹ-S和E-5ʹ-S,分别与NUP配对,反应程序:94℃ 5 min;94℃ 30 s,60℃ 30 s,72℃ 1 min,35个循环。扩增产物用E.Z.N.A-Gel Extraction Kit切胶回收纯化,将纯化后的PCR产物连接到pMD18-T载体,然后转入DH5α感受态细胞,经37℃活化45 min,涂板,过夜培养后,通过蓝白斑筛选,挑取阳性单克隆,菌落PCR检测后送公司测序。
|
|
表 1 本实验用到的引物 Table 1 Primers used in this study |
测序得到RACE结果序列,利用Contig Express软件,将其与三疣梭子蟹Drosha和Exportin 5基因EST序列拼接后,获得2个基因的cDNA全长序列。利用ORF Finder软件(http://www.ncbi.nlm.nih.gov/projects/gorf/orfig.cgi)预测其开放阅读框,利用Gene Tool软件进行序列分析。利用EditSeq软件进行氨基酸序列预测,利用NCBI Blastx软件(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列相似性比对,利用NCBI Conserved damain软件(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析其蛋白质功能结构域,利用ExPASy软件(http://web.expasy.org/compute_pi/)预测其分子量和等电点,利用SingalP 4.1 Server软件(http://www.cbs.dtu.dk/services/SignalP/)预测基因信号肽序列,利用SOSUI软件(http://harrier.nagahama-i-bio.ac.jp/sosui/)分析其跨膜区域。从GenBank中下载其他物种的Drosha和Exportin 5基因序列,利用DNAMAN软件进行同源分析,在此基础上,利用MAGE 5.0软件构建2个基因的NJ系统进化树。
1.6 组织表达分析用Trizol提取三疣梭子蟹的胃、眼柄、输精管、鳃、肠、肝胰腺、脑神经节、胸神经节、血淋巴、肌肉、心脏以及卵巢和精巢不同时期组织的总RNA(每个组织取3个个体)。使用PrimeScript RT Reagent Kit反转录成cDNA,-20℃保存。
根据已获得的三疣梭子蟹β-actin、Drosha和Exportin 5开放阅读框序列,设计qRT-PCR引物(表 1),分析Drosha和Exportin 5基因在三疣梭子蟹不同组织(胃、眼柄、输精管、鳃、肠、肝胰腺、脑神经节、胸神经节、血淋巴、肌肉、心脏)及精巢和卵巢不同时期的相对表达量(每个样品设3个重复)。反应体系为10 μl:5 μl SYBR Premix Ex Taq Ⅱ (2×)、0.4 μl正向引物(10 μmol/L)、0.4 μl反向引物(10 μmol/L)、0.2 μl ROX Reference Dye Ⅱ (50×)、1.0 μl cDNA、3.0 μl DEPC水。反应程序:95℃ 30 s;95℃ 5 s,60℃ 34 s,40个循环;95℃ 15 s,60℃ 1 min,95℃ 15 s。使用2-ΔΔCt法计算Drosha和Exportin 5基因相对表达量,用SPSS 19.0软件进行数据差异显著性分析。
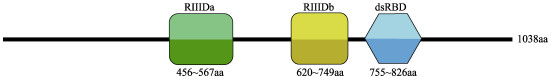
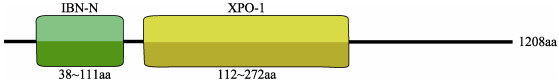
2 结果与分析 2.1 三疣梭子蟹Drosha和Exportin 5基因cDNA全长的获得和序列分析将测序得到的3ʹ和5ʹ RACE序列,分别与三疣梭子蟹Drosha和Exportin 5基因EST序列拼接后,得到其cDNA全长,GenBank登录号分别为KY625632和KY402080。序列分析显示,Drosha基因cDNA序列全长3443 bp,包括5ʹ非编码区184 bp和3ʹ非编码区142 bp,其中,开放阅读框3117 bp,编码1038个氨基酸,理论等电点为8.22,预测蛋白质的分子量为121.09 kDa;Exportin 5基因cDNA序列全长5000 bp,包括3627 bp的ORF,1204 bp的3ʹ端UTR以及169 bp的5ʹ端UTR。编码1208个氨基酸,预测分子量为136.15 kDa,理论等电点为5.86。NCBI Conserved domain软件分析发现,三疣梭子蟹Drosha基因包含2个相邻的RNA酶Ⅲ结构域(RⅢDa)(456~567aa)和RⅢDb(620~749aa)和1个双链RNA结合结构域(dsRBD)(755~826aa)(图 3)。Exportin 5基因包含1个IBN-N(38-111aa)结构域和1个XPO-1(112-272aa)结构域(图 4)。同时,在Drosha基因RⅢDa和RⅢDb两结构域中分别发现了RNA酶Ⅲ家族所特有的,由9个氨基酸残基组成的特征性标记位点ERLEFLGDA和QRLEFLGDT(图 1、图 2)。相关在线软件分析发现,三疣梭子蟹Drosha和Exportin 5基因的编码蛋白均没有跨膜结构和信号肽序列,为水溶性蛋白。

|
图 1 三疣梭子蟹Drosha基因cDNA序列全长以及其编码的氨基酸序列 Figure 1 The nucleotide sequence and deduced amino acids sequence of the swimming crab Drosha gene 起始密码子ATG和终止密码子TGA均用粗线方框标出,RⅢDa、RⅢDb和dsRBD结构域分别用下划线、细线方框和阴影标出。特征性标记位点(ERLEFLGDA和QRLEFLGDT)加粗标出 Start codon (ATG) and stop codon (TGA) are marked with thick boxes, the RⅢDa, RⅢDb and dsRBD domains are marked with underline, filament box and shadow, respectively. The signature motifs (ERLEFLGDA, QRLEFLGDT) are bolded |

|
图 2 三疣梭子蟹Exportin 5基因cDNA序列全长以及其编码的氨基酸序列 Figure 2 The nucleotide sequence and deduced amino acids sequence of the swimming crab Exportin 5 gene 起始密码子ATG和终止密码子TGA均用方框标出,IBN-N和XPO-1结构域分别用阴影和下划线标出 Start codon (ATG) and stop codon (TGA) are marked with square frame; IBN-N and XPO-1 domain are marked with underline and shadow, respectively |
|
图 3 三疣梭子蟹Drosha蛋白的结构域示意 Figure 3 Domain organization of the swimming crab Drosha protein |
|
图 4 三疣梭子蟹Exportin 5蛋白的结构域示意图 Figure 4 Domain organization of the swimming crab Exportin 5 protein |
利用NCBI blastx软件分析得知,三疣梭子蟹Drosha与日本对虾(Marsupenaeus japonicus)该蛋白的氨基酸序列同源性最高,为94%,与白蚁(Zootermopsis nevadensis)、达氏按蚊(Anopheles darlingi)和黑森瘿蚊(Mayetiola destructor)的同源性分别为79%、75%和73%。三疣梭子蟹Exportin 5与黑蚁(Lasius niger)和蓝莓蜂(Habropoda laboriosa)该蛋白氨基酸序列同源性最高,为44%。
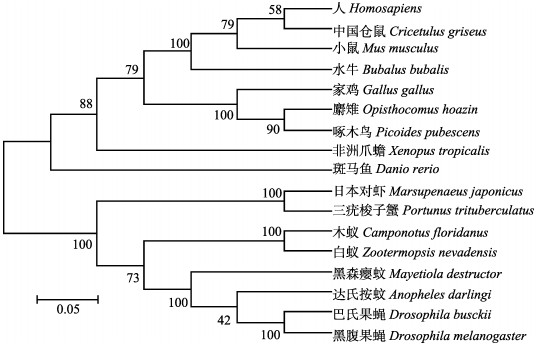
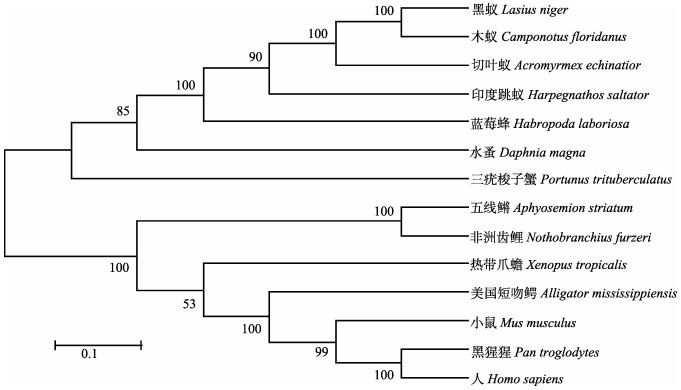
利用MEGA 5.0以邻接法(Neighbor-joining)构建三疣梭子蟹Drosha和Exportin 5系统进化树,结果显示,二者均可分为脊椎动物和节肢动物两大簇,且三疣梭子蟹Drosha和Exportin 5均在节肢动物一簇中,与鱼类和哺乳类等脊椎动物亲缘关系较远,其中,三疣梭子蟹Drosha与日本对虾紧密聚为一支,亲缘关系最近,其次为白蚁和木蚁(Camponotus floridanus),而三疣梭子蟹Exportin 5与水蚤(Daphnia magna)在进化上关系最近(图 5和图 6)。
|
图 5 基于Drosha氨基酸序列的NJ进化树 Figure 5 NJ tree based on Drosha amino acid sequences 各物种Drosha基因GenBank登录号:人(NP_037367.3)、中国仓鼠(ERE81427.1)、小鼠(NP_001123621.1)、水牛(BAS30507.1)、家鸡(NP_001006379.1)、麝雉(KFR12253.1)、啄木鸟(KFV67625.1)、非洲爪蟾(NP_001107152.1)、斑马鱼(NP_001103942.1)、日本对虾(ADB65770.2)、三疣梭子蟹(KY625632)、木蚁(EFN62400.1)、白蚁(KDR08017.1)、黑森瘿蚊(AFX89030.1)、达氏按蚊(ETN65888.1)、巴氏果蝇(ALC42743.1)、黑腹果蝇(NP_477436.1) The GenBank accession numbers of different species' Drosha gene were as follows: Homo sapiens(NP_037367.3), Cricetulus griseus(ERE81427.1), Mus musculus(NP_001123621.1), Bubalus bubalis(BAS30507.1), Gallus gallus(NP_001006379.1), Opisthocomus hoazin(KFR12253.1), Picoides pubescens(KFV67625.1), Xenopus tropicalis(NP_001107152.1), Danio rerio(NP_001103942.1), Marsupenaeus japonicus(ADB65770.2), Portunus trituberculatus(KY625632), Camponotus floridanus(EFN62400.1), Zootermopsis nevadensis(KDR08017.1), Mayetiola destructor(AFX89030.1), Anopheles darlingi(ETN65888.1), Drosophila busckii(ALC42743.1), Drosophila melanogaster(NP_477436.1) |
|
图 6 基于Exportin 5氨基酸序列的NJ进化树 Figure 6 NJ tree based on Exportin 5 amino acid sequences 各物种Exportin 5基因序列GenBank登录号:木蚁(EFN62948.1)、黑蚁(KMQ95474.1)、切叶蚁(EGI70297.1)、印度跳蚁(EFN89855.1)、蓝莓蜂(KOC65194.1)、水蚤(JAN75960.1)、三疣梭子蟹(KY402080)、五线鳉(SBP29565.1)、非洲齿鲤(SBP51127.1)、热带爪蟾(NP_989112.2)、美洲短吻鳄(KQL87848.1)、小家鼠(AAI31662.1)、黑猩猩(JAA34037.1)、人(NP_065801.1) The GenBank accession numbers of different species' Exportin 5 gene were as follows: Camponotus floridanus(EFN62948.1), Lasius niger(KMQ95474.1), Acromyrmex echinatior(EGI70297.1), Harpegnathos saltator(EFN89855.1), Habropoda laboriosa(KOC65194.1), Daphnia magna(JAN75960.1), Portunus trituberculatus(KY402080), Aphyosemion striatum(SBP29565.1), Nothobranchius furzeri(SBP51127.1), Xenopus tropicalis(NP_989112.2), Alligator mississippiensis(KQL87848.1), Mus musculus(AAI31662.1), Pan troglodytes(JAA34037.1), Homo sapiens(NP_065801.1) |
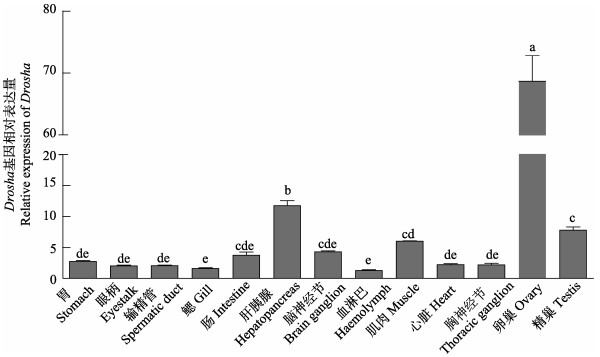
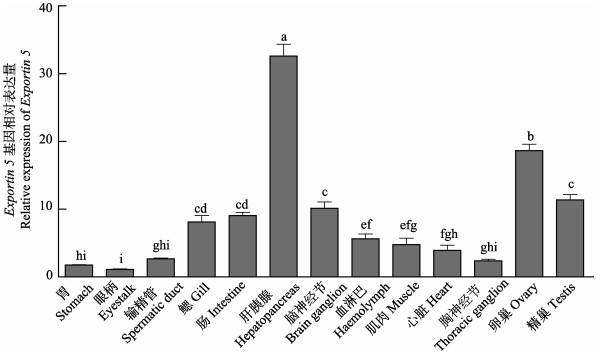
qRT-PCR分析三疣梭子蟹Drosha和Exportin 5基因在不同组织中的相对表达量(图 7和图 8),结果显示,2个基因在各种组织中均表达,其中,Drosha基因在卵巢中表达量最高(P<0.05),其次依次为肝胰腺和精巢,而Exportin 5基因高表达于肝胰腺(P<0.05),其次依次为卵巢和精巢。
|
图 7 三疣梭子蟹Drosha基因在不同组织中的表达分布状况 Figure 7 The distribution of Drosha gene in different tissues of the swimming crab 不同字母表示组间差异显著(P<0.05),下同 The different lowercase letters represented the significant difference in the different tissues of swimming crab (P<0.05), the same as below |
|
图 8 三疣梭子蟹Exportin 5基因在不同组织中的表达分布状况 Figure 8 The distribution of Exportin 5 gene in different tissues of the swimming crab |
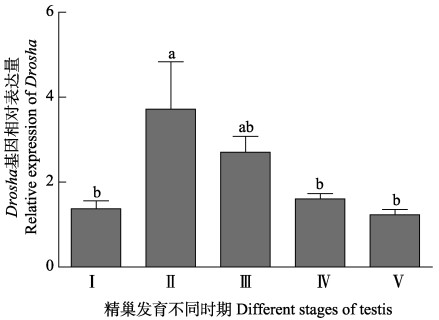
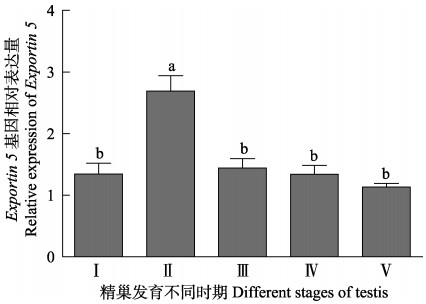
三疣梭子蟹Drosha和Exportin 5基因在精巢发育不同时期表达均存在差异(图 9和图 10),但变化趋势一致,且其在Ⅰ期、Ⅲ期、Ⅳ期和Ⅴ期表达量差异均不显著(P>0.05)。此外,2个基因都在精巢发育Ⅱ期高表达,之后呈下降趋势。
|
图 9 三疣梭子蟹Drosha基因在不同发育时期精巢组织中的表达 Figure 9 The expression of Drosha gene in testis of the swimming crab at different gonadal stages |
|
图 10 三疣梭子蟹Exportin 5基因在不同发育时期精巢组织中的表达 Figure 10 The expression of Exportin 5 gene in testis of the swimming crab at different gonadal stages |
三疣梭子蟹Drosha和Exportin 5基因在卵巢发育不同时期表达量变化趋势大致相同,均随着卵巢发育(Ⅱ~Ⅴ期)逐渐升高,且在卵巢发育Ⅴ期,2个基因的表达量均显著高于其他时期(P<0.05)(图 11和图 12)。其中,Drosha基因在Ⅳ期也显著高于Ⅰ期、Ⅱ期、Ⅲ期和Ⅵ期(P<0.05),而Exportin 5基因在Ⅰ期、Ⅲ期、Ⅳ期和Ⅵ期表达量差异不显著(P>0.05)。
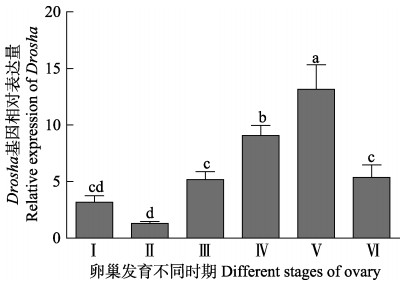
|
图 11 三疣梭子蟹Drosha基因在不同发育时期卵巢组织中的表达 Figure 11 The expression of Drosha gene in the ovary of swimming crab at different gonadal stages |
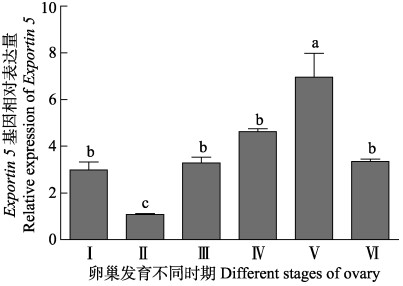
|
图 12 三疣梭子蟹Exportin 5基因在不同发育时期卵巢组织中的表达 Figure 12 The expression of Exportin 5 gene in the ovary of swimming crab at different gonadal stages |
Drosha和Exportin 5基因是miRNA合成通路中的重要基因,本研究克隆获得三疣梭子蟹Drosha和Exportin 5基因cDNA全长序列。序列分析发现,Drosha包含2个相邻的RNA酶Ⅲ结构域(RⅢDa和RⅢDb)和1个dsRBD,属于RNA酶Ⅲ家族(Lee et al, 2006; Filippov et al, 2000; Wu et al, 2000)。RⅢDa和RⅢDb结构域可各结合1个DGCR8,发挥其双链RNA剪切功能(Kwon et al, 2016; Huang et al, 2012)。Exportin 5基因包含IBN-N和XPO-1两个功能结构域,是核质蛋白β家族中的一员(Brownawell et al, 2002),XPO-1是其发挥核输出受体功能的关键结构域,IBN-N具体功能暂不清楚(Sharma et al, 2017)。同源分析结果显示,三疣梭子蟹Drosha氨基酸序列与其他无脊椎动物的相似性为67%~94%,序列高度保守。三疣梭子蟹Exportin 5与其他无脊椎动物的同源度较低,为39%~46%。但有研究发现,从低等真菌酵母菌到高等生物人类,Exportin 5与靶RNA的结合活性位点高度保守(Shibata et al, 2006)。
本研究对三疣梭子蟹Drosha和Exportin 5基因组织差异性表达分析结果显示,其在检测的各组织中均有表达,但在不同组织中的表达差异较大,在肝胰腺和卵巢中的表达量均显著高于其他组织,其次为精巢。该结果暗示三疣梭子蟹Drosha和Exportin 5基因可能在性腺发育过程中发挥调控作用。性腺不同发育时期的表达分析结果显示,在性腺发育过程中,Drosha和Exportin 5基因表达量的变化趋势大致相同,暗示这2个基因可能协同调控三疣梭子蟹性腺发育过程。在精巢发育过程中,Drosha和Exportin 5基因在精巢发育Ⅱ期的表达量最高,之后均呈依次下降趋势,表明两个基因可能在精巢发育Ⅱ期发挥重要调控作用。精巢发育Ⅱ期是精子发生的重要时期,期间初级精母细胞大量分化,并减数分裂生成次级精母细胞,细胞代谢旺盛(管卫兵等, 2009b)。Wu等(2012)敲除小鼠Drosha基因发现,其精母细胞存在大量基因表达失调现象,导致精母细胞减数分裂中断,精子发生困难;Iwasaki等(2013)发现抑制小鼠胚胎纤维细胞Exportin 5基因表达,导致细胞增殖缺陷,表现为从G1期到S期过渡延迟,同时发现,其细胞周期开始前,整体miRNA水平的提高与Exportin 5的调控密不可分。由此可见,Drosha和Exportin 5基因可能参与了精子发生过程的调控,同时也暗示miRNA可能在三疣梭子蟹精母细胞的分化和减数分裂过程中发挥重要作用。在卵巢不同发育时期,三疣梭子蟹Drosha和Exportin 5基因表达量从Ⅱ期到Ⅴ期均逐渐升高,且在Ⅴ期达到峰值(P<0.05),期间卵母细胞大量积累其减数分裂所需营养物质,并逐渐发育成熟(吴旭干等, 2007)。其中,Drosha基因在Ⅳ期也显著高于Ⅰ期、Ⅱ期、Ⅲ期和Ⅵ期(P<0.05),而Exportin 5基因在Ⅰ期、Ⅲ期、Ⅳ期和Ⅵ期表达量差异不显著(P>0.05),暗示2个基因在三疣梭子蟹卵巢发育过程中的不同功能。有研究表明,在海胆成熟卵巢中Drosha和Exportin 5基因均有较高表达(Song et al, 2007)。Yang等(2016)发现,Drosha能够调控果蝇原始生殖细胞的分化和卵巢的发育。因此,推测这2个基因可能通过调节miRNAs的合成在三疣梭子蟹卵母细胞成熟过程中发挥着重要的作用。
本研究首次成功克隆三疣梭子蟹Drosha和Exportin 5基因cDNA全长序列,分析了其在三疣梭子蟹不同组织和精卵巢不同时期的表达情况,暗示Drosha和Exportin 5基因可能在三疣梭子蟹性腺发育过程中发挥协同调控作用,为进一步深入研究miRNA在三疣梭子蟹性腺发育过程的分子机制提供了基础资料。
| Brownawell AM, Macara IG. Exportin-5, a novel karyopherin, mediates nuclear export of double-stranded RNA binding proteins. Journal of Cell Biology, 2002, 156(1): 53-64 DOI:10.1083/jcb.200110082 | |
| Denli AM, Tops BB, Plasterk RH, et al. Processing of primary microRNAs by the microprocessor complex. Nature, 2004, 432(7014): 231-235 DOI:10.1038/nature03049 | |
| Filippov V, Solovyev V, Filippova M, et al. A novel type of RNase Ⅲ family proteins in eukaryotes. Gene, 2000, 245(1): 213-221 DOI:10.1016/S0378-1119(99)00571-5 | |
| Grossman H, Shalgi R. A Role of MicroRNAs in cell differentiation during gonad development. Springer International Publishing, 2016, 58: 309-336 | |
| Guan WB, Xuan FJ, Chen HH, et al. Reproductive characteristics and condition status of adult swimming crab Portunus trituberculatus (Brachyura:Portunidae) in East China Sea. Marine Fisheries, 2009a, 31(2): 120-127 [管卫兵, 宣富君, 陈辉辉, 等. 东海三疣梭子蟹成熟群体生殖特征和条件状况. 海洋渔业, 2009a, 31(2): 120-127] | |
| Guan WB, Xuan FJ, Dai XJ, et al. Spermatogenesis and spermatophore formation of swimming crab (Portunus trituberculatus Miers, 1876) in East China Sea. Journal of Fishery Sciences of China, 2009b, 16(2): 173-182 [管卫兵, 宣富君, 戴小杰, 等. 东海三疣梭子蟹精子发生及精荚形成. 中国水产科学, 2009b, 16(2): 173-182] | |
| Guarnieri DJ, Dileone RJ. MicroRNAs:A new class of gene regulators. Annals of Medicine, 2008, 40(3): 197-208 DOI:10.1080/07853890701771823 | |
| Han Y, Liu Y, Gui Y, et al. Inducing cell proliferation inhibition and apoptosis via silencing Dicer, Drosha, and Exportin 5 in urothelial carcinoma of the bladder. Journal of Surgical Oncology, 2013, 107(2): 201-205 DOI:10.1002/jso.23214 | |
| Huang T, Xu D, Zhang X. Characterization of shrimp Drosha in virus infection. Fish & Shellfish Immunology, 2012, 33(3): 575-581 | |
| Huszar JM, Payne CJ. MicroRNA 146 (Mir146) modulates spermatogonial differentiation by retinoic acid in mice. Biology of Reproduction, 2013, 88(1): 15 | |
| Iwasaki YW, Kiga K, Kayo H, et al. Global microRNA elevation by inducible Exportin 5 regulates cell cycle entry. RNA, 2013, 19(4): 490-497 DOI:10.1261/rna.036608.112 | |
| Kaneda M, Tang F, O'Carroll D, et al. Essential role for Argonaute2 protein in mouse oogenesis. Epigenetics & Chromatin, 2009, 2(1): 1-8 | |
| Kim YJ, Ku SY, Kim YY, et al. MicroRNAs transfected into granulosa cells may regulate oocyte meiotic competence during in vitro maturation of mouse follicles. Human Reproduction, 2013, 28(11): 3050-3061 DOI:10.1093/humrep/det338 | |
| Kim YK, Kim B, Kim VN. Re-evaluation of the roles of DROSHA, Exportin 5, and DICER in microRNA biogenesis. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(13): 1881-1889 DOI:10.1073/pnas.1602532113 | |
| Kuehbacher A, Urbich C, Zeiher AM, et al. Role of Dicer and Drosha for endothelial microRNA expression and angiogenesis. Circulation Research, 2007, 101(1): 59-68 DOI:10.1161/CIRCRESAHA.107.153916 | |
| Kwon SC, Nguyen TA, Choi YG, et al. Structure of human DROSHA. Cell, 2016, 164(1-2): 81-90 DOI:10.1016/j.cell.2015.12.019 | |
| Lee Y, Han J, Yeom KH, et al. Drosha in primary microRNA processing. Cold Spring Harbor Symposia on Quantitative Biology, 2006, 71: 51-57 DOI:10.1101/sqb.2006.71.041 | |
| Li Y, Wang X, He B, et al. Downregulation and tumor-suppressive role of XPO5 in hepatocellular carcinoma. Molecular and Cellular Biochemistry, 2016, 415(1): 1-9 | |
| Modzelewski AJ, Hilz S, Crate EA, et al. Dgcr8 and Dicer are essential for sex chromosome integrity during meiosis in males. Journal of Cell Science, 2015, 128(12): 2314-2327 DOI:10.1242/jcs.167148 | |
| Nothnick WB, Healy C, Hong X. Steroidal regulation of uterine miRNAs is associated with modulation of the miRNA biogenesis components Exportin-5 and Dicer1. Endocrine, 2010, 37(2): 265-273 DOI:10.1007/s12020-009-9293-9 | |
| Ott CA, Linck L, Kremmer E, et al. Induction of exportin-5 expression during melanoma development supports the cellular behavior of human malignant melanoma cells. Oncotarget, 2016, 7(38): 62292-62304 | |
| Passon N, Gerometta A, Puppin C, et al. Expression of Dicer and Drosha in triple-negative breast cancer. Journal of Clinical Pathology, 2012, 65(4): 320-326 DOI:10.1136/jclinpath-2011-200496 | |
| Romero-Cordoba SL, Salido-Guadarrama I, Rodriguez-Dorantes M, et al. miRNA biogenesis:Biological impact in the development of cancer. Cancer Biology & Therapy, 2014, 15(11): 1444-1455 | |
| Sharma C, Mohanty D. Sequence-and structure-based analysis of proteins involved in miRNA biogenesis. Journal of Biomolecular Structure and Dynamics, 2017: 1-13 | |
| Shigeyasu K, Okugawa Y, Toden S, et al. Exportin-5 functions as an oncogene and a potential therapeutic target in colorectal cancer. Clinical Cancer Research, 2017, 23(5): 1312-1322 DOI:10.1158/1078-0432.CCR-16-1023 | |
| Shibata S, Sasaki M, Miki T, et al. Exportin-5 orthologues are functionally divergent among species. Nucleic Acids Research, 2006, 34(17): 4711-4721 DOI:10.1093/nar/gkl663 | |
| Song JL, Wessel GM. Genes involved in the RNA interference pathway are differentially expressed during sea urchin development. Developmental Dynamics, 2007, 236(11): 3180-3190 DOI:10.1002/dvdy.v236:11 | |
| Wu H, Xu H, Miraglia LJ, et al. Human RNase Ⅲ is a 160-kDa protein involved in preribosomal RNA processing. Journal of Biological Chemistry, 2000, 275(47): 36957-36965 DOI:10.1074/jbc.M005494200 | |
| Wu J, Bao J, Wang L, et al. MicroRNA-184 downregulates nuclear receptor corepressor 2 in mouse spermatogenesis. BMC Developmental Biology, 2011, 11(1): 1-10 DOI:10.1186/1471-213X-11-1 | |
| Wu Q, Song R, Ortogero N, et al. The RNase Ⅲ enzyme DROSHA is essential for microRNA production and spermatogenesis. Journal of Biological Chemistry, 2012, 287(30): 25173-25190 DOI:10.1074/jbc.M112.362053 | |
| Wu XG, Yao GG, Yang XZ, et al. A study on the ovarian development of Portunus trituberculatus in East China Sea during the first reproductive cycle. Acta Oceanologica Sinica, 2007, 29(4): 120-127 [吴旭干, 姚桂桂, 杨筱珍, 等. 东海三疣梭子蟹第一次卵巢发育规律的研究. 海洋学报, 2007, 29(4): 120-127] | |
| Yang H, Li M, Hu X, et al. MicroRNA-dependent roles of Drosha and Pasha in the Drosophila larval ovary morphogenesis. Developmental Biology, 2016, 416(2): 312-323 DOI:10.1016/j.ydbio.2016.06.026 | |
| Yan M, Huang H, Wang T, et al. Dysregulated expression of Dicer and Drosha in breast cancer. Pathology & Oncology Research, 2012, 18(2): 343-348 |



