2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071
蜕皮贯穿甲壳动物的整个生活史,与其生长、发育和繁殖息息相关。蜕皮主要受互为拮抗的Y-器官分泌蜕皮激素(MH, 蜕皮甾类)和眼柄内X-器官窦腺复合体分泌蜕皮抑制激素(MIH)影响。蜕皮激素是调控甲壳动物蜕皮及变态发育的重要激素,它的活性形式是20-羟基蜕皮酮(20-hydroxyecdysone, 20E)。在昆虫体内,蜕皮激素受体(Ecdysteroid receptor, ECR)与超气门蛋白(Ultraspiracle, USP)形成异源二聚体,在甲壳动物体内,ECR与维甲酸X受体(Retinoid X receptor, RXR)形成异源二聚体(Yao et al, 1994; Verhaegen et al, 2011)。这些二聚体与20E结合后形成的受体复合物直接激活E75等蜕皮激素早期应答因子,形成初级应答反应(Riddiford et al, 2003; Thummel et al, 2002)。早期应答因子诱导表达的蜕皮激素次级应答基因级联放大蜕皮激素信号,对蜕皮、变态、生殖等生理过程起调控作用(Dubrovsky et al, 2005)。在昆虫中,E75基因首先在果蝇(Drosophilidae)中分离得到,Crossgrove等(2008)研究表明,E75对果蝇的蜕皮、变态及成虫雌蝇的卵室发育有重要作用。果蝇存在E75A、E75B和E75C三种亚型,E75A突变使幼体发育过程中蜕皮激素水平降低,导致发育迟缓、蜕皮缺陷等现象(Bialecki et al, 2002)。E75B突变对生长发育和繁殖无明显影响,E75C突变导致果蝇在成虫期死亡。在甲壳动物中,E75首先在刀额新对虾(Metapenaeus ensis)中分离鉴定,而后又在中国明对虾(Fenneropenaeus chinensis)、三疣梭子蟹(Portunus trituberculatus)、凡纳滨对虾(Litopenaeus vannamei)、黑背陆地蟹(Gecarcinus lateralis)和大型蚤(Daphnia magna)等物种中被分离鉴定(Chan, 1998; Priya et al, 2010; Qian et al, 2014; Kim et al, 2005; Hannas et al, 2010; Litoff et al, 2014)。对E75在甲壳动物蜕皮激素信号转导中的作用研究较少。
学者们对脊尾白虾(Exopalaemon carinicauda)的蜕皮相关基因进行了研究,梁俊平等(2015)克隆了ECR基因,并分析其在卵巢和胚胎发育过程中的表达情况。柳飞等(2016)克隆了RXR基因,并分析其在温度、盐度胁迫和蜕皮周期中的表达情况。张美等(2016)克隆了MIH基因,并分析其在pH和氨氮胁迫中的表达规律。而关于脊尾白虾的E75基因的研究至今尚未见报道。因此,本研究结合脊尾白虾转录组数据库,运用RT-PCR和RACE技术克隆EcE75基因cDNA全长,通过荧光定量PCR分析了该基因在脊尾白虾的组织表达分布及在不同蜕皮分期EcRXR、EcECR和EcE75的表达规律,以比较生长蜕皮和生殖蜕皮的不同,为了解虾蟹类蜕皮机制提供参考信息。
1 材料与方法 1.1 实验材料脊尾白虾取自山东省日照海辰水产有限公司,均为活力好、大小均匀的健康个体,体长为(5.42± 0.34) cm,体重为(1.58±0.22) g。将实验用虾暂养于200 L的PVC桶中,每桶50尾,暂养海水盐度为31,pH为8.2,水温为24℃,24 h持续通气充氧。每天早晚2次饲喂蛤蜊肉,吸污换水1/3,连续养殖7 d后开始实验。
1.2 实验方法 1.2.1 2种蜕皮的区分及样品处理未性成熟的脊尾白虾处于生长期,该组实验虾的蜕皮为生长蜕皮。性成熟的实验虾分为卵巢发育虾和抱卵虾,其中,卵巢发育且带有腹蓝者的蜕皮为生殖蜕皮。根据中华锯齿米虾(Neocaridina denticulata sinensis)(王战芳, 2014)和凡纳滨对虾(Gao et al, 2015)的方法进行蜕皮分期,分为蜕皮间期(C期)、蜕皮后期(软皮期,AB期)、蜕皮前期(D期,取自特征明显的D2亚期)。实验用虾暂养1周后,挑选活力好、无残缺的个体,用解剖剪剪取尾节末端,置于倒置显微镜,观察分期并拍照。将确定分期的脊尾白虾每尾虾取0.1 ml血与0.1 ml预冷的抗凝剂混合,于4℃下5000 r/min离心10 min,弃上清液后加入1 ml Trizol,然后,再采集肝胰腺、鳃、肌肉、肠、胃、眼柄、大颚器、表皮、外壳、腹神经节、卵巢和心共12种组织。液氮研磨后,称取50 mg样品,置于无RNA酶离心管中,并加入1 ml Trizol,震荡混匀后,置于–80℃冰箱保存。每个平行取3尾,每个蜕皮分期共3个平行。
1.2.2 总RNA提取和cDNA合成采用Trizol试剂法提取各组织的总RNA,核酸定量仪(NanoDrop 2000, Thermo)测定纯度,用1.0%琼脂糖凝胶电泳检测浓度及完整性。利用SMARTTM RACE Amplifica- tion Kit (Clontech)合成cDNA模板。
1.2.3 EcE75基因cDNA全长的克隆根据实验室脊尾白虾转录组数据和NCBI上Blast的比对结果,利用Primer Premier 5.0软件设计特异性引物E75-F和E75-R(表 1),以1.2.2中合成的cDNA为模板,使用HiFi酶进行EcE75基因中间片段的扩增。扩增体系:cDNA模板1 μl,HiFi Buffer 2 μl,dNTP (2.5 mmol/L) 1.6 μl,引物(上、下游)各1 μl,HiFi酶(5 U/ml) 0.2 μl,灭菌水补至20 μl。
|
|
表 1 研究所用的引物序列 Table 1 Primers used in this study |
PCR反应程序:95℃ 5 min;94℃ 40 s,58℃ 40 s,72℃ 1 min,35个循环;72℃ 10 min;4℃保温。PCR产物经1.0%的TAE琼脂糖凝胶电泳初步检测后,使用NucleoSpin Gel and PCR Clean-Up Kit (TaKaRa)切胶回收,并纯化检测。切胶回收产物与pEASY-T1载体连接后,将5 μl连接产物转化到50 μl Trans1-T1感受态细胞中。在含AMP、IPTG和X-gal的LB固体培养基中,37℃培养过夜,挑取单一白斑菌落继续培养6 h。菌落PCR鉴定后,将含有目的条带的菌液送往生工生物工程(上海)股份有限公司进行测序。测序结果经NCBI比对,此序列片段与其他物种的E75基因同源性较高,确定为脊尾白虾的E75基因序列片段。
根据上述扩增得到的中间片段利用Primer Premier 5.0软件设计3′和5′RACE引物E75 3′RACE和E75 5′RACE(表 1),用于RACE扩增E75基因cDNA全长序列。使用Smart Race试剂盒(TaKaRa)进行3xRACE和5′RACE PCR扩增,扩增体系为3′/5′RACE cDNA 0.5 μl,10×Advantage 2 PCR Buffer 2.0 μl,dNTP(10 mmol/L) 0.2 μl,3′/5′引物格0.2 μl,50× Advantage 2 Polymerase 0.2 μl,UPM(10×) 1 μl,PCR级H2O 6.9 μl。PCR反应程序为95℃ 3 min;95℃ 30 s,68℃ 30 s,72℃ 3 min,10个循环,每个循环退火温度降0.5℃;95℃ 30 s,63℃ 30 s,72℃ 3 min,20个循环;72℃ 10 min;4℃保存。PCR扩增产物的纯化、克隆、测序与基因中间片段的克隆方法相同。
1.3 EcE75基因的生物信息学分析使用DNAStar软件中的SeqMan程序将测序所得的中间片段和3′、5′末端序列进行拼接,得到EcE75基因全长。利用ExPASy(http://www.expasy.ch/tools/)和Gene Tool软件预测开放阅读框(ORF)和蛋白质序列。使用Predict Protein和SMART服务器进行蛋白质理化性质预测和功能结构域分析。使用TMpred进行跨膜结构域预测。使用ClustalX和DNAman软件对脊尾白虾和其他物种的E75氨基酸序列进行比对分析,并用MEGA 6.07软件以邻接法(Neighbour- Joining,NJ)构建系统进化树,1000次Bootstrap重复检验进化树的置信度。
1.4 EcE75基因组织表达分析根据已获得的基因序列,用Primer Premier 5.0软件设计1对特异引物E75RT-F和E75RT-R,并以脊尾白虾18S rRNA为内参基因设计引物18sF与18sR (表 1)。利用Real-time PCR检测脊尾白虾不同组织E75基因的相对表达量。反应体系和程序参考FS Universal SYBR Green Master(ROX)说明书,采用10 μl反应体系。反应程序:95℃ 10 min;95℃ 10 s,60℃ 34 s,40个循环;95℃ 15 s;60℃ 1 min;95℃ 15 s。实验中每3尾混合作为1个样品,每个样品进行3次重复,数据为3个重复的平均值±标准差,采用2‒∆∆Ct法计算脊尾白虾E75基因在各组织中的相对表达量。
1.5 EcRXR、EcECR和EcE75基因在不同蜕皮分期的组织表达分析根据GenBank的基因序列EcRXR (GenBank No. KU647675),EcECR (GenBank No. KC506600),EcE75 (GenBank No. KY471317),利用Primer Premier 5.0软件分别设计特异引物RXRRT-F、RXRRT-R;EcRRT-F、EcRRT-R;E75RT-F、E75RT-R,并以脊尾白虾18S rRNA为内参基因设计引物18sF与18sR,对生长蜕皮和生殖蜕皮不同分期眼柄中基因的表达进行检测。
1.6 数据处理实验数据采用平均值±标准差(Mean±SD)表示,使用SPSS 19.0软件进行单因素方差分析(One-way ANOVA),Duncan多重比较进行差异显著性检验,P<0.05认为差异显著。
2 结果与分析 2.1 EcE75基因cDNA全长克隆及序列分析通过RACE技术成功获得EcE75基因cDNA序列全长。该基因全长为3944 bp,包括2520 bp的开放阅读框(ORF),450 bp的5′非编码区(5xUTR)和974 bp的3′非编码区(3′UTR)。经PredictProtein软件预测,EcE75基因编码一个由839个氨基酸组成的蛋白质,分子量为92.307 kDa,理论等电点为7.48,脂溶系数为77.07,亲水指数(GRAVY)为–0.417。经SMART服务器预测,EcE75蛋白包含1个C4锌指结构(位于第32~104个氨基酸之间)、1个配体结合结构域(位于第207~366个氨基酸之间)及6个低组分复杂区域(分别位于第396~408个氨基酸、第419~443个氨基酸之间、第499~514个氨基酸之间、第574~627个氨基酸之间、第685~691个氨基酸之间以及第716~736个氨基酸之间),具体位置见图 1。经TMpred软件预测,EcE75基因有1个明显的跨膜螺旋,跨膜方向为由内向外,由第297~313个氨基酸构成,得分为580,预测结果见图 2。

|
图 1 EcE75的cDNA核苷酸序列及其预测的氨基酸序列 Figure 1 Prediction of amino acid sequence based on the cDNA sequence of EcE75 虚线下划线表示C4锌指结构区域,方框表示配体结合结构域,小方框(ATG)表示起始密码子,小方框*表示终止密码子,其他下划线表示6个低组分复杂区域 The dotted line represents the C4 zinc finger structure, the box represents the ligand binding domain, the small box (ATG) represents the start codon, the small box * indicates the stop codon, and the underlines represent the 6 low component complex regions |

|
图 2 EcE75跨膜螺旋预测 Figure 2 The prediction of transmembrane helice of EcE75 |
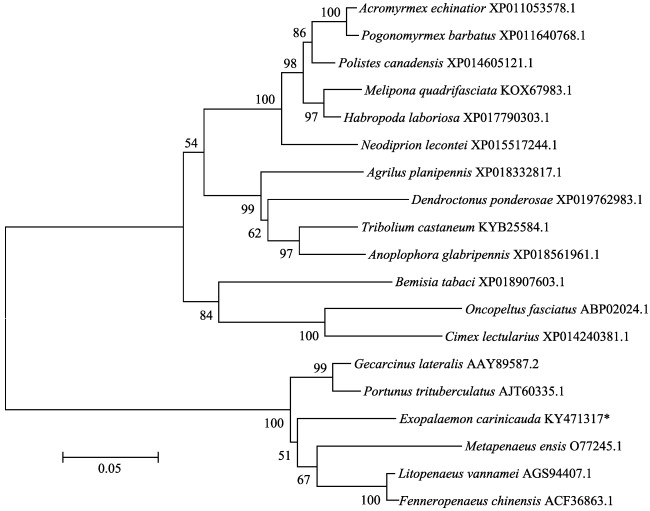
经过多序列比对发现,EcE75蛋白与三疣梭子蟹、凡纳滨对虾、中国明对虾、黑背陆地蟹、刀额新对虾同源性较高,可以证明此序列就是脊尾白虾的E75蛋白(图 3)。系统进化树分析表明,EcE75蛋白在进化上具有一定的保守性,与甲壳动物聚为一支,与三疣梭子蟹、黑背陆地蟹、凡纳滨对虾、中国明对虾等亲缘关系最近,相似性分别为79%、79%、78%和78%(图 4)。

|
图 3 EcE75与其他物种的氨基酸序列比对 Figure 3 Multiple alignment of the deduced amino acid sequences of EcE75 with other species Exopalaemon carinicauda KY471317; Fenneropenaeus chinensis ACF36863.1; Gecarcinus lateralis AAY89587.2; Litopenaeus vannamei AGS94407.1; Metapenaeus ensis O77245.1; Portunus trituberculatus AJT60335.1 |
|
图 4 脊尾白虾与其他物种的E75蛋白系统进化树 Figure 4 NJ phylogenetic tree of amino acid sequences of EcE75 and other species 标尺长度表明每个位点发生0.05次置换 The length of the ruler indicates 0.05 replacements at each site |
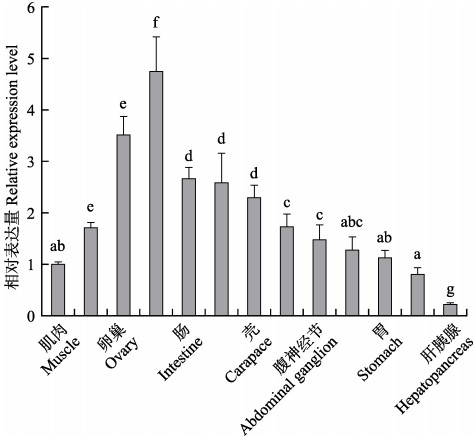
采用RT-PCR方法检测脊尾白虾肝胰腺、鳃、肌肉、肠、血、胃、心、表皮、壳、卵巢、眼柄、大颚器和腹神经节共13种组织中EcE75基因的表达情况。结果显示,EcE75基因在13种组织中均有表达,眼柄表达量最高,卵巢次之,肌肉和血细胞表达量较低,肝胰腺中最低(图 5)。
|
图 5 EcE75基因在不同组织中的表达 Figure 5 Relative expression level of EcE75 in different tissues 不同字母代表组织间差异显著(P<0.05) Different letters mean significant difference among tissues at 0.05 level |
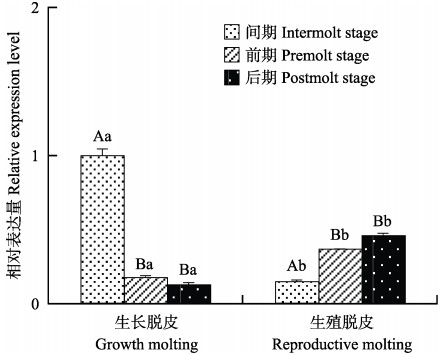
EcRXR基因在不同蜕皮分期的脊尾白虾眼柄中的表达情况如图 6所示。生长蜕皮时,EcRXR的表达量呈逐渐降低的趋势,蜕皮间期显著高于蜕皮前期和后期(P<0.05),蜕皮前期和后期差异不显著;生殖蜕皮时,EcRXR的表达量呈逐渐升高的趋势,蜕皮间期显著低于蜕皮前期和后期(P<0.05),蜕皮前期和后期差异不显著;蜕皮间期时,生长蜕皮显著高于生殖蜕皮(P<0.05);蜕皮前期和后期时,生长蜕皮显著低于生殖蜕皮(P<0.05)。
|
图 6 EcRXR基因在不同蜕皮分期的表达 Figure 6 Relative expression level of EcRXR in different molting stages 同种蜕皮的不同分期用大写字母表示差异显著(P<0.05),不同种蜕皮的同一分期用小写字母表示差异显著(P<0.05)。下同 Capital letters indicate significant difference among different stages of the same type of molting, while lower case letters indicate significant difference between different types of molting in the same stages. The same as below |
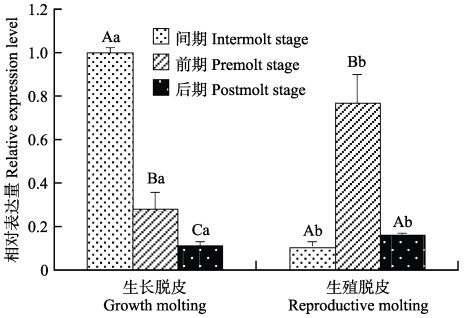
EcECR基因在不同蜕皮分期的脊尾白虾眼柄中的表达情况如图 7所示,生长蜕皮时,EcECR的表达量呈逐渐降低的趋势,蜕皮间期显著高于蜕皮前期(P<0.05),蜕皮前期显著高于蜕皮后期(P<0.05);生殖蜕皮时,蜕皮前期显著高于蜕皮间期和后期(P<0.05),蜕皮间期与后期差异不显著;间期时,生长蜕皮显著高于生殖蜕皮(P<0.05);前期和后期时,生长蜕皮显著低于生殖蜕皮(P<0.05)。
|
图 7 EcECR基因在不同蜕皮分期的表达 Figure 7 Relative expression level of EcECR in different molting stages |
EcE75基因在不同蜕皮分期的脊尾白虾眼柄中的表达情况如图 8所示。EcE75的表达量在生长蜕皮时,蜕皮后期显著低于蜕皮间期和前期(P<0.05),蜕皮间期和前期差异不显著;生殖蜕皮时,蜕皮前期显著高于蜕皮间期和后期(P<0.05),蜕皮间期与后期差异不显著;蜕皮间期和后期时,生长蜕皮显著高于生殖蜕皮(P<0.05),前期时,生长蜕皮显著低于生殖蜕皮(P<0.05)。

|
图 8 EcE75基因在不同蜕皮分期的表达 Figure 8 Relative expression level of EcE75 in different molting stages |
本研究首次克隆得到脊尾白虾的E75基因,该基因全长为3944 bp,包括2520 bp的开放阅读框(ORF),该开放阅读框编码839个氨基酸。EcE75基因编码的蛋白包含1个C4锌指结构、1个配体结合结构域及6个低组分复杂区域。氨基酸序列比对结果显示,克隆得到的EcE75基因具有E75基因典型的结构域。系统进化树分析表明,脊尾白虾E75在进化上具有一定的保守性,与甲壳动物聚为一支,与三疣梭子蟹、黑背地蟹、凡纳滨对虾、中国明对虾等亲缘关系最近,表明本研究克隆得到的E75基因确定为脊尾白虾E75基因。EcE75基因在脊尾白虾的13种组织中均有表达,其中,眼柄的表达量最高,卵巢次之。不同物种的组织表达量有差异,如凡纳滨对虾肌肉中表达量最高,眼柄次之(钱昭英, 2014);三疣梭子蟹眼柄中表达量最高,表皮次之(Xi et al, 2016)。而脊尾白虾E75基因在眼柄和卵巢中的高表达,暗示其可能与蜕皮及卵巢发育相关,需要进一步进行功能验证。
眼柄对于甲壳动物蜕皮调控具有重要作用,且ECR、RXR和E75基因在眼柄中都具有高的表达量,因此,本研究检测了不同蜕皮分期EcRXR、EcECR和EcE75的表达规律。结果显示,EcRXR与EcECR的表达规律基本一致,生殖蜕皮的蜕皮前期和后期表达量都比生长蜕皮高。Lachaise等(1981)和Chan(1995)研究表明,在卵巢的发育过程中,随着卵巢的逐渐成熟,蜕皮激素的浓度随之增加,在产卵前下降,而蜕皮激素的活性形式20E需要结合ECR和RXR形成的二聚体,从而导致ECR和RXR表达量升高。EcRXR在生殖蜕皮的蜕皮后期表达量高于蜕皮前期,这与Durica等(1996)的研究结果相似,推测可能与卵巢发育有关。EcE75基因在生殖蜕皮的表达规律与EcECR一致,而生长蜕皮前期,表达量与蜕皮间期表达量差异不显著,与EcECR和EcRXR的表达规律不一致,原因有待进一步研究。生长蜕皮与生殖蜕皮的主要区别在于生殖蜕皮伴随卵巢发育,而本研究中,相较于生长蜕皮,EcECR、EcRXR和EcE75在生殖蜕皮的高表达,说明其可能与脊尾白虾的卵巢发育有关。通过对脊尾白虾E75、ECR和RXR基因在不同蜕皮分期的表达研究,为虾蟹类蜕皮机制及苗种育成提供了参考信息。
Bialecki M, Shilton A, Fichtenberg C, et al. Loss of the ecdysteroid-inducible E75A orphan nuclear receptor uncouples molting from metamorphosis in Drosophila. Developmental Cell, 2002, 3(2): 209-220 DOI:10.1016/S1534-5807(02)00204-6 |
Chan SM. Cloning of a shrimp (Metapenaeus ensis) cDNA encoding a nuclear receptor superfamily member:An insect homologue of E75 gene. FEBS Letters, 1998, 436(3): 395-400 DOI:10.1016/S0014-5793(98)01148-X |
Chan SM. Possible roles of 20-hydroxyecdysone in the control of ovary maturation in the white shrimp Penaeus vannamei, (Crustacea:Decapoda). Comparative Biochemistry and Physiology Part C:Pharmacology, Toxicology and Endocrinology, 1995, 112(1): 51-59 DOI:10.1016/0742-8413(95)00073-9 |
Crossgrove K, Maina CV, Robinsonrechavi M, et al. Orthologues of the Drosophila melanogaster E75 molting control gene in the filarial parasites Brugia malayi and Dirofilaria immitis. Molecular and Biochemical Parasitology, 2008, 157(1): 92-97 DOI:10.1016/j.molbiopara.2007.08.010 |
Dubrovsky EB. Hormonal cross talk in insect development. Trends in Endocrinology and Metabolism, 2005, 16(1): 6-11 DOI:10.1016/j.tem.2004.11.003 |
Durica DS, Hopkins PM. Expression of the genes encoding the ecdysteroid and retinoid receptors in regenerating limb tissues from the fiddler crab, Uca pugilator. Gene, 1996, 171(2): 237-241 DOI:10.1016/0378-1119(96)00047-9 |
Gao Y, Zhang X, Wei J, et al. Whole transcriptome analysis provides insights into molecular mechanisms for molting in Litopenaeus vannamei. PLoS One, 2015, 10(12): e0144350 DOI:10.1371/journal.pone.0144350 |
Hannas BR, LeBlanc GA. Expression and ecdysteroid responsiveness of the nuclear receptors HR3 and E75 in the crustacean Daphnia magna. Molecular and Cellular Endocrinology, 2010, 315(2): 208-218 |
Kim HW, Lee SG, Mykles DL. Ecdysteroid-responsive genes, RXR and E75, in the tropical land crab, Gecarcinus lateralis:Differential tissue expression of multiple RXR isoforms generated at three alternative splicing sites in the hinge and ligand-binding domains. Molecular and Cellular Endocrinology, 2005, 242(1-2): 80-95 DOI:10.1016/j.mce.2005.08.001 |
Lachaise F, Goudeau M, Hetru C, et al. Ecdysteroids and ovarian development in the shore crab, Carcinus maenas. Hoppe-Seyler's Zeitschrift fur Physiologische Chemie, 1981, 362(5): 521-529 |
Liang JP, Wang Y, Duan YF, et al. Molecular cloning of ecdysteroid receptor and its expression during the ovarian development and embryogenesis of Exopalaemon carinicauda. Journal of Fisheries of China, 2015, 39(7): 942-952 [ 梁俊平, 王芸, 段亚飞, 等. 脊尾白虾EcR基因的克隆及其在卵巢和胚胎发育过程中的表达分析. 水产学报, 2015, 39(7): 942-952] |
Litoff EJ, Garriott TE, Ginjupalli GK, et al. Annotation of the Daphnia magna nuclear receptors:Comparison to Daphnia pulex. Gene, 2014, 552(1): 116-125 DOI:10.1016/j.gene.2014.09.024 |
Liu F, Li J, Li JT, et al. Cloning and expression of retionid X receptor under temperature and salinity stresses and molting cycles in Exopalaemon carinicauda. Oceanologia et Limnologia Sinica, 2016, 47(4): 828-837 [ 柳飞, 李健, 李吉涛, 等. 脊尾白虾(Exopalaemon carinicauda)维甲酸X受体基因克隆及其在温盐胁迫和蜕皮周期中的表达分析. 海洋与湖沼, 2016, 47(4): 828-837 DOI:10.11693/hyhz20160300065] |
Priya TAJ, Li FH, Zhang JQ, et al. Molecular characterization of an ecdysone inducible gene E75 of Chinese shrimp Fenneropenaeus chinensis and elucidation of its role in molting by RNA interference. Comparative Biochemistry and Physiology Part B Biochemistry and Molecular Biology, 2010, 156(3): 149-157 DOI:10.1016/j.cbpb.2010.02.004 |
Qian Z, He S, Liu T, et al. Identification of ecdysteroid signaling late-response genes from different tissues of the Pacific white shrimp, Litopenaeus vannamei. Comparative Biochemistry and Physiology Part A Molecular and Integrative Physiology, 2014, 172(6): 10-30 |
Qian ZY. Isolation, identification, expression and function studies of growth performance candidate genes in Litopenaeus vannamei. Doctoral Dissertation of North West Agriculture and Forestry University, 2014[钱昭英. 凡纳滨对虾生长性能相关候选基因的分离鉴定、表达规律及调控功能研究. 西北农林科技大学博士研究生学位论文, 2014]
|
Riddiford LM, Hiruma K, Zhou X, et al. Insights into the molecular basis of the hormonal control of molting and metamorphosis from Manduca sexta, and Drosophila melanogaster. Insect Biochemistry and Molecular Biology, 2003, 33(12): 1327-1338 DOI:10.1016/j.ibmb.2003.06.001 |
Thummel CS, Chory J. Steroid signaling in plants and insects-common themes, different pathways. Genes and Development, 2002, 16(24): 3113 DOI:10.1101/gad.1042102 |
Verhaegen Y, Parmentier K, Swevers L, et al. The heterodimeric ecdysteroid receptor complex in the brown shrimp Crangon crangon:EcR and RXR isoform characteristics and sensitivity towards the marine pollutant tributyltin. General and Comparative Endocrinology, 2011, 172(1): 158-169 DOI:10.1016/j.ygcen.2011.02.019 |
Wang ZF. Study on the characteristics of reproduction and molting in Neocaridina denticulata sinensis. Masterxs Thesis of Hebei University, 2014, 22-27[王战芳. 中华锯齿米虾繁殖及蜕皮特征的研究. 河北大学硕士研究生学位论文, 2014, 22-27]
|
Xi X, Zhou Y, Liu M, et al. The nuclear receptor E75 from the swimming crab, Portunus trituberculatus:cDNA cloning, transcriptional analysis, and putative roles on expression of ecdysteroid-related genes. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 2016, 200: 69-77 DOI:10.1016/j.cbpb.2016.06.004 |
Yao TP, Forman BM, Jiang Z, et al. Functional ecdysone receptor is the product of EcR and ultraspiracle genes. Nature, 1994, 366(6454): 476-479 |
Zhang M, Liu P, Li JT, et al. Cloning and expression of molt-inhibiting hormone gene from Exopalaemon carinicauda under environmental stresses. Oceanologia et Limnologia Sinica, 2015, 46(4): 764-775 [ 张美, 刘萍, 李吉涛, 等. 脊尾白虾蜕皮抑制激素基因全长cDNA的克隆及其对环境胁迫的响应. 海洋与湖沼, 2015, 46(4): 764-775] |



