瘤背石磺(Onchidium struma)是生活在潮间带高潮区滩涂的一种肺螺类,隶属于软体动物门(Mollusca)、腹足纲(Gastropoda)、肺螺亚纲(Pulmonata)、石磺科(Onchidioidea),是一种具有重要营养价值和药用价值的两栖性贝类,广泛分布于赤道两侧的热带和亚热带海区(吴旭峰等, 2010),国内主要分布在江苏以南沿海地区。石磺科贝类呈现从海洋向陆地扩展的梯度分布状态(Dayrat, 2009),深入研究其辐射过程会更好地了解海洋无脊椎动物由海栖向陆栖的宏观进化模式(Bouchet et al, 2005)。近年来,其线粒体基因组(Sun et al, 2014; 王冬凤等, 2015)、群体多样性分析(姚理想等, 2015)以及分子标记开发(王成暖等, 2014)的研究较多,但与性状相关功能基因的研究报道较少。石磺的真皮含有大量纵横排列的肌纤维,能显著增强皮肤的弹性和韧性(Liu et al, 2014)。作为两栖性最为明显(不能长时间淹没在水中,也不能长期在干燥环境中生活)的瘤背石磺,腹足中含有的大量肌纤维,在其及时快速躲避海水淹没等运动过程中起着非常重要的作用。
肌球蛋白(Myosin)是骨骼肌纤维粗肌丝的基本组成单位,约占肌原纤维蛋白的二分之一以上,具有结构蛋白和功能蛋白的双重性质,是动物体内重要的功能性马达蛋白(Knight et al, 2000),在机体的信号传导、肌肉收缩、细胞器运动等方面起着重要的调控作用(Hooper et al, 2005)。肌球蛋白分子为“Y”形不对称六聚体结构,由2条重链亚基和4条轻链亚基组成,重链亚基由位于氨基端球状结构的肌球蛋白头部和位于羧基端具有α-螺旋结构的轻酶解肌球蛋白构成,分子量约为200 kDa,肌球蛋白头部含有1个ATP和肌动蛋白(actin)结合位点(Craig et al, 2006),轻链亚基具有使肌球蛋白在生理条件下形成丝状的能力(Pett et al, 2000),分子量约为16~20 kDa。单个肌球蛋白分子没有功能,不能参与细胞生理过程,必须聚合成排列有序的粗肌纤维才能完成一个完整的生命活动。
近年来,随着生命科学技术的发展,生物学界对各类物种的相关结构基因,特别是肌球蛋白重链(MyHC)基因的研究也越来越多。目前,在对肌球蛋白基因的研究中,大多数的研究对象是脊椎动物,而有关无脊椎动物的研究报道甚少。目前,已经对多个物种的肌球蛋白重链基因进行了克隆及表达,包括人(Homo sapiens) (Jaenicke et al, 1990)、大鼠(Rattus norvegicus) (Strehler et al, 1986)、鸡(Gallus gallus) (Molina et al, 1987)、鹅(Anser anser) (李艳艳等, 2015)以及鱼类中的大麻哈鱼(Oncorhynchus ketaf) (Iwami et al, 2002)、鲤鱼(Cyprinus carpio) (Muramatsu et al, 2005)、翘嘴鳜(Siniperca chuatsi) (陈之航等, 2017)等,与这些物种MyHC基因相对应的核酸分子杂交探针也陆续制备出来,已达到对MyHC进一步深入细致研究的效果(Lied et al, 1985)。
本研究是在实验室团队对石磺肌肉理化性质研究的基础上,选择在我国分布广泛、产量大、两栖性最为明显的瘤背石磺作为实验对象,以石磺科贝类转录组数据库为基础,差异分析后筛选、克隆了瘤背石磺MyHC基因的cDNA全长,对其序列进行同源性分析,并测定分析其在不同组织中的表达状态,为研制MyHC基因核酸杂交探针提供了基础资料,也为进一步分析石磺科贝类的肌肉生长规律、探究无脊椎动物从海生到陆生的适应性进化过程及分子机制作有益尝试。
1 材料与方法 1.1 材料本实验所用瘤背石磺采自上海市崇明岛,采回样品放置于北向实验室暂养箱中进行暂养,定时向暂养箱中投放玉米粉,清理排泄物,增添清水,并及时清除死亡个体。实验进行前体重为(10.68±0.11) g,肌肉舒展状态下体长为(5.82±0.12) cm。
1.2 方法 1.2.1 RNA提取和cDNA合成取瘤背石磺的背部皮肤、腹部皮肤、口球、腹足、肝胰腺、蛋白腺、两性腺、肺囊部位,采用Trizol(TaKaRa, 大连)试剂提取总RNA,提取方法参照使用说明书,用NanoDrop 2000 (Thermo Scientific, 美国)检测提取的RNA浓度,并用1%琼脂糖凝胶电泳检测RNA的完整性。以瘤背石磺腹足总RNA为模板,用SMARTer™ RACE cDNA Amplification Kit(Clontech, 日本)按照操作指南合成RACE cDNA第一条链。
1.2.2 MyHC基因克隆根据已有的瘤背石磺转录组数据,通过基因差异表达分析、GO功能显著性富集分析和KEGG pathway显著性分析,从转录组数据库中筛选出一条与肌肉生长相关的关键序列(S_Unigene106_c0_seq1),在NCBI进行BLAST比对,结果为MyHC基因。利用Primer Premier5.0软件设计引物MyHC-F1、MyHC-R1(表 1),扩增目的基因片段,25 μl体系:10×PCR Buffer 2.5 μl,dNTP Mixture (各2.5 mmol/L) 0.5 μl,上下游引物(10 μmol/L)各1 μl,Taq (5 U/μl) 0.3 μl,ddH2O 18.7 μl,cDNA 1 μl。PCR反应程序:94℃预变性4 min;94℃ 30 s,55℃ 30 s,72℃ 30 s,35个循环;72℃延伸10 min。扩增产物经1.5%琼脂糖凝胶电泳检测后,用胶回收试剂盒(天根, 北京)割胶回收目的条带,并与pGEM-T Easy (Promega, 美国)载体连接构建重组质粒,热转化感受态DH5α大肠杆菌后涂布于含氨苄的选择性培养基,37℃过夜培养,经蓝白斑筛选和菌液PCR检测后将阳性克隆送生工生物工程(上海)股份有限公司测序。
|
|
表 1 本研究用到的引物 Table 1 Primers used in this study |
根据获得的MyHC基因序列,分别设计3′和5′RACE特异性引物(表 1),按照Clontech RACE说明书分别进行3′ RACE和5′ RACE扩增,PCR反应产物经电泳、胶回收、连接和转化后,通过菌落PCR,挑选阳性克隆样品送生工生物工程(上海)股份有限公司测序,将所获序列和已知序列进行拼接,获得MyHC基因的全长,并通过重测序消除不确定碱基。
1.2.3 序列分析利用在线工具ProtParam、NetPhos 2.0 Server、ORF Finder、Signal 4.1等分别对蛋白质理化性质进行预测分析;用Phyre2程序预测蛋白质的二级结构和三级结构;以日本三角涡虫(Dugesia japonica)为外群采用MrBayes-3.2.6和MEGA 6.0软件用邻位法(Neighbor-Joining)构建系统进化树,1000次bootstraps,其他参数均使用默认值。
1.2.4 实时荧光定量PCR实验前随机挑选9个目测健康、大小均一的瘤背石磺,分别取其背部皮肤、腹部皮肤、口球、腹足、肝胰腺、蛋白腺、两性腺和肺囊8个组织,将每3个个体的相同器官或组织等量混合作为一个样品,共制成3个样品组,提取总RNA并将其反转为cDNA,–20℃保存备用。
根据已获得的MyHC基因序列,设计荧光定量引物MyHC-RT-F和MyHC-RT-R(表 1),以18S rRNA为内参。实时荧光定量PCR采用SYBR premix Ex TaqTM Ⅱ试剂盒(TaKaRa, 大连)在CFX-96(Bio-Rad, 美国)实时荧光定量PCR仪上进行。采用相对定量2-ΔΔCt法(Livak et al, 2001)计算目的基因的相对表达量。基因的表达量用平均值±标准误(Mean±SE)表示,使用SPSS 19.0进行单因素方差分析(One-way ANOVA),P<0.05表示有显著性差异。
2 结果与分析 2.1 MyHC cDNA的全长序列瘤背石磺MyHC的cDNA(GenBank登录号:KU550708)全长为7566 bp,包括5895 bp的开放阅读框,228 bp的5′端非翻译区(UTR),1443 bp的3′端非翻译区(UTR)。3′UTR区含有典型的多聚核苷酸加尾信号序列AATAAA和Poly(A)尾巴(图 1)。NCBI进行Blast比对,显示该序列与光滑双脐螺(Biomphalaria glabrata)相似性为92%,与加州海兔(Aplysia californica)相似性为89%,与二者的相似性均较高。

|
图 1 瘤背石磺MyHC cDNA序列和推测的氨基酸序列 Figure 1 Nucleotide and deduced amino acid sequences of O. struma MyHC 基因序列编码区用大写字母表示,非编码区用小写字母表示。下划线表示Poly(A)尾巴,双下划线表示Myosin_tail _l结构域,阴影部分为AATAAA加尾信号 The UTR and ORF regions are shown in lowercase and uppercase letters, respectively; underline is the polyadenylation signal; Myosin_tail _l domain is double underlined; the shadow is the putative eukaryotic polyadenylation signal (AATAAA) |
推导该序列共编码1964个氨基酸,分子量约为225.28 kDa,理论等电点为5.56。谷氨酸(Glu)、亮氨酸(Leu)和赖氨酸(Lys)含量最高,分别为13.2%、10.5%和10.1%;色氨酸(Trp)、半胱氨酸(Cys)和组氨酸(His)含量最少,分别为0.5%、0.6%和1.4%(表 2)。带负电荷氨基酸残基(Asp+Glu)366个,带正电荷氨基酸残基(Arg+Lys)324个。原子总量31713,分子式为C9765H15897N2849O3150S52。不稳定指数为47.48。
|
|
表 2 瘤背石磺MyHC多肽链的氨基酸组分 Table 2 Amino acid composition of MyHC from O. struma |
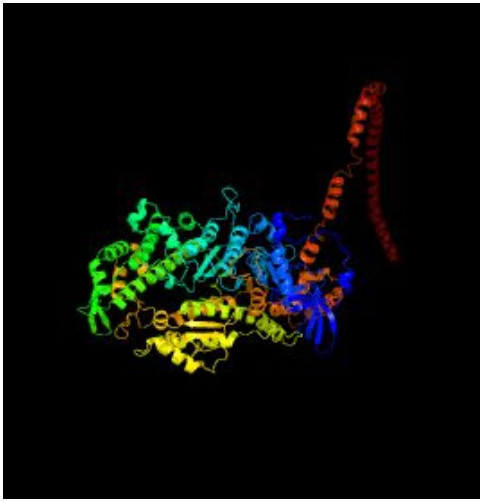
信号肽的预测结果显示,信号肽切割位点存在于第29和30个氨基酸残基之间,表明N-端具有一个由29个氨基酸组成的信号肽。磷酸化位点预测结果显示有74个Ser、41个Thr、9个Tyr,可能是磷酸化位点,糖基化位点预测结果显示有4个N糖基化位点。利用Blastp将蛋白序列与GenBank数据库所收录的全部蛋白序列进行比对,结果显示,瘤背石磺肌球蛋白重链按其结构有4个ATP结合位点,1个P-loop、1个SH1 helix、1个switch Ⅱ region、1个switch Ⅰ region、2个converter subdomain、1个purine-binding loop、1个relay loop,同时包括2个保守结构域MYSc_class_Ⅱ和Myosin_tail_l结构域,MyHC Myosin_tail_l结构域与其他物种MyHC序列进行比对见图 2,肌球蛋白重链三级预测结构如图 3。

|
图 2 瘤背石磺MyHC和其他物种MyHC Myosin_tail _l结构域氨基酸序列的多重比对 Figure 2 Alignment of the deduced amino acid sequences of O.struma and other reported MyHC Myosin_tail _l domain sequences “*”表示保守的氨基酸残基,“.”表示相似性氨基酸 "*" indicates highly conserved amino acid residue; "." represents similar amino acid |
|
图 3 预测的瘤背石磺MyHC三级结构 Figure 3 The predicted tertiary structures of O.struma MyHC |
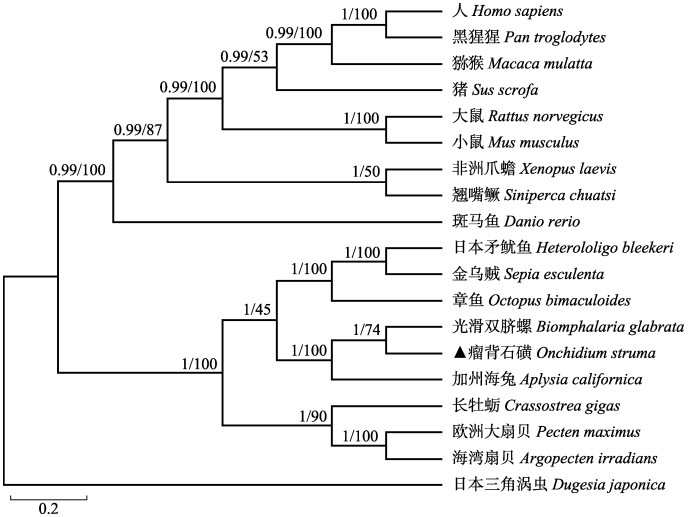
为分析瘤背石磺MyHC的进化地位,根据MyHC氨基酸序列,使用MrBayes-3.2.6和MEGA6.0(前者为MrBayes分析结果,后者为MEGA分析结果)构建了包括瘤背石磺在内的19个物种MyHC序列(表 3)的系统进化树。系统进化树结果显示,无脊椎动物和脊椎动物分为两支,瘤背石磺和光滑双脐螺的MyHC聚类成一个分支,然后与加州海兔聚在一起,海湾扇贝(Argopecten irradians)、欧洲大扇贝(Pecten maximus)和长牡蛎(Crassostrea gigas)聚在一起,日本矛鱿鱼(Heterololigo bleekeri)、金乌贼(Sepia esculenta)和章鱼(Octopus bimaculoides)聚在一起,脊椎动物方面按照形态学分类地位聚类(图 4)。
|
|
表 3 其他物种MyHC的NCBI登录号 Table 3 Accession numbers of MyHC from other species |
|
图 4 基于MyHC氨基酸序列构建的系统树 Figure 4 Phylogenetic analysis of O.struma MyHC and other reported MyHC sequences |
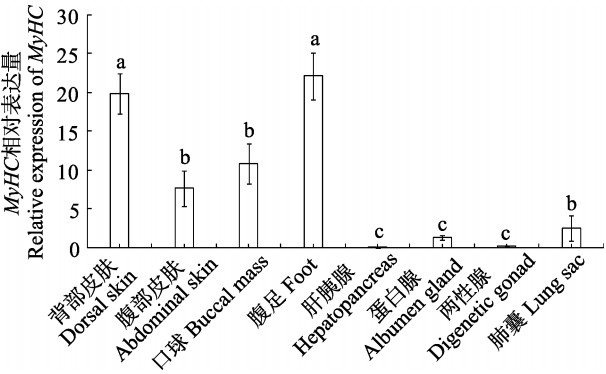
以18S rRNA为内参基因,荧光定量PCR检测瘤背石磺MyHC基因在背部皮肤、腹部皮肤、口球、腹足、肺囊、肝胰腺、蛋白腺和两性腺8个组织中的表达,结果显示,MyHC基因在各个组织中均有表达;腹足和背部皮肤表达量最高,腹部皮肤、口球和肺囊中高表达,肝胰腺、蛋白腺和两性腺中微量表达且表达差异显著(P<0.05) (图 5)。
|
图 5 MyHC在瘤背石磺不同组织的表达 Figure 5 qRT-PCR analysis of the expression profiles of MyHC in different tissues of O.struma 相同字母表示差异不显著(P>0.05),不同字母表示差异显著(P<0.05) Same letters above the bars indicate no significant difference (P>0.05), statistical significance is presented by different letters (P<0.05) |
系统进化树显示,瘤背石磺的MyHC与光滑双脐螺的MyHC聚成一个分支,其次是加州海兔,这与它们的形态学分类结果一致,而且从系统进化树也能看出各物种从低等到高等、从水生到陆生的进化规律,表明肌球蛋白重链是研究生物进化良好的备选基因。
同时有研究显示,脊椎动物中大部分都含有多种肌球蛋白重链基因和亚型,在禽类中至少有9种MyHC基因被鉴定(Kropp et al, 1987; Bandman et al, 2000),在哺乳动物中至少也有13种,其中,8种来自于横纹肌,5种来自于光滑肌和非肌肉组织(Weiss et al, 1999; Flück et al, 2003)。在鱼类肌球蛋白重链多基因家族研究中,分别从虹鳟(Oncorhynchus mykiss)的红肌、白肌和胸肌中获得3种不同的MyHC cDNA片断(Weaver et al, 2001);从翘嘴鳜中克隆得到红肌肌球蛋白重链cDNA片段(易潭等, 2014)。Nakaya等(1995)发现肌球蛋白分子α-螺旋结构会因基因亚型的差异出现显著的不同,Imai等(1997)和Huriaux等(1991)发现了在不同温度下的不同类型的MyHC cDNA。斑马鱼(Danio rerio)的骨骼肌纤维也存在3种不同的类型,应存在不同的MyHC基因的亚型,而不同的MyHC基因的亚型在不同的肌纤维中的表达也存在较大的差异。瘤背石磺中是否存在MyHC基因亚型,以及存在哪几种MyHC基因亚型还有待进一步研究判断。
3.2 瘤背石磺MyHC基因蛋白结构分析本研究克隆获得瘤背石磺MyHC具有典型的保守结构组件,4个ATP结合位点,1个SH1螺旋,3个Loop环,2个转换区域,2个转换子区域,这些氨基酸残基组成了瘤背石磺MyHC两个保守结构域MYSc_class_Ⅱ和Myosin_tail_l的特性。其中,MYSc_ class_Ⅱ位于第33~256位氨基酸,Myosin_tail _l位于第881~1925位氨基酸,位于蛋白尾部,由卷曲的肌球蛋白重链尾部区域构成,为粗肌丝提供了结构支撑。已知MyHC家族蛋白的典型结构主要是由N端球状盘绕的头部和C端α-螺旋型的尾部组成,头部有ATP结合位点、Loop1和Loop2环区,是MyHC家族中的保守区,同时也是肌球蛋白动力产生的主要区域,Loop1表面环状结构决定肌纤维收缩速率以及持续的动力,而Loop2表面环状结构则影响MyHC上游肌动蛋白激活的ATP酶水解活性(Holmes et al, 2002)。另外,在MyHC中还发现了SH1螺旋。SH1修饰肌球蛋白亚片段能大大提高其热稳定性,SH1可以大大减少整个肌球蛋白亚片段的改变(Lubov et al, 2007),其对肌纤维运动等功能有待进一步研究验证。本研究获得的瘤背石磺MyHC的蛋白理化性质的预测结果发现,瘤背石磺MyHC蛋白MYSc_class_Ⅱ结构域疏水性氨基酸所占的比例较大。目前,刘希良等(2012)根据已经克隆的鳜鱼MyHC序列制备出原核表达载体和多克隆抗体。瘤背石磺肌球蛋白重链Myosin_tail _l保守区域中亲水性氨基酸较多,属于亲水性氨基酸密集区域,该实验结果对深入研究石磺肌球蛋白重链基因的原位杂交实验、Western blot检测奠定了基础。
在对目前哺乳动物骨骼肌中MyHC基因进行研究时,已经发现有多种途径可以调控该基因的同型异构体表达,其中有两条途径研究较多,分别是通过肌源调节因子(Myogenic regulatory factor, MRF)和活化T细胞核因子(Nuclear factor of activated T cells, NFAT)(Devoto et al, 1996)。前者由MyoD、myogenin、myfS和MRF4这4个蛋白组成,后者调控途径的主要方式是通过调节Ca2+的浓度的变化。研究表明,当肌肉收缩时,释放Ca2+数量增多,相应的细胞浆内Ca2+浓度就会上升,而升高的Ca2+浓度会通过一系列的化学反应来激活下游相应的蛋白,由此使得细胞质中某些蛋白质进行脱磷酸化,随后进入细胞核中,与一个保守的DNA结合位点相结合,以此来调节相应转录水平的变化(Tao et al, 2004)。Kubis等(1997)在对兔培养的肌细胞进行研究时,将Ca2+载体加入培养基中,使肌浆中Ca2+浓度缓慢升高,发现此时肌细胞中慢型肌球蛋白重链和轻链的比例较高。在对石磺MyHC基因构成的蛋白质进行结构分析时,发现钙调素结合区段以及许多磷酸化位点,例如蛋白激酶C的活性必须依赖Ca2+的存在。虽然现在没有确切的证据表明石磺内有相似的调控机制来控制MyHC基因的特异性表达,但可以为今后探究石磺的肌球蛋白的调控机制提供基础依据。
3.3 MyHC基因的表达与环境适应性分析研究证实,MyHC基因的表达量会显示出对于周围环境变化的适应和调节,Altringham等(1985)发现鲤鱼会通过MyHC基因的表达量的不同来调节ATP酶的活性以适应温度的变化。Nihei等(2006)通过对鲤鱼胚胎和肌肉组织中的MyHC基因表达量的研究发现,鲤鱼MyHC基因不仅在不同发育阶段、不同肌肉类型表现出表达量的差异,而且在不同温度下也会存在这种差异。此外,在其他鱼类品种中,Weaver等(2001)利用RT-PCR检测幼鱼阶段虹鳟的平滑肌、骨胳肌和心肌中MyHC基因表达,显示平滑肌表达MyHC基因的表达略高于骨骼肌和心肌,在进一步对鱼苗期和幼鱼期的平滑肌中MyHC基因表达量进行比较时发现,幼鱼期MyHC基因表达量高于鱼苗期。本研究中,MyHC在腹足部位高表达,同时在其他各个组织中都进行表达,充分证明肌球蛋白既是一种不可缺少的结构蛋白,同时又能作为功能蛋白发挥作用;腹足作为瘤背石磺的运动器官,MyHC在该器官中表达量最高,增强了瘤背石磺的运动能力,为瘤背石磺逐渐适应陆生环境提供了有力的条件。
4 结论本研究从瘤背石磺转录组差异表达基因中筛选到MyHC Unigenes,进行了cDNA序列克隆、结构分析、进化分析以及qRT-PCR表达水平分析。结果表明,瘤背石磺MyHC基因的结构特征主要有:存在多个N-糖基化位点,MYSc_class_Ⅱ和Myosin_tail_l两个保守结构域,N末端具有多个ATP结合位点等特异性功能区域。系统进化树显示,瘤背石磺MyHC基因与光滑双脐螺聚成一支,其符合形态学分类且物种等级相近;表达水平分析显示,瘤背石磺MyHC基因具有不同的组织分布,在组织运动最发达的腹足有高水平的表达。通过对瘤背石磺MyHC基因结构、功能预测、组织表达以及分子进化的分析,为进一步探究石磺肌球蛋白的调控机制提供基础依据;为探究肌肉运动对瘤背石磺湿地环境两栖适应性的相关联系;对研究瘤背石磺肌球蛋白重链基因进行原核表达及多克隆抗体的制备提供基础资料,更为深入探讨海洋无脊椎动物从海洋向陆地进化的研究提供有参考意义的分子依据。
Altringham JD, Johnston IA. Changes in tension generation and ATPase activity in skinned muscle fibres of the carp following temperature acclimation. Pflugers Archiv-European Journal of Physiology, 1985, 403(4): 449-451 DOI:10.1007/BF00589261 |
Bandman E, Rosser BWC. Evolutionary significance of myosin heavy chain heterogeneity in birds. Microscopy Research and Technique, 2000, 50(6): 473-491 DOI:10.1002/(ISSN)1097-0029 |
Bouchet P, Rocroi JP. Classification and nomenclator of gastropod families. Malacologia:International Journal of Malacology (Hackenheim, Germany:ConchBooks), 2005, 47(1): 1-397 |
Chen ZH, Dong JJ, Sun CF, et al. cDNA cloning and analyses of two myosin heavy chain isoforms of mandarin fish (Siniperca chuatsi) based on transcriptome sequencing. Progress in Fishery Sciences, 2017, 38(3): 51-61 [ 陈之航, 董浚键, 孙成飞, 等. 基于转录组测序对翘嘴鳜(Siniperca chuatsi)2种肌球蛋白重链基因的克隆与分析. 渔业科学进展, 2017, 38(3): 51-61 DOI:10.11758/yykxjz.20160414001] |
Craig R, Woodhead JL. Structure and function of myosin filaments. Current Opinion in Structural Biology, 2006, 16(2): 204-212 DOI:10.1016/j.sbi.2006.03.006 |
Dayrat B. Review of the current knowledge of the systematics of Onchidiidae (Mollusca:Gastropoda:Pulmonata) with a checklist of no minal species. Zootaxa, 2009, 2068(2068): 1-26 |
Devoto SH, Melancon E, Eisen JS, et al. Identification of separate slow and fast muscle precursor cells in vivo, prior to somite formation. Canadian Metallurgical Quarterly, 1996, 122(11): 3371-3380 |
Flück M, Hoppeler H. Molecular basis of skeletal muscle plasticity-from gene to form and function. Reviews of Physiology, Biochemistry and Pharmacology, 2003, 146(1): 159-216 |
Holmes JM, Whiteley NM, Magnay JL, et al. Comparison of the variable loop regions of myosin heavy chain genes from Antarctic and temperate isopods. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 2002, 131(3): 349-359 DOI:10.1016/S1096-4959(01)00509-7 |
Hooper SL, Thuma JB. Invertebrate muscles:Muscle specific genes and proteins. Physiological Reviews, 2005, 85(3): 1001-1060 DOI:10.1152/physrev.00019.2004 |
Huriaux F, Vandcalle P, Focant B. Myosin heavy chain isoforms in white, red and ventricle muscles of barbel (Barbus barbus L). Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 1991, 100(2): 309-312 DOI:10.1016/0305-0491(91)90380-V |
Imai J, Hirayama Y, Kikuchi K, et al. cDNA cloning of myosin heavy chain isoforms from carp fast skeletal muscle and their gene expression associated with temperature acclimation. Journal of Experimental Biology, 1997, 200(1): 27-34 |
Iwami Y, Ojima T, Jnoue A, et al. Primary structure of myosin heavy chain from fast skeletal muscle of Chum salmon Oneorhynehus keta. Comparative Biochemistry & Physiology Part B Biochemistry & Molecular Biology, 2002, 133(2): 257-267 |
Jaenicke T, Diederich KW, Haas W, et al. The complete sequence of the human beta-myosin heavy chain gene and a comparative analysis of its product. Genomics, 1990, 8(2): 194-206 DOI:10.1016/0888-7543(90)90272-V |
Knight AE, Molloy JE. Muscle, myosin and single molecules. Essays in Biochemistry, 2000, 35(35): 43-59 |
Kropp KE, Gulick J, Robbins J. Structural and transcriptional analysis of a chicken myosin heavy chain gene subset. Journal of Biological Chemistry, 1987, 262(34): 16536-16545 |
Kubis HP, Haller EA, Wetzel P, et al. Adult fast myosin pattern and Ca2+ induced slow myosin pattern in primary skeletal muscle culture. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(8): 4205-4210 DOI:10.1073/pnas.94.8.4205 |
Li YY, Shao F, Wu XJ, et al. Molecular cloning, sequence and expression characterization of myosin heavy chain 1 gene (MyHC1) in goose (Anser anser) at embryonic period. Journal of Agricultural Biotechnology, 2015, 23(3): 359-368 [ 李艳艳, 邵芳, 吴小娟, 等. 鹅肌球蛋白重链1基因MyHC1全长cDNA克隆、序列及胚胎期表达特征分析. 农业生物技术学报, 2015, 23(3): 359-368] |
Lied E, von der Decken A. Purification of fish muscle myosin heavy chain and quantification of the specific polyribosome-bound polypeptide. Biochemical Journal, 1985, 232(2): 467-470 DOI:10.1042/bj2320467 |
Liu C, Wei LL, Shen HD, et al. Complete mitochondrial genome of Peronia verruculata (Gastropoda:Pulmonata:Systellom'matophora:Onchidiidae). Mitochondrial DNA, 2014, 27(2): 918-920 |
Liu XL, Wang KZ, Cheng J, et al. Prokaryotic expression and preparation of polyclonal antibody of myosin heavy chain genes in Siniperca chuatsi. Journal of Natural Science of Hunan Normal University, 2012, 35(6): 67-73 [ 刘希良, 王开卓, 成嘉, 等. 鳜鱼肌球蛋白重链基因的原核表达及多克隆抗体的制备. 湖南师范大学自然科学学报, 2012, 35(6): 67-73] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method. Methods, Weaver, 25(4): 402-408 |
Lubov S, Valeria M, Elena S, et al. Nucleotide-induced and actin-induced structural changes in SH1-SH2-modified myosin subfragment 1. Journal of Muscle Research and Cell Motility, 2007, 28(1): 67-78 DOI:10.1007/s10974-007-9108-7 |
Molina MI, Kropp KE, Gulick J, et al. The sequence of an embryonic heavy chain gene and isolation of its corresponding cDNA. Journal of Biological Chemistry, 1987, 262(14): 6478-6488 |
Muramatsu UM, Kikuchi KH, Ikeda D, et al. The complete genomic sequence of the carp fast skeletal myosin heavy chain gene. Gene, 2005, 349(2): 143-151 |
Nakaya M, Watabe S, Ooi T. Differences in the thermal stability of acclimation temperature-associated types of carp myosin and its rod on differential scanning calorimetry. Biochemistry, 1995, 34(9): 3114-3120 DOI:10.1021/bi00009a043 |
Nihei Y, Kobiyama A, Ikeda D, et al. Molecular cloning and mRNA expression analysis of carp embryonic, slow and cardiac myosin heavy chain isoforms. Journal of Experimental Biology, 2006, 209(1): 188-198 DOI:10.1242/jeb.01978 |
Pette D, Staron RS. Myosin isoform, muscle fiber types and transitions. Microscopy Research & Technique, 2000, 50(6): 500-509 |
Strehler EE, Strehler-Page MA, Perriard JC, et al. Complete nucleotide and encoded amino acid sequence of a mammalian myosin heavy chain gene. Evidence against intron-dependent evolution of the rod. Journal of Molecular Biology, 1986, 190(3): 291-317 DOI:10.1016/0022-2836(86)90003-3 |
Sun BN, Chen C, Shen HD, et al. Species diversity of Onchidiidae (Eupulmonata:Heterobranchia) on the mainland of China based on molecular data. Molluscan Research, 2014, 34(1): 62-70 DOI:10.1080/13235818.2013.868860 |
Tao Y, Kobayashi M, Liang CS, et al. Temperature-dependent expression patterns of grass carp fast skeletal myosin heavy chain genes. Comparative Biochemistry & Physiology Part B:Biochemistry and Molecular Biology, 2004, 139(4): 649-656 |
Wang CN, Shen HD, Zheng P. Evaluation of genetic diversity in the Onchidium struma from different geographical populations in China by inter simple sequence repeat (ISSR). Biotechnology Bulletin, 2014(9): 208-212 [ 王成暖, 沈和定, 郑培. 中国沿海瘤背石磺不同地理群体遗传多样性的ISSR标记研究. 生物技术通报, 2014(9): 208-212] |
Wang DF, Shen HD, Wu X. Comparative histology of the skin from three species of Onchidiidae. Chinese Journal of Zoology, 2015, 50(3): 437-444 [ 王冬凤, 沈和定, 吴欣. 石磺科3种贝类皮肤显微结构比较. 动物学杂志, 2015, 50(3): 437-444] |
Weaver FE, Stauffer KA, Coughlin DJ. Myosin heavy chain expression in the red, white, and ventricular muscle of juvenile stages of rainbow trout. Journal of Experimental Zoology, 2001, 290(7): 751-758 DOI:10.1002/(ISSN)1097-010X |
Weiss A, McDonough D, Wertman B, et al. Organization of human and mouse skeletal myosin heavy chain gene clusters is highly conserved. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(6): 2958-2963 DOI:10.1073/pnas.96.6.2958 |
Wu XF, Shen HD, Wu WJ, et al. Comparison on morphology of Onchidiidae in eastern coast of China. Chinese Journal of Zoology, 2010, 45(6): 92-100 [ 吴旭峰, 沈和定, 吴文健, 等. 我国华东沿海4种石磺形态学比较. 动物学杂志, 2010, 45(6): 92-100] |
Yao LX, Zhou N, Shen HD, et al. Genetic diversity and differentiation of Platevindex mortoni from the coastal area of China based on mitochondrial COI gene. Journal of Fishery Sciences of China, 2015, 22(4): 729-739 [ 姚理想, 周娜, 沈和定, 等. 中国沿海平疣桑椹石磺COI基因的遗传多样性与遗传分化. 中国水产科学, 2015, 22(4): 729-739] |
Yi T, Liu XL, Bin SY, et al. Molecular cloning and expression analysis of slow myosin heavy chain 1(sMyHC1) gene cDNA in mandarin fish (Siniperca chuatsi). Journal of Guangxi Normal University, 2014, 32(2): 175-180 [ 易潭, 刘希良, 宾石玉, 等. 鳜鱼Siniperca chuatsi红肌sMyHC1基因cDNA的克隆及其表达分析. 广西师范大学学报, 2014, 32(2): 175-180] |



