2. 农业农村部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
4. 南京农业大学无锡渔业学院 无锡 214081;
5. 上海海洋大学水产与生命学院 上海 201306;
6. 青岛金沙滩水产开发有限公司 青岛 266000
2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071;
4. Wuxi Fisheries College, Nanjing Agricultural University, Wuxi 214081;
5. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306;
6. Qingdao Jinshatan Aquatic Product Development Co., Ltd., Qingdao 266000
DNA甲基化为表观遗传学的重要组成部分,DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达。脊椎动物DNA甲基化最常见的是CpG二核苷酸中胞嘧啶的甲基化修饰,即由S-腺苷甲硫氨酸提供甲基,在DNA甲基转移酶的催化下,将甲基基团以共价键形式结合到基因组CpG岛的二核苷酸胞嘧啶5'碳位上,并可在细胞分裂过程中传递给子细胞的表观遗传现象;植物和真菌的甲基化还会发生在非CpG岛区域,如CG、CHH、CHG位点(H为除G外的其他碱基)(李艳等, 2017; 王玉龙, 2016)。这种在不改变基因序列条件下产生可遗传的基因表达,已成为当前的研究热点。诸多研究表明,DNA甲基化与基因表达调控(De et al, 2015; Rhee et al, 2002)、基因组印迹(Hata et al, 2002)、X染色体失活(Zhang et al, 2010; Mohandas et al, 1981; Bird et al, 2002)、细胞功能丧失(Golbus et al, 1990)、细胞衰老(Berdyshev et al, 1967)、肿瘤发生(魏绍峰等, 2016)等有着密不可分的关系。
近年来,水产动物的DNA甲基化研究也不断深入。左之良等(2016)研究发现,刺参(Apostichopus japonicus)的自然群体与白化群体相同组织间甲基化水平和模式存在差异,同一群体不同组织间的DNA甲基化水平和模式也不同;杜盈等(2013)对中国明对虾(Fenneropenaeus chinensis)的自然群体和人工选育品种“黄海1号”不同组织基因组DNA甲基化的研究表明,DNA甲基化水平和模式因群体和组织的不同而存在差异;李思平等(2015)发现,在面对相同的胁迫环境,雌、雄半滑舌鳎(Cynoglossus semilaevis Günther)的不同组织DNA甲基化水平变化并不完全相同。DNA甲基化具组织差异性已在多个物种中证实,但关于头足类DNA甲基化组织差异性研究鲜见报道。
金乌贼(Sepia esculenta Hoyle, 1885)是我国沿海重要的经济头足类品种,属软体动物门(Mollusca)、头足纲(Cephalopoda)、鞘亚纲(Coleoidea)、乌贼目(Sepiida)、乌贼科(Sepiidae)、乌贼属(Sepia),具有生命周期短、世代更新快、生长迅速、食性广、肉质鲜美、蛋白质含量高、综合利用范围广等优点,曾是我国海洋渔业重要的捕捞对象(董正之等, 1991; 郝振林等, 2007)。但自20世纪80年代以来,由于过度捕捞和海洋环境的破坏等,其资源量明显衰退,产量急剧下降,目前,金乌贼在许多海域已绝迹。近年来,为了恢复和开发利用金乌贼资源,金乌贼资源增殖等研究工作已引起科研工作者的高度重视,陆续从遗传育种学、胚胎发育学、生理学等多方面展开相关研究(刘长琳等, 2009、2016)。为探讨生长期金乌贼不同性别、不同组织DNA甲基化水平和模式的差异,本研究利用荧光标记的甲基化敏感扩增多态性(Fluorescence- labeled Methylation Sensitive Amplified Polymorphism, F-MSAP)技术对生长期金乌贼雌雄个体肌肉、心脏、胰脏和性腺组织基因组DNA甲基化水平和模式进行检测,在分子水平上探讨金乌贼不同组织间DNA甲基化水平差异,认识处于快速生长阶段的金乌贼基因组DNA甲基化水平和模式,分析生长发育、组织分化与DNA甲基化之间的关系,以期为深入研究金乌贼衰老死亡等提供基础数据。
1 材料与方法 1.1 材料2015年9月在山东青岛近海捕捞金乌贼野生幼体,经过1年人工培育,于2016年8月性成熟并自然交配获得受精卵。受精卵经30 d孵化后得到初孵仔鱼,随机采集生长期(约90日龄)健康个体3雌3雄,体重为181.02~231.40 g,胴背长为105.67~124.51 mm。分别保存6个个体的外套膜肌肉、心脏、胰脏和性腺4种组织共24份样品,液氮速冻后,转至-80℃冰箱备用。
1.2 方法 1.2.1 基因组DNA的提取取各组织样品50~100 mg,用液氮研磨至粉末,采用传统的酚-氯仿抽提法提取基因组DNA,1%琼脂糖凝胶电泳检测DNA是否降解,并且使用NanoDrop2000(Thermo)检测DNA的浓度和纯度,选择条带清晰且纯度高的样品稀释至100 ng/μl,-20℃中保存备用。
1.2.2 F-MSAP原理F-MSAP是无参考基因组物种DNA甲基化检测的常用技术。该技术是在MSAP技术(Xiong et al, 1999)上发展起来的,与后者相比,其结合荧光标记引物的可显性和DNA测序仪的自动化,分辨率、准确性、安全性和检测通量均得到提升(杨春等, 2016)。F-MSAP的工作原理:使用1对甲基化敏感的限制性内切酶HpaⅡ和MspⅠ,识别基因组CpG岛的CCGG位点,但2种酶对不同DNA甲基化模式的敏感程度不同,酶切产生的条带不同。HpaⅡ可以对非甲基化和半甲基化序列进行酶切,而对胞嘧啶的全甲基化序列无酶切活性;MspⅠ则可以对非甲基化和内侧胞嘧啶全甲基化序列进行酶切,而对半甲基化和外侧胞嘧啶全甲基化序列无酶切活性。
1.2.3 酶切和连接体系将限制性内切酶HpaⅡ和MspⅠ分别与EcoRⅠ组合成双酶切反应体系:EcoRⅠ+HpaⅡ和EcoRⅠ+MspⅠ。酶切反应体系20 μl,包括2 μl DNA (100 ng/μl),2 U的FastDigest EcoRⅠ和FastDigest HpaⅡ/MspⅠ,4 μl 10×Buffer (Thermo),补ddH2O至20 μl。酶切体系37℃水浴0.5 h。
连接反应:根据Xu等(2000)的方法进行接头引物设计(表 1),将EcoRⅠ对应的E-A1-2单链接头分别稀释为10 pmol/L,HpaⅡ/MspⅠ对应的HM-A1-2单链接头分别稀释为100 pmol/L,各取等体积的单链接头引物混合后,94℃变性5 min,缓慢冷却到室温形成双链接头E和HM。连接反应体系20 μl,包括10 μl酶切产物,50 pmol双链接头E和HM,1U T4 DNA Ligase,2 μl 10×Buffer(TaKaRa),补ddH2O至20 μl。连接体系16℃过夜。连接产物用TE缓冲液(Solarbio)稀释10倍后用于预扩增反应体系。
|
|
表 1 金乌贼F-MSAP甲基化分析所用接头和引物序列 Table 1 Sequence of adapters and primers used for F-MSAP analysis of S. esculenta |
预扩增反应体系20 μl,包括2 μl稀释后连接产物,1 U EasyTaqDNA Polymerase,2 μl 10×Buffer(TransGen),1 μl dNTP (2.5 mmol/L) (TransGen),5 pmol HM0 Primer,5 pmol E0 Primer(表 1),补灭菌ddH2O至20 μl。PCR反应条件:94℃预变性5 min;94℃变性30 s,56℃退火1 min,72℃延伸1 min,25个循环;72℃延伸10 min。预扩增产物用TE溶液稀释20倍后用于选择性扩增反应体系。
1.2.5 选择性扩增反应体系参考太平洋牡蛎(Crassostrea gigas) (姜群, 2015; 张鑫等, 2017)和大珠母贝(Pinctada fucata)(Yao et al, 2015)DNA甲基化检测的选择性扩增引物,筛选出4条HpaⅡ/MspⅠ选择性扩增引物HM(1-4)和7条FAM荧光标记的EcoRⅠ选择性扩增引物E(1-7),从这28对引物组合中筛选出12对扩增条带清晰且效率高的选择性扩增引物(表 1)。选择性扩增反应体系20 μl,包括2 μl稀释后预扩增产物,1 U EasyTaq DNA Polymerase,2 μl 10×buffer,1 μl dNTP(2.5 mmol/L),5 pmol HpaⅡ/MspⅠ选择性扩增引物,5 pmol EcoRⅠ选择性扩增引物,补灭菌ddH2O至20 μl。PCR反应条件:第一阶段为13个Touch down循环,94℃预变性5 min;94℃变性30 s,65℃退火(每个循环递减0.7℃),72℃延伸2 min;72℃延伸10 min。第二阶段为27个普通PCR循环,94℃变性30 s,56℃退火30 s,72℃延伸2 min;最后72℃延伸10 min,4℃保存。
1.2.6 数据分析使用ABI 3130 Genetic Analyzer检测选择性扩增产物的甲基化多态性,获得峰值图,使用Gene Mapper V4.0对所得峰值图进行扫描。同一DNA不同酶切组合的相同引物扩增互为一组,分别记为EH、EM(图 1)。根据上述酶切原则,所得数值可分为3种条带形式,分别代表DNA甲基化的3种模式(图 1):Ⅰ半甲基化位点,甲基化修饰仅在DNA一条链,经EH和EM分别处理后,EH酶切有带而EM无带;Ⅱ全甲基化位点,DNA双链发生甲基化修饰,EH酶切无带而EM有带;Ⅲ非甲基化位点,EH和EM酶切均出现条带。
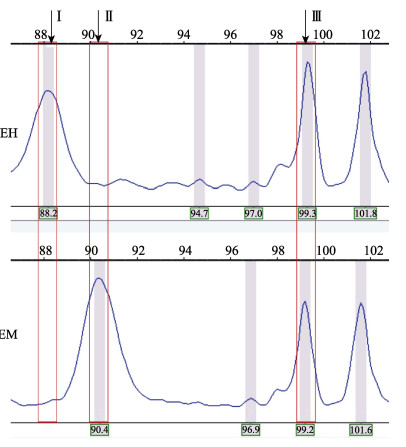
|
图 1 金乌贼的3种DNA甲基化模式 Figure 1 Three DNA methylation types of S. esculenta EH为EcoRⅠ+HpaⅡ酶切;EM为EcoRⅠ+MspⅠ酶切;Ⅰ表示半甲基化位点,Ⅱ表示全甲基化位点,Ⅲ表示非甲基化位点 EH: Digested by EcoRⅠ/HpaⅡ; EM: Digested by EcoRⅠ/MspⅠ; Ⅰ: Hemi-methylated sites; Ⅱ: Full-methylated sites; Ⅲ: Non-methylated sites |
整合统计数据(表 2),采用SPSS 16.0软件单因素方差分析法(One-way ANOVA)对雌雄金乌贼的4种组织基因组DNA总甲基化水平、半甲基化水平和全甲基化水平进行显著性分析,结果以平均值±标准误(Mean±SE)在GraphPad Prism 5中绘制(图 4~图 8)。
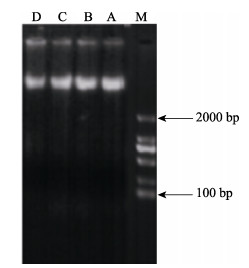
|
图 2 金乌贼基因组DNA电泳 Figure 2 Electropherograms of genomic DNA in S. esculenta A:肌肉; B:心脏; C:胰脏; D:性腺; M: DL2000 DNA marker A: Muscle; B: Heart; C: Pancreas; D: Gonad |

|
图 3 金乌贼基因组DNA酶切反应电泳 Figure 3 Electropherograms of enzyme cleavage in genomic DNA of S. esculenta a, c, e, g分别代表金乌贼肌肉、心脏、胰脏和性腺组织的EcoRⅠ+HpaⅡ酶切条带; b, d, f, h分别代表金乌贼肌肉、心脏、胰脏和性腺组织的EcoRⅠ+MspⅠ酶切条带 m: DL2000 DNA marker; a, c, e, g represent the EcoRⅠ+HpaⅡ digestion of four tissues of S. esculenta; b, d, f, h represent the EcoRⅠ+MspⅠ digestion of four tissues of S. esculenta |
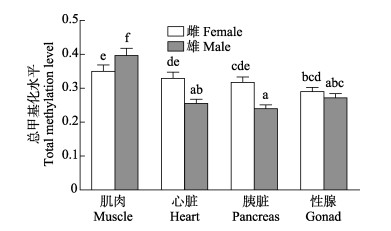
|
图 4 雌雄金乌贼不同组织基因组DNA总甲基化水平比较 Figure 4 Comparison of total methylation level of genomic DNA of female and male S. esculenta 柱上不同字母表示不同组织间差异显著(P < 0.05),下同 Columns with different letters were significantly different (P < 0.05). The same as below |
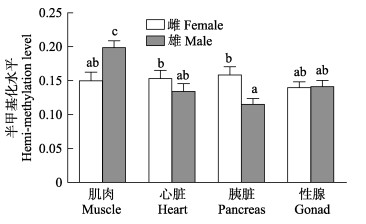
|
图 5 雌雄金乌贼不同组织基因组DNA半甲基化水平比较 Figure 5 Comparison of hemi-methylation level of genomic DNA of female and male S. esculenta |
|
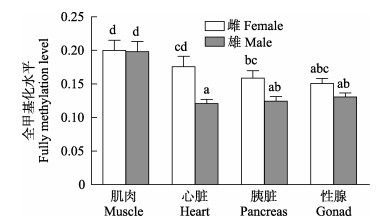
图 6 雌雄金乌贼不同组织基因组DNA全甲基化水平比较 Figure 6 Comparison of fully methylation level of genomic DNA of female and male S. esculenta |
|
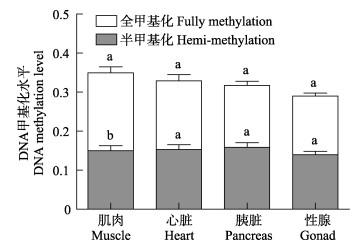
图 7 雌性金乌贼不同组织基因组DNA总甲基化、半甲基化、全甲基化水平对比 Figure 7 Comparison of total/hemi-/fully methylation level of genomic DNA of female S. esculenta |
|
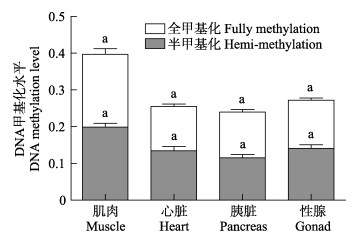
图 8 雄性金乌贼不同组织基因组DNA总甲基化、半甲基化、全甲基化水平对比 Figure 8 Comparison of total/hemi-/fully methylation level of genomic DNA of male S. esculenta |
|
|
表 2 金乌贼雌雄不同组织基因组DNA甲基化的检测结果 Table 2 Methylation detection on various tissues of female and male S. esculenta (%, Mean±SE) |
基因组DNA提取与酶切反应是F-MSAP技术成功的关键,提取的基因组DNA完整无断裂决定检测结果是否可靠;酶切是否完全直接决定能否完全检测DNA胞嘧啶甲基化位点。来自金乌贼肌肉、心脏、胰脏和性腺的4种组织基因组DNA(图 2),DNA带型整齐,泳带无拖尾,说明所提取的金乌贼基因组DNA质量好,可用于后续酶切反应。对4种组织基因组DNA,采用快速酶切体系,经EcoRⅠ+HpaⅡ和EcoRⅠ+MspⅠ两组双酶切反应后,酶切反应产物经琼脂糖凝胶电泳,呈弥散状,无明显条带,表明酶切反应比较充分(图 3)。
2.1 金乌贼甲基化水平和模式的性别差异根据DNA甲基化模式的分类原则(图 1),整合24个样品与12对引物组合的分析结果,统计金乌贼生长期不同组织的基因组DNA甲基化各模式比例(表 2),可见生长期金乌贼的非甲基化水平显著高于总甲基化水平(P < 0.05),表明金乌贼基因组DNA序列的CCGG位点虽发生了甲基化修饰,仍以非甲基化位点居多。
在总甲基化水平上,相同组织间的性别差异表现为雌性金乌贼心脏和胰脏的总甲基化水平显著高于雄性(P < 0.05);但雌性肌肉的总甲基化水平却显著低于雄性(P < 0.05);性腺的总甲基化水平不同性别差异不显著(图 4)。在半甲基化水平上,相同组织间的性别差异与总甲基化水平的趋势基本一致(图 5)。在全甲基化水平上,比较相同组织的性别差异,仅有雌性金乌贼心脏的全甲基化水平显著高于雄性(P < 0.05),其他组织差异均不显著(图 6)。
2.3 金乌贼甲基化水平和甲基化模式的组织差异 2.3.1 雌性金乌贼不同组织间的DNA甲基化比较雌性金乌贼生长期阶段不同组织基因组DNA甲基化程度不完全相同。各组织的总甲基化水平从高到低依次为肌肉 > 心脏 > 胰脏 > 卵巢(表 2和图 7),仅有肌肉与性腺组织存在显著差异(P < 0.05);在半甲基化水平上,各组织间差异均不显著(P > 0.05);在全甲基化水平上,肌肉与胰脏、肌肉与性腺均存在显著性差异(P < 0.05)。可见,金乌贼基因组DNA的CCGG位点上全甲基化水平在不同组织中的高低趋势与总甲基化水平接近。通过比较分析还发现,各组织的全甲基化水平均高于半甲基化水平,但仅有肌肉组织的全甲基化与半甲基化水平差异显著(P < 0.05)。再次说明,雌性金乌贼基因组DNA的全甲基化水平更能代表总甲基化水平,且肌肉组织的甲基化程度更高。
2.3.2 雄性金乌贼不同组织间的DNA甲基化比较雄性金乌贼生长期的基因组DNA甲基化水平同样存在组织差异,但各组织的总甲基化水平高低顺序却不同于雌性金乌贼,其顺序依次为肌肉 > 精巢 > 心脏 > 胰脏(表 2和图 8),且肌肉组织与其他各组织间均存在显著差异(P < 0.05)。雄性金乌贼半甲基化水平和全甲基化水平在组织间的差异性规律与总甲基化水平基本一致,但各组织的半甲基化和全甲基化水平间差异不显著(P > 0.05),且半甲基化和全甲基化水平均不能代表雄性总甲基化水平。上述结果表明,金乌贼基因组DNA半甲基化和全甲基化的发生,在不同性别和组织间均存在差异。
3 讨论 3.1 金乌贼基因组DNA甲基化水平和模式与其他物种的比较DNA甲基化作为表观遗传修饰的重要方式,对生长发育、环境适应具有重要意义。与脊椎动物相比,对无脊椎动物DNA甲基化发生和作用机制了解甚少,可能与无脊椎模式生物秀丽隐杆线虫(Caenorhabditis elegans)的DNA甲基化缺失及果蝇(Drosophila melanogaster)的DNA甲基化水平过低(0.1%~0.6%)有关(Yi et al, 2009; Urieli-Shoval et al, 1982)。统计已有研究报道发现,水产无脊椎动物的总甲基化水平变化范围(15%~40%)小于水产脊椎动物(20%~65%)(表 3)。本研究监测到金乌贼总甲基化水平为23.97%~ 39.70%,在水产无脊椎动物中处于较高水平。此外,对比相同组织的总甲基化水平后发现,头足类(金乌贼、真蛸Octopus vulgaris)的外套膜肌肉组织总DNA甲基化水平(34.97%和66%),远高于双壳贝类(大珠母贝和马氏珠母贝Pinctada martensii)的外套膜组织(15.48%~19.61%),金乌贼和真蛸DNA总甲基化水平如此之高可能与头足类动物属于软体动物门中发育最完善、最高级的一大类群密切相关。在检测DNA总甲基化水平时,不同物种的生长环境、生活方式以及发育阶段等的差别也会造成一定的影响。
|
|
表 3 主要经济水产动物基因组DNA总甲基化水平比较 Table 3 Comparison of total methylation level of genomic DNA from major economic aquatic animals |
DNA的甲基化状态与生长发育调控密切相关,不同物种的全甲基化和半甲基化所发挥作用的大小不同。亲代母本橙色莫桑比克罗非鱼(Oreochromis mossambicus)、父本荷那龙罗非鱼(Oreochromis hornorum)以及子代荷那龙罗非鱼及其杂交种(尚慧文等, 2017)、太平洋牡蛎(姜群等, 2014)、虾夷扇贝(Patinopecten yessoensis) (吴彪等, 2015)等动物中半甲基化水平低于全甲基化水平;虾夷扇贝选育群体—玉贝(吴彪等, 2015)半甲基化和总甲基化水平相当;家禽动物中,鸡(Gallina)和鸭(Anatinae)(唐韶青等, 2006)则表现出半甲基化水平高于全甲基化水平。本研究结果显示,金乌贼基因组DNA半甲基化和全甲基化在不同性别和组织间均存在差异。其中,雌性金乌贼基因组DNA的全甲基化水平更能代表总甲基化水平,且肌肉组织的甲基化程度更高;雄性金乌贼半甲基化和全甲基化的组织间差异规律与总甲基化水平基本一致,但各组织的半甲基化和全甲基化间差异不显著(P > 0.05)。
3.2 性别间的甲基化差异分析性别对DNA甲基化水平有影响,同时DNA甲基化在调控性别分化中起重要作用。Nugent(2015)研究表明,小鼠雌性全甲基化水平显著高于雄性,通过高DNA甲基化水平可维持脑部女性化,当DNA Methyltransferase活性降低,视前区和性行为男性化。林蛙(Rana chensinensis)显现出因组织差异而造成雌雄总甲基化水平高低的差异,雄性林蛙肝脏和脑干中DNA总甲基化水平高于雌性,肌肉和皮肤雄性低于雌性(李雅芳等, 2014);莆田黑猪的公猪心脏、肝脏、脂肪、耳和尾DNA甲基化水平高于母猪,而肌肉低于母猪(张金耀, 2016);但在荣昌仔猪耳组织中显示,公猪总甲基化水平比母猪高,但差异不显著(白小青等, 2010);人类男性甲基化水平显著高于女性(Fuke et al, 2004)。本研究也发现,金乌贼甲基化水平和模式的性别差异,雌性金乌贼肌肉组织DNA总甲基化水平显著低于雄性,心脏和胰脏组织DNA总甲基化水平却显著高于雄性。推测金乌贼的DNA甲基化水平可能受发育阶段、取样部位等多种因素共同作用,从而造成不同组织的总甲基化水平在雌雄中的高低不相同。
3.3 组织间的甲基化差异分析同一个体不同组织间存在基因组甲基化水平的差异是生物界普遍存在的现象,且组织DNA的甲基化程度与基因的功能关系密切。李金龙等(2014)对北京油鸡的研究显示,肌肉甲基化程度与体重呈显著负相关,体重越高,肌肉组织的甲基化程度越低,其推测可能与DNA甲基化参与体重相关基因的表达和物种特异性有关。本研究显示,金乌贼不同组织间总甲基化水平变化差异达15.73%,且雌雄个体总甲基化水平最高的组织均是肌肉。生长期肌肉的主要功能是增加体重和增强运动,肌肉组织中的基因组DNA甲基化水平较高,则会导致相关基因的表达量较低。生长期金乌贼各组织的DNA甲基化水平与该阶段相应组织的生理功能是否存在相关性?这一问题还有待于进一步研究探讨。
在总甲基化水平上,除肌肉与性腺组织外,金乌贼其他组织间差异均不显著(P > 0.05)。表明基因表达调控、性状分化等过程不仅有DNA甲基化参与,这些过程还受多种其他调控机制的作用,如RNA干扰、组蛋白修饰等。并非全部组织特异性表达的基因都与甲基化有关(Song et al, 2005; 姜群等, 2014)。
Bai XQ, Wang JY, Chen Y, et al. Study on the difference of DNA methylation levels in Rongchang piglets. Chinese Journal of Animal Science, 2010, 46(13): 12-13 [ 白小青, 王金勇, 陈英, 等. 荣昌猪仔猪性别间DNA甲基化水平的差异研究. 中国畜牧杂志, 2010, 46(13): 12-13] |
Berdyshev GD, Korotaev GK, Boiarskikh GV, et al. Nucleotide composition of DNA and RNA from somatic tissues of humpback and its changes during spawning. Biokhimiia, 1967, 32(5): 988-993 |
Bird A. DNA methylation patterns and epigenetic memory. Genes & Development, 2002, 16(1): 6-21 |
Cao ZM, Yang J. Analysis of the methylation in genome DNA from different tissues of Anodonta woodiana. Ecology and Environmental Sciences, 2009, 18(6): 2011-2016 [ 曹哲明, 杨健. 背角无齿蚌不同组织的基因组DNA甲基化分析. 生态环境学报, 2009, 18(6): 2011-2016] |
De BL, Riedel L, Schmitt I, et al. DNA methylation levels of α-synuclein intron 1 in the aging brain. Neurobiology of Aging, 2015, 36(12): 3334.e7 DOI:10.1016/j.neurobiolaging.2015.08.028 |
Dong ZZ. The biology of world ocean economic cephalopod. Jinan: Shandong Science and Technology Press, 1991: 197-207 [ 董正之. 世界大洋经济头足类生物学. 济南: 山东科学技术出版社, 1991: 197-207]
|
Du Y, He YY, Li J, et al. MSAP analysis of genomic DNA in the tissues of wild and "Huanghai No.1" Fenneropenaeus chinensis. Journal of Fishery Sciences of China, 2013, 20(3): 536-543 [ 杜盈, 何玉英, 李健, 等. 野生和"黄海1号"中国明对虾不同组织基因组DNA的MSAP分析. 中国水产科学, 2013, 20(3): 536-543] |
Eva DF, Camino G, Sheila CM, et al. The role of DNA methylation on Octopus vulgaris development and their perspectives. Frontiers in Physiology, 2014, 5: 62 |
Fuke C, Shimabukuro M, Petronis A, et al. Age related changes in 5-methylcytosine content in human peripheral leukocytes and placentas:An HPLC-based study. Annals of Human Genetics, 2004, 68(3): 196-204 DOI:10.1046/j.1529-8817.2004.00081.x |
Gao S, Yang AF, Dong Y, et al. Analysis of DNA methylation in the body wall of sea cucumber Apostichopus japonicus with skin ulceration by the methylation-sensitive amplified polymorphism MSAP. Acta Hydrobiologica Sinica, 2017, 41(3): 637-642 [ 高杉, 杨爱馥, 董颖, 等. 仿刺参"化皮病"体壁组织DNA甲基化的MSAP分析. 水生生物学报, 2017, 41(3): 637-642 DOI:10.7541/2017.81] |
Golbus J, Palella TD, Richardson BC. Quantitative changes in T cell DNA methylation occur during differentiation and ageing. European Journal of Immunology, 1990, 20(8): 1869-1872 DOI:10.1002/(ISSN)1521-4141 |
Guo TT, Sun GH, Yang JM, et al. MSAP analysis of genome DNA methylation in different tissues of Apostichopus japonicus. Oceanologia et Limnologia Sinica, 2013, 44(1): 77-82 [ 郭婷婷, 孙国华, 杨建敏, 等. 刺参(Apostichopus japonicus)不同组织基因组甲基化状态MSAP分析. 海洋与湖沼, 2013, 44(1): 77-82 DOI:10.11693/hyhz201301012012] |
Hao ZL, Zhang XM, Zhang PD. Biological characteristics and multiplication techniques of Sepia esculenta. Chinese Journal of Ecology, 2007, 26(4): 601-606 [ 郝振林, 张秀梅, 张沛东. 金乌贼的生物学特性及增殖技术. 生物学杂志, 2007, 26(4): 601-606] |
Hata K, Okano M, Lei H, et al. Dnmt3L cooperates with the Dnmt3 family of de novo DNA methyltransferases to establish maternal imprints in mice. Development, 2002, 129(8): 1983-1993 |
Jiang Q, Li Q, Yu H, et al. Genetic and epigenetic variation in mass selection populations of Pacific oyster Crassostrea gigas. Genes & Genomics, 2013, 35(5): 641-647 |
Jiang Q, Yu H, Kong LF, et al. Analysis of DNA methylation in different tissues of the Pacific oyster (Crassostrea gigas) with the fluorescence-labeled methylation-sensitive amplified polymorphism (F-MSAP). Journal of Fishery Sciences of China, 2014, 21(4): 676-683 [ 姜群, 于红, 孔令锋, 等. 太平洋牡蛎不同组织DNA甲基化的F-MSAP分析. 中国水产科学, 2014, 21(4): 676-683] |
Jiang Q. DNA methylation studies on the Pacific oyster Crassostrea gigas. Doctoral Dissertation of Ocean University of China, 2015 [姜群. 长牡蛎DNA甲基化研究. 中国海洋大学博士研究生学位论文, 2015] http://cdmd.cnki.com.cn/Article/CDMD-10423-1015716143.htm
|
Li JL, Tang SQ, Zhao M, et al. Detection of DNA methylation in whole genomes of muscle and ovary tissues of Beijing You chicken. Acta Veterinaria et Zootechnica Sinica, 2014, 45(11): 1784-1792 [ 李金龙, 唐韶青, 赵萌, 等. 北京油鸡肌肉和卵巢组织基因组DNA甲基化状态检测与分析. 畜牧兽医学报, 2014, 45(11): 1784-1792] |
Li SP, He F, Wen HS, et al. Analysis of DNA methylation levels by methylation-sensitive amplification polymorphism in half smooth tongue sole Cynoglossus semilaevis subjected to salinity stress. Annual Meeting of Chinese Society of Fisheries, 2015 [李思平, 何峰, 温海深, 等. 低盐胁迫下半滑舌鳎不同组织基因组甲基化状态MSAP分析. 中国水产学会学术年会, 2015]
|
Li Y, Qian WQ. Mechanisms of DNA methylation and demethylation in plants. Chinese Bulletin of Life Sciences, 2017, 29(3): 302-309 [ 李艳, 钱伟强. 植物中DNA甲基化及去甲基化研究进展. 生命科学, 2017, 29(3): 302-309] |
Li YF, Wang N, Bai JW, et al. In vivo DNA methylation levels of Rana chensinensis in gold mining area. Asian Journal of Ecotoxicology, 2014, 9(5): 956-963 [ 李雅芳, 王宁, 柏建雯, 等. 金矿区中国林蛙体内DNA甲基化水平. 生态毒理学报, 2014, 9(5): 956-963] |
Liu CL, Ruan FT, Qin B, et al. Analysis and evaluation of nutritive composition of the muscle of wild adult Sepia esculenta. Marine Sciences, 2016, 40(8): 42-48 [ 刘长琳, 阮飞腾, 秦搏, 等. 野生金乌贼成体肌肉的营养成分分析及评价. 海洋科学, 2016, 40(8): 42-48 DOI:10.11759//hykx20141223002] |
Liu CL, Zhuang ZM, Chen SQ, et al. Study on the brood stock domestication and propagation characteristics of Sepia esculenta Hoyle. Fishery Modernization, 2009, 36(2): 34-37 [ 刘长琳, 庄志猛, 陈四清, 等. 金乌贼亲体驯养与繁殖特性研究. 渔业现代化, 2009, 36(2): 34-37] |
Luo SJ, Deng YW, Zheng ZH, et al. Analysis of genomic DNA methylation on different regions of mantle tissue from Pinctada martensii by methylation-sensitive amplification polymorphism. Journal of Fishery Sciences of China, 2016, 23(6): 1227-1235 [ 罗少杰, 邓文, 郑哲, 等. 马氏珠母贝外套膜不同区域基因组DNA甲基化MSAP分析. 中国水产科学, 2016, 23(6): 1227-1235] |
Lv J, Hou R, Li N, et al. Application of MSAP technique for investigation of genome-wide DNA methylation level in scallops. Periodical of Ocean University of China (Natural Science), 2013, 43(10): 48-53 [ 吕佳, 侯睿, 李宁, 等. 应用MSAP技术研究扇贝全基因组DNA甲基化水平. 中国海洋大学学报(自然科学版), 2013, 43(10): 48-53] |
Mohandas T, Sparkes RS, Shapiro LJ. Reactivation of an inactive human X chromosome:Evidence for X inactivation by DNA methylation. Science, 1981, 211(4480): 393-396 DOI:10.1126/science.6164095 |
Nugent BM, Wright CL, Shetty AC, et al. Brain feminization requires active repression of masculinization via DNA methylation. Nature Neuroscience, 2015, 18(5): 690-697 DOI:10.1038/nn.3988 |
Pablo GF, Danie GS, Eduardo A, et al. Epigenetic DNA methylation mediating Octopus vulgaris early development:Effect of essential fatty acids enriched diet. Frontiers in Physiology, 2017, 8: 292 DOI:10.3389/fphys.2017.00292 |
Rhee I, Bachman KE, Park BH, et al. DNMT1 and DNMT3b cooperate to silence genes in human cancer cells. Nature, 2002, 416(6880): 552-556 DOI:10.1038/416552a |
Shang HW, Liu ZG, Zhu HP, et al. Analysis of DNA methylation difference between "Mohe" hybrid tilapia and its parents. Journal of Fisheries of China, 2017, 41(11): 1699-1709 [ 尚慧文, 刘志刚, 朱华平, 等. 莫荷罗非鱼"广福1号"与其亲本间DNA甲基化的差异分析. 水产学报, 2017, 41(11): 1699-1709] |
Song F, Smith JF, Kimura MT, et al. Association of tissue-specific differentially methylated regions (TDMs) with differential gene expression. Proceeding of the National Academy of Sciences, 2005, 102(9): 3336-3341 DOI:10.1073/pnas.0408436102 |
Tang SQ, Zhang Y, Xu Q, et al. Analysis of methylation level of genome in various tissues of different animal species. Journal of Agricultural Biotechnology, 2006, 14(4): 507-510 [ 唐绍青, 张沅, 徐青, 等. 不同动物部分组织基因组甲基化程度的差异分析. 农业生物技术学报, 2006, 14(4): 507-510] |
Urieli-Shoval S, Gruenbaum Y, Sedat J, et al. The absence of detectable methylated bases in Drosophila melanogaster DNA. FEBS Letters, 1982, 146(1): 148-152 DOI:10.1016/0014-5793(82)80723-0 |
Wang YL. DNA methylation in the sexual stage of Cordyceps militaris and functions of DNA methyltransferases in Metarhizium robertsii. Doctoral Dissertation of Anhui Agricultural University, 2016 [王玉龙. 蛹虫草有性阶段DNA甲基化及绿僵菌DNA甲基转移酶功能研究. 安徽农业大学博士研究生学位论文, 2016] http://cdmd.cnki.com.cn/Article/CDMD-10364-1016258361.htm
|
Wei GH, Cao ZM, Ding WD, et al. Effects of light application time on genomic DNA methylation of different tissues in rice field eel (Monopterus albus) by MSAP analysis. Journal of Huazhong Agricultural University, 2015, 34(6): 101-107 [ 魏国华, 曹哲明, 丁炜东, 等. 不同光照周期下黄鳝不同组织DNA甲基化的MSAP分析. 华中农业大学学报, 2015, 34(6): 101-107] |
Wei SF, Zhang AH. Research progress on DNA hypomethylation and its relationship with tumor. Carcinogenesis, Teratogenesis & Mutagenesis, 2016, 28(3): 243-245 [ 魏绍峰, 张爱华. DNA低甲基化及其与肿瘤关系的研究进展. 癌变·畸变·突变, 2016, 28(3): 243-245] |
Wu B, Yang AG, Dong YH, et al. MSAP analysis on genome-wide DNA methylation in selected and wild Janpanese scallop Patinopecten yessoensis. Oceanologia et Limnologia Sinica, 2015, 46(4): 976-982 [ 吴彪, 杨爱国, 董迎辉, 等. 虾夷扇贝选育群体与野生群体基因组DNA甲基化的MSAP分析. 海洋与湖沼, 2015, 46(4): 976-982] |
Xiong LZ, Xu CG, Saghai Maroof MA, et al. Patterns of cytosine methylation in an elite rice hybrid and its parental lines, detected by a methylation-sensitive amplification polymorphism technique. Molecular & General Genetics Mgg, 1999, 261(3): 439-446 |
Xu M, Li X, Korban SS. AFLP-based detection of DNA methylation. Plant Molecular Biology Report, 2000, 18(4): 361-368 DOI:10.1007/BF02825064 |
Yang C, Chu WH, Lu X, et al. Comparison the methods using in detect genome-wide DNA methylation of Antler stem cell. Journal of Jilin Agricultural University, 2016, 38(1): 97-101 [ 杨春, 褚文辉, 路晓, 等. 鹿茸干细胞基因组DNA甲基化的检测方法. 吉林农业大学学报, 2016, 38(1): 97-101] |
Yao GL, Yun YG, Qin L, et al. Analysis of DNA methylation in tissues and development stages of pearl oyster Pinctada fucata. Genes & Genomics, 2015, 37: 263-270 |
Yi SV, Goodisman MAD. Computational approaches for understanding the evolution of DNA methylation in animals. Epigenetics, 2009, 4(8): 551-556 DOI:10.4161/epi.4.8.10345 |
Zhang H, Zhang X, Clark E, et al. TET1 is a DNA-binding protein that modulates DNA methylation and gene transcription via hydroxylation of 5-methylcytosine. Cell Research, 2010, 20(12): 1390-1393 DOI:10.1038/cr.2010.156 |
Zhang JY. Study of the difference on DNA methylation of Putian black pig. Masterxs Thesis of Fujian Agriculture and Forestry University, 2016 [张金耀. 莆田黑猪DNA甲基化差异研究. 福建农林大学硕士研究生学位论文, 2016] http://cdmd.cnki.com.cn/Article/CDMD-10389-1016133733.htm
|
Zhang X, Li Q, Yu H, et al. Effects of air exposure on genomic DNA methylation in the Pacific oyster (Crassostrea gigas). Journal of Fishery Sciences of China, 2017, 24(4): 690-697 [ 张鑫, 李琪, 于红, 等. 干露胁迫对长牡蛎基因组DNA甲基化的影响. 中国水产科学, 2017, 24(4): 690-697] |
Zuo ZL, Tan J, Wu B, et al. MSAP analysis of genomic DNA in the tissues of Apostichopus japonicus and white A. japonicus. Progress in Fishery Sciences, 2016, 37(3): 93-100 [ 左之良, 谭杰, 吴彪, 等. 普通刺参(Apostichopus japonicus)和白刺参不同组织基因组DNA的MSAP研究. 渔业科学进展, 2016, 37(3): 93-100 DOI:10.11758/yykxjz.20151010001] |



