2. 中国水产科学研究院南海水产研究所 农业农村部南海渔业资源开发利用重点实验室 广州 510300;
3. 广州大学生命科学学院 广州 510006
2. Key Laboratory of South China Sea Fishery Resources Exploitation & Utilization, Ministry of Agriculture and Rural Affairs, South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510300;
3. School of Life Science, Guangzhou University, Guangzhou 510006
无乳链球菌(Streptococcus agalactiae)也称B簇链球菌(Group B streptococci, GBS),为兼性革兰氏阳性球菌(Rosinski-Chupin et al, 2013; Soto et al, 2015)。该菌宿主广泛,不仅能感染包括人在内的哺乳动物,还是养殖鱼类的重要病原(Schuchat et al, 1998)。罗非鱼为重要的世界性养殖鱼类,是联合国粮农组织(FAO)向全球推广养殖的优良品种,中国罗非鱼养殖年产量约为178万t,占世界罗非鱼总产量的三分之一以上,是水产品出口量最大的品种之一(2016中国渔业统计年鉴)。近年来,罗非鱼链球菌病日益严重,无乳链球菌作为其主要病原,传染力强,致死率高,由此产生的病害给罗非鱼养殖产业带来了巨大的经济损失(Evans et al, 2008; Kayansamruaj et al, 2015)。
罗非鱼无乳链球菌病发病较快,部分鱼从发病到死亡无明显病症,较难确定鱼体是否携带该病原菌,从而影响对病害的有效处理(程世亮等, 2016)。传统的病原菌分离和生理生化鉴定等方法,由于操作繁琐、检测周期长,不能达到快速检测目的(张新艳等, 2008; 卢迈新等, 2010);普通PCR方法,一次仅能检测1个基因,可能会造成漏检或误检(Wu et al, 2008);环介导等温扩增(LAMP)方法,具有灵敏度高、反应时间短、临床使用不需要特殊的仪器、操作简单等优点,但对引物设计要求比较高,有些基因可能不适用,还容易形成气溶胶污染,造成假阳性(甘文佳等, 2010);荧光定量PCR虽然具有较高的灵敏度,但检测仪器昂贵,对实验操作技术水平要求较高(Su et al, 2016)。因此,建立快速、精准、高效且适用于生产一线的无乳链球菌检测技术势在必行。
多重PCR在一次反应中能同时扩增多个目的片段的靶序列,具有高效、快捷、高度特异、敏感等优势,已经成为诸多实验室的常规诊断方法(毕艳妮等, 2014)。目前,多重PCR方法已用于养殖对虾中同时检测6种病毒(刘飞等, 2014)。本研究以无乳链球菌的毒力基因即透明质酸酶基因hylB、编码表面相关青霉素结合蛋白的ponA基因和CAMP因子的cfb基因为靶标,设计3对特异性引物,通过引物组合优化建立一种快速、高效、稳定、特异性好、灵敏度高的无乳链球菌三重PCR检测方法,旨在为无乳链球菌的快速检测和早期预警提供一种准确、高效的检测技术。
1 材料与方法 1.1 实验菌株鱼源无乳链球菌标准株(ATCC51487)和人源无乳链球菌标准株(ATCC BAA-1138)购于中国微生物菌种保藏中心。来源于不同年份和地区的无乳链球菌分离株TZQ0901(2009年分离自广东省肇庆市)、TGZ1001(2010年分离自广东省高州市)、TYC1101 (2011年分离自广东省阳春市)、THZ1301(2013年分离自广东省惠州市惠东区)、GZN1501(2015年分离自广东省广州市)、TLC1601(2016年分离自广东省龙川县)均从宿主罗非鱼分离获得,海豚链球菌(S. iniae)、美人鱼发光杆菌杀鱼亚种(Photobacterium damselae subsp. piscicida)、粪肠球菌(Enterococcus faecalis)、金黄色葡萄球菌(Staphylococcus aureus)、迟缓爱德华氏菌(Edwardsiella tarda)、创伤弧菌(Vibrio vulnificus)均由实验室分离、鉴定并保存。
1.2 引物的设计与合成根据GenBank中无乳链球菌3个特异性hylB、ponA和cfb基因序列,通过Primer Premier 6.0软件设计相应的引物对(表 1),由上海生工生物工程技术服务有限公司合成。
|
|
表 1 3对引物的信息 Table 1 The information of the three pairs of primers |
各实验菌株的DNA提取按照细菌基因组DNA提取试剂盒的操作说明,并保存于-20℃备用。罗非鱼组织中细菌DNA提取步骤,参照Su等(2016)的方法。首先取鱼组织约50 mg,放入组织匀浆器中研磨,加入1000 μl TE缓冲液,制成组织匀浆液。取上述匀浆液180 μl,加入20 μl浓度为50 mg/ml的溶菌酶,30℃孵育10 min,后续步骤均参照细菌DNA提取试剂盒进行。即得到高纯度的鱼(鱼包括体内细菌)基因组DNA,保存于-20℃备用。
1.4 反应条件的优化与建立单一引物扩增采用25 μl反应体系:2×PCR DsMix 12.5 μl,引物F(10 μmol/L) 1.0 μl,引物R(10 μmol/L) 1.0 μl,DNA模板1.0 μl,ddH2O 9.5 μl。利用3对引物进行PCR扩增,反应体系组成:2×PCR DsMix 12.5 μl,各引物上、下游(10 μmol/L)分别为0.2~1.0 μl,DNA模板1.0 μl,补充ddH2O至25 μl。反应条件:94℃预变性4 min;94℃变性30 s,55~60℃退火30 s,72℃延伸1 min,扩增30个循环;72℃延伸10 min。然后取5 μl扩增产物,在恒压120 V条件下,1%的琼脂糖凝胶电泳25 min检测结果。其中,无菌超纯水代替DNA模板的扩增结果为阴性对照。根据扩增结果,选择最佳的引物比例和退火温度。同时,将三重PCR方法所扩增的特异性片段送北京六合华大基因科技有限公司(广州)进行测序验证。
1.5 特异性实验分别以鱼源无乳链球菌标准株(ATCC51487)、人源无乳链球菌标准株(ATCC BAA-1138)、6株罗非鱼无乳链球菌(TZQ0901、TGZ1001、TYC1101、THZ1301、GZN1501和TLC1601)、海豚链球菌、美人鱼发光杆菌杀鱼亚种、粪肠球菌、金黄色葡萄球菌、创伤弧菌和迟缓爱德华菌的基因组DNA为模板,根据优化后的多重PCR反应条件和体系进行PCR扩增。
1.6 敏感性实验测定提取的鱼源无乳链球菌标准株(ATCC51487)基因组DNA浓度,将样品浓度按照50、5-1、5-2、5-3、5-4、5-5、5-6和5-7进行梯度稀释后分别为模板DNA,采用优化的多重PCR反应体系和条件进行PCR扩增,确定三重PCR检测方法所需模板的最低浓度。
1.7 应用利用构建好的三重PCR方法,检测来自广东省廉江市、高州市、吴川市、开平市、惠城区和龙川县6个不同罗非鱼养殖区188个罗非鱼组织样品。同时,利用常规细菌分离鉴定法对每份组织样品进行检测,比较这2种检测方法的结果。
1.8 无乳链球菌三重PCR检测方法的评价根据世界动物卫生组织(OIE)《水生动物疾病诊断手册》(2016)所规定的原则,在95%以上的置信区间、允许误差5%范围内对188个罗非鱼组织样品进行检测,以常规细菌分离鉴定法为标准计算此三重PCR检测方法的诊断敏感性(Dse)和诊断特异性(Dsp)。
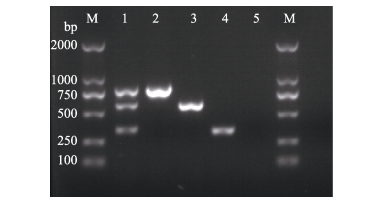
2 结果 2.1 三重PCR反应体系的优化分别对各引物加入体系的体积和退火温度进行优化,优化后的最佳退火温度为57℃,反应体系优化结果见表 2。利用优化后的反应体系和反应条件进行无乳链球菌检测,出现3条特异性条带;使用单一引物分别扩增hylB、ponA和cfb基因,均出现1条特异性条带,条带位置与预期结果一致(图 1)。此外,对送测序的特异性条带序列进行BLAST比对,与无乳链球菌hylB、ponA和cfb基因的同源性均达到或超过99%,表明构建的三重PCR所扩增的特异性条带是准确的。
|
|
表 2 三重PCR反应体系试剂组成 Table 2 The content of the reagent in the reaction system of a triple PCR |
|
图 1 3对引物和单一引物PCR扩增结果 Figure 1 The amplification of the single primer and three pairs of primers M: DL2000 bp marker;1:三重PCR的结果;2~4分别表示hylB、ponA和cfb基因的扩增片段;5:阴性 M: DL2000 bp marker; Lane 1: the result of three pairs of primers; Lanes 2~4: Amplified fragments of hylB, ponA, and cfb; Lane 5: Negative control |
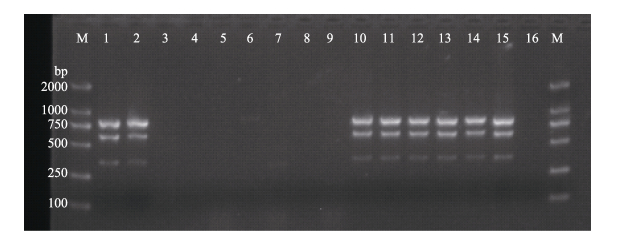
特异性实验结果显示,鱼源无乳链球菌(ATCC51487)、人源无乳链球菌(ATCC BAA-1138)以及不同年份和地区的无乳链球菌分离株扩增结果一致,均扩增出3条目的条带。而以罗非鱼、美人鱼发光杆菌杀鱼亚种、粪肠球菌、金黄色葡萄球菌、迟缓爱德华氏菌、海豚链球菌、创伤弧菌的基因组DNA为模板均未扩增出任何条带,与阴性对照结果相一致(图 2)。
|
图 2 三重PCR特异性检测结果 Figure 2 The pecificity result of triple PCR assay M:DL2000 bp marker;1:鱼源无乳链球菌(ATCC51487);2:人源无乳链球菌(ATCC BAA-1138);3:罗非鱼基因组;4:美人鱼发光杆菌杀鱼亚种;5:粪肠球菌;6:金黄色葡萄球菌;7:迟缓爱德华氏菌;8:海豚链球菌;9:创伤弧菌;10~15:扩增使用的模板为TZQ0901、TGZ1001、TYC1101、THZ1301、GZN1501和TLC1601;16:阴性对照 M: DL2000 bp marker; Lane 1: S. agalactiae ATCC51487; Lane 2: S. agalactiae ATCC BAA-1138; Lane 3: Tilapia genomic DNA; Lane 4: P. damselae subsp. piscicida; Lane 5: E. faecalis; Lane 6: S. aureus; Lane 7: E. tarda; Lane 8: S. iniae; Lane 9: V. vulnificus; Lanes 10~15: TZQ0901, TGZ1001, TYC1101, THZ1301, GZN1501, and TLC1601 of S. agalactiae; Lane 16: Negative control |
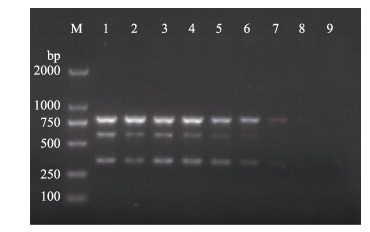
以无乳链球菌基因组DNA浓度分别为5.65、1.13、2.26×10-1、4.52×10-2、9.04×10-3、1.81×10-3、3.62×10-4和7.24×10-5 ng/μl为模板进行PCR扩增,结果显示,三重PCR能检测到的无乳链球菌基因组DNA最低浓度为1.81×10-3 ng/μl(图 3)。
|
图 3 三重PCR敏感性检测结果 Figure 3 The sensitivity result of the triple PCR assay M:DL2000 bp marker;1~8:无乳链球菌模板浓度分别为5.65、1.13、2.26×10-1、4.52×10-2、9.04×10-3、1.81×10-3、3.62×10-4和7.24×10-5 ng/μl;9:阴性对照 M: DL2000 bp marker; Lanes 1~8: Template concentration of S. agalactiae as 5.65, 1.13, 2.26×10-1, 4.52×10-2, 9.04×10-3, 1.81×10-3, 3.62×10-4, and 7.24×10-5 ng/μl; Lane 9: Negative control |
采自廉江、龙川、高州、吴川、开平和惠东的罗非鱼样品阳性检出率分别为100%(4/4)、100%(8/8)、57.14%(4/7)、50%(2/4)、1.23%(2/159)和0(0/6);总体来看,188尾罗非鱼组织样品的阳性检出率为10.6% (20/188),均与常规细菌分离鉴定结果相一致(表 3)。
|
|
表 3 组织样品检测结果 Table 3 The detection result of the tissue samples |
以常规细菌分离鉴定方法为标准,根据Dse和Dsp计算公式得出本研究所建立的三重PCR检测方法的Dse和Dsp均为100%(表 4)。
|
|
表 4 两种不同的方法对组织样品的检测 Table 4 Two different methods for detection of the tissue samples |
罗非鱼是我国主要的水产品养殖和出口品种之一,近几年,由于罗非鱼产品不合格(农兽药残留、品质不合格、微生物污染、食品添加剂超标等),出口严重受阻(赵海军等, 2015)。其中,由无乳链球菌带来的病害给罗非鱼养殖产业带来的损失最为严重。水产品中微生物的检测方法主要有生化培养法、免疫学法和核酸检测法等(应晓国等, 2017),王远微等(2008)利用三重PCR对临床疑似样本的检出率为81.8%,对人工攻毒样本的检出率为87.5%,两组数据均高于用细菌分离的检出率(40.9%和67.5%),说明多重PCR可避免单个毒力基因进行检测时漏检情况的发生,同时也暗示多重PCR比细菌分离技术更灵敏。
基于PCR技术的无乳链球菌检测方法大多选取靶标基因为16S~23S rRNA间区序列、sip和荚膜多糖类编码基因等(王均等, 2011; Forsman et al, 1997)。无乳链球菌普遍存在20多种毒力基因(Lin et al, 2011),透明质酸酶(Hyl)是无乳链球菌重要的毒力因子,有助于链球菌在巨噬细胞内存活并影响巨噬细胞促炎性因子的表达,在无乳链球菌致病过程中起关键作用(Wang et al, 2014)。青霉素结合蛋白(PBP1a)在GBS抗吞噬清除中起重要作用(Hamilton et al, 2006; Jones et al, 2000),蔡朝阳等(2006)发现青霉素结合蛋白的改变与金黄色葡萄球菌对甲氧西林等β-内酰胺类抗生素以及万古霉素等糖肽类抗生素耐药有密切关系,影响药物作用的靶位,阻止抗生素的杀菌作用。CAMP因子认为是具有成孔性质的分泌蛋白,在GBS致病过程中发挥重要作用(Udo et al, 2013)。通过基因组的数据检索确认,不同来源的(包括人及其他动物源)无乳链球菌均包含上述3种毒力基因,说明它们在病原体的侵入、繁殖与扩散及抵抗机体防御中起关键作用(Casadevall et al, 2001),因此,选择hylB、ponA和cfb毒力基因设计引物检测罗非鱼无乳链球菌十分必要。
多重PCR方法具有特异性好、灵敏度高的优点,被广泛应用于水产病原的检测。曾伟伟等(2013)针对草鱼呼肠孤病毒(Grass carp reovirus, GCRV)各类型毒株的保守序列,建立了GCRV的三重PCR检测方法;危芙蓉等(2010)根据广州管圆线虫核糖体小亚基rDNA基因序列设计特异性引物,并与小管福寿螺16S rDNA的特异性引物组合,建立了检测小管福寿螺体内广州管圆线虫的多重PCR方法;李晨等(2010)根据水产上常见病原菌鳗弧菌(Vibrio anguillarum)、嗜水气单胞菌(Aeromonas hydrophila)、迟缓爱德华氏菌(E. tarda)的毒力相关基因,建立了同时检测3种菌的单管多重PCR方法。本研究中,用单一引物分别扩增hylB、ponA和cfb基因,可获得特异性扩增条带,将上述3对引物组合并优化出多重PCR条件和体系,可同时扩增出3条特异性条带。Su等(2016)研究发现,以无乳链球菌的一些毒力基因(如sip和cpsE)为靶标设计引物进行PCR扩增,存在跟其他病原菌基因组DNA交叉反应的现象,而本研究中未能在其他水产动物病原菌基因组中扩增出条带,表明所建立的三重PCR检测方法特异性强。灵敏度测试发现,本研究所构建的方法可检测到浓度为1.81×10-3 ng/μl的细菌基因组DNA,比黄锦炉等(2012)构建的三重PCR检测方法灵敏度提高了近200倍。运用本研究构建的三重PCR方法检测广东省各地区罗非鱼组织样品的阳性检出率为10.6%(20/188),与细菌分离鉴定的结果吻合,说明三重PCR在罗非鱼样品检测过程中具有广阔的应用前景。该三重PCR检测方法准确性高、特异性强、灵敏度高,为罗非鱼无乳链球菌病的诊断、大规模检疫、流行病学调查等提供了一种快速、准确而有效的方法。
Bi YN, Song Y, Ma SQ, et al. Progress in the application of multiplex PCR in detection of pathogenic microorganisms. Chinese Journal of Microecology, 2014, 26(12): 1476-1478 [ 毕艳妮, 宋宇, 马淑青, 等. 多重PCR技术在病原微生物检测中的应用进展. 中国微生态学杂志, 2014, 26(12): 1476-1478] |
Cai ZY. Relationship between penicillin binding protein and drug resistance of Staphylococcus aureus. International Journal of Laboratory Medicine, 2006, 27(4): 361-363 [ 蔡朝阳. 青霉素结合蛋白与金黄色葡萄球菌耐药性关系研究. 国际检验医学杂志, 2006, 27(4): 361-363 DOI:10.3969/j.issn.1673-4130.2006.04.027] |
Casadevall A, Pirofski L. Host-pathogen interactions: The attributes of virulence. Journal of Infectious Diseases, 2001, 184(3): 337-344 DOI:10.1086/jid.2001.184.issue-3 |
Cheng SL, Liu XF, Zheng W, et al. Streptococcus agalactiae infection in fish. Progress in Veterinary Medicine, 2016, 37(2): 105-109 [ 程世亮, 刘新风, 郑文, 等. 鱼类无乳链球菌病. 动物医学进展, 2016, 37(2): 105-109 DOI:10.3969/j.issn.1007-5038.2016.02.021] |
Evans JJ, Klesius PH, Bohnsack JF, et al. Genomic diversity of Streptoccocus agalactiae isolates from multiple hosts and their infectivity in Nile tilapia. International Symposium on Talipia in Aquaculture, 2008(8): 109 |
Forsman P, Tilsala-Timisjä rvi A, Alatossava T. Identification of staphylococcal and streptococcal causes of bovine mastitis using 16S-23S rRNA spacer regions. Microbiology, 1997, 143(11): 3491-3500 DOI:10.1099/00221287-143-11-3491 |
Gan WJ, Hu XC. LAMP and its use in gene diagnosis of pathogens. Journal of Pathogen Biology, 2010, 5(2): 143-145 [ 甘文佳, 胡旭初. LAMP技术及其在病原基因诊断中的应用. 中国病原生物学杂志, 2010, 5(2): 143-145] |
Hamilton A, Popham DL, Carl DJ, et al. Penicillin-binding protein 1a promotes resistance of group B streptococcus to antimicrobial peptides. Infection & Immunity, 2006, 74(11): 6179-6187 |
Huang JL, Wang KY, Xiao D, et al. The development and application of a triplex PCR method for rapid detection of Streptococcus agalactiae. Oceanologia et Limnologia Sinica, 2012, 43(2): 254-261 [ 黄锦炉, 汪开毓, 肖丹, 等. 无乳链球菌(Streptococcus agalactiae)三重PCR快速检测方法的建立与应用. 海洋与湖沼, 2012, 43(2): 254-261] |
Jones AL, Knoll KM, Rubens CE, et al. Identification of Streptococcus agalactiae virulence genes in the neonatal rat sepsis model using signature-tagged mutagenesis. Molecular Microbiology, 2000, 37(6): 1444-1455 DOI:10.1046/j.1365-2958.2000.02099.x |
Kayansamruaj P, Pirarat N, Kondo H, et al. Genomic comparison between pathogenic Streptococcus agalactiae isolated from Nile tilapia in Thailand and fish-derived ST7 strains. Infection Genetics & Evolution Journal of Molecular Epidemiology & Evolutionary Genetics in Infectious Diseases, 2015, 36: 307-314 |
Li C, Wang XH, Huang J. Multiplex PCR for the detection of three main aquatic pathogens. Progress in Fishery Sciences, 2010, 31(3): 100-106 [ 李晨, 王秀华, 黄倢. 3种主要水产病原菌多重PCR检测方法的建立. 渔业科学进展, 2010, 31(3): 100-106 DOI:10.3969/j.issn.1000-7075.2010.03.014] |
Lin FP, Lan R, Sintchenko V, et al. Computational bacterial genome-wide analysis of phylogenetic profiles reveals potential virulence genes of Streptococcus agalactiae. PLoS One, 2011, 6(4): e17964 DOI:10.1371/journal.pone.0017964 |
Liu F, Zhang BC, Zhang XH, et al. Multiplex PCR assay for simultaneous detection of six viruses in shrimp. Progress in Fishery Sciences, 2014, 35(1): 60-67 [ 刘飞, 张宝存, 张晓华, 等. 对虾6种病毒多重PCR检测方法的建立. 渔业科学进展, 2014, 35(1): 60-67 DOI:10.3969/j.issn.1000-7075.2014.01.009] |
Lu MX, Li J, Ye X, et al. Identification and characterizations of Streptococcus agalactiae isolated from tilapia cultured in Guangdong and Hainan provinces. Microbiology, 2010, 37(5): 766-774 [ 卢迈新, 黎炯, 叶星, 等. 广东与海南养殖罗非鱼无乳链球菌的分离、鉴定与特性分析. 微生物学通报, 2010, 37(5): 766-774] |
Rosinski-Chupin I, Sauvage E, Mairey B, et al. Reductive evolution in Streptococcus agalactiae and the emergence of a host adapted lineage. BMC Genomics, 2013, 14(1): 252 DOI:10.1186/1471-2164-14-252 |
Schuchat A. Epidemiology of group B streptococcal disease in the United States: Shifting paradigms. Clinical Microbiology Reviews, 1998, 11(3): 497-513 |
Soto E, Wang R, Wiles J, et al. Characterization of isolates of from diseased farmed and wild marine fish from the U. S. Gulf Coast, Latin America, and Thailand. Journal of Aquatic Animal Health, 2015, 27(2): 123-134 |
Su YL, Feng J, Li YW, et al. Development of a quantitative PCR assay for monitoring Streptococcus agalactiae, colonization and tissue tropism in experimentally infected tilapia. Journal of Fish Diseases, 2016, 39: 229-238 DOI:10.1111/jfd.2016.39.issue-2 |
Udo EE, Boswihi SS, Alsweih N. Genotypes and virulence genes in group B streptococcus isolated in the maternity hospital, Kuwait. Medical Principles and Practice, 2013, 22(5): 453-457 DOI:10.1159/000349932 |
Wang J, Wang KY, Xiao D, et al. Development of double PCR for rapid detection of Streptococcus agalactiae isolated from tilapia. Chinese Veterinary Science, 2011, 41(5): 496-502 [ 王均, 汪开毓, 肖丹, 等. 罗非鱼源无乳链球菌双重PCR快速检测方法的建立. 中国兽医科学, 2011, 41(5): 496-502] |
Wang YW, Tang C, Yu XH, et al. Detecting pathogenic Aeromonas hydrophila in fish by triplex PCR. Acta Microbiologica Sinica, 2008, 48(7): 947-951 [ 王远微, 汤承, 于学辉, 等. 三重PCR检测鱼类致病性嗜水气单胞菌. 微生物学报, 2008, 48(7): 947-951 DOI:10.3321/j.issn:0001-6209.2008.07.018] |
Wang Z, Guo C, Xu Y, et al. Two novel functions of hyaluronidase from Streptococcus agalactiae are enhanced intracellular survival and inhibition of proinflammatory cytokine expression. Infection & Immunity, 2014, 82(6): 2615-2625 |
Wei FR, Liu HX, Lv S, et al. Multiplex PCR assay for the detection of Angiostrongylus cantonensis larvae in Pomacea canaliculata. China Journal of Parasitology and Parasitic Diseases, 2010, 28(5): 355-358 [ 危芙蓉, 刘和香, 吕山, 等. 用多重PCR技术检测小管福寿螺体内广州管圆线虫幼虫. 中国寄生虫学与寄生虫病杂志, 2010, 28(5): 355-358] |
Wu J, Liu Y, Hu S, et al. Development of a rapid PCR test for identification of Streptococcus agalactiae in milk samples collected on filter paper disks. Asian Australasian Journal of Animal Sciences, 2008, 21(1): 124-130 DOI:10.5713/ajas.2008.70076 |
Ying XG, Yuan N, Deng SG, et al. Research progress on detection techniques of aquatic products. Journal of Food Safety and Quality, 2017, 8(4): 1196-1202 [ 应晓国, 袁宁, 邓尚贵, 等. 水产品质量安全检测技术研究进展. 食品安全质量检测学报, 2017, 8(4): 1196-1202] |
Zeng WW, Wang Q, Wang YY, et al. Establishment of multiplex PCR for detection of grass carp reovirus and its application. Journal of Fishery Sciences of China, 2013, 20(2): 419-426 [ 曾伟伟, 王庆, 王英英, 等. 草鱼呼肠孤病毒三重PCR检测方法的建立及其应用. 中国水产科学, 2013, 20(2): 419-426] |
Zhang XY, Fan HP, Zhong QF, et al. Isolation, identification and pathogenicity of Streptococcus agalactiae from tilapia. Journal of Fisheries of China, 2008, 32(5): 772-779 [ 张新艳, 樊海平, 钟全福, 等. 罗非鱼无乳链球菌的分离、鉴定及致病性研究. 水产学报, 2008, 32(5): 772-779] |
Zhao HJ, Yang BB, Hao Y, et al. The actualities and countermeasures of export tilapia in China. Journal of Food Safety and Quality, 2015, 6(12): 5100-5106 [ 赵海军, 杨彬彬, 郝跃, 等. 我国罗非鱼出口现状及对策. 食品安全质量检测学报, 2015, 6(12): 5100-5106] |



