2. 青岛大学生命科学学院 青岛 266071;
3. 农业农村部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;
4. 杭州千岛湖鲟龙科技股份有限公司 杭州 311701
2. College of Life Sciences Qingdao University, Qingdao 266071;
3. Key Laboratory for Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
4. Hangzhou Qiandaohu Xunlong Sci-Tech Co. Ltd. Hangzhou 311701
防御素是一类富含半胱氨酸的阳离子抗菌肽,广泛分布于植物、昆虫和脊椎动物中。根据对6个保守的半胱氨酸的位置和二硫键连接方式不同将防御素分为3个亚家族:α、β和θ-防御素(Zhu et al, 2013)。β-防御素是动物防御素系统中最大的成员,具有广谱的抗菌活性及不受传统抗生素耐药性菌株影响的抗菌机理,同时,与典型抗生素具有协同作用,能中和内毒素、调节机体免疫应答和炎症反应、调节组织创伤修复等而备受人们关注(张伟等, 2006)。
目前,鱼类中只发现了β-防御素,相对于其他脊椎动物,研究处于起步阶段。2007年首次在斑马鱼(Danio rerio)和河鲀(Takifugu rubripes)中分离鉴定出β-防御素(Zou et al, 2007)以来,迄今仅在金头鲷(Sparus aurata) (Cuesta et al, 2011)、团头鲂(Megalobrama amblycephala)(Liang et al, 2013)、斑点叉尾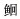
鲟鱼被称为“水中活化石”,近年来,俄罗斯鲟(Acipenser gueldenstaedti)养殖业在我国发展较快,由于其性成熟周期较长,养殖过程一旦发生病害,将对养殖业产生巨大影响,而嗜水气单胞菌(Aeromonas hydrophila)是致病的主要病原体之一(杨治国, 2001)。但俄罗斯鲟的免疫基因发掘及免疫机制鲜有报道。本研究在前期俄罗斯鲟性腺转录组(Chen et al, 2016)中挖掘到β-防御素部分序列的基础上,克隆了俄罗斯鲟β-防御素cDNA序列,推导出包含信号肽的完整蛋白序列,研究其组织表达及响应病原菌刺激后在免疫组织中的时空表达模式,为俄罗斯鲟免疫机理研究、病害防治和健康养殖提供有价值的研究资料。
1 材料与方法 1.1 实验材料健康1.5龄俄罗斯鲟,体长为(47.5±2.5) cm,体重为(450±10) g。取自杭州千岛湖鲟龙集团有限公司,暂养于直径2 m、水深0.5 m的圆形玻璃钢养殖缸中,至少15 d后方可进行实验。养殖用水引自乌溪江,水温为17~20℃。嗜水气单胞菌为实验室保存菌种,分离自俄罗斯鲟病鱼组织,经16S rRNA测序进行菌种鉴定。
1.2 组织收集分别收集3条健康俄罗斯鲟的肝、肠、脾、头肾、胃、鳃、血液、脑、皮肤、肌肉和性腺11种组织,立即放入液氮中,随后将组织转移至–80℃冰箱中保存,用于总RNA提取。采用腹腔注射方法按照半致死剂量(2.07×104 CFU/g)进行病原感染。对照组注射同等体积的1×PBS溶液。收集感染0、6、12、24、48、72 h实验组和对照组,各取3条俄罗斯鲟的肝、脾、鳃、血液、头肾和肠组织,保存方式同上所述。
1.3 总RNA提取及cDNA的合成采用液氮研磨收集的组织,参照总RNA提取试剂盒(天根, 北京)说明书进行组织总RNA提取。使用Agilent 2100生物分析仪(Applied Biosystems, 美国)检测其质量和浓度,通过琼脂糖凝胶电泳检测RNA的完整性。按照Prime Script TM RT reagent kit (Perfect Real time) (宝生物, 大连)试剂盒合成cDNA,-20℃保存备用。
1.4 俄罗斯鲟β-防御素基因cDNA片段克隆从俄罗斯鲟性腺转录组中筛选出β-防御素基因部分cDNA序列,采用Primer Premier 5.0在线软件(www.bio-soft.net)设计特异性引物P1和P2(表 1),以俄罗斯鲟11个组织混合cDNA为模板,进行PCR扩增。扩增产物连接T载体,转化到T1感受态细胞中,对阳性克隆进行测序。
|
|
表 1 俄罗斯鲟β-防御素基因克隆和表达分析所用引物 Table 1 Primers used for cloning and expression of A. gueldenstaedti β-defensin |
将测序拼接获得的基因序列运用NCBI(http://blast.ncbi.nlm.nih.gov/) Protein Blast进行β-防御素氨基酸同源性比对,使用在线软件SMART(http://smart.embl-heidelberg.de/)预测蛋白质结构域;信号肽预测采用SignalP3.0Server(http://www.cbs.dtu.dk/services/SignalP/),应用ORF finder(https://www.ncbi.nlm.nih.gov/orffinder/)、DNAstar(http://www.dnastar.com/)、ClustalX1.83 (http://www.genome.jp/tools/clustalx/)和DNAMAN(http://www.lynnon.com/)等软件进行俄罗斯鲟β-防御素基因开放阅读框的查找及氨基酸多重序列比对分析。利用MEGA 5.1重复1000次构建N-J进化树。应用CPHmodels3.2server(http://www.cbs.dtu.dk/services/CPHmodels/)在线软件预测β-防御素蛋白的三维结构。
1.6 俄罗斯鲟β-防御素基因表达的实时定量分析基因实时定量表达分析参照已报道的方法进行(路飏等, 2013; 于孟君等, 2017)。根据测序得到的俄罗斯鲟β-防御素cDNA序列设计qRT-PCR引物为P3、P4(表 1),以俄罗斯鲟18S rRNA基因为内参设计特异引物18S-F和18S-R(表 1)。用SYBR Green荧光定量试剂盒(天根, 北京)按照说明书在ABI PRISM 7500Fast实时定量扩增仪上(ABI,美国)进行该基因的实时定量分析(qRT-PCR)。每个样品设置3个生物学重复,使用2-ΔΔCt法计算目的基因相对表达量,Ct值取3个平行样品的平均值。qRT-PCR检测结果使用SPSS 19.0统计软件进行单因素方差分析(One-way ANOVA)的Duncan法对多组样本均数进行两两比较分析,P < 0.05时,认为存在显著性差异。
2 结果 2.1 俄罗斯鲟β-防御素基因序列分析克隆测序得到的俄罗斯鲟β-防御素基因部分cDNA片段长度为333 bp,用ORF finder软件分析其ORF长度为213 bp,编码71个氨基酸(图 1),与其他物种的同源序列所包含的ORF长度相当。信号肽和结构域分析结果显示,该基因序列包含23个氨基酸的信号肽、完整的β-防御素-2结构域(82~234)。

|
图 1 俄罗斯鲟β-防御素cDNA序列和推测的氨基酸序列 Figure 1 cDNA sequence and deduced amino acid sequence of A. gueldenstaedti β-defensin 推导的氨基酸序列显示在核苷酸序列下方,用大写字母表示;双下划线(=)所示为信号肽;方框所示为起始密码子(ATG)、星号(*)所示为终止密码子(TAG);下划线(—)为defensin-β-2结构域 Translated amino acid sequence was shown under nucleotide sequence as uppercase; The iniation codon and termination codon were marked by a box and an asterisk, respectively; Signal peptide was shown with double solid line(=); Defensin-β-2 domain was shown with underline(—) |
推导的俄罗斯鲟β-防御素氨基酸序列和其他物种的β-防御素氨基酸序列如斑马鱼(Danio rerio, NP_001075024.1)、青鳉(Oryzias latipes, ACG55699.1)、鳜(Epinephelus coioides, AFA41485.1)、鲤(Cyprinus carpio, AGZ03658.1)、梭鱼(Liza haematocheila, AIK66783.1)、点带石斑鱼(Epinephelus coioides, AFA41485.1)、福鼎蝾螈(Cynops fudingensis, AHF22421.1)、原鸡(Gallus gallus, ALI16124.1)、小鼠(Mus musculus, EDL32923.1)、大鼠(Rattus norvegicus, AAC61871.1)、恒河猴(Macaca mulatta, AAC61871.1)和人(Homo sapiens, AAH93985.1)等进行同源比对。结果显示,俄罗斯鲟β-防御素与其他物种的氨基酸序列同源性在39%~60%之间,其中,与斑马鱼和鲤鱼的同源性最高,为60%,其次是鳜鱼59%,与青鳉的同源性最低,为54%。但在不同的物种之间,都具有6个保守的半胱氨酸残基(图 2)。

|
图 2 推导的俄罗斯鲟β-防御素氨基酸与其他物种已知同源序列比对 Figure 2 Multiple alignment of A. gueldenstaedti β-defensin deduced amino acid sequence with other known orthologs 图中标黑处表示6个保守的半胱氨酸残基及位置,分别是C38、C44、C48、C60、C66和C67 Six conserved Cysteine residues (C38, C44, C48, C60, C66 and C67) in β-defensin protein were highlighted by black color |
利用MEGA 5.1以N-J法构建基于上述物种β-防御素氨基酸序列的分子系统进化树。结果显示,系统进化树分为两个大的分支,俄罗斯鲟β-防御素与斑马鱼、青鳉、鳜、梭鱼、点带石斑鱼聚为一大支,其中俄罗斯鲟单独聚为一个亚支,与斑马鱼和鲤进化关系最近;小鼠、大鼠和人聚为另外一个大支,其中,鼠类按亲缘关系远近聚成相应的亚支;两栖类福鼎蝾螈单独聚为一个分支(图 3)。

|
图 3 俄罗斯鲟与其他物种的β-防御素系统进化分析 Figure 3 Phylogenetic tree of β-defensin of A. gueldenstaedti and other vertebrates |
预测的俄罗斯鲟β-防御素的3D结构如图 4所示。结果显示,C38和C66、C44和C60及C48和C67之间分别形成3对二硫键,6个保守的半胱氨酸残基按照箭头方向形成反向平行的β片层折叠结构,对β-防御素结构稳定具有重要作用。该结构与斑马鱼β-防御素-2非常相似,从结构上判断,获得的俄罗斯鲟β-防御素基因应属于β-防御素-2基因。

|
图 4 预测的俄罗斯鲟β-防御素蛋白质三维结构 Figure 4 Three-dimensions structure of deduced protein of A. gueldenstaedti β-defensin 三个二硫键:C38和C66(绿色),C44和C60(蓝色)和C48和C67(红色),箭头方向表示β-片层折叠 Three disulfied bonds were formed C38-C66 (green), C44-C60 (blue) and C48-C67 (red), respectively Arrow mark indicates β-sheet |
qRT-PCR结果显示,β-防御素基因在检测的健康俄罗斯鲟11种组织(肝、肠、脾、头肾、胃、鳃、血液、脑、皮肤、肌肉和性腺)中均有不同程度的表达(图 5)。以表达量最低的血液(1.0)为对照,相对表达量最高的组织是性腺(21.75),其次是皮肤(10.05)、胃(2.5)、肌肉(2.4),在肝(1.5)和脾(1.5)、鳃(1.05)、肠(1.2)、脑(1.25)及头肾(1.1)中的表达量相对较低,β-防御素基因在不同组织中表达存在特异性。
|
图 5 β-防御素基因在健康俄罗斯鲟组织中的表达 Figure 5 β-defensin gene expressions in 11 tissues of healthy A. gueldenstaedti L:肝; In:小肠; Sp:脾; HK:头肾; Sk:皮肤; Mu:肌肉; Gi:鳃; Br:脑; Bl:血液; Go:性腺; St:胃。字母“a,b,c”代表SPSS多重分析的不同分组,有相同字母表示差异不显著(P > 0.05),无相同字母表示差异显著(P < 0.05)。下同 L: Liver; In: Intestine; Sp: Spleen; HK: Head-Kidney; Sk: Skin; Mu: Muscle; Gi: Gill; Br: Brain; Bl: Blood; Go: Gonad; St: Stomach. The letters 'a, b, c, d' are subsets by Duncan algorithm. The same letters indicate unsignificant difference (P > 0.05), the different letters indicate significant difference (P < 0.05). The same as below |
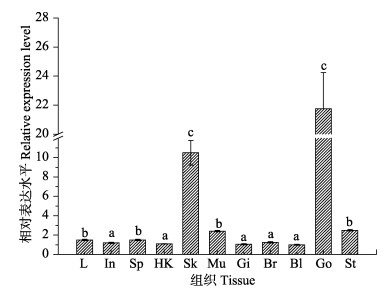
嗜水气单胞菌感染俄罗斯鲟0、6、12、24、48和72 h后,β-防御素基因在6种免疫组织(肝、肠、脾、头肾、鳃和血液)中呈现不同的表达特征,结果如图 6所示。嗜水气单胞菌感染俄罗斯鲟后,β-防御素在头肾、血液、肠、脾和鳃5种组织中均呈现上调表达,只是各组织中峰值出现的时间点不同,在肝中则呈现下调趋势。其中,β-防御素基因在头肾中呈现最为显著的上调表达,与0 h相比,在感染后6 h后即出现了10倍的增加,在感染后的12~48 h,基因表达表现为几十倍到上百倍不等的上调,到感染72 h达到峰值,为0 h表达量的700倍;β-防御素基因在小肠、血液和脾中整体上表现为上调表达,最大表达峰值达到几倍到十几倍,在鳃中表达量变化不大,最高的表达量出现在感染后12 h,为0 h表达量的2倍,其他时间点表达量与0 h相当;β-防御素基因在肝脏中则总体呈现下调表达模式,感染12~72 h后,表达量与0 h相比,呈现近5倍的明显下调表达。
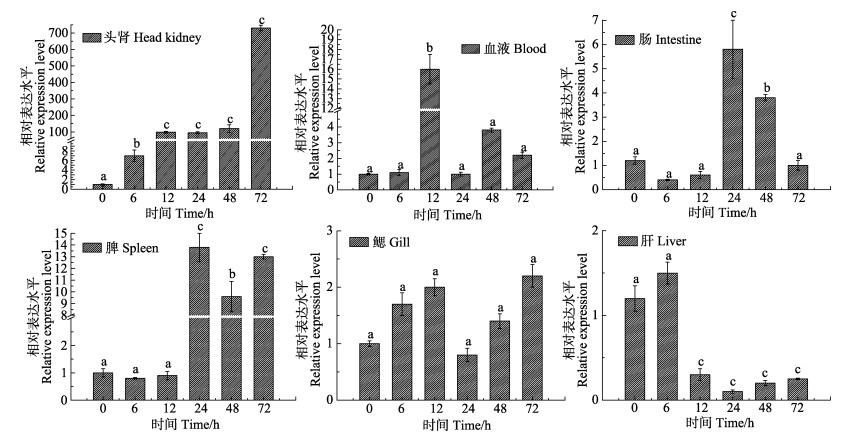
|
图 6 俄罗斯鲟感染嗜水气单胞菌后β-防御素基因在免疫组织中的表达 Figure 6 β-defensin gene expressions in immune tissues of A. gueldenstaedti after Aeromonas hydrophila challenge 每个时间点的β-防御素的表达量为感染组除以PBS组的相对表达量 The expression level of β-defensin at each timepoint was presented as relative expression in which the infected group divided by the PBS group |
养殖环境的生态环保对鱼类健康养殖产业提出了严格要求,发展替代抗生素的绿色无污染的渔药如抗菌肽等势在必行。鱼类β-防御素已被证明具有明显的抗菌、抗病毒功能(Jin et al, 2010; Casadei et al, 2009),具有广阔的应用前景。但目前鱼类β-防御素的研究不深入,亟待在更多的鱼种和功能分析上进行更深入的研究。俄罗斯鲟是生产鱼子酱的主要鱼种之一,经济价值很高,开展免疫机理和绿色病害防治研究,对发展和稳定鲟鱼养殖产业具有重要意义。本研究关于俄罗斯鲟β-防御素基因的克隆和表达研究,是俄罗斯鲟宿主免疫的首次报道。
俄罗斯鲟β-防御素基因cDNA长度为333 bp,ORF为213 bp,编码71个氨基酸,包含23个氨基酸的信号肽和48个成熟肽,目前报道的不同鱼类的β-防御素前体分子一般由62~77个氨基酸残基组成,信号序列和前导序列含有22~38个氨基酸残基,成熟肽序列由39~45个氨基酸残基组成(Casadei et al, 2009),本研究结果与报道结果吻合。另外,俄罗斯鲟β-防御素C端包含6个保守的半光氨酸残基,形成3个二硫键结构,与报道的鱼类β-防御素结构一致(Dong et al, 2015; Ruangsri et al, 2013; Guo et al, 2012; Ellis, 2001)。俄罗斯鲟β-防御素蛋白质空间结构预测形成3个β-片层折叠,与斑马鱼BD-2的结构(Ellis, 2001)很类似,SMART结构域预测,俄罗斯鲟β-防御素包含β-防御素-2结构域,同源序列比对的结果也证实其与斑马鱼BD-2的相似度最高,为60%,这些证据都指明所获得俄罗斯鲟β-防御素基因应属于β-防御素-2,在进化上还是属于相对原始的类型。
鱼类β-防御素是抗菌肽的重要成员,在各组织中广泛表达,罗非鱼β-防御素在检测的9种组织(心、肝、脾、鳃、头肾、肠、胃、肌肉和皮肤)中都有表达,其中,在皮肤中表达量最高,其次是在脾、头肾和肌肉中有较高的表达,在肝、心、肠、胃和鳃中的表达量最低(Dong et al, 2015)。β-防御素基因在健康斑点叉尾
目前,对鱼类β-防御素生物学活性的研究主要集中在抗菌和抗病毒等免疫活性方面,石斑鱼的防御素对革兰氏菌(阳性或阴性)均具有抗菌活性,并且还具有抗RGV病毒活性(Jin et al, 2010)。爱德华菌(Edwardsiella ictaluri)感染斑点叉尾
本研究初步研究了俄罗斯鲟的克隆、组织表达及相应病原刺激的免疫应答,后续研究尚需进一步深入。由于俄罗斯鲟的基因资源还远远不足,缺乏基因组及多数组织的转录组数据,加之俄罗斯鲟的基因组又极其复杂,染色体数目多达236条(尹洪滨等, 2006),β-防御素有可能存在较多的同源基因和重复基因,在一定程度上对俄罗斯鲟β-防御素基因的克隆和精准分型造成困难。由于获得的俄罗斯鲟cDNA片段长度只有333 bp,缺乏合适的位点设计RACE引物,我们尝试多次克隆,仍无法获得完整的5'和3'全长,但我们获得了完整的ORF,对进一步开展功能研究奠定了良好基础。在后续研究中,将进一步验证其对不同病原(细菌、病毒)等的体内和体外抗菌活性的研究,为俄罗斯鲟的绿色病害防治途径探讨可行之路。
Cuesta A, Meseguer J, Esteban M. Molecular and functional characterization of the gilthead seabream β-defensin demonstrate its chemotactic and antimicrobial activity. Molecular Immunology, 2011, 48(12-13): 1432-1438 DOI:10.1016/j.molimm.2011.03.022 |
Casadei E, Wang T, Zou J, et al. Characterization of three novel β-defensin antimicrobial peptides in rainbow trout (Oncorhynchus mykiss). Molecular Immunology, 2009, 46(16): 3358-3366 DOI:10.1016/j.molimm.2009.07.018 |
Chen Y, Xia Y, Shao C, et al. Discovery and identification of candidate sex-related genes based on transcriptome sequencing of Russian sturgeon (Acipenser gueldenstaedtii) gonads. Physiological Genomics, 2016, 48(7): 464 DOI:10.1152/physiolgenomics.00113.2015 |
Dong JJ, Wu F, Ye X, et al. B-defensin in Nile tilapia (Oreochromis niloticus): Sequence, tissue expression, and anti-bacterial activity of synthetic peptides. Gene, 2015, 566(1): 23-31 DOI:10.1016/j.gene.2015.04.025 |
Ellis AE. Innate host defense mechanisms of fish against viruses and bacteria. Developmental & Comparative immunology, 2001, 25(8-9): 827-839 |
Guo M, Wei J, Huang X, et al. Antiviral effects of β-defensin derived from orange-spotted grouper (Epinephelus coioides). Fish & Shellfish Immunology, 2012, 32(5): 828-838 |
Jin JY, Zhou L, Wang Y, et al. Antibacterial and antiviral roles of a fish β-defensin expressed both in pituitary and testis. PLoS One, 2010, 5(12): e12883 DOI:10.1371/journal.pone.0012883 |
Liang T, Wang DD, Zhang GR, et al. Molecular cloning and expression analysis of two beta-defensin genes in the blunt snout bream (Megalobrama amblycephala). Comparative Biochemistry & Physiology Part B Biochemistry & Molecular Biology, 2013, 166(1): 91-98 |
Lu Y, Wang QL, Li M, et al. Expression analysis of TLR20 and TLR21 genes in channel catfish Ictalurus punctatus challenged by different bacteria and virus. Progress in Fisheries Science, 2013, 34(6): 44-51 [ 路飏, 王启龙, 李敏, 等. 斑点叉尾  TLR20和TLR21基因在不同细菌和病毒感染后的表达特征>. 渔业科学进展, 2013, 34(6): 44-51 DOI:10.3969/j.issn.1000-7075.2013.06.007] TLR20和TLR21基因在不同细菌和病毒感染后的表达特征>. 渔业科学进展, 2013, 34(6): 44-51 DOI:10.3969/j.issn.1000-7075.2013.06.007] |
Qi Z, Xu W, Meng F, et al. Cloning and expression of β-defensin from soiny mullet (Liza haematocheila), with insights of its antibacterial mechanism. PLoS One, 2016, 11(6): e0157544 DOI:10.1371/journal.pone.0157544 |
Ruangsri J, Kitani Y, Kiron V, et al. A novel beta-defensin antimicrobial peptide in Atlantic cod with stimulatory effect on phagocytic activity. PLoS One, 2013, 8(4): e62302 DOI:10.1371/journal.pone.0062302 |
Yang ZG. Isolation and identification of Aeromonas hydrophila from sturgeons. Freshwater Fisheries, 2001, 31(5): 43-44 [ 杨治国. 鲟鱼嗜水气单胞菌的分离鉴定. 淡水渔业, 2001, 31(5): 40-41 DOI:10.3969/j.issn.1000-6907.2001.05.017] |
Yin HB, Sun ZW, Sun DJ, et al. Cytogenetic analysis of Acipenser gueldenstaedti Brandt. Journal of Fisheries of China, 2006, 30(2): 181-184 [ 尹洪滨, 孙中武, 孙大江, 等. 俄罗斯鲟的细胞遗传学分析. 水产学报, 2006, 30(2): 181-184] |
Yu MJ, Sun LM, Chen YD, et al. Cloning and expression of the c-Jun gene in immune response to pathogen challenge in half-smooth tongue sole (Cynoglossus semilaevis). Progress in Fisheries Science, 2017, 38(2): 31-39 [ 于孟君, 孙璐明, 陈亚东, 等. 半滑舌鳎(Cynoglossus semilaevis) c-Jun基因的克隆及免疫应答分析. 渔业科学进展, 2017, 38(2): 31-39] |
Zhu S, Gao B. Evolutionary origin of beta-defensins. Developmental and Comparative Immunology, 2013, 39: 79-84 DOI:10.1016/j.dci.2012.02.011 |
Zou J, Mercier C, Koussounadis A, et al. Discovery of multiple beta-defensin like homologues in teleost fish. Molecular Immunology, 2007, 44(4): 638-647 DOI:10.1016/j.molimm.2006.01.012 |
Zhu J, Wang H, Wang J, et al. Identification and characterization of a β-defensin gene involved in the immune defense response of channel catfish, Ictalurus punctatus. Molecular Immunology, 2017, 85: 256 DOI:10.1016/j.molimm.2017.03.009 |
Zhang W, Zhu LQ, Zhao FL, et al. Prongress on β-defensin. Progress in Veterinary Medicine, 2006, 27(5): 47-50 [ 张伟, 朱连勤, 赵风立, 等. β-防御素研究进展. 动物医学进展, 2006, 27(5): 47-50 DOI:10.3969/j.issn.1007-5038.2006.05.013] |



