2. 农业农村部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋渔业科学 与食物产出过程功能实验室 青岛 266071
2. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071
三疣梭子蟹(Portunus trituberculatus)是我国海水养殖的主导品种之一,具有重要的经济价值。盐度是三疣梭子蟹养殖中的重要环境因子,直接影响其生长、发育和存活(隋延鸣等, 2012;路允良等, 2012;江山等, 2011;郑萍萍等, 2010;王冲等, 2010;吕富等, 2010)。
三疣梭子蟹最适的生长盐度为28.7(隋延鸣等,2012),我们在实际生产和实验研究中发现,由于暴雨或换水等因素导致养殖水盐度骤降后,不同家系间因耐低盐性状差异导致成活率参差不齐。这一特性为三疣梭子蟹养殖业带来巨大隐患,有可能造成重大的经济损失。另外,我国有广袤的低盐海域,如大江、大河入海口,其海水盐度并不适合三疣梭子蟹的正常生长。因此,耐低盐性状是重要的育种性状,解析低盐适应机理对于培育三疣梭子蟹耐低盐良种具有重要意义。
甲基转移酶基因(DNMT1)是维持基因组甲基化状态的重要基因,该基因具有特异性的N末端结构域和保守的C末端催化结构域,DNA甲基化由DNA甲基转移酶催化而成,DNA甲基化整体水平在一定程度上与甲基转移酶的表达呈现正相关(杨晓丹等, 2012)。本实验室通过三疣梭子蟹转录组测序得到PtDNMT1基因片段,发现该基因在低盐胁迫后的鳃组织中显著差异表达,暗示其在盐度适应发挥重要作用。本研究利用RACE技术,首次克隆了PtDNMT1基因,通过qRT-PCR分析了其在三疣梭子蟹不同组织中的表达水平,并对其在盐度胁迫下的表达变化规律进行了研究,以此探讨DNA甲基化在三疣梭子蟹低盐适应中的功能。
1 材料与方法 1.1 材料实验所需三疣梭子蟹均取自中国水产科学研究院黄海水产研究所实验基地昌邑海丰水产有限公司。实验所用个体为120日龄蟹,所选取三疣梭子蟹均健康有活力,平均体重为(120±10.5) g,在室内养殖池塘(5 m×3 m×2 m)中暂养,暂养期间持续充氧,控制水温为(20±2)℃,pH为7.5。
1.2 盐度胁迫实验随机挑取暂养7 d的三疣梭子蟹分为2组:对照组(正常海水30)、低盐组(12),每个处理组设3个平行,每个平行30只个体。利用自然海水和淡水配制各盐度实验用水,使用YSI盐度仪进行盐度校准(隋延鸣等, 2012)。每天少量投喂饵料,实验期间定时测量海水盐度,每天少量换水,以维持海水盐度的稳定。在低盐胁迫过程中分别取0、3、6、12、24、48和72 h的肝胰腺、鳃和肌肉等组织,每个时间点选取3个个体。样品置于液氮中保存,用于RNA的提取。
1.3 总RNA的提取及cDNA的合成利用TRIzol试剂提取三疣梭子蟹各组织总RNA,通过核酸定量仪检测RNA浓度,用琼脂糖凝胶电泳检测RNA的完整性,检测完成后将其放置于超低温冰箱中保存。利用SMART RACE Amplification Kit合成3'和5' RACE模板。
1.4 cDNA全长的克隆及测序根据本实验室转录组测序所得的PtDNMT1基因的片段,利用Primer Premier 5.0软件设计出2对3'RACE和5'RACE特异性引物,所有引物均由青岛擎科生物有限公司合成,序列等信息见表 1。利用Trizol法提取健康三疣梭子蟹肌肉、鳃、肝胰腺组织的RNA,利用RACE模板合成试剂盒合成三疣梭子蟹RACE模板。3'和5'末端使用Advantage2 PCR Kit扩增,运用巢式PCR方法,第一轮反应使用引物Dt-3'-F,Dt-5'-F,分别与UPM配对,反应程序:95℃ 5 min,95℃ 30 s,63℃ 30 s,75℃ 2 min,30个循环。第二轮反应以第一轮PCR扩增产物为模板,使用引物Dt-3'-R和Dt-5'-R,分别与NUP配对,反应程序:95℃ 5 min,95℃ 30 s,63℃ 30 s,75℃ 2 min,30个循环。利用pMD18-T载体连接目的片段,然后利用DH5α感受态细胞,将连接液转入感受态细胞中,经过37℃活化1 h,然后进行涂板,经过12 h培养,挑菌摇床培养,M13引物检测菌液PCR,挑选所需的阳性单克隆,经过菌落PCR鉴定后,送青岛擎科生物有限公司测序。
|
|
表 1 本研究用到的引物 Tab.1 The primers used in this study |
利用ORF Finder软件(http://www.ncbi.nlm.nih.gov/projects/gorf/orfig.cgi)预测其开放阅读框,利用EditSeq软件预测其氨基酸序列,利用ExPASy软件(http://web.expasy.org/compute_pi/)预测其分子量和等电点,利用NCBI Blastx软件(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列同源性比对,利用NCBI Conserved damain软件(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析该基因的蛋白质功能结构域,从GenBank(https://blast.ncbi.nlm.nih.gov/Blast.cgi)中下载其他物种的DNMT1基因蛋白质序列,利用MAGE 6.0软件构建该基因的NJ系统进化树。
1.6 PtDNMT1组织表达分析挑选出三疣梭子蟹的肌肉、鳃、肝胰腺、血液、卵巢、心脏、胃并提取总RNA(每个组织选取3个个体)。使用PrimeScriptTM RT reagent kit with gDNA Eraser (TaKaRa)将上述组织的RNA反转录成cDNA,放置于-20℃保存。取出不同组织cDNA,根据该基因的开放阅读框(ORF)设计1对荧光定量引物(PtDNMT1-DF、PtDNMT1-DR)以及内参引物(β-actin-F、β-actin-R),使用ABI 7500 Real Time PCR仪和TaKaRa SYBR Premix Ex TaqⅡ试剂分析DNMTI在三疣梭子蟹不同组织中的表达情况。反应体系为10 μl,包括5 μl SYBR Premix Ex TaqⅡ (2×)、0.4 μl正向引物(10 μmol/L)、0.4 μl反向引物(10 μmol/L)、0.2 μl ROX Reference DyeⅡ (50×)、1.0 μl cDNA、另加3.0 μl DEPC水补足10 μl。反应程序:95℃ 30 s; 95℃ 5 s,60℃ 34 s,35个循环; 95℃ 15 s,60℃ 1 min,95℃ 15 s。
2 结果与分析 2.1 三疣梭子蟹PtDNMT1基因cDNA全长的获得和序列分析采用RACE技术扩增获得PtDNMT1基因的全长cDNA,GenBank登录号:MF770486。分析显示,PtDNMT1 cDNA序列全长5919 bp。氨基酸序列分析发现,PtDNMT1包括4832 bp的开放阅读框(ORFL),编码1610个氨基酸,理论等电点为4.68,预测蛋白质的分子量为148.15 kDa。PtDNMT1有2个特殊的结构域,分别是zf -CXXC锌指结构域和甲基转移酶家族特有的Dcm结构域。PtDNMT1基因的cDNA序列全长以及其编码的氨基酸序列见图 1。

|
图 1 三疣梭子蟹PtDNMT1基因cDNA序列全长及其编码的氨基酸序列 Fig.1 Nucleotide sequence and the deduced amino acids sequence of P. trituberculatus PtDNMT1 gene 起始密码子ATG和终止密码子TAA均用粗线方框标出,zf-CXXC锌指结构域和Dcm结构域分别用细线方框和阴影标出 Start codon (ATG) and stop codon (TAA) are marked with thick boxes, and the zf-CXXC and Dcm domain are separately marked with filament box and shadow |
利用DNAMAN软件进行同源性分析,结果显示,PtDNMT1与合浦珠母贝(Pinctada fucata)的DNMT1最为相似,同源性为59%,与斑马鱼(Danio rerio)的同源性为56%,与欧洲熊蜂(Bombus terrestris)的同源性为57%。利用MEGA5.0以邻接法(Neighbor-joining)构建三疣梭子蟹PtDNMT1系统进化树,结果显示,三疣梭子蟹与欧洲熊蜂、芜菁叶蜂进化上关系聚为一支,与昆虫类亲缘关系最近(图 2)。
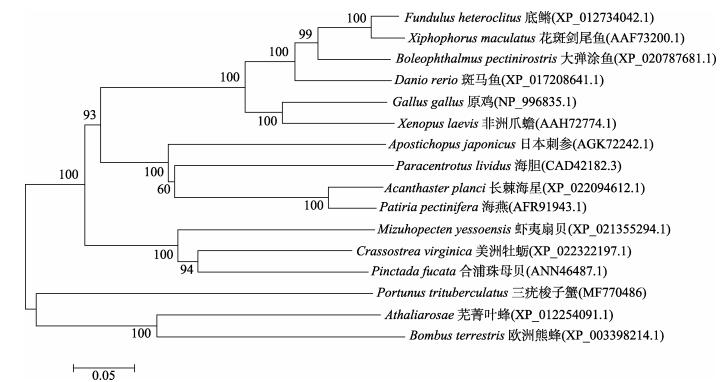
|
图 2 基于PtDNMT1氨基酸序列的NJ进化树 Fig.2 NJ tree based on PtDNMT1 amino acid sequences |
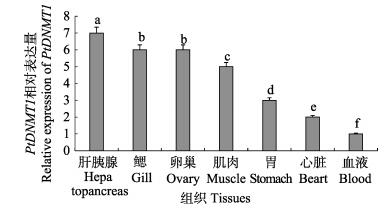
利用qRT-PCR技术分析三疣梭子蟹PtDNMT1基因在肝胰腺、鳃、卵巢、肌肉、胃、心脏和血液组织中的相对表达量(图 3),结果显示,PtDNMT1基因在各种组织中广泛表达,但表达量存在显著差异。其中,在肝胰腺中表达量最高(P < 0.05),鳃和卵巢次之,在血液中表达量最低。
|
图 3 PtDNMT1基因在三疣梭子蟹不同组织中的表达
Fig.3 The expression of PtDNMT1 gene in different tissues of P. trituberculatus
相同字母代表差异不显著(P>0.05), 不同字母代表差异极显著(P < 0.05) Same letters indicated no significant difference (P > 0.05); different letters indicated significant difference (P < 0.05) |
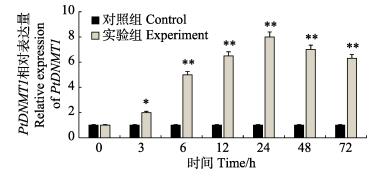
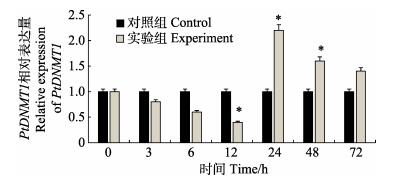
盐度胁迫后鳃组织中PtDNMT1基因呈现显著上调表达趋势,在低盐胁迫后的6 h出现峰值(5.3倍),并极显著高于对照组(P < 0.01)(图 4)。在肝胰腺组织中(图 5),低盐胁迫后6~72 h间一直呈极显著的上调表达,并在低盐胁迫24 h达到最大值(8倍)。在肌肉组织中(图 6),低盐胁迫后PtDNMT1基因表达开始出现降低趋势,但降低趋势较为缓慢,在低盐胁迫24 h出现上升趋势,并在24 h表达量达到峰值(2.2倍)。

|
图 4 低盐胁迫后三疣梭子蟹PtDNMT1基因在鳃组织中的表达 Fig.4 The expression of PtDNMT1 in gills of P. trituberculatus during low salinity stress *代表同一时间实验组与对照组差异显著(P < 0.05);**代表同一时间实验组与对照组差异极显著(P < 0.01),下同 * indicated significant difference (P < 0.05); ** indicated highly significant difference (P < 0.01). The same as below |
|
图 5 低盐胁迫后三疣梭子蟹PtDNMT1基因在肝胰腺组织中的表达 Fig.5 The expression of PtDNMT1 in the hepatopancreas of P. trituberculatus during low salinity stress |
|
图 6 低盐胁迫后三疣梭子蟹PtDNMT1基因在肌肉组织中的表达 Fig.6 The expression of PtDNMT1 in the muscle of P. trituberculatus during low salinity stress |
甲基转移酶基因(DNMT1)是维持基因组甲基化状态的重要基因,DNA甲基化水平在一定程度上与甲基转移酶表达呈现正相关(杨晓丹等, 2012)。本研究克隆获得了三疣梭子蟹PtDNMT1基因cDNA全长序列,发现PtDNMT1有2个特殊的结构域,分别是zf-CXXC锌指结构域和甲基转移酶家族特有的Dcm结构域。
有关三疣梭子蟹盐度适应研究主要集中于国内,目前已查明了盐度耐受范围以及最适生长盐度范围,明确盐度胁迫对胚胎、幼体发育、摄食、免疫等的影响(隋延鸣等, 2012;路允良等, 2012;江山等, 2011;郑萍萍等, 2010;王冲等, 2010;吕富等, 2010)。由于研究基础相对薄弱和缺少基因组信息,其盐度适应机制研究开展较晚,一些学者通过候选基因法筛选具代表性的离子转运酶基因并分析其在盐度适应中的功能(马金武等, 2016);覃烨等(2012)利用芯片技术发掘417条盐度胁迫差异基因; 吕建建等(2013)通过高通量转录组研究发掘了1705条盐度胁迫后差异表达的基因。并在此基础上克隆了水通道蛋白、氯离子通道蛋白等重要离子转运基因(王瑜等, 2014)。然而,总体而言,三疣梭子蟹盐度适应机制的研究仍处于起始阶段,盐度适应相关基因的发掘及验证均主要在转录水平上开展。
DNA甲基化是表观遗传学最为重要的一种调控方式,相关研究在脊椎动物和植物中广泛开展(李娜等, 2012;周翠兰等, 2005;黄韫宇等, 2013),而在甲壳动物中少有报道。DNA甲基化在水产动物生长、发育、性逆转以及环境适应中发挥举足轻重的作用(何暮春等, 2018)。研究发现,虾夷扇贝(Patinopecten yessoensis)生长和耐热性状的累代选育会导致DNA甲基化水平和模式的改变(吴彪等, 2015);尼罗罗非鱼(Oreochromis niloticus)耐寒品系的总甲基化水平、全甲基化和半甲基化水平较对照组均存在一定差异(朱华平等, 2013);尤为值得注意的是,经氨氮和pH胁迫后的中国对虾(Fenneropenaeus chinensis)体内整体甲基化水平呈显著上升趋势(杜盈等, 2013),且随着胁迫时间的延长,体内甲基化水平会相应出现持续升高的现象,暗示甲基化修饰在甲壳动物逆境胁迫中具有重要作用。然而,DNA甲基化修饰在甲壳动物盐度适应中是否也具有一定功能,尚未见相关研究报道。
本研究发现,PtDNMT1基因在所检测的7个组织中均有表达,但在不同的组织中相对表达量存在差异,其中,在肝胰腺中表达量最高,在鳃、卵巢和肌肉组织中表达次之,这与张蕾等(2010)在鲫鱼(Carassius auratus)中的研究结果基本一致。鳃、肝胰腺和肌肉是三疣梭子蟹体进行盐度适应的主要器官(Rosa et al, 2014),而PtDNMT1基因在以上3个组织中均有较高表达,暗示DNA甲基化可能在三疣梭子蟹盐度适应中发挥一定的作用。
为了继续探索DNA甲基化在三疣梭子蟹盐度适应中的作用机制,我们分析了低盐胁迫过程中PtDNMT1基因在鳃、肝胰腺和肌肉组织中的表达变化规律。鳃是三疣梭子蟹最重要的渗透压调节器官之一,位于鳃上皮细胞的离子转运基因具有维持体内外渗透压和离子平衡的重要作用(马金武等, 2016)。本研究发现,低盐胁迫6 h时鳃组织中PtDNMT1基因的表达即达到峰值(8倍),并一直持续到12 h (6倍),随后逐步下降,在72 h时仍显著高于对照组(3倍)。马金武等(2016)研究发现,盐度胁迫12 h后其血液渗透压和主要离子浓度均达到了相对稳定的水平,暗示盐度胁迫后的0~12 h是三疣梭子蟹进行体内外渗透压和离子平衡的重要时期。PtDNMT1基因在盐度胁迫后6~12 h间极显著上调表达,暗示DNA甲基化修饰在三疣梭子蟹平衡体内外渗透压的关键时期发挥重要的功能。
甲壳动物进行主动的渗透压调节需要消耗大量能量,而肝胰腺是重要的消化和能量供给器官(Wang et al, 2014)。本研究发现,盐度胁迫后PtDNMT1基因在肝胰腺中的表达规律非常类似于在鳃中的表达,只是其达到峰值的时间稍晚于鳃(24 h),且上调倍数相对高于鳃,在胁迫后6~72 h间一直极显著的上调表达。推测低盐胁迫过程需要肝胰腺提供大量能量用于鳃器官进行渗透压调节,而DNA甲基化在调控肝胰腺能量供给过程中发挥重要功能。
自由氨基酸(FAA)途径也是甲壳动物进行渗透压调节的重要方式,甲壳动物的肌肉组织是FAA发挥渗透压调节的主要场所(付萍等, 2017)。低盐胁迫后,PtDNMT1基因在肌肉中的表达最初呈现下调趋势,但降低趋势并不明显且较为缓慢。在低盐胁迫24 h后,该基因上调表达至峰值(2.2倍),且上调趋势一直持续至72 h。相对于鳃中的表达情况,其在肌肉中的表达峰值相对滞后,且上调表达倍数亦相对较低。研究结果显示,DNA甲基化在FAA中具有一定功能,然而根据上调幅度推测,其发挥的调控功能相对弱于在鳃和肝胰腺中所发挥的功能。
本研究首次克隆了三疣梭子蟹PtDNMT1基因cDNA全长序列,分析了其在三疣梭子蟹不同组织、低盐胁迫不同时期的表达情况,暗示PtDNMT1基因可能在三疣梭子蟹盐度适应中发挥重要作用,为进一步深入解析DNA甲基化在三疣梭子蟹盐度适应中的分子机制提供了依据。
Du Y, He YY, Li J, et al. MSAP analysis of genomic DNA in the tissues of wild and "Huanghai No.1" Fenneropenaeus chinensis. Journal of Fishery Sciences of China, 2013, 8(3): 536-543 [ 杜盈, 何玉英, 李健, 等. 野生和"黄海1号"中国明对虾不同组织基因组DNA的MSAP分析. 中国水产科学, 2013, 8(3): 536-543] |
Fu P, Lü JJ, Liu P, et al. Effects of different salinity level on free amino acid composition in muscle and hemolymph of the swimming crab Portunus trituberculatus. Journal of Fisheries of China, 2017, 41(3): 374-381 [ 付萍, 吕建建, 刘萍, 等. 盐度胁迫对三疣梭子蟹肌肉和血淋巴中游离氨基酸含量的影响. 水产学报, 2017, 41(3): 374-381] |
He MC, Zhang JY, Liu CL, et al. DNA methylation level and pattern in growing sepia esculenta hoyle. Progress in Fishery Sciences, 2018, 39(4): 46-55 [ 何暮春, 张金勇, 刘长琳, 等. 金乌贼生长期DNA甲基化水平和模式. 渔业科学进展, 2018, 39(4): 46-55] |
Huang WY, Zhang HJ, Xing YX, et al. Effects of NaCl stress on seed germination and DNA methylation status detected by MSAP analysis in cucumber. Scientia Agricultura Sinica, 2013, 46(8): 1646-1656 [ 黄韫宇, 张海军, 邢燕霞, 等. NaCl胁迫对黄瓜种子萌发的影响及DNA甲基化的MSAP分析. 中国农业科学, 2013, 46(8): 1646-1656 DOI:10.3864/j.issn.0578-1752.2013.08.014] |
Jiang S, Xu QH. Influence of salinity stress on the activity of gill Na+/K+-ATPase in swimming crab (Portunus trituberculatus). Journal of Fisheries of China, 2011, 35(10): 1475-1480 [ 江山, 许强华. 盐度胁迫对三疣梭子蟹鳃Na+/K+-ATPase活力的影响. 水产学报, 2011, 35(10): 1475-1480] |
Li N, Zhang Y, Xie LN, et al. Research progress in DNA methylation in plants. Plant Physiology Journal, 2012, 48(11): 1027-1036 [ 李娜, 张旸, 解莉楠, 等. 植物DNA甲基化研究进展. 植物生理学报, 2012, 48(11): 1027-1036] |
Lu YL, Wang F, Zhao ZY, et al. Effects of salinity on growth, molt and energy utilization of juvenile swimming crab Portunus trituberculatus. Journal of Fishery Sciences of China, 2012, 19(2): 237-245 [ 路允良, 王芳, 赵卓英, 等. 盐度对三疣梭子蟹生长、蜕壳及能量利用的影响. 中国水产科学, 2012, 19(2): 237-245] |
Lü F, Huang JT, Yu YB, et al. Effects of salinity on growth, muscle composition, and protease activity of Portunus trituberculatus. Transactions of Oceanology and Limnology, 2010(4): 137-142 [ 吕富, 黄金田, 於叶兵, 等. 盐度对三疣梭子蟹生长、肌肉组成及蛋白酶活性的影响. 海洋湖沼通报, 2010(4): 137-142 DOI:10.3969/j.issn.1003-6482.2010.04.020] |
Lü J, Liu P, Wang Y, et al. Transcriptome analysis of Portunus trituberculatus in response to salinity stress provides insights into the molecular basis of osmoregulation. PLoS One, 2013, 8(12): e82155 DOI:10.1371/journal.pone.0082155 |
Ma JW, Lü JJ, Liu P, et al. Na+/H+-exchanger in swimming crab (Portunus trituberculatus): Cloning, characterization and mRNA expression under salinity stress. Acta Hydrobiologica Sinica, 2016, 40(5): 902-906 [ 马金武, 吕建建, 刘萍, 等. 三疣梭子蟹Na+/H+-exchanger基因克隆鉴定及在盐度胁迫下的表达分析. 水生生物学报, 2016, 40(5): 902-907] |
Rosa R, Nunes M. Biochemical changes during the reproductive cycle of the deep-sea decapod Nephrops norvegicus on the south coast of Portugal. Marine Biology, 2002, 141(6): 1001-1009 DOI:10.1007/s00227-002-0911-9 |
Sui YM, Gao BQ, Liu P, et al. The tolerance to and optimal salinity for growth in swimming crab Portunus trituberculatus "Huangxuan No.1". Journal of Dalian Fisheries University, 2012, 27(5): 398-401 [ 隋延鸣, 高保全, 刘萍, 等. 三疣梭子蟹"黄选1号"盐度耐受性及适宜生长盐度分析. 大连海洋大学学报, 2012, 27(5): 398-401 DOI:10.3969/j.issn.2095-1388.2012.05.003] |
Sui YM, Gao BQ, Liu P, et al. Preliminary studies on the salinity tolerance of "Huangxuan No.1" Portunus trituberculatus. Progress in Fishery Sciences, 2012, 33(2): 63-68 [ 隋延鸣, 高保全, 刘萍, 等. 三疣梭子蟹"黄选1号"盐度耐受性分析. 渔业科学进展, 2012, 33(2): 63-68 DOI:10.3969/j.issn.1000-7075.2012.02.010] |
Tan Y, Xu QH. The prokaryotic expression of recombinant heat shock protein HSP90a of Portunus trituberculatus under salinity stress. Journal of Fisheries of China, 2012, 36(5): 681-684 [ 覃烨, 许强华. 盐度胁迫下三疣梭子蟹热休克蛋白HSP90a的原核表达. 水产学报, 2012, 36(5): 681-684] |
Wang C, Jiang LX, Wang RJ, et al. Effect of abrupt and gradual changes in salinity on development and feeding in juvenile swimming crab (Portunus trituberculatus). Fisheries Science, 2010, 29(9): 510-514 [ 王冲, 姜令绪, 王仁杰, 等. 盐度骤变和渐变对三疣梭子蟹幼蟹发育和摄食的影响. 水产科学, 2010, 29(9): 510-514 DOI:10.3969/j.issn.1003-1111.2010.09.002] |
Wang Y, Lü JJ, Liu P, et al. Cloning and characterization of aquaporins 1 and its expression analysis under salinity stress in Portunus trituberculatus. Journal of Fishery Sciences of China, 2014, 21(5): 893-901 [ 王渝, 吕建建, 刘萍, 等. 三疣梭子蟹水通道蛋白1 cDNA及其盐度胁迫下的表达分析. 中国水产科学, 2014, 21(5): 893-901] |
Wang Y, Lü JJ, Liu P, et al. Cloning and characterization of chloride intracellular channel gene and its expression under low salinity stress in Portunus trituberculatus. Oceanologia et Limnologia Sinica, 2014, 45(6): 1359-1366 [ 王瑜, 吕建建, 刘萍, 等. 三疣梭子蟹胞内氯离子通道蛋白基因克隆及其表达分析. 海洋与湖沼, 2014, 45(6): 1359-1366] |
Wu B, Yang AG. MSAP analysis on genome-wide DNA methylation in selected and wild Japanese scallop Patinopecten yessoensis. Oceanologla et Limnologla Sinica, 2015, 46(4): 976-981 [ 吴彪, 杨爱国. 虾夷扇贝选育群体与野生群体基因组DNA甲基化的MSAP分析. 海洋与湖沼, 2015, 46(4): 976-982] |
Yang XD, Han W, Liu F, et al. DNA methylation in vertebrate embryogenesis. Hereditas, 2012, 34(9): 1108-1113 [ 杨晓丹, 韩威, 刘峰, 等. DNA甲基化与脊椎动物胚胎发育. 遗传, 2012, 34(9): 1108-1113] |
Zhang L, Xie BH, Zhang QY, et al. The cloning and expression analysis of the DNMT1 gene cDNA of Crucian carp. Acta Hydrobiologica Sinica. Journal of Fisheries of China, 2010, 34(2): 230-234 [ 张蕾, 谢冰花, 张琼宇, 等. 鲫鱼DNMT1基因cDNA的克隆及表达分析. 水产学报, 2010, 34(2): 229-235] |
Zheng PP, Wang CL, Song WW, et al. Effect of salinity stress on serum non-specific immune factors in swimming crab Portunus trituberculatus. Fisheries Science, 2010, 29(11): 634-638 [ 郑萍萍, 王春琳, 宋微微, 等. 盐度胁迫对三疣梭子蟹血清非特异性免疫因子的影响. 水产科学, 2010, 29(11): 634-638 DOI:10.3969/j.issn.1003-1111.2010.11.002] |
Zhou CL, Yin YF, Zhang J, et al. Biological implications of DNA methylation and DNA methylation assays. Journal of Nanhua University (Medical Edition), 2005, 33(2): 148-152 [ 周翠兰, 殷宇芳, 张佳, 等. DNA甲基化的生物学意义及其检测方法. 南华大学学报(医学版), 2005, 33(2): 148-153 DOI:10.3969/j.issn.2095-1116.2005.02.003] |
Zhu HP, Lu MX, Huang ZH, et al. Effect of low temperature on genomic DNA methylation in Nile tilapia (Oreochromis niloticus). Journal of Fisheries of China, 2013, 37(10): 1460-1467 [ 朱华平, 卢迈新, 黄樟翰, 等. 低温对罗非鱼基因组DNA甲基化的影响. 水产学报, 2013, 37(10): 1460-1467] |



