2. 中国水产科学研究院 农业农村部水生动物基因组学重点实验室 渔业生物技术北京市重点实验室 北京 100141;
3. 中国水产科学研究院黑龙江水产研究所 哈尔滨 150070;
4. 四川农业大学动物科技学院 雅安 625014;
5. 厦门大学海洋与地球学院 福建省海洋生物资源开发利用协同创新中心 厦门 361102
2. Key Laboratory of Aquatic Genomics, Ministry of Agriculture and Rural Affairs, Beijing Key Laboratory of Fishery Biotechnology, Chinese Academy of Fishery Sciences, Beijing 100141;
3. Heilongjiang River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Haerbin 150070;
4. College of Animal Science and Technology, Sichuan Agricultural University, Yaan 625014;
5. Fujian Collaborative Innovation Center for Exploitation and Utilization of Marine Biological Resources, College of Ocean and Earth Sciences, Xiamen University, Xiamen 361102
鲑鳟是鲑科(Salmonidae)多种鱼类的统称,是冷水鱼主要养殖品种,在中高端水产品市场中占据了重要份额。我国有良好的冷水资源以及多样化的鲑鳟养殖品种,除年产量最高的虹鳟(Oncorhynchus mykiss)外,还有银鲑(Oncorhynchus kisutch) (又称“银大麻哈鱼”)、山女鳟(Oncorhynchus masou masou)、美洲红点鲑(Salvelinus fontinalis)(商品名“七彩鲑”)、白斑红点鲑(商品名“白点鲑”)(Salvelinus leucomaenis)等均已形成了一定的养殖规模(孙大江等, 2010; 户国等, 2012)。
由于各种鲑鳟养殖品种对生境的要求类似,我国鲑鳟养殖的地域比较集中,往往同一育苗场或养殖场同时养殖多种鲑科鱼类,这些养殖物品种主要集中于大麻哈鱼属(Oncorhynchus)和红点鲑属(Salvelinus),但除虹鳟外,其他鲑鳟养殖种现有的基因组资源和遗传分析工具十分有限,以往的遗传分析主要基于线粒体DNA(Yu et al, 2010)和微卫星标记(Small et al, 2010; Naish et al, 2013)来开展,通常标记数量较少,基因组覆盖度较低,并且针对不同的物种需要开发不同的标记,通用性较低。
本研究基于虹鳟57K单核苷酸多态性(Single nucleotide polymorphism, SNP)芯片(Palti et al, 2015),致力于开发常见鲑鳟养殖种间通用的分子标记,形成小型SNP芯片,从而利用同一套分子标记和检测方法,实现对多个鲑鳟养殖物种的遗传分析,将有利于显著降低遗传检测成本、简化技术人员培训流程、提升科学养殖管理效率。
1 材料与方法 1.1 实验材料山女鳟、美洲红点鲑、白斑红点鲑采自中国水产科学研究院黑龙江水产研究所渤海冷水性鱼试验站,银鲑采自四川省都江堰市三文鱼养殖基地和中国水产科学研究院房山试验基地。每个群体或家系各采样20尾,剪取尾鳍贴于干燥的滤纸上,56℃烘干6 h,常温保存。
1.2 DNA提取使用海洋动物基因组DNA提取试剂盒(天根生化)提取基因组DNA,1%琼脂糖凝胶电泳检测DNA完整性,NanoDrop 8000超微量紫外可见光分光光度计(Thermo Fisher)检测DNA纯度和浓度。
1.3 高通量SNP芯片分型对山女鳟、美洲红点鲑、白斑红点鲑、银鲑4个群体进行线粒体控制区测序,检测个体单倍型,每个群体选取DNA质量高、具有不同单倍型的8个样本用于57K虹鳟SNP芯片(Affymetrix)分型,分别记为OK1~8(银鲑),OM1~8(山女鳟),SF1~8(美洲红点鲑),SL1~8(白斑红点鲑)。分型实验由纽勤生物科技(上海)有限公司完成,使用SNPolisher软件(Affymetrix)读取分型数据。使用PLINK 1.09(http://pngu.mgh.harvard.edu/purcell/plink/)(Purcell et al, 2007)进行分型结果过滤、统计和连锁不平衡(Linkage disequilibrium, LD)检验,个体质控标准设为Call rate (CR) > 80%,位点质控标准设为CR > 97%。
1.4 低通量SNP芯片分型96个SNP位点由Fluidigm公司设计合成探针,使用EP1平台及96.96动态芯片(Fluidigm)开展分型检测。检测样本共计96尾,包括进行过高通量SNP芯片检测的样本32尾,以及新增银鲑样本64尾,其中,新增样本包含银鲑养殖家系6个,每个家系样本各8尾(记为OK9~56),以及养殖家系候选亲本16尾(记为OK57~72),其中,部分家系互为半同胞,其真实亲本为5尾雌鱼和3尾雄鱼。通过Fluidigm SNP genotyping analysis software 4.1软件读取分型结果,质控阈值设为85,对缺失和低质量分型数据进行人工复检。使用Cervus 3.0.7进行遗传多态性统计,并对48尾银鲑样本(OK9~56)进行亲权鉴定(Kalinowski et al, 2007)。使用Structure 2.3.4 (http://pritch.bsd.uchicago.edu/structure.html),对32尾鲑鳟群体样本(OK1~8、OM1~8、SF1~8和SL1~8)进行群体遗传结构分析(Falush et al, 2003; Hubisz et al, 2009)。
2 结果 2.1 虹鳟高通量SNP芯片在国内代表性鲑鳟养殖群体中的多态性分析使用虹鳟57K SNP芯片,对国内4个代表性鲑鳟养殖群体样本进行分型检测,包括大麻哈鱼属的山女鳟、银鲑,以及红点鲑属的美洲红点鲑和白斑红点鲑,每个群体选取8尾个体。该芯片共包含虹鳟多态性位点57, 501个,32尾个体全部通过CR > 80%的质控标准,即每尾个体分型成功的位点数均大于80%。位点质控标准设为CR > 97%,即仅统计基因型无缺失的位点。各群体多态性位点统计结果如表 1所示,其中,群体间分型位点比例波动较小,在65.96%~74.19%范围内,但多态性位点比例差异很大,银鲑群体内仅有9.01%的多态性位点,而美洲红点鲑群体内多态性位点比例达到45.99%。
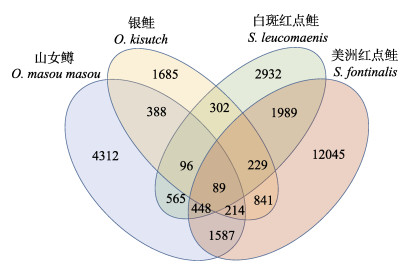
2.2 鲑鳟群体间共享多态性位点筛选图 1以韦恩图的形式展示了4个鲑鳟群体间多态性SNP位点的分布,包括群体间共享的和群体特异性的多态位点,其中,4个群体共享的多态性位点有89个。为了避免基因组内紧密连锁的位点同时入选,在32尾鲑鳟个体中对共享多态性位点进行LD检验,剔除R2值高于0.3的位点4个。根据共享多态性位点侧翼序列设计Fluidigm分型探针,进一步剔除探针设计失败位点6个,获得用于构建鲑鳟通用SNP芯片的共享多态性位点79个。由于位点数目不足96个,从山女鳟、美洲红点鲑和白斑红点鲑3个群体共享的448个多态性位点中选取补充位点17个,选择标准为:经LD检验,与已入选位点间R2值低于0.3,并能够成功设计分型探针。96个入选位点的侧翼序列及基因组定位信息见表 1。最终入选的芯片位点中,编号OS-79及以前的位点,为4群体共享多态位点,编号OS-80及以后的位点,为3群体共享多态位点。
|
图 1 国内4个代表性鲑鳟养殖群体的多态性SNP位点韦恩图 Fig.1 Venn diagram for distribution of shared polymorphic SNPs among 4 representative salmonid populations in China |
|
|
表 1 鲑鳟代表性群体多态性SNP位点数目和比例 Tab.1 Number and percentage of polymorphic SNPs in representative Salmonid populations |
使用构建的鲑鳟通用型低通量SNP芯片对96尾鲑鳟养殖个体进行分型检测,在总计9216个分型反应中,分型成功率为97.48%,检测样本中,32尾个体与高通量芯片检测样本相同,其检测结果一致性为96.55%。各位点遗传多态性统计结果见表 2。
|
|
表 2 鲑鳟通用型低通量SNP芯片位点遗传多态性统计 Tab.2 Genetic polymorphism of markers in the salmonid 96 SNP array |
基于鲑鳟通用型低通量SNP芯片分型数据,应用Cervus 3.0.7软件对来自6个家系的48尾银鲑个体进行亲权鉴定,将同批次检测的其他48尾鲑鳟样本均作为待分析亲本群体,结果表明,无论是单亲本鉴定还是双亲本鉴定,基于96个位点分型数据的亲权鉴定结果均与真实系谱相符。
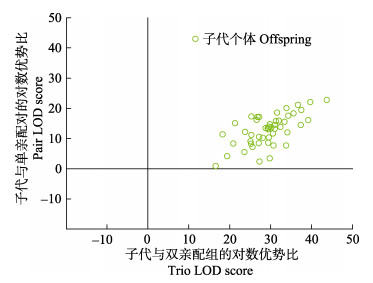
根据表 2中列出的各位点第一亲本非排除率(Non-exclusion probability for first parent, NE-1P)和双亲非排除率(Non-exclusion probability for parent pair, NE-PP),计算得该芯片用于单亲本亲权鉴定的NE-1P值为4.120×10-4,用于双亲本亲权鉴定的NE-PP值为6.219×10-12。图 2展示了该芯片用于48尾银鲑子代亲权鉴定的对数优势比(Logarithm of odds, LOD)值,由图 2可见,所有子代样本的检测结果都落在可靠象限内。
|
图 2 48尾银鲑家系样本的亲权鉴定准确性分析 Fig.2 Parent assignments analysis for 48 O. kisutch offsprings 亲权鉴定基于鲑鳟通用型低密度SNP芯片分型数据。每个点代表一尾子代个体。横坐标表示双亲本亲权鉴定数据,纵坐标表示单亲本亲权鉴定数据 The parent assignments analysis was based on genotyping data determined by the salmonid 96 SNP array. Each dot represents one offspring. Horizontal ordinate indicates data for parent pair assignment. Longitudinal ordinate indicates data for single parent assignment |
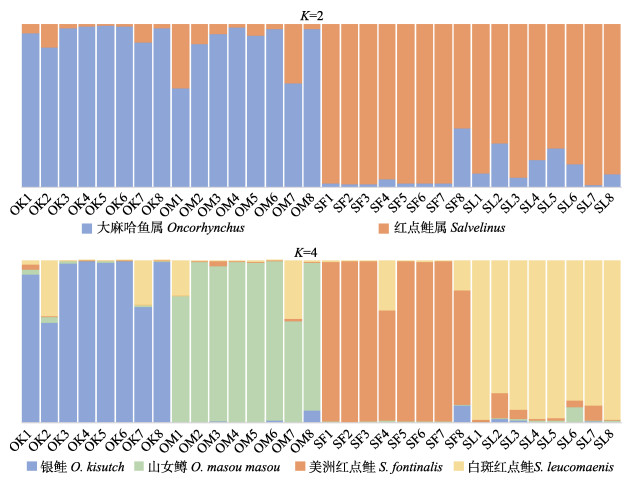
基于鲑鳟通用型低通量SNP芯片分型数据,应用Structure软件对来自国内4个代表性鲑鳟养殖群体的32尾个体开展遗传结构分析。图 3展示了假定祖源群体数(K)分别为2和4时的Structure遗传结构图,图中不同颜色代表不同的祖源成分。
|
图 3 国内4个代表性鲑鳟养殖群体遗传结构分析 Fig.3 Structure analysis for 4 representative salmonid populations in China 遗传结构分析基于鲑鳟通用型低密度SNP芯片分型数据。每种颜色代表一种假定的祖源成分。每条柱形代表一尾个体 The structure analysis was based on genotyping data determined by the salmonid 96 SNP array. Each color represents one putative ancestral fraction. Each bar represents one individual |
K=2的假设下,所有样本以属为分类阶元进行聚类,而K=4的假设下,样本以种为分类阶元进行聚类。每尾样本中占主导地位的遗传组分与个体所属的分类阶元相符,同时包含少量其他遗传组分,2组分析结果都显示,银鲑样本OK2、OK7,山女鳟样本OM1、OM7,美洲红点鲑样本SF8、白斑红点鲑样本SL2、SL6为非主导遗传组分占比较高的个体。
3 讨论各种鲑科鱼类物种间亲缘关系较近,基因组序列相似性较高,因而具有大量共享的分子标记。梁利群等(2004)从50个虹鳟微卫星标记中筛选出12个在黑龙江乌苏里白鲑(Coregonus ussuriensis)自然群体中具有多态性的标记用于遗传多样性分析。鲁翠云等(2016)从150个虹鳟微卫星标记中筛选出48个在白斑红点鲑养殖群体中具有多态性的标记,并开展了群体遗传结构分析。目前,虹鳟的全基因组测序已经完成,并开发了多种通量的SNP分型芯片等遗传分析工具,建立了基于基因组信息的现代化育种、育苗体系。本研究基于这些新开发的虹鳟基因组信息资源,开展了共享标记筛查,使用虹鳟高通量SNP芯片,在国内代表性鲑鳟养殖群体中对57, 501个标记进行了分型检测。
结果显示,在跨物种检测中,标记分型成功比例和多态比例都明显低于虹鳟样本。在相同的质控标准(CR > 97%)下,该芯片在不同虹鳟群体中的分型位点数为49, 299~49, 468个(Palti et al, 2015),而在其他鲑鳟养殖物种中的分型位点数仅为37, 926~42, 658个;各虹鳟群体中多态性位点比例在21.5%~92.8%范围内波动(Palti et al, 2015),而在其他鲑鳟养殖群体中的多态性位点比例仅为9.01%~45.99%。但由于SNP标记的总量巨大,基因组覆盖度高,在基因组编码区和非编码区都有大量分布,这些物种间共享的SNP仍能提供较为丰富的遗传多态信息,鲤高通量SNP芯片也曾被成功应用于鲫(Carassius auratus)、草鱼(Ctenopharyngodon idellus)等8种近缘鱼类遗传分析中(Xu et al, 2014)。由表 1可见,跨物种分型成功位点数目与目标物种间的亲缘关系相关,山女鳟、银鲑与虹鳟同属于大麻哈鱼属,亲缘关系较近,因此,分型位点数略高于美洲红点鲑和白斑红点鲑;而多态性位点数目主要取决于群体本身的遗传多样性,虽然银鲑与虹鳟亲缘关系更近,但其多态位点比例远低于与虹鳟亲缘关系较远的美洲红点鲑,表明采样的银鲑群体遗传多样性较低,亟待改善。
Fluigidm低通量分型平台有48.48和96.96两种芯片,分别适用于48和96个位点的同时分型。Xu等(2017)使用48.48芯片构建了鲤(Cyprinus carpio)低密度SNP芯片,该芯片在用于家系亲权鉴定时的准确率为94%。Liu等(2016)构建虹鳟低密度SNP芯片则使用了96.96芯片,结果表明,当使用其中68个位点开展家系亲权鉴定时,结果准确率为100%,仅使用48个位点时,准确率为99.2%,当位点数下降到36个时,准确率降至92.5%。Harlizius等(2011)就SNP芯片在杜洛克猪商品群体亲权鉴定中的应用开展过更为细化的统计分析,当候选亲本中仅包含真实亲本(n=66)时,60个SNP位点即可实现100%准确的亲权鉴定,而当候选亲本中混入了大量干扰个体(n=304)时,则需要80个SNP位点来实现100%准确的亲权鉴定。鉴定的准确性受到子代样本数目、候选亲本数目,以及群体遗传结构、分型位点在群体中的多态性等多种因素影响,而本研究试图构建多物种通用的SNP芯片,还需要考虑部分位点可能在待测物种或群体中分型失败的可能性,因此,采用96.96芯片,通过检测略高于必要数目的SNP位点,来保障分析结果的可靠性。
本研究经过高通量SNP芯片分型筛查,获得4个鲑鳟养殖群体共享的多态性标记共89个,涵盖了大麻哈鱼属和红点鲑属各2个物种,推测这些位点在鲑科常见养殖物种中都具有较高的通用性,可以作为鲑鳟通用型SNP芯片构建的候选位点。考虑到共享位点筛查所用的银鲑群体遗传多样性较低,可能导致多态性位点发掘不充分,从其他3个群体共享的多态性位点中选取了补充位点,补足96个位点用于芯片构建。
为了验证所构建的低通量SNP芯片分型结果准确性,使用该芯片对高通量SNP芯片检测过的32尾样本进行了重复检测,结果一致性为96.55%,不一致的位点主要表现为分型失败,结果缺失。由此可见,Fluidigm低通量SNP芯片在鲑鳟样本检测中的分型准确性较高。
高通量芯片分型结果显示,OK1~8样本所属的银鲑群体遗传多样性不足,因此,在低通量芯片样本选择时,从另一个银鲑养殖群体中采集了亲本及子代样本共计64尾,即OK9~72,以验证该芯片在银鲑中的适用性。分型结果显示,初筛银鲑群体中不具有多态性的17个位点中,有12个在新的银鲑样本中表现出多态性,证明芯片的位点选择是成功的,入选位点在鲑科不同物种中均具有较高的多态性。
虹鳟低通量芯片的研究结果显示(Liu et al, 2016),在用于亲权鉴定时,Cervus软件的准确率略高于其他同类软件。将同批次分型的其他鲑鳟样本均作为候选亲本,应用Cervus 3.0.7软件开展亲权鉴定,结果表明,基于该芯片分型结果能够准确重现复杂家系的真实系谱。亲权鉴定的准确性通过LOD值来反映,表示假设为真与假设为假的概率之比的Log10对数值,当LOD值为0时,假设成立的概率为50%,当LOD为正值时,认为假设成立,即存在亲子关系。由图 2可见,多数子代个体与软件预测的亲本间配对或配组的LOD值均远大于零,特别是双亲鉴定时,所有配组的LOD值均高于18以上,表明亲权鉴定结果准确性很高。
除系谱鉴定外,该芯片还能够应用于群体遗传资源分析,如图 3中的示例,基于96个SNP位点的基因频率分布,反映群体间的遗传组分构成和遗传关系,筛查群体内部具有独特遗传组成的离群个体。在水产养殖业中,准确的系谱鉴定为科学制种和育种方案的规划奠定了基础(李东宇等, 2016),基于分子标记的群体遗传分析还可应用于种质资源评估(王军等, 2018)、增殖放流效果评估(司飞等, 2017)等多个领域,因此,本研究构建的鲑鳟通用型低密度SNP具有较为广泛的应用前景,能够为鲑鳟制种、育种和引种等科学决策提供基因组信息参考。
Falush D, Stephens M, Pritchard JK. Inference of population structure using multi locus genotype data: Linked loci and correlated allele frequencies. Genetics, 2003, 164(4): 1567-1587 |
Harlizius B, Lopes MS, Duijvesteijn N, et al. A single nucleotide polymorphism set for paternal identification to reduce the costs of trait recording in commercial pig breeding. Journal of Animal Science, 2011, 89(6): 1661-1668 DOI:10.2527/jas.2010-3347 |
Hu G, Gu W, Bai QL, et al. Research advances in genetics and breeding in salmonids. Chinese Journal of Fisheries, 2012, 25(3): 58-62 [ 户国, 谷伟, 白庆利, 等. 主要养殖鲑科鱼类遗传育种的研究进展. 水产学杂志, 2012, 25(3): 58-62 DOI:10.3969/j.issn.1005-3832.2012.03.013] |
Hubisz MJ, Falush D, Stephens M, et al. Inferring weak population structure with the assistance of sample group information. Molecular Ecology Resources, 2009, 9(5): 1322-1332 DOI:10.1111/men.2009.9.issue-5 |
Kalinowski ST, Taper ML, Marshall TC. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 2007, 16(5): 1099-1106 DOI:10.1111/j.1365-294X.2007.03089.x |
Li DY, Kong J, Meng XH, et al. Development of multiplex PCR systems of microsatellite markers for Pacific white shrimp (Litopenaeus vannamei) and its application for parentage identification. Progress in Fishery Sciences, 2016, 37(3): 58-67 [ 李东宇, 孔杰, 孟宪红, 等. 凡纳滨对虾(Litopenaeus vannamei)微卫星多重PCR体系的建立及其在家系亲权鉴定中的应用. 渔业科学进展, 2016, 37(3): 58-67] |
Liang LQ, Chang YM, Dong CZ. Analysis of genetic diversity for Coregonus usssruensis Berg in Heilongjiang River. Journal of Fishery Sciences of China, 2004, 11(6): 501-505 [ 梁利群, 常玉梅, 董崇智. 黑龙江乌苏里白鲑遗传多样性分析. 中国水产科学, 2004, 11(6): 501-505 DOI:10.3321/j.issn:1005-8737.2004.06.003] |
Liu S, Palti Y, Gao G, et al. Development and validation of a SNP panel for parentage assignment in rainbow trout. Aquaculture, 2016, 452: 178-182 DOI:10.1016/j.aquaculture.2015.11.001 |
Lu CY, Cao DC, Li YF, et al. Evaluation of genetic potential of Siberian char Salvelinus leucomaenis cultured population using microsatellite markers from rainbow trout Oncorhynchus mykiss. Chinese Journal of Fisheries, 2016, 29(1): 1-7 [ 鲁翠云, 曹顶臣, 李永发, 等. 利用虹鳟微卫星标记评价白斑红点鲑养殖群体的遗传结构. 水产学杂志, 2016, 29(1): 1-7 DOI:10.3969/j.issn.1005-3832.2016.01.001] |
Naish KA, Phillips RB, Brieuc MS, et al. Comparative genome mapping between Chinook salmon (Oncorhynchus tshawytscha) and rainbow trout (O. mykiss) based on homologous microsatellite loci. G3 (Bethesda), 2013, 3(12): 2281-2288 DOI:10.1534/g3.113.008003 |
Palti Y, Gao G, Liu S, et al. The development and character- rization of a 57K single nucleotide polymorphism array for rainbow trout. Molecular Ecology Resources, 2015, 15(3): 662-672 DOI:10.1111/1755-0998.12337 |
Purcell S, Neale B, Todd-Brown K, et al. PLINK: A tool set for whole-genome association and population-based linkage analyses. American Journal of Human Genetics, 2007, 81(3): 559-575 DOI:10.1086/519795 |
Si F, Sun ZH, Yu SS, et al. Analysis of the genetic variability of the captured population of Japanese flounder Paralichthys olivaceus in the coastal releasing area of Qinhuangdao. Progress in Fishery Sciences, 2017, 38(6): 18-24 [ 司飞, 孙朝徽, 于姗姗, 等. 秦皇岛近海增殖放流区牙鲆(Paralichthys olivaceus)回捕群体的遗传多样性分析. 渔业科学进展, 2017, 38(6): 18-24] |
Small MP, Beacham TD, Withler RE, et al. Discriminating coho salmon (Oncorhynchus kisutch) populations within the Fraser River, British Columbia, using microsatellite DNA markers. Molecular Ecology, 2010, 7(2): 141-155 |
Sun DJ, Wang BQ. Aquaculture of salmonids in China. Chinese Journal of Fisheries, 2010, 23(2): 56-63 [ 孙大江, 王炳谦. 鲑科鱼类及其养殖状况. 水产学杂志, 2010, 23(2): 56-63 DOI:10.3969/j.issn.1005-3832.2010.02.014] |
Wang J, Wang QY, Kong J, et al. SSR analysis on genetic diversity in breeding and wild populations of Fenneropenaeus chinensis. Progress in Fishery Sciences, 2017, 38(6): 18-24 |
王军, 王清印, 孔杰, 等. 中国对虾人工选育群体与野生群体遗传多样性的SSR分析. 渔业科学进展, 2018, 39(2): 1-8 |
Xu J, Feng J, Peng W, et al. Development and evaluation of a high-throughput single nucleotide polymorphism multiplex assay for assigning pedigrees in common carp. Aquaculture Research, 2017, 48(4): 1866-1876 DOI:10.1111/are.13024 |
Xu J, Zhao Z, Zhang X, et al. Development and evaluation of the first high-throughput SNP array for common carp (Cyprinus carpio). BMC Genomics, 2014, 15(1): 307 DOI:10.1186/1471-2164-15-307 |
Yu JN, Azuma N, Yoon M, et al. Genetic structure and phylogeography of masu salmon (Oncorhynchus Masou masou) inferred from mitochondrial and microsatellite DNA Analyses. Zoological Science, 2010, 27(5): 375-385 DOI:10.2108/zsj.27.375 |



