2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食品产出过程功能实验室 青岛 266071;
3. 农业农村部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071;
3. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071
十足目动物是甲壳动物中种类最多(8000~10000种)、进化位阶最高、经济价值最大的一个生物类群(堵南山, 1993)。近年来,随着人们对优质水产品的需求增加,包括龙虾、鳌虾、蟹和对虾等的腹胚亚目和枝鳃亚目在内的十足目动物在水产养殖中的地位日益突出,因此,有必要加强种质鉴定和群体遗传学研究。发展十足目甲壳动物基因组中大量有效的分子标记,可以帮助保护种质资源,改善影响重要性状基因和分子标记辅助基因的识别和开发。作为优良遗传标记的微卫星(Microsatellite)与其他分子标记相比,具有较高稳定性、共显性表达、多态性信息丰富、易于检测等特点,被大量应用于构建遗传图谱(Guo et al, 2017)、基因定位(Zhou et al, 2017)、遗传多样性分析(王日芳等, 2017)及亲缘关系鉴定(李东宇等, 2016)等。
研究各物种基因组特征,第一步需要生成表达序列标签(Expressed sequence tag, EST)信息。EST在物种间高度保守,具有良好的通用性(Varshney et al, 2005),目前已广泛应用于多种植物(Yumurtaci et al, 2017; Singh et al, 2013)和动物(白俊艳等, 2016)。在甲壳类动物如中国明对虾(Fenneropenaeus chinensis) (Wang et al, 2005)、日本沼虾(Macrobrachium nipponense) (周巧, 2013)等中也有报道,例如EST-SSR在EST中的分布频率、大小、引物开发等。
本研究运用生物信息学方法,从GenBank的EST数据库中收集21种十足目甲壳动物EST数据,发掘EST-SSR信息,通过比较不同十足目甲壳动物间EST的特点及不同基元EST-SSR的分布频率和类型,加深对十足目甲壳动物SSR分布规律的认识,为甲壳动物EST-SSR标记的开发及实际应用提供参考。此外,对脊尾白虾(Exopalaemon carinicauda)含有SSR的EST进行GO分析,进一步对脊尾白虾功能基因研究提供重要信息。
1 材料与方法 1.1 数据来源EST数据来源于GenBank(www.nchi.nih.gov)中的dbEST数据库,截止于2017年9月1日。包括21种十足目甲壳动物:美国龙虾(Homarus americanus)、淡水螯虾(Pacifastacus leniusculus)、日本龙虾(Panulirus japonicus)、拟穴青蟹(Scylla paramamosain)、中华绒螯蟹(Eriocheir sinensis)、三疣梭子蟹(Portunus trituberculatus)、克氏原螫虾(Procambarus clarkii)、招潮蟹(Uca pugilator)、红蟹(Gecarcoidea natalis)、珍宝蟹(Metacarcinus magister)、蓝蟹(Callinectes sapidus)、岸蟹(Carcinus maenas)、脆壳蟹(Petrolisthes cinctipes)、脊尾白虾、日本沼虾、罗氏沼虾(Macrobrachium rosenbergii)以及凡纳滨对虾(Litopenaeus vannamei)、斑节对虾(Penaeus monodon)、中国明对虾、日本囊对虾(Marsupenaeus japonicus)、白滨对虾(Litopenaeus setiferus)。物种及EST信息详列于表 1。
|
|
表 1 腹胚亚目EST、Unigenes及SSR在Unigenes序列中的丰度分布 Tab.1 The EST, Unigenes and abundance of SSR in Unigenes of Pleocyemata |
本实验采用EMBOSS软件中的TRIMEST工具,去除尾部包含Poly-A或T的EST序列,利用CROSS_MATCH软件截去过短的序列片段;运行CAP3软件对EST序列进行聚类和拼接处理,获得非冗余EST,包括Contigs和Singlets两类序列(黄启星等, 2012)。
1.3 EST-SSR分析采用自行编写的Perl语言脚本程序对处理后的EST序列进行SSR筛选。筛选标准为二核苷酸重复次数≥6次,三、四、五和六核苷酸重复次数≥5次。同时,筛选重复基元之间没有插入序列的复合型SSR位点(Compound microsatellite),即复合(Ⅰ)型;筛选重复基元之间存在插入序列长度≤100 nt的复合型SSR,即复合(Ⅱ)型。
1.4 脊尾白虾EST的功能注释使用BLAST将Unigenes与NCBI的NR蛋白数据库和Uniprot蛋白数据库进行比对,设置E值为“10-5”(孙政等, 2013),利用Interproscan搜索得到的GO编号对Unigenes按照分子功能、细胞组分和生物学过程进行分类(Ye et al, 2006)。
2 结果与分析 2.1 甲壳动物EST长度及SSR位点数从GenBank数据库下载了21种十足目甲壳动物已发布的EST。由表 1可见,16种腹胚亚目的EST共242178条,碱基总长度约为183.50 Mb。处理后获得85647条非冗余EST序列,包括30120条Contigs和55527条Singlets序列,碱基总长度约为66.72 Mb。从27536条Unigenes序列发掘34601个SSR位点,占EST总数的32.15%。其中,脊尾白虾含有SSR位点的EST最丰富(43.00%),分布频率为868.02个/Mb;拟穴青蟹含有SSR位点的EST所占百分比最小(12.70%),分布频率为286.48个/Mb。
由表 2可见,5种枝鳃亚目的EST共231130条,碱基总长度约为141.28 Mb。获得的59605条Unigenes序列中包括20506条Contigs和39099条Singlets序列,碱基总长度约为37.89 Mb。从5种枝鳃亚目的18475条Unigenes序列中发掘出21911个SSR位点,占EST总数的31.00%。其中,斑节对虾含有SSR位点的EST占总数的百分比最大,为35.49%,分布频率为641.19个/Mb;白滨对虾含有SSR位点的EST所占百分比最小(10.56%),分布频率为166.96个/Mb。
|
|
表 2 枝鳃亚目EST、Unigenes及SSR在Unigenes序列中的丰度分布 Tab.2 The EST, Unigenes and abundance of SSR in Unigenes of Dendrobranchiata |
表 3和表 4列出了十足目甲壳动物不同类型EST-SSR的数量和分布频率。在腹胚亚目和枝鳃亚目EST序列中,复合(Ⅰ)型远高于其他类型,分别占总数的51.38%、65.13%;二核苷酸、三核苷酸和四核苷酸基元的SSR数目相差不多,多于五核苷酸和六核苷酸基元的SSR数目,三者在腹胚亚目中占总数的41.11%,在枝鳃亚目中占总数的28.00%。在2种亚目中,五核苷酸和六核苷酸基元的SSR数目占有很小的比例,在腹胚亚目中分别占总数的3.67%、0.99%,在枝鳃亚目中分别占总数的2.01%、1.33%。克氏原螯虾和淡水鳌虾的EST序列中没有搜索到六核苷酸基元的SSR位点。
|
|
表 3 腹胚亚目不同重复单元EST-SSR的数量和分布频率 Tab.3 The number and frequency of SSR containing different size of repeat unit of EST sequences in Pleocyemata |
|
|
表 4 枝鳃亚目不同重复单元EST-SSR的数量和分布频率 Tab.4 The number and frequency of SSR containing different size of repeat unit of EST sequences in Dendrobranchiata |
表 5和表 6列出了二、三核苷酸基元的SSR在各物种内的数量和分布频率。21种十足目甲壳动物中,各种类型的基元分布频率不一,有的基元甚至没有出现。不同基元的SSR在21种甲壳动物中的分布频率具有很大的差异。16种腹胚亚目中,二核苷酸基元的SSR中分布频率最高的类型为AC/GT,远高于CG/CG的分布频率,AG/CT和AT/AT次之;三核苷酸基元的SSR中,分布频率最高的集中于ACC/GGT和AAT/ATT,远高于ACG/CGT的分布频率。5种枝鳃亚目中,二核苷酸基元SSR中,AG/CT的分布频率最高;三核苷酸基元的SSR中,AAT/ATT的分布频率远高于其他类型的三核苷酸基元。从整体来看,21种十足目甲壳动物的SSR中多为富含A/T的类型。
|
|
表 5 腹胚亚目中二、三核苷酸基元SSR的数量和频率分布 Tab.5 The number and frequency of EST-SSR with di- and tri-nucleotide motifs in Pleocyemata |
|
|
表 6 枝鳃亚目中二、三核苷酸基元SSR的数量和频率分布 Tab.6 The number and frequency of EST-SSR with di- and tri-nucleotide motifs in Dendrobranchiata |
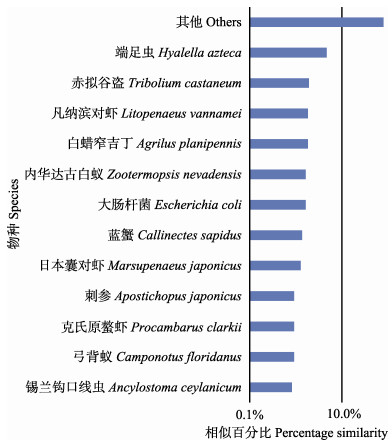
通过Blast2GO功能注释工具,比对脊尾白虾含SSR的序列与NCBI数据库中已知物种的基因(图 1),脊尾白虾EST-SSR注释过程中引用已知注释基因的物种主要为端足虫(Hyalella azteca)(4.67%),此外,白滨对虾为1.83%,蓝蟹为1.37%,日本囊对虾和克氏原螯虾分别为1.28%和0.92%。
|
图 1 脊尾白虾EST-SSR与其他物种相似度统计图 Fig.1 Percentage similarity of Top-Blast hits for EST-SSR in E. carinicauda against other species |
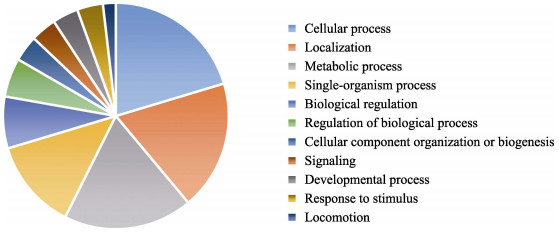
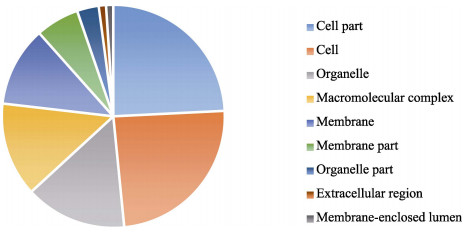
将脊尾白虾含有SSR的EST序列从生物学过程、分子功能和细胞组分3类进行GO分析。其中47条(5.87%)序列有一个或多个已经预测的功能。由图 2可以看出,在11个生物学过程中细胞代谢过程所占比例最大,达到20%,其次,定位和代谢过程的比例为18%。如图 3所示,5种分子功能被记录47次,其中,链接产物的比例最大为47%,其次,催化活性的比例为28%。如图 4所示,9个细胞组分中,细胞组分和细胞比例为24%。
|
图 2 GO分析-生物学过程 Fig.2 GO analysis-Biological process |

|
图 3 GO分析-分子功能 Fig.3 GO analysis-Molecular function |
|
图 4 GO分析-细胞组分 Fig.4 GO analysis-Cellular component |
本研究分析了21种十足目甲壳动物EST序列中SSR的分布频率和重复基元的特点,发现腹胚亚目中含有SSR位点的非冗余EST序列占总数的32.15%,枝鳃亚目中为31.00%,二者比例均高于其他水产动物,如牙鲆(Paralichthys olivaceus)中的比例为7.95% (陈松波等, 2010),在斑点叉尾 
在腹胚亚目和枝鳃亚目中,不同重复基元SSR的分布频率有着非常明显且一致的分布特征,均是复合(Ⅰ)型远高于其他类型,斑鳢中复合SSR类型也占有相当的比例(占总数的7.07%)(谢楠等, 2016)。二、三、四核苷酸基元的数目多于五、六核苷酸基元,在其他的水产动物如牙鲆(陈松波等, 2010)和金头鲷(Vogiatzi et al, 2011)中,同样是二、三核苷酸基元的数目较多,Wang等(2005)研究发现,中国对虾EST-SSR中三核苷酸基元的数目最多,与本研究对中国对虾的结果相一致;十足目甲壳动物中四核苷酸基元的数目也占很大比例,特别是在凡纳滨对虾中,四核苷酸数目最多,相似的报道较少。
腹胚亚目中二核苷酸重复中分布频率最高的类型为AC/GT,同Vogiatzi等(2011)对金头鲷的研究中发现AC/GT占优势的结果相一致;枝鳃亚目中较高的类型为AG/CT,同样在南亚野鲮(Labeo rohita)的研究中发现AG/CT占优势(Dinesh et al, 2013)。2种亚目中均为CG/CG的分布频率最低,其中,中国明对虾、脊尾白虾、白滨对虾、克氏原螫虾、红蟹和珍宝蟹中没有CG/CG类型,大部分鱼类的研究中,也没有CG/CG类型(Ju et al, 2005)。不同三核苷酸基元的SSR的分布频率在不同物种中差异较大,整体上来看,在枝鳃亚目中分布较腹胚亚目集中,枝鳃亚目中为AAT/ATT最多,腹胚亚目中较多的类型集中于ACC/GGT、AAT/ATT,3种核苷酸基元均为富含A/T的SSR,在许氏平
比对脊尾白虾含SSR的序列与NCBI数据库中已知物种的基因,发现脊尾白虾同虾蟹类的基因相似度较低,Wang等(2017)的研究发现,脊尾白虾与凡纳滨对虾和斑节对虾等物种的基因相似比例也较低,主要原因是NCBI数据库中有关十足目甲壳动物的数据资源较少。GO分析后发现,细胞代谢过程、链接产物、细胞组分和细胞的比例分别在3个类型的注释中占优势,这与凡纳滨对虾(孙政等, 2013)和梭鲈(Sander lucioperca)(Han et al, 2016)的研究结果相一致。
4 结论本研究发现十足目甲壳动物中EST-SSR出现频率较高,2种亚目中二、三、四核苷酸基元的SSR较多,且类型丰富。EST-SSR信息的挖掘有利于加深人们对于十足目甲壳动物SSR分布规律的认识,为甲壳动物EST-SSR标记的开发及实际应用提供参考,推动甲壳动物种质资源的创新利用。针对脊尾白虾的基因功能注释分析发现,在3个类型的注释中,细胞代谢过程、链接产物、细胞组分和细胞的比例占有优势,为脊尾白虾在基因功能方面的深入研究提供参考资料。
Bai JY, Pang YZ, Yang YB, et al. Development and application of EST-SSR markers in the black quail. Animal Husbandry and Veterinary Medicine, 2016, 48(11): 49-51 [ 白俊艳, 庞有志, 杨又兵, 等. 黄羽鹌鹑EST-SSR标记的开发与应用. 畜牧与兽医, 2016, 48(11): 49-51] |
Chen SB, Gong L, Liu HJ. Analysis of SSR information in EST resources of Japanese flounder (Paralichthys olivaceus). Journal of Northeast Agricultural University, 2010, 42(42): 1215-1220 [ 陈松波, 龚丽, 刘海金. 牙鲆EST资源的SSR信息分析. 东北农业大学学报, 2010, 42(42): 1215-1220] |
Dinesh KS, Soumya PP, Sujata P, et al. Identification of reproduction-related genes and SSR-markers through expressed sequence tags analysis of a monsoon breeding carp rohu, Labeo rohita (Hamilton). Gene, 2013, 524(1): 1-14 DOI:10.1016/j.gene.2013.03.111 |
Du NS. Crustacean biology. Beijing: Science Press, 1993: 675-760 [ 堵南山. 甲壳动物学. 北京: 科学出版社, 1993: 675-760]
|
Guo Q, Guo LL, Zhang L, et al. Construction of a genetic linkage map in tree peony (Paeonia Sect. Moutan) using simple sequence repeat (SSR) markers. Scientia Horticulturae, 2017, 219: 294-301 |
Han X, Ling Q, Li C, et al. Characterization of pikeperch (Sander lucioperca) transcriptome and development of SSR markers. Biochemical Systematics and Ecology, 2016, 66: 188-195 DOI:10.1016/j.bse.2016.04.001 |
Huang QX, Zuo J, Kong H, et al. Data-mining and diversity analysis of EST-SSRs from 11 tropical plant species. Chinese Journal of Tropical Crops, 2012, 33(7): 1208-1214 [ 黄启星, 左娇, 孔华, 等. 11种热带植物EST-SSR标记的开发和多样性分析. 热带作物学报, 2012, 33(7): 1208-1214 DOI:10.3969/j.issn.1000-2561.2012.07.012] |
Ju Z, Wells MC, Martinez A, et al. An in silico mining for simple sequence repeats from expressed sequence tags of zebrafish, medaka, fundulus, and xiphophorus. In Silico Biology, 2005, 5: 439-463 |
Li DY, Kong J, Meng XH, et al. Development of multiplex PCR systems of microsatellite markers for pacific white shrimp (Litopenaeus vannamei) and its application for parentage identification. Progress in Fishery Sciences, 2016, 37(3): 58-67 [ 李东宇, 孔杰, 孟宪红, 等. 凡纳滨对虾(Litopenaeus vannamei)微卫星多重PCR体系的建立及其在家系亲权鉴定中的应用. 渔业科学进展, 2016, 37(3): 58-67] |
Serapion J, Kucuktas H, Feng JN, et al. Bioinformatic mining of type Ⅰ microsatellites from expressed sequence tags of channel catfish (Ictalurus punctatus). Marine Biotechnology, 2004, 6(4): 364-377 DOI:10.1007/s10126-003-0039-z |
Singh RK, Jena SN, Khan S, et al. Development, cross- species/genera transferability of novel EST-SSR markers and their utility in revealing population structure and genetic diversity in sugarcane. Gene, 2013, 524(2): 309-329 DOI:10.1016/j.gene.2013.03.125 |
Sun Z, Liu CZ, Li FH, et al. Bioinformatic processing of a large number of Litopenaeus vannamei ESTs and analysis of tissue-specific gene expression. Acta Oceanologica Sinica, 2013, 35(5): 128-136 [ 孙政, 柳承璋, 李富花, 等. 凡纳滨对虾大量EST的生物信息学分析. 海洋学报, 2013, 35(5): 128-136 DOI:10.3969/j.issn.0253-4193.2013.05.014] |
Varshney RK, Graner A, Sorrells ME. Genic microsatellite markers in plants: Features and applications. Trends in Biotechnology, 2005, 23(1): 48-55 |
Vogiatzi E, Lagnel J, Pakaki V, et al. In silico mining and characterization of simple sequence repeats from gilthead sea bream (Sparus aurata) expressed sequence tags (EST- SSRs): PCR amplification, polymorphism evaluation and multiplexing and cross-species assays. Marine Genomics, 2011, 4(2): 83-91 DOI:10.1016/j.margen.2011.01.003 |
Wang HX, Li FH, Xiang JH. Polymorphic EST-SSR markers and their mode of inheritance in Fenneropenaeus chinensis. Aquaculture, 2005, 249(1-4): 107-114 DOI:10.1016/j.aquaculture.2005.03.041 |
Wang JJ, Ge QQ, Li JT, et al. Identification of novel EST-SSR markers by transcriptome sequencing in ridgetail white prawn Exopalaemon carinicauda. Genes and Genomics, 2017, 40(1): 1-9 |
Wang RF, Li J, Li JT, et al. Genetic analysis of 33 microsatellite loci in 3 families of Exopalaemon carinicauda inbred line. Progress in Fishery Sciences, 2017, 38(4): 78-86 [ 王日芳, 李健, 李吉涛, 等. 脊尾白虾(Exopalaemon carinicauda)近交系3个家系33个微卫星座位的遗传分析. 渔业科学进展, 2017, 38(4): 78-86] |
Wang YP, Guo XM. Development and characterization of EST-SSR markers in the eastern oyster Crassostrea virdinica. Marine Biotechnology, 2007, 9(4): 500-511 DOI:10.1007/s10126-007-9011-7 |
Xie N, Liu K, Feng XY, et al. Analysis of simple sequence repeats information in expressed sequence tags of Channa argus and Channa maculatus. Freshwater Fisheries, 2016, 44(3): 16-22 [ 谢楠, 刘凯, 冯晓宇, 等. 乌鳢和斑鳢EST序列微卫星信息分析. 淡水渔业, 2016, 44(3): 16-22 DOI:10.3969/j.issn.1000-6907.2016.03.003] |
Xue R. Development of EST-SSR markers and genetics assessment of fast-growing breeding line for the black rockfish, Sebastes schlegelii. Masterxs Thesis of Shanghai Ocean University, 2015 [薛蕊.许氏平鮋(Sebastes schlegelii) EST-SSR标记开发及快速生长选育系遗传结构分析.上海海洋大学硕士研究生学位论文, 2015]
|
Ye J, Fang L, Zheng H, et al. WEGO: A web tool for plotting GO annotations. Nucleic Acids Research, 2006, 34: 293-297 DOI:10.1093/nar/gkl031 |
Yumurtaci A, Sipahi H, Al-Abdallat A, et al. Construction of new EST-SSRs for Fusarium resistant wheat breeding, Computational Biology and Chemistry, 2017, 68: 22-28
|
Zhou L, Liu T, Cheng YK, et al. Molecular mapping of a stripe rust resistance gene in Chinese wheat landrace "Hejiangyizai" using SSR, RGAP, TRAP, and SRAP markers. Crop Protection, 2017, 94: 178-184 DOI:10.1016/j.cropro.2016.12.008 |
Zhou Q. Screening of highly polymorphic EST-SSR markers and investigation of genetic diversity in Macrobrachium Nipponensis. Masterxs Thesis of Nanjing Agricultural University, 2013 [周巧.青虾高多态性EST-SSR标记的筛选及遗传多样性的研究.南京农业大学硕士研究生学位论文, 2013]
|




