2. 中国水产科学研究院黄海水产研究所 青岛海洋科学与技术试点 国家实验室海洋渔业科学与食物产出过程功能实验室 农业农村部海水养殖病害防治重点实验室 青岛市海水养殖流行病学与生物安保重点实验室 青岛 266071
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences; Laboratory for Marine Fisheries Science and Food Production Processes, Polit National Laboratory for Marine Science and Technology (Qingdao); Key Laboratory of Maricultural Organism Disease Control, Ministry of Agriculture and Rural Affairs; Qingdao Key Laboratory of Mariculture Epidemiology and Biosecurity, Qingdao 266071
凡纳滨对虾(Litopenaeus vannamei)是中国主要的对虾养殖品种,2016年全国的凡纳滨对虾海淡水养殖总产量为167.2万t,占对虾类养殖产量的91% (中国渔业统计年鉴, 2017)。高密度集约化养殖是凡纳滨对虾养殖的主要模式,随着集约化养殖模式的剧增,对虾养殖生产的病害问题日益严重,先后有白斑综合征(White spot disease, WSD)、急性肝胰腺坏死病(Acute hepatopancreatic necrosis disease, AHPND)、偷死野田村病(Covert mortality nodavirus disease, CMNVD)、虾肝肠胞虫病(Enterocytozoon hepatopenaei disease, EHP)等疫病发生与流行(Flegel, 1997; Zhang et al, 2016; 中国水生动物卫生状况报告, 2016)。
益生菌作为养殖对虾病害生物防控的重要手段,已经在拮抗细菌或抗病毒感染方面展示出了较好的保护效果,国内外水产养殖使用较多的益生菌是光合细菌和芽孢杆菌(Ninawe et al, 2009; Wen et al, 2015; 杜佗等, 2017)。芽孢杆菌不仅可以改善肠道微生物生态平衡、促进对虾对饲料的消化和吸收能力、提高对虾免疫和抗逆水平,还可以抑制养殖水体和肠道病原微生物的生长和繁殖、提高对虾抗病力(孙艳等, 2012; Lakshmi et al, 2013; Zokaeifar et al, 2014)。但目前研究与应用的多为单一芽孢杆菌,多种类的芽孢杆菌混合使用还鲜有报道(Ravi et al, 2007; Sánchez-Ortiz et al, 2016)。
本研究以枯草芽孢杆菌(Bacillus subtilis)、地衣芽孢杆菌(B. licheniformis)和短小芽孢杆菌(B. pumilus)为候选益生菌,采用投喂单一和复合配伍形式包裹饲料免疫凡纳滨对虾,3周后进行白斑综合征病毒(White spot syndrome virus, WSSV)人工感染实验,统计实验组和对照组的累积死亡率,验证复合芽孢杆菌对凡纳滨对虾抗病毒能力的影响;并通过荧光定量PCR技术分析各实验组和对照组对虾组织病毒拷贝数变化,以及含半胱氨酸的天冬氨酸蛋白酶(Cysteinyl aspartate specific proteinase, Caspase)基因和硫氧还原蛋白(Thioredoxin, Trx)基因在免疫阶段和WSSV感染阶段的相对表达情况,用于探究益生菌提高对虾抗病能力的机制。
1 材料与方法 1.1 对虾与菌种 1.1.1 对虾实验用凡纳滨对虾购自山东省海阳市某对虾养殖场[生长环境盐度为20±1、温度为(25±1)℃],对虾进入实验室前先行采样,确认WSSV、CMNV等主要对虾病原检测阴性。购买的对虾在实验室养殖条件下暂养1周左右,使其适应实验室养殖环境,暂养期间投喂商品饲料,定期处理残饵和更换养殖水体。
1.1.2 菌种及培养短小芽孢杆菌、枯草芽孢杆菌和地衣芽孢杆菌均分离于健康凡纳滨对虾肠道,纯化后经16S rDNA序列分析及Biolog碳源利用等生理生化特征分析完成菌株鉴定,并于–80℃保存。菌种活化后经发酵罐发酵培养,离心收集菌体,短小芽孢杆菌活菌量达到(1~2)×1011 CFU/ml,地衣芽孢杆菌和枯草芽孢杆菌活菌数量达到(1~2)×1012 CFU/ml。
1.2 饲料制备实验全过程选用大海跃牌饲料作为基础饲料。免疫饲料按一定比例将菌液与无菌PBS混合后,喷洒在基础饲料上,混合均匀,阴干后于–4℃冰箱中保存。每周涂布含益生菌的饲料于固体2216E培养基中,检测饲料中的细菌数是否达到所需菌种浓度。基础饲料喷洒等量PBS为空白对照。
1.3 实验分组将暂养稳定后的凡纳滨对虾随机挑取个体均匀、活力较强的1200尾分配在整理箱中。共5组,每组6个重复,每个重复40尾对虾。5组分别投喂不同饲料:A组含枯草芽孢杆菌活菌(1~2)×107 CFU/g,B组含地衣芽孢杆菌活菌(1~2)×107 CFU/g,C组含短小芽孢杆菌活菌(1~2)×107 CFU/g,D组含枯草芽孢杆菌、地衣芽孢杆菌和短小芽孢杆菌的等量混合活菌(1~2)×107 CFU/g,PBS组为投喂不含益生菌的基础饲料的对照组。实验期间,各实验组和对照组每日投喂3次相应饲料,日投喂量为凡纳滨对虾体重的10%(依据对虾摄食情况做适量调整),并按相同条件进行养殖管理。
1.4 WSSV感染实验将实验室保存的感染WSSV的鳌虾去除肝胰腺的头胸组织,加入适量4℃预冷的PBS,20000 r/min冰浴匀浆;匀浆液4℃、3000 g离心10 min,上清液4℃反复离心3次(4000 g,6000 g,8000 g各10 min);上清液经400目筛绢过滤除去杂质后,用0.45 μm滤膜过滤除细菌,分装保存于-80℃。制备的病毒粗提液浓度为0.16 g/ml,注射感染前进行预实验,确定最适感染剂量为50 μl/尾。
在免疫21 d后,从各实验组和对照组中随机挑取240尾对虾进行感染实验,每组6个重复,每个重复40尾对虾。其中,3个重复用于统计累积死亡率,3个重复用于采样。感染前1 d停止投喂饲料,次日,于每尾对虾第2腹节注射WSSV粗提液50 μl,以PBS溶液为阴性对照组。攻毒后,实验组和对照组依然投喂相应饲料。每天及时捡出死亡或濒死对虾,记录死亡数量,14 d后结束感染实验。
1.5 样品采集与处理免疫期间的样品采集与处理:实验开始前取样作为0 h,免疫实验开始后分别在1、3、7和15 d随机采集对照组(PBS)和实验组(A、B、C、D组)对虾。无菌操作取出对虾肠道放于1.5 ml无RNase离心管中,于-80℃超低温冰箱中保存。
感染期间的样品采集与处理:感染实验开始前,取样作为0 h,攻毒实验开始后,分别在6、18、96和192 h随机采集对照组和实验组(A、B、C、D组)对虾。分别无菌操作采集对虾鳃和肠道置于1.5 ml无RNase离心管中,于-80℃超低温冰箱中保存。
1.6 RNA提取利用总RNA提取试剂盒(TaKaRa)提取RNA,方法参考使用说明书。用NanoDrop 2000c (Thermo)测定RNA OD260 nm/OD280 nm值,进行定量测定,分装保存于-80℃备用。进行反转录前,去除总RNA中残留的基因组DNA。
1.7 cDNA合成使用Prime Script RT reagent Kit With gDNA Eraser试剂盒(TaKaRa)进行cDNA合成,在20 μl反应体系中加入1 ml (≤1 mg/ml)的总RNA,4 μl 5×PrimeScript Bμffer 2,1 μl Prime Script Mix,10 μl基因组DNA去除反应液,4 μl无RNase水,37℃孵育15 min。85℃加热5 s,合成cDNA模板。
1.8 病毒定量检测 1.8.1 检测引物病毒定量检测引物见程晓艳等(2010),WSSV-FP(CTCTTGTGGTTCATCAGGGGC);WSSV-RP(CTGGATTTTCTCTCAGGGTCTTTAGT)。
1.8.2 标准品制备制备含有WSSV目的扩增片段的质粒作为标准品。按照世界动物卫生组织(OIE)《水生动物疫病诊断手册》对WSSV进行PCR扩增,得到256 bp的扩增产物。将扩增产物连接到pMD18-T载体,转化到大肠杆菌DH5α,构建重组质粒,在LB培养基(含100 μg/ml氨苄青霉素)上增殖,提取质粒DNA,用NanoDrop 2000c (Thermo)测定质粒DNA的浓度,每对碱基的平均分子量为649 g/mol,再根据阿佛加德罗常数6.02×1023,计算出标准品的拷贝数为8×1011 copies/μl。按照100倍稀释梯度,绘制标准曲线。
1.8.3 WSSV绝对定量实时荧光定量PCR反应体系:Real-time PCR按TransStart Top Green qPCR Super Mix试剂盒(Trans Gen Biotech)说明书进行,反应体系为25 μl:DNA模板1 µl (10~20 ng/μl),总量为200~400 ng;正、反向引物各0.5 µl (10 μmol/L),总量为5 pmol;2×TransStartTM Green qPCR Super Mix 12.5 µl;1×Passive Reference Dye 0.5 µl;无RNase水9 µl。每组设3个平行,无RNase水作为阴性对照。扩增程序:94℃预变性5 min,1个循环;94℃预变性30 s,61℃退火30 s,72℃延伸30 s,40个循环,并且在此步72℃时收集荧光,72℃延伸10 min。
1.9 抗病毒相关基因表达量的定量检测 1.9.1 引物参照GenBank中凡纳滨对虾相应序列,利用Primer Premier 5.0进行目的基因Caspase和Trx的引物设计,引物序列见表 1,内参基因与目的基因在扩增时加入等量模板,扩增后熔解曲线无非特异性扩增。
|
|
表 1 Real-time PCR引物序列 Tab.1 Primer used for real-time PCR |
依据SYBR Premix Ex TaqTMⅡ试剂盒(TaKaRa)配制25 μl反应体系:2×SYBR Premix Ex TaqTMⅡ 12.5 μl,正、反向引物各10 μmol/L 2 μl,DNA模板1 ml(≤100 ng),无RNase水9.5 μl;于Rotor Gene3000 (Corbett, 澳大利亚)进行实时荧光定量PCR,反应程序:95℃预变性10 min,1个循环;95℃预变性5 s、60℃退火(由每个基因确定)20 s、72℃延伸20 s,45个循环。
1.9.3 熔解曲线测定实时荧光定量PCR反应完成后,在Rotor Gene 3000中每完成1个循环进行1次信息采集。采用Rotor-Gene Analysis Software 6.0进行分析,2种免疫相关基因的相对表达量采用2–∆∆Ct法来进行计算(Livak et al, 2001)。
1.10 统计分析本研究所有数据均采用SPSS16.0统计软件进行单因素方差分析(One-way ANOVA),若出现显著差异时(P < 0.05, P < 0.01),用LSD法做多重比较。
2 结果与分析 2.1 益生菌对凡纳滨对虾感染WSSV后累积死亡率的影响凡纳滨对虾感染WSSV后14 d内的累积死亡率统计结果见表 2,由表 2可知,枯草芽孢杆菌+地衣芽孢杆菌+短小芽孢杆菌复合益生菌组(D组)到感染实验结束时,对虾累积死亡率最低,只有(50.0±5.3)%;其次为地衣芽孢杆菌组(B组),对虾累积死亡率为(63.3±5.5)%;投喂含枯草芽孢杆菌和短小芽孢杆菌饲料实验组(A组和C组)对虾的累积死亡率分别为(73.3±7.0)%和(75.0±7.9)%;对照组(PBS组)对虾的累积死亡率为100%,显著高于各实验组的累积死亡率。
|
|
表 2 口服不同益生菌包裹饲料的凡纳滨对虾感染WSSV后的累积死亡率 Tab.2 Cumulative mortality of L. vannamei fed with different probiotic preparations after injection with WSSV |
利用绝对定量PCR技术,测定了凡纳滨对虾感染WSSV后在不同感染时间段的鳃组织病毒拷贝数(图 1)。在整个感染阶段,各实验组的病毒拷贝数呈先上升后下降趋势,但对照组病毒拷贝数一直呈现上升趋势,且病毒拷贝数均高于实验组,差异显著(P < 0.05)或极显著(P < 0.01)。病毒感染6 h后,对虾鳃组织的病毒拷贝数达到2.73×105~1.59×106 copies/µl,对照组最高,与A、B组差异显著,与C、D组差异极显著;18 h各组对虾鳃组织的病毒拷贝数继续增加,对照组最高,与A、B组差异显著,与C、D组差异极显著;到96 h各组的病毒拷贝数均达到最大值,对照组最高,与B组差异显著,与A、、C和D组差异极显著;192 h对照组病毒拷贝数略下降,实验组下降更加突出,D组最低,为1.69×105 copies/µl,对照组与实验组间差异极显著。此结果证实,饲料中添加益生菌,在一定程度上能延缓病毒复制速度,降低病毒拷贝数,复合益生菌组延缓病毒复制效果更佳。
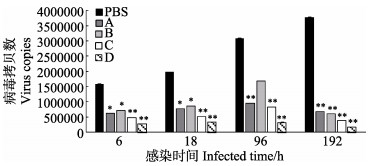
|
图 1 凡纳滨对虾感染WSSV后鳃组织内病毒拷贝数 Fig.1 The virus copies in gill of L. vannamei during WSSV infection A:枯草芽孢杆菌组;B:地衣芽孢杆菌组;C:短小芽孢杆菌组;D:复合芽孢杆菌组(枯草芽孢杆菌、短小芽孢杆菌和地衣芽孢杆菌);PBS组:对照组。*表示差异显著(P < 0.05),**表示差异极显著(P < 0.01)。下同 A: B. subtilis group, B: B. licheniformis group, C: B. pumilus group, D: Mixed Bacillus group, PBS: Control group. * denote significant difference (P < 0.05), **denote highly significant difference (P < 0.01). The same as below |
通过荧光定量PCR技术测定了对虾肠道Caspase基因在免疫期间的相对表达量,结果显示,免疫期间实验组Caspase基因相对表达量在第1天上调明显,随后其相对表达量随着免疫时间的延长逐渐恢复到正常水平,各时间节点Caspase基因相对表达量差异不显著(图 2)。
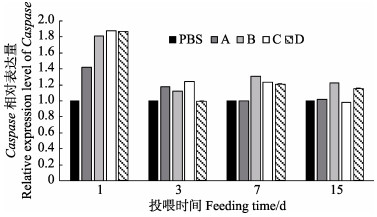
|
图 2 益生菌包裹饲料投喂期间对虾肠道中免疫基因Caspase的相对表达量 Fig.2 Relative expression of immune gene Caspase in the gut of L. vannamei fed with different probiotic preparations |
通过荧光定量PCR技术测定了对虾肠道Caspase基因在WSSV感染后的相对表达量,结果显示,WSSV感染后各组对虾肠道Caspase相对表达量随感染时间的延长呈先上调再下调的趋势,且在18 h各组对虾肠道Caspase表达量达到最大值。在6 h时,复合芽孢杆菌组的对虾肠道Caspase表达量极显著高于对照组,地衣芽孢杆菌的对虾肠道Caspase表达量显著高于对照组;18 h时,所有实验组的对虾肠道Caspase表达量均极显著高于对照组,而96 h时,只有复合芽孢杆菌组的对虾肠道Caspase表达量显著高于对照组(图 3)。
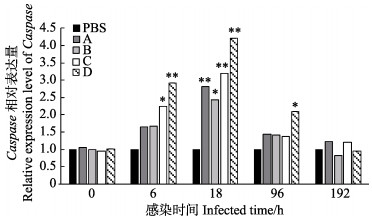
|
图 3 WSSV攻毒后各组对虾肠道中免疫基因Caspase的相对表达量 Fig.3 Relative expression of immune gene Caspase in the gut of L. vannamei fed with different probiotic preparations during WSSV infection |
通过荧光定量PCR技术测定了对虾肠道Trx基因在免疫期间的相对表达量,结果显示,各实验组Trx相对表达量在整个免疫期间均呈现上调趋势,且以第1天值最高,各实验组对虾肠道中Trx基因相对表达量均极显著高于对照组,其中,复合芽孢杆菌组表达量最高。第3天,对虾肠道中Trx基因相对表达量略有下降,A、B组显著高于对照组,C、D组极显著高于对照组;第7天又有所下降,C组显著高于对照组,D组极显著高于对照组;到第15天,只有C组显著高于对照组(图 4)。
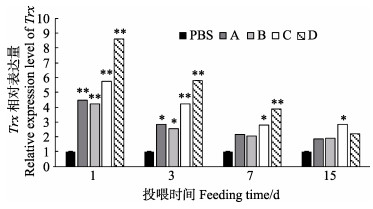
|
图 4 益生菌包裹饲料投喂期间对虾肠道中免疫基因Trx的相对表达量 Fig.4 Relative expression of immune genes Trx in the gut of L. vannamei fed with different probiotic preparations |
通过荧光定量PCR技术测定了对虾肠道Trx基因在WSSV感染后的相对表达量,结果显示,WSSV感染后,各实验组对虾肠道Trx相对表达量在6 h内没有变化,18 h时,陡升到最大值,且极显著高于对照组;96 h时,复合芽孢杆菌组的对虾肠道Trx表达量极显著高于对照组,其余3个实验组显著高于对照组;到192 h时,3个实验组的对虾肠道Trx表达量仍显著高于对照组(图 5)。
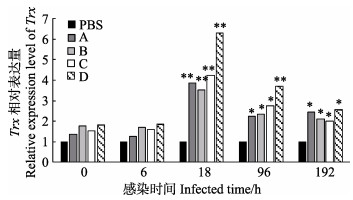
|
图 5 WSSV攻毒后各组对虾肠道中Trx基因的相对表达 Fig.5 Relative expression of immune gene Trx in the gut of L. vannamei fed with different probiotic preparations during WSSV infection |
枯草芽孢杆菌、地衣芽孢杆菌和短小芽孢杆菌普遍存在于土壤及植物体表,是人、动物肠道内常见的共生菌。它们均在1989年美国食品药物管理局(FDA)和美国饲料公定协会(AAFCO)公布的44种“可直接饲喂且通常认为是安全的微生物”范围之内,也属于2008年12月中国农业部1126号公告《饲料添加剂品种目录》中规定的16种可以直接饲喂动物的饲料级微生物添加剂菌种。芽孢杆菌和双歧杆菌、乳酸菌等传统益生菌相比,具有营养体–芽孢–营养体的独特生活史周期,抗逆能力强、繁殖速度快、营养要求简单,具有可抵抗酸、高温、干燥等不良环境条件等优势,适宜大规模产业化发酵生产。芽孢杆菌作为饲料微生物可以100%活菌状态到达肠道,因此,以饲料添加剂方式在水产动物养殖生产中有很好的应用前景(Ninawe et al, 2009; Lakshmi et al, 2013)。
对虾肠道中存在着大量微生物,仅可培养细菌其总数通常在106~109 CFU/g之间,弧菌属(Vibrio sp.)、乳球菌属(Lactococcus sp.)、芽孢杆菌属(Bacillus sp.)、葡萄球菌属(Staphylococcus sp.)、希瓦氏菌属(Shewanella sp.)、节杆菌属(Arthrobacter sp.)、发光杆菌属(Photobacterium sp.)、微小杆菌属(Microbacterium sp.)等为对虾肠道的主要(可培养)优势菌(张盛静等, 2015)。复合益生菌制剂突破了单一益生菌的局限,能够相互协调作用,发挥更好效果(刘淑兰等, 2017)。Salinas等(2005)应用枯草芽孢杆菌和乳酸菌以复合和单一添加形式投喂金头鲷(Sparus aurata),结果显示,复合菌比单一菌有更好的效果,认为不同细菌在肠道微生物环境所处生态位不同,因而复合菌比单一菌组免疫力高。Ravi等(2007)在斑节对虾(Penaeus monodon)幼体养殖期间,向养殖水体中添加芽孢杆菌可以有效地抑制水体中如副溶血弧菌(Vibrio parahaemolyticus)、哈维氏弧菌(V. harveyi)等致病菌的生长,并可以显著地降低死亡率。Sánchez-Ortiz等(2016)报道,将从毛蚶(Anadara tuberculosa)中分离的地衣芽孢杆菌和枯草芽孢杆菌混合到饲料中投喂凡纳滨对虾,结果降低了WSSV和传染性皮下和造血组织坏死病毒(IHHNV)感染后的死亡率,增强了对虾的免疫相关基因表达。本研究选用的芽孢杆菌均分离自健康对虾肠道,以单独和复合的方式添加到对虾饲料中,饲喂21 d后,通过WSSV感染实验来验证其抗病效果。结果证实,饲料中添加单一或复合芽孢杆菌均可提高凡纳滨对虾的抗WSSV感染能力,以复合芽孢杆菌抗病毒能力最显著。WSSV感染后的几个时间节点对虾鳃组织中病毒拷贝数与对虾累积死亡率的结果相吻合,证实芽孢杆菌进入对虾消化道通过某种或多种机制间接影响了病毒在对虾体内的复制和增殖,或是提高了对虾对病毒的清除作用。高剂量投入益生菌可在环境中形成优势菌群,一方面抑制其他病原微生物的繁殖,将继发性感染控制在较低水平;另一方面当环境遭遇病原微生物侵染,威胁到微生物区系的平衡时,有助于维持或建立宿主微生物生态平衡,间接控制对虾传染性疾病的发生和发展(Tannock, 1998;Chai et al, 2016; Sha et al, 2016)。研究证实,来自细菌细胞壁成分可作为甲壳动物免疫增强剂使用,因而,有益微生物的作用机理可能是通过菌种本身或细胞壁成分刺激动物非特异性免疫系统发挥作用,从而提高动物免疫力和抗病力(Rengpipat et al, 2000;Zhou et al, 2006; Sruthy et al, 2015)
含半胱氨酸的天冬氨酸蛋白酶(Caspase)是古老进化史的多成员家族,具有进化上的保守性,并在细胞凋亡中起重要作用。Caspase在细胞凋亡过程中,不仅是接受外界信号,使细胞发生凋亡,还是细胞凋亡的执行者,完成对特定蛋白底物的水解,而使细胞显示出一系列凋亡特征(Wakiyama et al, 2006)。Wu等(2004)推测,当日本对虾(Penaeus japonicus)感染WSSV后,会启动一个高效的程序化细胞死亡机制来控制WSSV的入侵,且细胞凋亡的程度与感染程度密切相关。Phongdara等(2006)克隆了斑节对虾(Penaeus monodon) Caspase基因,证实该基因具有Caspase-3特征,且发现由病毒刺激或处于垂死状态的对虾中,Caspase基因表达量升高。宋光年等(2010)克隆了中国明对虾(Fenneropenaeus chinensis)Caspase基因,注射感染WSSV 3 h后,对虾肝胰腺中Caspase的表达呈现显著上调,推测该基因与细胞凋亡相关。本研究同样证实,益生菌日常饲喂凡纳滨对虾,不会对对虾肠道Caspase基因的表达产生影响,而当对虾感染WSSV后6 h Caspase基因表达量有所上升,18 h Caspase基因表达量最高,而且经过复合芽孢杆菌饲料免疫的对虾较单一芽孢杆菌饲料的Caspase在同一时刻表达量差异显著。20~45 h是对虾注射感染WSSV后病毒的指数生长期(宋晓玲等, 2002),Caspase的高表达必然引发对虾鳃细胞的凋亡,从而减缓了病毒在对虾鳃细胞的增殖速度。但是,Caspase基因的表达首先影响了WSSV的复制,而对虾累积死亡率到感染后的第8天呈现显著差异,原因尚待进一步研究。
硫氧还原蛋白(Trx)是广泛存在于原核细胞和真核细胞中的具有多种重要功能、在进化上保守的蛋白,1964年在大肠杆菌(Escherichia coli)中首先被发现。Trx参与细胞的氧化还原调控,维持细胞蛋白处于还原状态,是一种重要的抗病抗逆蛋白(Jin et al, 1997)。Garcia-Orozco等(2012)比较了WSSV和IHHNV感染时凡纳滨对虾Trx (LVTrx)在mRNA和蛋白质水平上的异同,结果证实病毒感染会提高对虾Trx基因的表达水平,且Trx对WSSV比IHHNV有更明显的反应,WSSV或IHHNV感染对虾还能诱导LVTrx蛋白的差异反应。本研究证实,益生菌摄取和WSSV感染都能刺激Trx的表达,相比较而言,益生菌的刺激相对平缓;而WSSV感染后,各实验组对虾肠道Trx相对表达量在18 h时陡升到最大值,且以复合芽孢杆菌组的激活能力最强。由此可见,不仅不同的病毒感染对虾使其Trx表达水平不同,不同功能微生物刺激对虾使其Trx表达水平也不相同,这可能是由所研究对虾种类、组织类别和实验处理方式不同造成的。另外,虽然实验组Trx自病毒感染后的18~192 h一直维持较高水平,但实验组对虾累积死亡率到感染后的第8天(192 h)才呈现显著差异,推测Trx基因表达的影响只是对虾抗病毒感染的一个环节,对虾的抗病毒感染能力的提高是由多种原因和多个环节实现的。
综上所述,自健康凡纳滨对虾肠道筛选的枯草芽孢杆菌、地衣芽孢杆菌、短小芽孢杆菌通过添加到饲料中,投喂给予凡纳滨对虾,能够提高对虾的抗病毒感染能力,且以这3株菌的复合形式投喂对虾的免疫保护率最高,可作为抗白斑综合征的候选益生菌株。对虾抗病力的提高可能与芽孢杆菌减缓了病毒在靶组织的增殖速率,提高了Caspase基因和Trx基因的表达水平相关。
Chai PC, Song XL, Chen GF, et al. Dietary supplementation of probiotic Bacillus PC465 isolated from the gut of Fenneropenaeus chinensis improves the health status and resistance of Litopenaeus vannamei against white spot syndrome virus. Fish and Shellfish Immunology, 2016, 54: 602-611 DOI:10.1016/j.fsi.2016.05.011 |
Cheng XY, Liu QH, Huang J. Establishment of real-time fluorescent quantitative PCR assay for detecting white spot syndrome virus. Journal of Anhui Agricultural Sciences, 2010, 38(26): 14265-14267 [ 程晓艳, 刘庆慧, 黄倢. 实时荧光定量PCR检测对虾白斑综合症病毒方法的建立. 安徽农业科学, 2010, 38(26): 14265-14267 DOI:10.3969/j.issn.0517-6611.2010.26.028] |
Du T, Li B, Wang YG, et al. Screening and characteristic analysis of potential probiotics from large water ponds used for sea cucumber (Apostichopus japonicus) farming. Progress in Fishery Sciences, 2017, 38(3): 180-186 [ 杜佗, 李彬, 王印庚, 等. 刺参(Apostichopus japonicus)大水面养殖池塘环境中优势益生菌筛选及其特性分析. 渔业科学进展, 2017, 38(3): 180-186] |
Flegel TW. Major viral diseases of the black tiger prawn (Penaeus monodon) in Thailand. World Journal of Microbiology and Biotechnology, 1997, 13(4): 433-442 DOI:10.1023/A:1018580301578 |
Garcia-Orozco KD, Sanchez-Paz A, Aispuro-Hernandez E, et al. Gene expression and protein levels of thioredoxin in the gills from the whiteleg shrimp (Litopenaeus vannamei) infected with two different viruses: The WSSV or IHHNV. Fish and Shellfish Immunology, 2012, 32(6): 1141-1147 DOI:10.1016/j.fsi.2012.03.020 |
Jin DY, Chae HZ, Rhee SG, et al. Regulatory role for a novel human thioredoxin peroxidase in NF-κB activation. Journal of Biological Chemistry, 1997, 272(49): 30952-30961 DOI:10.1074/jbc.272.49.30952 |
Lakshmi B, Viswanath B, Sai Gopal DV. Probiotics as antiviral agents in shrimp aquaculture. Journal of Pathogens, 2013, 424123 |
Liu SL, Chen J, Li J, et al. Effects of complex-probiotic- preparation on the growth, digestive enzymes and the nonspecific immune indices of Atlantic salmon (Salmo salar). Progress in Fishery Sciences, 2017, 38(5): 100-106 [ 刘淑兰, 陈娟, 李杰, 等. 复合益生菌制剂对大西洋鲑(Salmo salar)生长、消化酶和非特异性免疫指标的影响. 渔业科学进展, 2017, 38(5): 100-106] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using rea-time quantitative PCR and 2-△△CT method. Methods, 2001, 25: 402-408 DOI:10.1006/meth.2001.1262 |
Ninawe AS, Selvin J. Probiotics in shrimp aquaculture: Avenues and challenges. Critical Reviews in Microbiology, 2009, 35(1): 43-66 DOI:10.1080/10408410802667202 |
Phongdara A, Wanna W, Chotigeat W. Molecular cloning and expression of caspase from white shrimp Penaeus merguiensis. Aquaculture, 2006, 252(2-4): 114-120 DOI:10.1016/j.aquaculture.2005.07.024 |
Ravi AV, Musthafa KS, Jegathammbal G, et al. Screening and evaluation of probiotics as a biocontrol agent against pathogenic Vibrios in marine aquaculture. Letters in Applied Microbiology, 2007, 45(2): 219-223 DOI:10.1111/lam.2007.45.issue-2 |
Rengpipat S, Rukpratanporn S, Piyatiratitivorakul S, et al. Immunity enhancement on black tiger shrimp (Penaeus monodon) by a probiont bacterium (Bacillus S11). Aquaculture, 2000, 191(4): 271-288 DOI:10.1016/S0044-8486(00)00440-3 |
Salinas I, Cueste A, Esteban M, et al. Dietary administration of Lactobacillus delbreeckii and Bacillus subtilis, single or combined, on gilthead seabream cellular innate immune responses. Fish and Shellfish Immunology, 2005, 19(1): 67-77 DOI:10.1016/j.fsi.2004.11.007 |
Sánchez-Ortiz AC, Angulo C, Luna-González A, et al. Effect of mixed-Bacillus spp isolated from pustulose ark Anadara tuberculosa, on growth, survival, viral prevalence and immune-related gene expression in shrimp Litopenaeus vannamei. Fish and Shellfish Immunology, 2016, 59: 95-102 DOI:10.1016/j.fsi.2016.10.022 |
Sha Y, Liu M, Wang B, et al. Bacterial population in intestines of Litopenaeus vannamei fed different probiotics or probiotic supernatant. Journal of Microbiology and Biotechnology, 2016, 26(10): 1736-1745 DOI:10.4014/jmb.1603.03078 |
Song GN, Jin SJ, Zhang JQ, et al. Cloning and expression of a caspase gene in Chinese shrimp Fenneropenaeus chinensis. Marine Sciences, 2010, 34(12): 1-7 [ 宋光年, 金松君, 张继泉, 等. 中国明对虾caspase基因的克隆与表达分析. 海洋科学, 2010, 34(12): 1-7] |
Song XL, Huang J, Tang KFL, et al. The construction and analysis for cDNA clones of white spot syndrome virus of shrimp. Acta Hydrobiologica Sinica, 2002, 26(5): 444-451 [ 宋晓玲, 黄倢, Tang KFL, 等. 对虾白斑综合症病毒重组cDNA克隆的构建与分析. 水生生物学报, 2002, 26(5): 444-451 DOI:10.3321/j.issn:1000-3207.2002.05.004] |
Sruthy KS, Chaithanya ER, Sathyan N, et al. Molecular characterization and phylogenetic analysis of novel isoform of anti-lipopolysaccharide factor from the mantis shrimp, Miyakea nepa. Probiotics and Antimicrobial Proteins, 2015, 7(4): 275-283 DOI:10.1007/s12602-015-9198-2 |
Sun Y, Liu F, Song XL, et al. Effects of adding probiotics in the feed on non-specific immune gene expression and disease resistance of Litopenaeus vannamei. Oceanologia et Limnologia Sinica, 2012, 43(4): 845-851 [ 孙艳, 刘飞, 宋晓玲, 等. 饲料中添加益生菌对凡纳滨对虾(Litopenaeus vannamei)非特异免疫基因表达量和抗病力的影响. 海洋与湖沼, 2012, 43(4): 845-851] |
Tannock GW. Studies of the intestinal microflora: A prerequisite for the development of probiotics. International Dairy Journal, 1998, 8(5-6): 527-533 DOI:10.1016/S0958-6946(98)00082-X |
Wakiyama M, Kaitsu Y, Yokoyama S. Cell-free translation system from Drosophila S2 cells that recapitulates RNAi. Biochemical and Biophysical Research Communications, 2006, 343(4): 1067-1071 DOI:10.1016/j.bbrc.2006.03.076 |
Wen CQ, Xue M, Liang HF, et al. Beneficial effects of Ectothiorhodospira shaposhnikovii WF on larval cultivation ofLitopenaeus vannamei. Beneficial Microbes, 2015, 6(4): 525-533 DOI:10.3920/BM2014.0116 |
Wu J, Muroga K. Apoptosis does not play an important role in the resistance of 'immune' Penaeus japonicus against white spot syndrome virus. Journal of Fish Diseases, 2004, 27(1): 15-21 DOI:10.1046/j.1365-2761.2003.00491.x |
Zhang SJ, Zhao XJ, Song XL, et al. Analysis of the culturable bacteria's quantity and composition in the intestinal tract of cultivation shrimp. Journal of Shanghai Ocean University, 2015, 24(2): 211-218 [ 张盛静, 赵小金, 宋晓玲, 等. 人工养殖对虾肠道内可培养细菌数量及组成分析. 上海海洋大学学报, 2015, 24(2): 211-218] |
Zhang XJ, Song XL, Huang J. Impact of Vibrio parahaemolyticus and white spot syndrome virus (WSSV) co-infection on survival of penaeid shrimp Litopenaeus vannamei. Chinese Journal of Oceanology and Limnology, 2016, 34(6): 1278-1286 DOI:10.1007/s00343-016-5165-3 |
Zhou J, Song XL, Huang J, et al. Effects of dietary supplementation of A3α-peptidoglycan on innate immune responses and defense activity of Japanese flounder (Paralichthys olivaceus). Aquaculture, 2006, 251(1-4): 172-181 |
Zokaeifar H, Babaei N, Saad CR, et al. Administration of Bacillus subtilis strains in the rearing water enhances the water quality, growth performance, immune response, and resistance against Vibrio harveyi infection in juvenile white shrimp, Litopenaeus vannamei. Fish and Shellfish Immunology, 2014, 36(1): 68-74 DOI:10.1016/j.fsi.2013.10.007 |



