2. 农业农村部长江中上游渔业资源环境科学观测实验站 中国水产科学研究院长江水产研究所 武汉 430223
2. Scientific Observing and Experimental Station of Fishery Resources and Environment in the Upper and Middle Reaches of Yangtze River, Ministry of Agriculture and Rural Affairs; Yangtze River Fishery Resources Institute, Chinese Academy of Fishery Sciences, Wuhan 430223
长江是世界第三、中国第一大河流,宜昌至湖口县为中游段(29°30'~30°36'N, 113″24'~115″58'E)。中游江宽流曲,河网发达,水质肥沃,饵料生物丰富(Yi et al, 2011),与通江湖泊形成了独特而完整的江湖复合生态系统。水流湍急和江河缓流中的鱼类相互衔接和交替,形成了具有较广种类的江河平原区系复合体鱼类区系(胡军华等, 2008)。中游水域孕育着丰富的鱼类资源,其中不乏珍稀、名贵以及重要的经济鱼类,是我国宝贵的淡水鱼类种质资源库。近半个世纪以来,环境污染、水工建设、围湖造田以及过渡捕捞等造成了鱼类生境片段化、栖息地逐渐丧失,严重影响了其生存和繁殖(Yi et al, 2011)。《长江鱼类志》记录中游鱼类119种(湖北省水生生物研究所鱼类研究室等, 1976),根据新的研究,删除同种异名、偶见种,整理得到长江中游鱼类97种,隶属14目22科68属。2003~2008年,在长江中游及洞庭湖、鄱阳湖共监测到鱼类14目22科86种(胡军华等, 2008);2009~2010年的调查结果为6目10科55种(刘春池等, 2012)。可见,长江中游渔业资源严重衰退,鱼类种群结构在不断简化,生态系统功能逐步退化。有效利用自然资源、保护中游地区鱼类多样性成为紧迫而艰巨任务。然而,鱼类种类繁多,传统的鱼类分类主要依据外部形态特征,不同生长阶段其形态特征的差异给鉴定及分类带来了许多困难(柳淑芳等, 2016)。Hebert等(2003)提出的DNA条形码概念,即通过一段标准的DNA序列对物种进行鉴定,为鱼类的快速鉴定提供了新的思路。在加拿大、古巴、北美、阿根廷等国家不同种类的鱼中得到了成功应用(Hubert et al, 2008; Lara et al, 2010; April et al, 2011; Mabragana et al, 2011)。中国也开展了DNA条形码的鱼类鉴别研究和应用。彭居俐等(2009)成功鉴定了鲌属(Erythroculter)的4种鱼,Zhang等(2012)鉴别了中国南海的242种1336条鱼类。作者曾经以DNA条形码技术实现了鲀形目(Tetraodontiformes)鱼类的准确鉴定,证实COⅠ基因可以用于硬骨鱼类物种鉴定(郜星晨, 2005)。随着分子条形码技术的不断发展,结合电子芯片自身快速、灵敏及高通量等优点,DNA条形码芯片技术已成为一种识别鉴定物种及确定阶元归属的理想工具。但国内外相关的研究工作相对较少,Kochzius等(2008)将芯片技术用于鱼类鉴定,证明了16S rRNA基因用于设计微阵列探针区分欧洲海洋至少11种鱼类的实用性。柳淑芳等(2016)筛选了山东沿海常见的77种常见鱼类DNA条形码物种特异性探针,最终有64种鱼类可用于制作DNA条形码芯片。关于我国淡水鱼类的DNA芯片适用性研究尚未开展,因此,本研究拟根据长江中游鱼类种类分布情况,探讨DNA条形码技术的适用性,并筛选物种特异性探针,从而为长江中游常见鱼类的DNA条形码芯片研发工作提供基础资料和技术支撑。
1 材料与方法 1.1 实验材料根据中国知网数据库(CNKI)查到的100余篇主题为“长江中游鱼类”、“鱼类多样性”、“鱼类物种组成”和“渔业资源调查”的调查文献和相关科学考察集,并参考往年监测记录作补充(胡军华等, 2008; Yi et al, 2011; 刘春池等, 2012),确定长江中游常见鱼类物种名录,即7目11科50属64种鱼类。物种的分类和名称通过Fishbase数据库(www.fishbase.org)、《中国动物志》(乐佩琦, 2000)及分类文献确认。从GenBank下载对应物种收录的168条COⅠ序列(表 1),尽量选取研究区域内不同的地理种群,每条序列需经BOLD数据库验证。
|
|
表 1 本研究中64种鱼类COⅠ基本信息 Tab.1 Information of COⅠ genes of 64 species |
Bioedit比对切齐序列,MEGA 6.0计算序列的序列变异、碱基组成及基于Kimura-2-paramater(K2P)模型不同分类阶元的遗传距离(Tamura et al, 2013)。进一步构建邻接(Neighbour-Joining, NJ)系统进化树,将Bootstrap值参数设为1000。Microsoft Excel 2007作遗传距离频率分布直方图。
1.2.2 DNA条形码芯片探针设计和分析建立本地Blast数据库,序列文件barcode.fasta格式化,形成文件barcode.fasta.nsq。由于序列数目较多,为了提高筛选效率,鲤形目48种鱼类130条序列划分为一个数据集,其余16种鱼类38条序列为另一数据集。各数据集分别利用OligoArray 2.1筛选探针,探针长度为23~27 bp,Tm值为70℃~82℃,GC含量为40%~ 55%,Na+和DNA浓度分别为1 mol/L和1 μmol/L,形成二级结构的最大Tm值和交叉杂交最小Tm值为65℃,两探针间距离为25 bp,最大探针数为10,连续出现的单一碱基数不超过4个。
选取该物种序列所共有且与其他物种序列无同源性的探针作为该物种的探针,在线软件OligoCalc (http://www.simgene.com/OligoCalc)优化,排除易形成发夹、茎环及自身二聚体结构的不合格探针。整理各数据集筛选合格的探针与切齐序列于在线软件OHM(http://bioinfo.unice.fr/softwares/ohm/)中进行虚拟杂交,查看杂交结果。
2 结果与分析 2.1 DNA条形码分析结果 2.1.1 DNA条形码序列特征经同源比对得到长度为624 bp的有效片段,其中,T=28.7%,C=28.2%,A=25.3%,G=17.8%(表 2)。A+T含量为54.0%,第1、2和3密码子的G+C含量分别为57.5%、42.7%和37.6%,表现出明显的反G偏倚,与其他硬骨鱼类线粒体的特点一致。在碱基变异中,不变位点为516个,占总位点数的82.7%。转换位点为66个,颠换位点为41个,86.4%的碱基转换和97.6%的碱基颠换发生在第3密码子位点。转换与颠换比(R)为3.58,表明此片段没有饱和,适合进行系统发育分析。
|
|
表 2 64种鱼类COⅠ基因序列组成统计 Tab.2 The content of COⅠ gene sequences of 64 species |
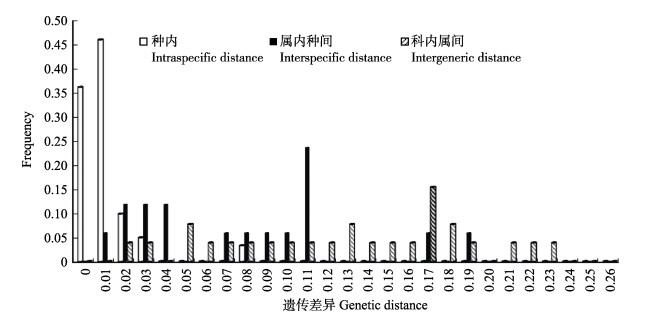
本研究中鱼类K2P遗传差异变化幅度较大,0~0.298不等。种内遗传差异在0~0.0776之间(P.prochilus),均值为0.0079。59种(占总类数92.1%)鱼类种内差异小于2%,其余5种鱼类,即马口鱼(O.uncirostris)、乌鳢(C.asiatica)、斑鳜(S.scherzeri)、泉水鱼(P.prochilus)等遗传距离大于2%(表 3和表 4)。属内种间遗传差异为0.003 (Rhinogobio)~0.19 (Rhodeinae),均值为0.084,该范围与种内遗传差异的分布范围重叠(图 1)。其中,圆筒吻





|
|
表 3 数据集1探针编号及序列 Tab.3 Probe IDs and sequences of dataset 1 |
|
|
表 4 数据集2探针编号及序列 Tab.4 Probe ids and sequences of dataset 2 |
|
图 1 不同分类阶元水平的遗传差异分布 Fig.1 Frequency distribution of K2P genetic distance within various taxonomic levels |
本研究中的64种鱼类不同个体均能聚类形成种的单系支,且支持率较高,50个属不同物种也能聚类成属的单系支。但是,科内属间、目内科间及目间聚类效果不明显,不能形成平行的独立分支,与传统分类存在一些分歧(图 2)。

|
图 2 基于线粒体CO I基因构建的NJ树 Fig.2 NJ tree resulting from analysis of CO I gene data |
初步筛选出46个物种的130条探针,OligoCalc优化后去除12条不合格探针,获得43个物种的118条探针(表 3)。所得探针与对应物种序列在OHM中虚拟杂交,结果显示:有些探针特异性不强,与靶标序列结合的同时还能与其他物种的序列结合,其中TZ83为花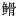
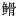
初步筛选出16个物种的80条探针,OligoCalc优化去除34条不合格探针,得到15个物种的46条探针(表 4)。探针与对应物种序列在OHM中虚拟杂交,结果显示,TZ154、TZ155、TZ162分别为粗唇


总之,本研究下载的64个物种的168条COⅠ序列,利用OligoArray 2.1软件初步筛选出62个物种的210条特异性探针,OligoCalc优化后选出58个种的164条探针,OHM中虚拟杂交后43个物种的112条探针可作为物种的鉴别探针,可鉴定的物种数占总物种数的67.2%(表 5)。
|
|
表 5 筛选探针及杂交情况 Tab.5 Probe selection and hybridization conditions |
DNA条形码是一项利用标准序列快速和准确鉴定的诊断技术,为分类学发展的新方向(Dayrat, 2005)。Hebert等(2003)对大量动物的线粒体COⅠ序列分析发现,2%是物种划分的参考值,且种间距离至少是种内距离的10倍。本研究中,59种鱼类(占总类数的92.1%)种内差异小于2%,种内平均遗传距离为0.0079,属内种间平均遗传距离为0.084,种间距离为种内距离的11倍,基本符合“10倍种内遗传距离”。构建的系统进化树中,64种鱼类均能形成种的单系支,且支持率较高。这些都充分证明了线粒体COⅠ序列的分子条形码对于长江中游常见鱼类有很好的分类效果,可进一步筛选DNA条形码芯片(Blanchet, 2012; Kim et al, 2011)。其中,49种(占总类数的76.6%)鱼类的种内遗传距离小于种间最小遗传距离(0.003),可形成明显的条形码间隙。其余种类种内与种间遗传差异存在重叠,未形成DNA条形码间隙,造成这种现象的原因可能是物种快速形成、渐渗杂交或不完全世系分选等(Zhang et al, 2012)。此外,本研究中,种内、属内种间、科内属间、目内科间及目间的遗传差异分别为0.0079、0.084、0.184、0.232和0.253,分类单元越高,遗传距离越大,为传统分类学的有效性提供了依据。但除了种间与种内距离差异明显外,物种以上阶元的增加明显变缓慢,且不同高阶分类单元遗传差异出现重叠,未形成明显的DNA条形码间隙。表明基于线粒体COⅠ序列的DNA条形码序列解析能力有限,可用于物种的分类鉴定,但不适合于高阶元分类单元的系统进化分析(Dayrat, 2005)。
3.2 电子芯片的适用性DNA条形码芯片是一种利用DNA条形码序列快速鉴定物种的技术,可用于构建物种分子监测网络,快速分析物种群落的演变(Hajibabaei et al, 2007)。本研究从64种鱼类中初步筛选得到62个种的210条物种特异性探针,2个物种没有筛选出探针。利用OligoCalc对筛选出的探针进行检验,4个种的探针不合格,最后获得58个物种的164条探针。OHM软件虚拟杂交后,仅43个物种的112条探针可以与其靶标序列特异性杂交。种内遗传距离小于2%的马口鱼、乌鳢、斑鳜、泉水鱼未筛选出物种特异性探针。其中,圆筒吻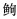
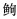


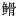


April J, Mayden RL, Hanner R, et al. Genetic calibration of species diversity among North America's freshwater fishes. Proceedings of the National Academy of Sciences of the USA, 2011, 108(26): 10602-10607 DOI:10.1073/pnas.1016437108 |
Blanchet S. The use of molecular tools in invasion biology: An emphasis on freshwater ecosystems. Fisheries Management and Ecology, 2012, 19(2): 120-132 DOI:10.1111/j.1365-2400.2011.00832.x |
Dayrat B. Towards integrative taxonomy. Biological Journal of the Linnean Society, 2005, 85(3): 407-415 DOI:10.1111/j.1095-8312.2005.00503.x |
Fish Research Laboratory of Institute of Hydrobiology. Fish of the Yangtze River. Beijing: Science Pres, 1976 [湖北省水生生物研究所鱼类研究室. 长江鱼类. 北京: 科学出版社, 1976]
|
Gao XC. DNA barcoding of Tetraodontiformes in coastal waters of China. Masterxs Thesis of Jinan University, 2015 [郜星晨.基于线粒体COⅠ序列的中国沿海鲀形目鱼类DNA条形码研究.暨南大学硕士研究生学位论文, 2015]
|
Hajibabaei M, Singer GA, Clare EL, et al. Design and applicability of DNA arrays and DNA barcodes in biodiversity monitoring. BMC Biology, 2007, 5(1): 24-24 DOI:10.1186/1741-7007-5-24 |
Hebert N, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes. Proceedings of the Royal Society B: Biological Sciences, 2003, 270(1512): 313-321 DOI:10.1098/rspb.2002.2218 |
Hu JH, Zhang CL, Hu HJ. Structure and variation of fish diversity of lake in the central Yangtze River. Journal of Hydroecologe, 2008, 1(1): 47-51 [胡军华, 张春兰, 胡慧建. 长江中游湖泊鱼类物种多样性结构及动态. 水生态学杂志, 2008, 1(1): 47-51] |
Hubert N, Hanner R, Holm E, et al. Identifying Canadian freshwater fishes through DNA barcodes. PLoS One, 2008, 3(6): e2490 DOI:10.1371/journal.pone.0002490 |
Kim S, Koo H, Kim J, et al. DNA chip for species identification of Korean freshwater fish: A case study. Biochip Journal, 2011, 5(1): 72-77 DOI:10.1007/s13206-011-5111-6 |
Kochzius M, Nolte M, Weber H, et al. DNA microarrays for identifying fishes. Marine Biotechnology, 2008, 10(2): 207-217 DOI:10.1007/s10126-007-9068-3 |
Kochzius M, Seidel C, Antoniou A, et al. Identifying fishes through DNA barcodes and microarrays. PLoS One, 2010, 5(9): e12620 DOI:10.1371/journal.pone.0012620 |
Lara A, De Leon JLP, Rodriguez R, et al. DNA barcoding of Cuban freshwater fishes: Evidence for cryptic species and taxonomic conflicts. Molecular Ecology Resources, 2010, 10(3): 421-430 DOI:10.1111/j.1755-0998.2009.02785.x |
Le PQ. China animal Scientific Osteichthyes. Beijing: Science Press, 2000 [乐佩琦. 中国动物志, 硬骨鱼纲. 北京: 科学出版社, 2000]
|
Liu CC, Gao X, Lin PC, et al. Fish community structure in Gezhouba reservior. Resources and Environment in the Yangtze Basin, 2012, 21(7): 843-849 [刘春池, 高欣, 林鹏程, 等. 葛洲坝水库鱼类群落结构特征研究. 长江流域资源与环境, 2012, 21(7): 843-849] |
Liu SF, Li XR, Du TF, et al. DNA barcoding and electronic microarray for common fish species in Shandong coastal water. Journal of Fishery Sciences of China, 2016, 23(4): 777-790 [柳淑芳, 李献儒, 杜腾飞, 等. 山东近海习见鱼类DNA条形码及其电子芯片分析. 中国水产科学, 2016, 23(4): 777-790] |
Mabragana E, De Astarloa JMD, Hanner R, et al. DNA barcoding identifies Argentine fishes from marine and brackish waters. PLoS One, 2011, 6(12): e28655 DOI:10.1371/journal.pone.0028655 |
Ouyang XY, Mo BH, Yu HL, et al. DNA barcoding identification comparion on accuracy of DNA barcodes in mosquito identification. Chinese Journal of Frontier Health and Quarantine, 2007, 30(6): 349-352 [欧阳小艳, 莫帮辉, 余华丽, 等. DNA条形码识别-DNA条形码与DNA芯片识别蚊媒准确性的比较. 中国国境卫生检疫杂志, 2007, 30(6): 349-352 DOI:10.3969/j.issn.1004-9770.2007.06.008] |
Peng JL, Wang XZ, Wang D, et al. Application of DNA barcoding based on the mitochondrial COⅠ gene sequences in classification of culter (Pisces: Cyprinidae). Acta Hydrobiologica Sinica, 2009, 33(2): 271-276 [彭居俐, 王绪祯, 王丁, 等. 基于线粒体COⅠ基因序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用. 水生生物学报, 2009, 33(2): 271-276] |
Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis Version 6. 0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729 DOI:10.1093/molbev/mst197 |
Yi Y, Yang Z, Zhang S. Ecological risk assessment of heavy metals in sediment and human health risk assessment of heavy metals in fishes in the middle reaches of the Yangtze River basin. Environmental Pollution, 2011, 159(10): 2575-2585 DOI:10.1016/j.envpol.2011.06.011 |
Zhang JB, Hanner R. Molecular approach to the identification of fish in the South China Sea. PLoS One, 2012, 7(2): e30621 DOI:10.1371/journal.pone.0030621 |




 Zacco platypus
Zacco platypus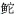 Gobiobotia filifer
Gobiobotia filifer 科Serranidae
科Serranidae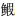 虎鱼科Gobiidae
虎鱼科Gobiidae