2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室 青岛 266071
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071;
3. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory for Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Qingdao 266071
甘露糖结合凝集素相关丝氨酸蛋白酶1(MASP1)属于丝氨酸蛋白酶(Serine proteases, SP)超家族成员,包括MASP1(Takahashi et al, 2013)、MASP2(Swierzko et al, 2014)和MASP3(Dahl et al, 2001)。MASP1是补体凝集素途径中起重要作用的激活蛋白,MASP1可被甘露糖结合凝集素(Mannan-binding lectin, MBL)激活以响应病原体感染,MBL-MASP复合物共同激活凝集素途径(Sunyer et al, 1998; Schwaebl et al, 2002)。
目前,关于MASP1的研究相对较少,2000年首次在人中克隆鉴定出MASP1(Chen et al, 2000),随后构建了人的MASP1 N端片段原核表达载体并在大肠杆菌中进行了表达(Cai et al, 2008)。迄今仅在文昌鱼(Branchiostoma belcheri)(Endo et al, 2003)、七鳃鳗(Lampetra fluviatilis)(Endo et al, 2003)、草鱼(Ctenopharyngodon idella)(Dang et al, 2017)和菊海鞘(Botryllus schlosseri)(Nicola et al, 2017)等水生生物中开展了MASP1的克隆表达及免疫应答相关研究。硬骨鱼MASP1的研究还极为有限。
半滑舌鳎(Cynoglossus semilaevis)是我国重要的海水养殖鱼类。近年来,随着水产养殖行业的快速发展,病害问题特别是弧菌病对半滑舌鳎养殖业造成严重的经济损失。有关半滑舌鳎免疫机制的研究和有效的病害防控手段还有待深入研究(Chen et al, 2015)。本研究通过PCR技术克隆了CsMASP1基因的cDNA,并分析了其基因结构及其进化关系,还研究了CsMASP1基因在半滑舌鳎11种健康组织中的表达特征,及鳗弧菌感染后该基因在6种免疫组织中的时空表达规律,为进一步深入了解CsMASP1基因在半滑舌鳎免疫防御中的作用奠定基础。
1 材料与方法 1.1 实验材料半滑舌鳎购自山东海阳黄海水产有限公司,鱼龄为2龄左右,平均体重为(400±5) g,平均体长为(40.3±0.1) cm。健康实验鱼在24℃水族箱中暂养7 d,每天换新鲜灭菌海水,尽量消除环境胁迫。鳗弧菌为本实验室保存菌种,经16S rRNA测序进行菌种鉴定。
1.2 样品处理和采集随机选取3条健康的半滑舌鳎,分别采集每条鱼的血液、脑、鳃、性腺、心脏、头肾、肠、肝、皮肤、脾和后肾共11种组织,并立即投入液氮中冷冻,然后转移至-80℃冰箱保存,用于总RNA提取。鳗弧菌感染参照(Sha et al, 2017)实验方法略作修改,采用腹腔注射方法对实验组进行鳗弧菌感染,感染剂量(半致死剂量)为3.18×105 CFU/g鱼体,对照组注射相同体积的无菌PBS溶液。在注射后0、6、12、24、48和72 h共6个时间点,每个时间点取3条鱼,分别收集实验组和对照组的血液、鳃、头肾、肠、肝脏和脾脏6种组织,保存方式同上所述。
1.3 RNA提取和cDNA制备采用液氮研磨收集的组织,按照RNA提取试剂盒(天根,北京)的说明书进行组织总RNA提取。提取的RNA用琼脂糖凝胶电泳(1.00%)检测其完整性。用Agilent 2100 biological analyzer (Applied Biosystems, 美国)检测其纯度和浓度。按照PrimeScript Ⅱ 1st Strand cDNA Synthesis Kit (TaKaRa, 大连)试剂盒反转录成cDNA,-20℃冰箱保存备用。
1.4 CsMASP1基因的cDNA序列的扩增通过对半滑舌鳎转录组和基因组数据进行生物信息学分析,筛选了CsMASP1基因的部分cDNA序列,并对该序列设计特异性引物P1和P2(表 1),以半滑舌鳎11个组织混合cDNA为模板,进行PCR扩增。PCR反应体系(20 μl):Trans TaqⓇHiFi Polymerase (5 U/μl) 0.2 μl, 10×Trans Taq HiFi BufferⅡ2 μl, 2.5 mmol/L dNTPs 1.6 μl, P1 1 μl, P2 1 μl, cDNA模板2 μl, ddH2O 12.2 μl。反应程序:95℃ 5 min;95℃ 30 s,58℃ 30 s,72℃ 30 s,38个循环;72℃ 10 min;4℃保存。1.00%琼脂糖凝胶电泳检测后,用DNA胶回收试剂盒(诺唯赞, 南京)纯化PCR扩增特异性产物,连接到pEASY-T1(全式金, 北京)载体上,重组载体转化到TransT1感受态细胞(全式金, 北京)中,挑取阳性克隆进行菌液PCR验证后测序(上海派森诺生物科技有限公司)。在克隆获得的序列5′端和3′端设计RACE (Rapid-Amplification of cDNA Ends)引物P3和P5(表 1)和巢式特异性引物P4和P6(表 1),使用SMARTerTM RACE cDNA Amplification Kit(TaKaRa, 大连)获得该基因的5′和3′端序列,将克隆的序列进行拼接获得CsMASP1基因的cDNA全长序列。
|
|
表 1 半滑舌鳎MASP1基因克隆和表达分析所用引物 Tab.1 PCR primers used for cloning and expression analysis of MASP1 |
将CsMASP1的全长cDNA序列提交到NCBI网站(http://blast.ncbi.nlm.nih.gov/blast.cgi),利用BLAST程序进行核苷酸同源性比对,利用ORF Finder (http://blast.ncbi.nlm.nih.gov/gorf/gorf.html)查找开放阅读框,并推导其相应的氨基酸序列;采用SMART(Simple Modular Architecture Research Tool)在线软件分析CsMASP1蛋白的结构域,利用ExPASy软件(http://www.expasy.org)预测其相对分子质量和等电点;用SignalP 4.0 Server (http://www.cbs.dtu.dk/services/SignalP/)软件分析CsMASP1蛋白的信号肽。应用Splign(http://www.ncbi.nlm.nih.gov/IEB/Research/Ostell/Spidey/)在线软件确定CsMASP1基因的内含子与外显子的数目,分析不同物种MASP1的基因结构以及其所在染色体的位置。
1.6 MASP1同源比对系统进化树构建从GenBank中查询不同物种MASP1基因的氨基酸序列,采用DNAMAN软件进行不同物种的MASP1蛋白的氨基酸序列多重比对,用MEGA 6.0软件中的Neighbor-joining法构建系统进化树。
1.7 CsMASP1基因表达的实时定量分析根据CsMASP1基因设计扩增引物P7和P8(表 1),以半滑舌鳎18S rRNA基因为内参设计特异引物Cs18s-F和Cs18s-R (表 1)。基因实时定量表达参照已报道的方法进行(Lu et al, 2013; Yu et al, 2017)。使用ChamQTMSYBRⓇ Color qPCR Master Mix试剂(诺唯赞, 南京),按照说明书于Applied Biosystems 7500 Real Time PCR仪上进行该基因的实时定量分析(qRT-PCR)。反应体系为20 μl:10 μl 2×ChamQ SYBR Color qPCR Master Mix,1 μl模板cDNA,0.4 μl引物P7,0.4 μl引物P8,0.4 μl 50×ROX Reference Dye 2,7.8 μl ddH2O。反应程序:预变性95℃ 3 min;95℃ 10 s,60℃ 30 s,40个循环;熔解曲线95℃ 15 s;60℃ 1 min;95℃ 15 s。每个样品设置3个技术重复,采用2-ΔΔCt方法计算目的基因的相对表达量,Ct值取3个平行样品的平均值。qRT-PCR检测结果使用SPSS 19.0统计软件进行单因素方差分析(One-way ANOVA)的Duncan法对多组样本平均值进行两两比较分析,P < 0.05时,认为存在显著性差异。
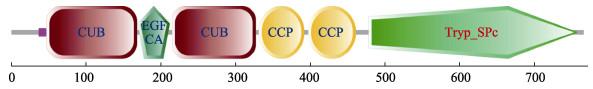
2 结果 2.1 CsMASP1基因序列分析和结构特征克隆获得的CsMASP1基因cDNA序列全长为2507 bp (登录号:XM_008309208.2),5′非编码区(Untranslated Region, UTR, 5′ UTR)长82 bp,3′ UTR长142 bp,开放阅读框(Open reading frame, ORF)长2283 bp (图 1)。推测CsMASP1编码氨基酸为760个,预测其分子量为84.95 kDa,理论等电点(pI)为5.33。SignalP 4.0 Server分析显示,此氨基酸序列无信号肽。SMART软件分析显示,CsMASP1蛋白含6个结构域:分别是2个类Clr/Cls蛋白(like Clr/Cls Protein domain, CUB)结构域(47~168 aa, 215~327 aa),1个表皮细胞生长因子样(Calcium-binding epidermal growth factor-like domain, EGF-CA)结构域(169~212 aa),2个补体控制蛋白区(Complement control protein domain, CCP)结构域(331~392 aa, 397~461 aa)和1个丝氨酸蛋白激酶区(Serine protease domain, SP)Tryp-SPc结构域(478~752 aa) (图 2)。

|
图 1 半滑舌鳎MASP1 cDNA序列和推测的氨基酸序列
Fig.1 Nucleotide and deduced amino acid sequences of Cynoglossus semilaevis MASP1
推导的氨基酸序列显示在核苷酸序列下方,用大写字母表示。方框所示为起始密码子(ATG)、星号(*)所示为终止密码子(TGA);实线( )所示为CUB结构域;圆点线( )所示为CUB结构域;圆点线( )所示为EGF-CA结构域;短划线( )所示为EGF-CA结构域;短划线( )所示为CCP结构域;双划线( )所示为CCP结构域;双划线( )所示为Tryp-SPc结构域;波浪线( )所示为Tryp-SPc结构域;波浪线( )所示为poly A结构
Translated amino acid sequence was shown under the nucleotide sequence as uppercase. The iniation codon and termination codon were marked by a box and an asterisk, respectively; CUB domain was shown in solid line( )所示为poly A结构
Translated amino acid sequence was shown under the nucleotide sequence as uppercase. The iniation codon and termination codon were marked by a box and an asterisk, respectively; CUB domain was shown in solid line( ); EGF-CA domain was shown in dot line( ); EGF-CA domain was shown in dot line( ); CCP domain was shown in dashed line( ); CCP domain was shown in dashed line( ); Tryp-SPc domain was shown in double solid line( ); Tryp-SPc domain was shown in double solid line( ); poly A was shown in wavy line( ); poly A was shown in wavy line( ) )
|
|
图 2 半滑舌鳎MASP1蛋白结构域预测 Fig.2 Domain structure prediction of Cynoglossus semilaevis MASP1 SMART软件在线预测CsMASP1蛋白的结构域,CsMASP1包含6个结构域,从C端到N端分别是CUB结构域(47~168 aa), EGF-CA结构域(169~212aa), CUB结构域(215~327 aa), CCP结构域(331~392 aa), CCP结构域(397~461 aa), Tryp-SPc结构域(478~752 aa) SMART software predicts the domain of CsMASP1 protein online. CsMASP1 contains six domains: CUB domain (47~168 aa), EGF-CA domain (169~212 aa), CUB domain (215~327 aa), CCP domain (331~392 aa), CCP domain (397~461 aa), Tryp-SPc domain (478~752 aa) |
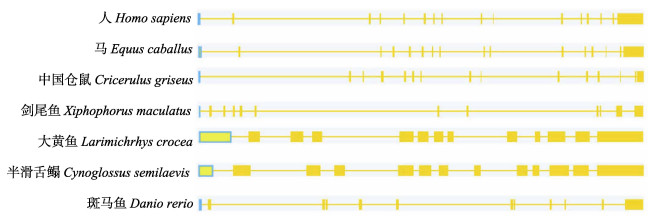
利用Splign软件分析了7个物种的MASP1基因结构并进行了比较(图 3),它们是半滑舌鳎(XM_ 008309208.2)、剑尾鱼(Xiphophorus maculatus)(XM_ 023351221.1)、大黄鱼(Larimichthys crocea)(XM_ 027280128.1)、斑马鱼(Danio rerio)(XM_001341900. 6)、马(Equus caballus) (XM_001499629.5)、人(Homo sapiens)(NM_001879.5)和中国仓鼠(Cricetulus griseus) (XM_003495604.2)。结果显示,CsMASP1含13个外显子,位于4号染色体上;剑尾鱼MASP1含13个外显子,位于18号染色体上;大黄鱼MASP1含13个外显子,位于7号染色体上;斑马鱼MASP1含12个外显子,位于15号染色体上;马MASP1含16个外显子,位于19号染色体上;人MASP1含16个外显子,位于3号染色体上;中国仓鼠MASP1含15个外显子,位于4号染色体上。半滑舌鳎MASP1基因结构与多数已知鱼类(剑尾鱼和大黄鱼)一致,与哺乳类(人、马和中国仓鼠)在外显子数目上有差异,说明该基因在鱼类保守。
|
图 3 不同物种的MASP1基因结构比较 Fig.3 Comparison of MASP1 gene structures of different species 7个物种的MASP1基因结构图,各个物种的MASP1基因序列号与所在染色体基因组序列号分别为:人NM_001879.5, NG_029440.1;马XM_001499629.5, NC_009162.3;中国仓鼠XM_003495604.2, NW_003613596.1;剑尾鱼XM_023351221.1, NC_036460.1;大黄鱼XM_027280128.1, NC_040017.1;半滑舌鳎XM_008309208.2, NC_024310.1;斑马鱼XM_001341900.6, NC_007126.7 The sequence numbers of the MASP1 gene and the genome numbers of the chromosomes in seven species were as follows: Homo sapiens NM_001879.5, NG_029440.1; Equus caballus XM_001499629.5, NC_009162.3; Cricetulus griseus XM_003495604.2, NW_003613596.1; Xiphophorus maculatus XM_023351221.1, NC_036460.1; Larimichthys crocea XM_027280128.1, NC_040017.1; Cynoglossus semilaevis XM_008309208.2, NC_024310.1; Danio rerio XM_001341900.6, NC_007126.7 |
通过BLAST比对,将CsMASP1氨基酸序列(XP_008307430.1)与金目鲈(Lates calcarifer)(XP_ 018560455.1)、牙鲆(Paralichthys olivaceus)(XP_ 019966501.1)、黄鳝(Monopterus albus)(XP_020477488.1)、伯氏朴丽鱼(Haplochromis burtoni)(XP_014184888.1)、非洲爪蟾(Xenopus laevis)(AAI70221.1)、海龟(Chelonia mydas)(EMP37646.1)、红喉潜鸟(Gavia stellata) (XP_009815055.1)、皇帝企鹅(Aptenodytes forsteri) (KFM09600.1)、白鹭(Egretta garzetta)(XP_009633907.1)、西伯利亚虎(Panthera tigris altaica) (XP_015393589.1)、羊驼(Vicugna pacos)(XP_ 006201039.1)、马(XP_ 005601936.1)、中国仓鼠(XP_007606939.1)、人(XP_ 016862358.1)的MASP1氨基酸序列进行同源比对,相似性范围为76%~42%。其中,CsMASP1与金目鲈的相似性最高,为76%;而与中国仓鼠的相似性最低,为42%。将半滑舌鳎和其他13个物种MASP1的氨基酸序列进行多重比对,可以看出不同物种MASP1存在高度保守的氨基酸残基,如图 4所示,保守氨基酸出现在丝氨酸蛋白酶活性中心处:组氨酸His526、天冬氨酸Asp582和丝氨酸Ser705,这3个氨基酸的保守定位对于维持丝氨酸蛋白酶的活性是十分重要的。

|
图 4 半滑舌鳎和其他物种MASP1氨基酸序列的多重比对 Fig.4 Multiple alignment of the deduced amino acids of MASP1 among half-smooth tongue sole and other different species MASP1氨基酸序列比对,丝氨酸蛋白酶结构域中保守氨基酸用黑框标出:组氨酸His526、天冬氨酸Asp582和丝氨酸Ser705 Amino acid sequence alignment of MASP1, conservative amino acids in serine protease domain were marked by the black box: His526, Asp582 and Ser705 |
利用MEGA 6.0软件对上述各个物种的MASP1氨基酸序列进行比对构建了系统进化树(图 5)。结果显示,半滑舌鳎与伯氏朴丽鱼、金目鲈和黄鳝等鱼类的MASP1聚为一支。两栖类海龟和非洲爪蟾、鸟类白鹭、皇帝企鹅和红喉潜鸟,哺乳类马、西伯利亚虎、羊驼、人和中国仓鼠等动物的MASP1聚为一支。但两栖类、鸟类和哺乳类又按亲缘关系远近聚成相应的亚类。MASP1在进化上与伯氏朴丽鱼的亲缘关系最近,与哺乳类、爬行类和鸟类等动物的MASP1的亲缘关系较远。从进化的结果看,几个关键节点的支持率较高,均在90%以上。例如:金目鲈和黄鳝,半滑舌鳎和伯氏朴丽鱼这几个物种聚类为一个分支,支持率分别为98%和100%。

|
图 5 MEGA 6.0软件构建的基于MASP1氨基酸序列的系统进化树 Fig.5 Phylogenetic tree based on MASP1 amino acid sequence by MEGA 6.0 |
利用qRT-PCR分析CsMASP1基因在11种健康组织中的表达特征。结果显示,CsMASP1基因在血液、脑、鳃、性腺、心脏、头肾、肠、肝、皮肤、脾和后肾中均有表达;其中,肝中相对表达量最高(185.4),其次为脾(138.1)和头肾(76.2),在脑、心脏、血液、肠、后肾、性腺、皮肤和鳃中表达量较低且表达量依次降低(图 6)。
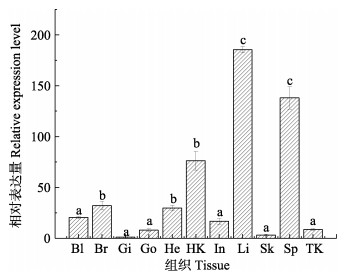
|
图 6 CsMASP1基因在半滑舌鳎健康组织中的相对表达量 Fig.6 The relative expression of CsMASP1 gene in normal tissues of C. semilaevis Bl:血液;Br:脑;Gi:鳃;Go:性腺;He:心脏;HK:头肾;In:肠;Li:肝;Sk:皮肤;Sp:脾;TK:后肾。字母“a, b, c”代表SPSS多重分析的不同分组,有相同字母表示差异不显著(P > 0.05),无相同字母表示差异显著(P < 0.05);下同 Bl: Blood; Br: Brain; Gi: Gills; Go: Gonad; He: Heart; Hk: Head kidney; In: Intestine; Li: Liver; Sk: Skin; Sp: Spleen; Tk: Trunk kidney. The letters of 'a, b, and c' indicated the Duncan grouping in SPSS 19.0 software. The same letters indicate no significant difference (P > 0.05), the different letters indicate significant difference (P < 0.05); the same applies below |
鳗弧菌感染半滑舌鳎后可诱导CsMASP1在免疫组织中的表达变化,在感染0、6、12、24、48、72 h后,CsMASP1在PBS对照组和感染组半滑舌鳎的血液、鳃、头肾、肠、肝和脾6种免疫组织中的表达水平如图 7所示。CsMASP1基因表达总体趋势表现为先升高后降低。在脾和鳃中,CsMASP1基因的表达呈现相同的上调趋势,其表达峰值出现在鳗弧菌感染后6 h,在脾中为对照组表达量的15.4倍;在鳃中则是对照组的4.6倍;在肝中CsMASP1基因表达上调,最大表达量出现在感染后24 h,是对照组的12.4倍;在肠中CsMASP1基因表达上调,最大表达量出现在感染后12 h,是对照组的7.7倍,72 h时依然保持较高的表达水平;在头肾和血液中,CsMASP1基因的表达峰值均出现在鳗弧菌感染后12 h,在头肾中为对照组表达量的9.7倍;在血液中则是对照组的6.8倍;随后基因表达量逐渐下降至略低于正常水平值。

|
图 7 鳗弧菌感染半滑舌鳎后不同时间点6种组织(血液、鳃、头肾、肠、肝和脾)中CsMASP1基因的相对表达水平 Fig.7 Relative expression of CsMASP1 gene in six tissues (blood, gill, head kidney, intestine, liver and spleen) of C. semilaevis at different time points after Vibrio anguillarum infection |
甘露聚糖结合凝集素相关丝氨酸蛋白酶是机体重要的天然免疫防御分子,通过激活补体和调理吞噬2种方式来清除病原体(Chen et al, 2000)。MASP1是形成C4bC2a转化酶的必要因子,是凝集素激活途径中的特异性启动酶(Oroszlan et al, 2016)。MASP最初是作为与Complement component 1s(Cls)功能相似可以裂解C4和C2的一种新型丝氨酸蛋白酶被发现的(Matsushita et al, 1992)。鉴于MASP与Cls相似,推测MBL-MASP1-MASP2与C1q-C1r2-C1s2相似,因此,MASP1和MASP2可能分别与类Complement component 1r (Clr)和类C1s丝氨酸蛋白酶有相似的功能(Matsushita et al, 1998、2000)。虽然在硬骨鱼的免疫系统中已经鉴定出了MASP1,但其对细菌感染的免疫功能尚不清楚(Endo et al, 2003; Dang et al, 2017)。本研究成功克隆了CsMASP1基因,预测其编码蛋白的分子量为84.95 kDa,与已报道的草鱼MASP1 (Dang et al, 2017)的分子量和人MASP1 (Matsushita et al, 1998)的分子量类似。CsMASP1编码的氨基酸序列经SignalP软件预测无信号肽,而草鱼MASP1蛋白序列中包含一段19 aa的信号肽(Dang et al, 2017)。研究表明,MASP的SP区(Serine protease domain)是催化功能区,含有所有丝氨酸蛋白酶活性中心高度保守的组氨酸(His)、丙氨酸(Asp)和丝氨酸(Ser)等3个氨基酸残基,与MASP的丝氨酸蛋白酶活性有关(Wallis et al, 2000、2007)。在本研究中,SMART结果显示,CsMASP1含有6个结构域,分别是2个CUB结构域(47~168 aa, 215~327 aa)、1个EGF结构域(169~212 aa)、2个CCP结构域(331~392 aa, 397~461 aa)和1个Tryp-SPc结构域(478~752 aa)。与文献报道的人MASP1(Matsushita et al, 1998; Stover et al, 2001; Dobo et al, 2016)结构域相同,与草鱼MASP1(Dang et al, 2017)结构域也相同。丝氨酸蛋白酶结构域(SP)活性中心有1个典型的组氨酸环。氨基酸序列比对结果显示,丝氨酸蛋白酶结构域完全相似,组氨酸His526、天冬氨酸Asp582和丝氨酸Ser705定位相当保守,与文献报道的文昌鱼和七鳃鳗(Endo et al, 2003)的比对结果一致,说明该活性中心在进化上相对保守。这些特性与MASP/C1r/C1s家族的进化相关(Jia et al, 2003)。系统进化分析结果显示,CsMASP1与伯氏朴丽鱼MASP1亲缘关系最近,与金目鲈和黄鳝等鱼类的MASP1亲缘关系次之。
3.2 CsMASP1在健康组织和鳗弧菌感染后的免疫组织中的表达分析本研究利用qRT-PCR检测CsMASP1在半滑舌鳎11种健康组织和感染后6种免疫组织中的表达谱,结果显示,在所检测的11种组织中分布广泛且具有组织特异性,在肝中表达水平最高,脾中表达水平次之,在头肾中的表达相对其他组织也较高,推测其可能在免疫调控中发挥作用。与草鱼的分布结果不相同,草鱼MASP1在心脏中表达水平最高,在肝和脑中表达水平次之(Dang et al, 2017)。
MASP1是构成先天性免疫防御体系的重要成员之一(Ammitzboll et al, 2013)。本研究报道了CsMASP1参与鳗弧菌感染后机体反应的免疫应答。鳗弧菌感染半滑舌鳎72 h内,CsMASP1在血液、鳃、头肾、肠、肝和脾组织中均出现上调表达的情况,总体趋势表现为表达量先升高,达到一个峰值后,随感染时间的延长表达量逐渐降低恢复至正常水平,说明CsMASP1在病原入侵后参与了机体的免疫应答。鳃和肠中的CsMASP1表达量在鳗弧菌感染后6 h大幅上调,鳃在6 h达到最大值,肠比鳃稍微滞后,肠在12 h达到最高峰,分别是对照组的7.7倍和4.6倍,随后下降到与对照组无显著差异。肠是最大的免疫器官,肠上分布着大量的肠相关淋巴组织,肠道组织和鳃是腹腔注射鳗弧菌后直接接触的部位,所以肠和鳃CsMASP1的表达量均先于其他免疫组织上调。随后,病原菌进入其他免疫组织,激活其他免疫组织CsMASP1基因的表达。与Dang等(2017)研究发现草鱼感染嗜水气单胞菌后,草鱼MASP1在鳃和肠中表达量短时快速增加结果相一致;但草鱼MASP1在肠中感染后4、24、72 h显示3个表达高峰与本研究的结果不太一致,推测草鱼MASP1在肠中感染后出现3个峰值可能是由于感染菌不同,也可能是由于鱼体差异等。脾CsMASP1的表达量随着时间增加而增加,在感染6 h后达到最大值,约是对照组的15倍,是所有免疫组织中增量最高的组织;这与肾、脾是鱼类免疫细胞生成的主要场所,也是产生免疫应答的主要部位这一观点是一致的(Wang et al, 2018)。6种免疫组织表现出不同的表达模式可能与不同时间点的信号转导有关,在我们之前的研究中,感染后,补体激活途径、凝血级联激活途径和弧菌感染相关途径均被激活来抵御病原菌的入侵(Zhang et al, 2015)。CsMASP1在免疫组织中的高表达暗示了CsMASP1在机体免疫反应中可能发挥着重要作用。本结果为深入研究MASP1基因在半滑舌鳎免疫防御系统中的作用奠定了基础。
Ammitzboll CG, Steffensen R, Nielsen HJ, et al. Polymorphisms in the MASP1 gene are associated with serum levels of MASP-1, MASP-3, and MAp44. PLoS One, 2013, 8(9): e73317 DOI:10.1371/journal.pone.0073317 |
Cai XM, Zhao N, Zuo DM, et al. Construction and expression of prokaryotic expression vector of human MASP1 N-terminal fragment. Cell and Molecular Immunology, 2008, 24(6): 546-549 [蔡学敏, 赵娜, 左大明, 等. 人MASPlN端片段原核表达载体的构建及其表达. 细胞与分子免疫学杂志, 2008, 24(6): 546-549 DOI:10.3321/j.issn:1007-8738.2008.06.002] |
Chen ZL, Lu X, Zhang LY, et al. Cloning and identification of human MBL-related serine protease 1 gene. Cellular and Molecular Immunology, 2000, 16(5): 417-419 [陈政良, 卢晓, 张丽芸, 等. 人MBL相关丝氨酸蛋白酶1 cDNA的克隆与鉴定. 细胞与分子免疫学杂, 2000, 16(5): 417-419 DOI:10.3321/j.issn:1007-8738.2000.05.026] |
Chen Y, Fan L, Liu TT, et al. Cloning and expression analysis of TRAF6 gene and TAK1 gene of half-smooth tongue sole. Chinese Aquatic Science, 2015, 22(5): 867-876 [陈燕, 樊琳, 刘田田, 等. 半滑舌鳎TRAF6基因和TAK1基因的克隆及表达分析. 中国水产科学, 2015, 22(5): 867-876] |
Dahl MR, Thiel S, Matsushita M, et al. MASP-3 and its association with distinct complexes of the mannan-binding lectin complement activation pathway. Immunity, 2001, 15(1): 127-135 DOI:10.1016/S1074-7613(01)00161-3 |
Dang YF, Shen YB, Xu XY, et al. Mannan-binding lectin- associated serine protease-1 (MASP-1) mediates immune responses against Aeromonas hydrophila in vitro and in vivo in grass carp. Fish and Shellfish Immunology, 2017(66): 93-102 |
Dobo J, Pal G, Cervenak L, et al. The emerging roles of mannose-binding lectin-associated serine proteases (MASPs) in the lectin pathway of complement and beyond. Immunological Reviews, 2016, 274(1): 98-111 |
Endo Y, Nonaka M, Saiga H, et al. Origin of mannose-binding lectin-associated serine protease (MASP)-1 and MASP-3 involved in the lectin complement pathway traced back to the invertebrate, amphioxus. Journal of Immunology, 2003, 170(9): 4701-4707 DOI:10.4049/jimmunol.170.9.4701 |
Jia TJ, Li P, Zhang SM. Research status of structure and function of MASPs. Medical Review, 2003, 9(4): 193-195 [贾天军, 李萍, 张庶民. MASPs的结构与功能研究现状. 医学综述, 2003, 9(4): 193-195 DOI:10.3969/j.issn.1006-2084.2003.04.001] |
Lu Y, Wang QL, Li M, et al. Expression analysis of TLR20 and TLR21 genes in channel catfish Ictalurus punctatus challenged by different bacteria and virus. Progress in Fisheries Science, 2013, 34(6): 44-51 [路飏, 王启龙, 李敏, 等. 斑点叉尾鮰TLR20和TLR21基因在不同细菌和病毒感染后的表达特征. 渔业科学进展, 2013, 34(6): 44-51 DOI:10.3969/j.issn.1000-7075.2013.06.007] |
Matsushita M, Fujita T. Activation of the classical complement pathway by mannose-binding protein in association with a novel Cls-like serine protease. Journal of Experimental Medicine, 1992, 176(6): 1497-1502 DOI:10.1084/jem.176.6.1497 |
Matsushita M, Endo Y, Nonaka M, et al. Complement-related serine proteases in tunicates and vertebrates. Current Opinion in Immunology, 1998, 10(1): 29-35 DOI:10.1016/S0952-7915(98)80027-7 |
Matsushita M, Thiel S, Jensenius JC, et al. Proteolytic activities of two types of mannose-binding lectin-associated serine protease. Journal of Immunology, 2000, 165(5): 2637-2642 DOI:10.4049/jimmunol.165.5.2637 |
Matsushita M, Endo Y, Fujita T. MASPl (MBL-associated serine protease 1). Immunobiology, 1998, 199(2): 340-347 DOI:10.1016/S0171-2985(98)80038-7 |
Nicola F, Loriano B. Morula cells as key hemocytes of the lectin pathway of complement activation in the colonial tunicate Botryllus schlosseri. Fish and Shellfish Immunology, 2017, 63: 157-164 DOI:10.1016/j.fsi.2017.02.003 |
Oroszlan G, Kortvely E, Szakacs D, et al. MASP-1 and MASP-2 do not activate pro-factor D in resting human blood, whereas MASP-3 is a potential activator: kinetic analysis involving specific MASP-1 and MASP-2 inhibitors. Journal of Immunology, 2016, 196(2): 857-865 |
Schwaebl W, Dalai MR, Thiel S, et al. The mannan-binding lectin-associated serine proteases (MASPs) and MAp19: Four components of the lectin pathway activation complex encoded by two genes. Immunobiology, 2002, 205(4-5): 455-466 DOI:10.1078/0171-2985-00146 |
Sha ZX, Wang LQ, Sun LM, et al. Isolation and characterization of monocyte/macrophage from peripheral blood of half- smooth tongue sole (Cynoglossus semilaevis). Fish and Shellfish Immunology, 2017, 65: 256-266 DOI:10.1016/j.fsi.2017.04.015 |
Stover C, Endo Y, Takahashi M, et al. The human gene for mannan binding lectin associated serine protease-2, the effector component of the lectin route of complement activation, is part of a tightly linked gene cluster on chromosome 1p36.2-3. Genes and Immunity, 2001, 2(3): 119-127 |
Sunyer JO, Lambris JD. Evolution and diversity of the complement system of poikilothermic vertebrates. Immunological Reviews, 1998, 166(1): 39-57 |
Swierzko AS, Szala A, Sawicki S, et al. Mannose-binding lectin (MBL) and MBL-associated serineprotease-2 (MASP-2) in women with malignant and benign ovarian tumours. Cancer Immunology Immunotherapy, 2014, 63(11): 1129-1140 DOI:10.1007/s00262-014-1579-y |
Takahashi M, Sekine H, Endo Y, et al. Comment on "Mannan-binding lectin-associated serine protease (MASP)-1 is crucial for lectin pathway activation in human serum, whereas neither MASP-1 nor MASP-3 is required for alternative pathway function". Journal of Immunology, 2013, 190(6): 2477 |
Wang LQ, Wang H, Chen YD, et al. Isolation and identification of T lymphocyte from peripheral blood of half-smooth tongue sole and analysis of TCRβ gene immune response. Acta Hydrobiologica Sinica, 2018, 42(3): 480-487 [汪林庆, 王航, 陈亚东, 等. 半滑舌鳎外周血T淋巴细胞的分离、鉴定及TCRβ基因免疫应答分析. 水生生物学报, 2018, 42(3): 480-487] |
Wallis R, Dodd RB. Interaction of mannose-binding protein with associated serine proteases: Effects of naturally occurring mutations. Journal of Biological Chemistry, 2000, 275(40): 30962-30969 DOI:10.1074/jbc.M004030200 |
Wallis R, Dodds AW, Mitchell DA, et al. Molecular interactions between MASP-2, C4, and C2 and their activation fragments leading to complement activation via the lectin pathway. Journal of Biological Chemistry, 2007, 282(11): 7844-7851 DOI:10.1074/jbc.M606326200 |
Yu MJ, Sun LM, Chen YD, et al. Cloning and expression of the c-Jun gene in immune response to pathogen challenge in half-smooth tongue sole (Cynoglossus semilaevis). Progress in Fisheries Science, 2017, 38(2): 31-39 [于孟君, 孙璐明, 陈亚东, 等. 半滑舌鳎(Cynoglossus semilaevis) c-Jun基因的克隆及免疫应答分析. 渔业科学进展, 2017, 38(2): 31-39] |
Zhang X, Wang SL, Chen SL, et al. Transcriptome analysis revealed changes of multiple genes involved in immunity in Cynoglossus semilaevis during Vibrio anguillarum infection. Fish and Shellfish Immunology, 2015, 43(1): 209-218 DOI:10.1016/j.fsi.2014.11.018 |



