2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071
凡纳滨对虾(Litopenaeus vannamei)的野生群体分布在太平洋东岸(墨西哥、厄瓜多尔、秘鲁等)。从20世纪末被引入我国开展养殖后,凡纳滨对虾已经发展成为国内最主要的海水养殖对虾种类,产量占海水养殖对虾总量的80%。国内养殖业对于凡纳滨对虾亲虾的依赖巨大。由于国内亲虾不足以满足生产,每年需要从国外进口大量成本高的亲虾。但是,通常这些亲虾的遗传背景和系谱信息无法得知。使用这些亲虾配对生产的后代苗种可能存在遗传多态性低的情况,随着培育代数的增加,近交变的更易发生,经济性状衰退明显。因此,培育国内优质凡纳滨对虾新品种意义重大。目前,已经证明遗传多态性的降低和近交情况能够增加隐性有害等位基因的表达,对选育群体的生长、存活、抗逆等性状造成近交衰退现象(Spielman et al, 2004; Moss et al, 2007; Goyard et al, 2008; Luo et al, 2014; 喻驰方等, 2013)。因此,在凡纳滨对虾遗传选育工作中,为维持育种群体的遗传多样性,避免近交,需要认清不同群体甚至个体间的分子遗传背景差异,为制定合理配种方案提供参考。
微卫星(SSR)标记是一种广泛存在于生物基因组内的共显性标记,拥有遗传多态性高的特点。研究表明,SSR适合于遗传多样性分析(Valles-Jimenez et al, 2004; Perez-Enriquez et al, 2009; Rezaee et al, 2016)。国内在21世纪初期已有利用SSR对黄渤海不同中国对虾(Fenneropenaeus chinensis)群体的遗传多样性进行调查研究,区分不同地理群体间的亲缘关系距离,为种质资源利用和新品种选育提供了很好的参考(刘萍等, 2004; 孟宪红等, 2008)。由于SSR位点信息量大、分型技术成本低的优点,近年来依然被应用在对虾遗传多样性分析中(王军等, 2018; 赵志英等, 2018)。本研究使用8对SSR引物,随机对采集于我国的6个凡纳滨对虾群体进行分析,为遗传育种工作中对不同遗传背景的凡纳滨对虾资源的利用提供一些参考。
1 材料与方法 1.1 实验动物随机从6家育苗场共收集133尾凡纳滨对虾,对其遗传背景进行分析。来自于6家育苗场的对虾分别编号为CP-1、CP-2、CP-3、CP-4、PRI和SIS群体。其中,CP-1、CP-2、CP-3、CP-4群体来自于同一个品牌。PRI和SIS群体分别来自2个不同品牌。21尾凡纳滨对虾来自CP-1群体;20尾来自CP-2群体;11尾来自CP-3群体;10尾来自CP-4群体;61尾来自PRI群体;10尾来自SIS群体。所有群体的对虾都孵化于同一年。在养殖场用消毒的剪刀取一条对虾游泳足,保存于95%酒精,带回实验室用于DNA提取和分析。
1.2 DNA提取使用海洋动物组织基因组DNA提取试剂盒(天根)从对虾游泳足的肌肉组织中提取DNA。分别使用分光光度计和琼脂糖电泳检测DNA的质量。合格的DNA用于后期SSR分型实验。
1.3 SSR分型使用8对已公开发表的凡纳滨对虾SSR引物(Meehan et al, 2003; Alcivar-Warren et al, 2007; Zhang et al, 2007)进行实验。引物序列信息详见表 1。PCR反应体系包括12.5 μl Premix Taq™ (TaKaRa Taq™ Version 2.0) (TaKaRa Taq 0.625 U、dNTPs 0.4 mmol/L、Tris-HCl 20 mmol/L、KCl 100 mmol/L、MgCl2 3 mmol/L) (TaKaRa),1 μl模板DNA (200 ng/μl),1 μl正向引物(10 μmol/L),1 μl反向引物(10 μmol/L),9.5 μl去离子水。反应程序如下:98℃ 3 min;98℃ 30 s,60℃ 10 s,72℃ 1 min,25个循环;72℃延伸3 min。使用ABI 3730XL测序仪(Applied Biosystems, 美国)获得每个SSR位点的基因型信息。
|
|
表 1 引物信息 Tab.1 Primer information |
观察等位基因数(Na)、有效等位基因数(Ne)、观察杂合度(Ho)、期望杂合度(He)、HWE检验、遗传一致度、遗传距离和F-统计量使用POPGene (Version 1.32)软件(https://sites.ualberta.ca/~fyeh/popgene.html)进行计算。使用NTSYSpc (Version 2.10)软件(http://www. exetersoftware.com/cat/ntsyspc/ntsyspc.html)进行UPGMA聚类分析。使用Cervus (Version 3.0.7)软件(http://www. fieldgenetics.com)计算多态性信息含量(PIC)。
2 结果 2.1 遗传多态性分析6个凡纳滨对虾群体的遗传多态性信息见表 2。其中,Na平均值为3.67~9.33。Ne平均值为2.20~5.67。Ho平均值为0.12~0.71。He平均值为0.41~0.81。除了在CP-4群体中的Lv12位点只检测到1种等位基因,其他SSR位点在6个群体中的等位基因数都大于1。8个SSR位点的PIC平均值为0.36~0.76。M1103位点在CP-1、CP-3和PRI群体中都显著偏离HWE (P < 0.05)。Lv12位点在CP-2和PRI群体中都极显著偏离HWE(P < 0.01)。TuMXLv10.33位点在CP-2、CP-3、CP-4和PRI群体中都极显著偏离HWE(P < 0.01)。TuMXLv8.256位点在CP-4群体中显著偏离HWE (P < 0.05);在CP-1、CP-2、CP-3和PRI群体中极显著偏离HWE(P < 0.01)。TuMXLv7.56位点在CP-4群体中显著偏离HWE(P < 0.05)。TuMXLv9.90位点在CP-3群体中显著偏离HWE(P < 0.05);在PRI群体中极显著偏离HWE(P < 0.01)。TuMXLv7.21和TuMXLv9.103位点在所有群体中都没有发生偏离HWE。
|
|
表 2 遗传多样性分析 Tab.2 Genetic diversity analysis |
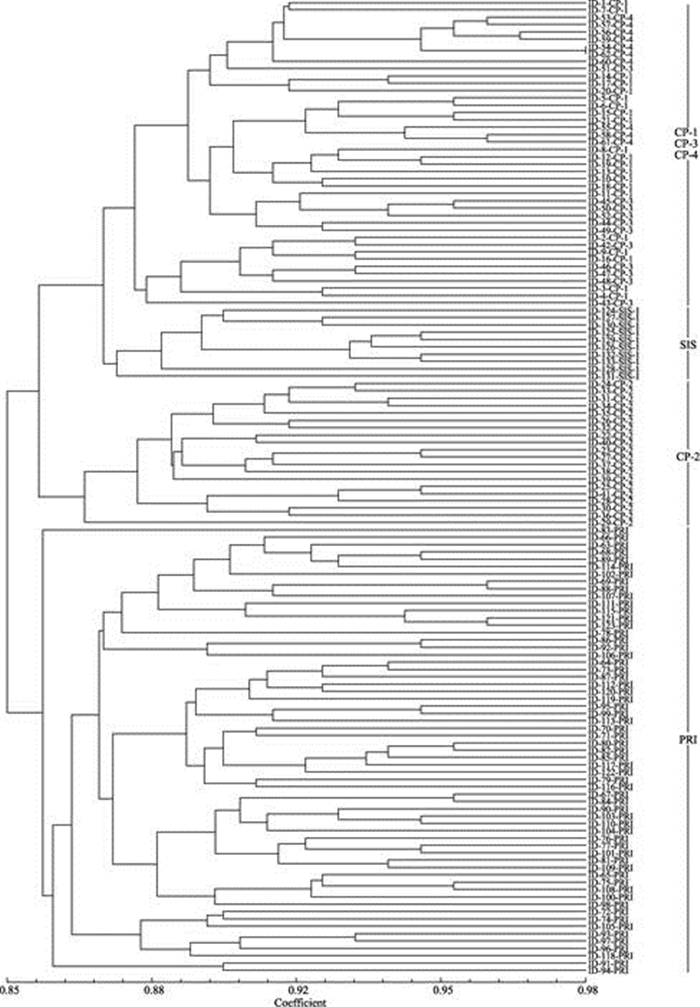
图 1是个体的聚类分析结果。所有来自于CP-1、CP-3和CP-4群体的凡纳滨对虾集中在第1分支。其余
|
图 1 个体水平聚类分析 Fig.1 The cluster analysis of individual L. vannamei from six populations |
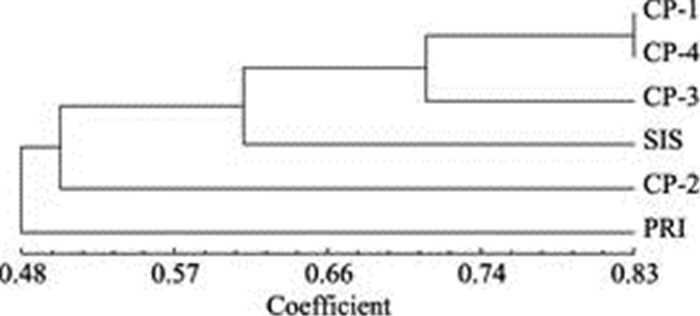
来自于CP-2、SIS和PRI群体的凡纳滨对虾分别集中于另外3个分支。除了个体水平,在群体水平也进行了聚类分析(图 2),在2个水平的聚类分析结果是相符的。6个群体间的遗传一致度和遗传距离结果见表 3。从表 3可以看出,遗传一致度为0.4229~0.8265。遗传距离为0.1905~0.8607。其中,CP-1和CP-4群体间的遗传一致度最高,遗传距离最近。CP-2和SIS群体间的遗传一致度最低,遗传距离最远。
|
图 2 群体水平聚类分析 Fig.2 The cluster analysis of six populations of L. vannamei |
|
|
表 3 遗传一致度和遗传距离 Tab.3 Genetic identity and genetic distance |
对6个凡纳滨对虾群体进行F-统计量分析(表 4)。结果表明,8个SSR位点的群体内近交系数(FIS)值为-0.0426~0.6943。多数位点的FIS > 0。FIS平均值是0.2514。8个SSR位点的总群体近交系数(FIT)值为0.0416~0.7665,FIT平均值为0.3889。8个SSR位点的遗传分化系数(FST)值为0.0808~0.3003。FST平均值为0.1837。
|
|
表 4 F-统计量分析 Tab.4 The F-statistics analysis |
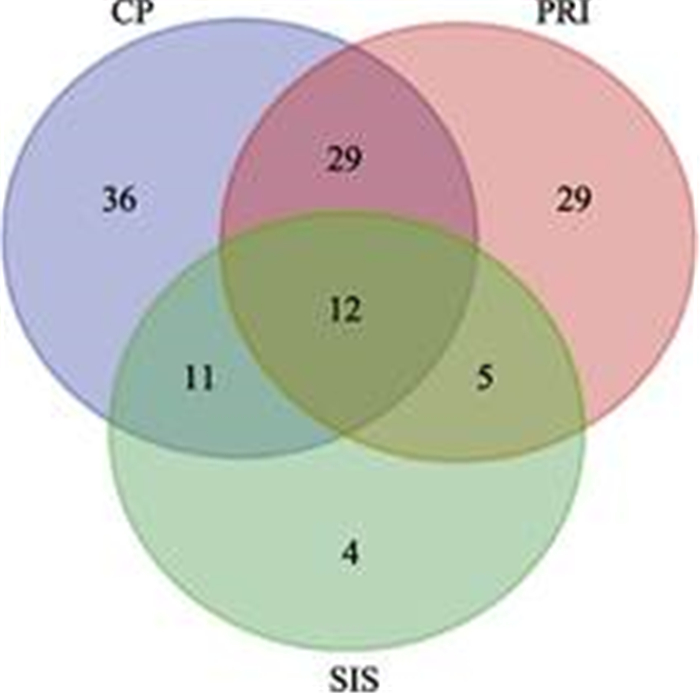
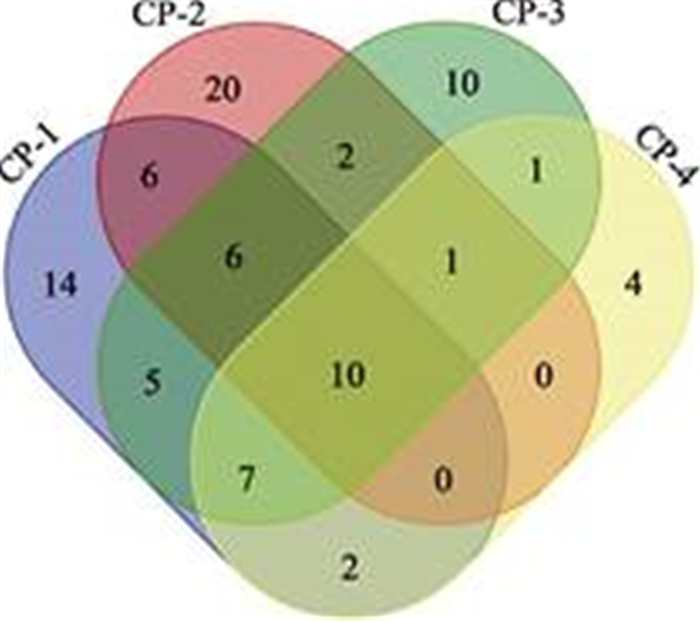
韦恩图(图 3)展示的是3个品牌凡纳滨对虾8个SSR位点的等位基因的分布情况。8个SSR位点共计获得等位基因个数是126。其中,只有12个等位基因类型是在3个品牌的凡纳滨对虾中都有发现。CP、PRI、SIS品牌的凡纳滨对虾各有36、29、4个等位基因类型是各自特有的,分别占各自所有等位基因类型的比例是40.9%、38.7%和12.5%。单独对CP的4个群体(CP-1、CP-2、CP-3和CP-4)进行分析(图 4),4个群体共计有88个等位基因类型,其中,4个群体共有的有10种。CP-1、CP-2、CP-3和CP-4群体各自拥有14、20、10、4种特有的等位基因,分别占各自所有等位基因类型的比例是28.0%、44.4%、23.8%和16.0%。
|
图 3 3个品牌间SSR等位基因类型分布 Fig.3 The allele distribution of SSRs of L. vannamei in the three different brands |
|
图 4 CP品牌的4个群体SSR等位基因类型分布 Fig.4 The allele distribution of SSRs of L. vannamei in the four populations from CP brand |
SSR标记广泛应用于生物的遗传多样性调查。在前期研究中,Xu等(2001)利用6对SSR引物对菲律宾的野生斑节对虾(Penaeus monodon)遗传多样性进行研究,结果显示,其中1个野生群体的遗传多样性要明显低于其他3个野生群体,研究结果为了解斑节对虾的野生资源情况提供了参考。Rumisha等(2017)利用SSR标记调查了斑节对虾的遗传多态性与当地水域金属污染水平的相关性,结果显示,遗传多态性的改变与金属污染存在显著的相关性。王军等(2018)利用SSR对中国对虾人工选育群体和野生群体进行比较分析,结果显示,人工选育群体与野生群体间发生弱遗传分化。
本研究中,Na平均值为3.67~9.33。在Perez-Enriquez等(2009)研究中,凡纳滨对虾SSR的Na平均值为8.98。Cruz等(2004)和Rezaee等(2016)的研究中,凡纳滨对虾SSR的Na值为5~10。上述研究结果和本研究结果相近。但是有趣的是,凡纳滨对虾与其他几种对虾相比,SSR的Na值显得偏低很多。例如,Wang等(2016)研究表明,中国对虾的Na为6~63。Zhang等(2015)研究表明,中国对虾SSR的Na平均值为36.5。Luan等(2006)研究表明,日本囊对虾(Marsupenaeus japonicas)SSR的Na平均值为7.5~ 13.5。Xu等(2001)关于斑节对虾的报道中,SSR的Na值为6~54。上述对虾种类都比凡纳滨对虾SSR的Na值高。考虑到凡纳滨对虾SSR位点普遍存在Na值偏低的情况,在进行遗传多样性分析时,最好能选用多态性高的SSR位点,或者适当增加分析位点的个数。已有报道中凡纳滨对虾SSR位点Na值偏低的现象是否与人工选择相关也值得探索。
聚类分析结果表明,来自6个群体的凡纳滨对虾主要分为4支。其中,来自CP-1、CP-3和CP-4群体的凡纳滨对虾组成第1分支,表明这3个群体来源的对虾遗传距离较近。来自CP-2、PRI和SIS群体的对虾分别聚在其他3个分支,提示这3个群体来源的对虾之间遗传距离较远。有趣的是,CP-1、CP-2、CP-3和CP-4群体都是来源于同一个品牌,但CP-2群体与CP-1、CP-3和CP-4群体的遗传距离较远,甚至比SIS群体与CP-1、CP-3和CP-4群体的距离还远,显示该品牌群体间遗传背景较为丰富,品牌内遗传距离甚至高于品牌间遗传距离。通过聚类分析结果可以看出,使用本研究的8对SSR引物,可以将不同品牌间的个体准确区分开。但同一个品牌的群体间个体,还不能被全部准确区分开,原因可能是这些群体的亲本亲缘关系太近。
本研究中,Ho值为0.12~0.71。8个位点的杂合度在不同群体中的差别较大。PIC值为0.36~0.76,SSR位点的多态性较高,达到中高度水平(Botstein et al, 1980),适合遗传多样性分析。FST平均值为0.1837,表明群体间已经达到高度遗传分化水平(0.15~0.25) (Balloux et al, 2002),18.37%的遗传分化存在于各群体间。
对于等位基因类型在不同品牌凡纳滨对虾中的分布情况,3个品牌共有的等位基因类型并不多。尤其是CP和PRI 2个品牌的凡纳滨对虾特有的等位基因类型比例较高。提示这2个品牌的对虾拥有更独特的遗传信息。但是与CP、PRI 2个品牌的对虾相比,SIS品牌的对虾特有的等位基因类型比例较低。CP品牌的4个群体中,CP-2特有的等位基因类型最高,达到44.4%,说明CP-2与其他3个CP品牌群体的遗传背景差异较大,这个结果和聚类分析的结果是一致的。假如在遗传选育中要将这6个群体引入基础选育群体,那么参考SSR分型结果,可将CP-1、CP-3、CP-4群体合并成1个群体看待,CP-2、SIS、PRI各自是3个独立群体。制定配种方案可优先考虑在这4个新定义的群体间进行个体间配对,尽量避免群体内部个体的配对。
已有研究表明,控制近交在对虾育种中的重要性。近交对于凡纳滨对虾生长、存活、抗病毒等性状具有显著影响(Moss et al, 2007)。在中国对虾的研究中同样也表明,近交对于生长和存活性状具有显著影响(Luo et al, 2014)。在没有系谱参考的情况下,经过几代人工选育群体内近交水平就可能达到极高水平。而遗传信息丰富的基础选育群体也是保证人工选育更好开展的前提。因此,在以未知遗传背景的群体作为选育对象时,借助分子标记技术对群体、个体间的分子遗传背景进行分析,可以为配种方案制定提供重要的参考。本研究中计算的FIS > 0,表明群体内存在杂合度降低现象;综合考虑Ho < He的结果,表明群体内已经存在一定程度的近交现象,提醒在人工选育过程中,可以适当增加基础群体的丰富性,避免遗传多样性的丢失,同时,要严格控制近交程度,避免性状衰退。
4 结论本研究表明,不同品牌来源的凡纳滨对虾间的分子遗传距离较远,利用SSR标记可以准确区分。但是相同品牌的对虾,即使来自于不同群体,个体间的分子遗传距离可能很近。本研究检测的群体内可能已经存在一定近交现象。在遗传选育或者生产扩繁苗种过程中,为了控制近交、提高遗传多样性,在缺少候选亲本遗传背景信息时,可以尝试利用SSR标记对群体或个体的分子遗传背景进行调查,并计算分子遗传距离用于配种方案参考。
Alcivar-Warren A, Meehan-Meola D, Park SW, et al. ShrimpMap: A low-density, microsatellite-based linkage map of the pacific whiteleg shrimp, Litopenaeus vannamei: Identification of sex-linked markers in linkage group 4. Journal of Shellfish Research, 2007, 26(4): 1259-1277 DOI:10.2983/0730-8000(2007)26[1259:SALMLM]2.0.CO;2 |
Balloux F, Lugon-Moulin N. The estimation of population differentiation with microsatellite markers. Molecular Ecology, 2002, 11(2): 155-165 DOI:10.1046/j.0962-1083.2001.01436.x |
Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314-331 |
Cruz P, Ibarra AM, Mejia-Ruiz H, et al. Genetic variability assessed by microsatellites in a breeding program of pacific white shrimp (Litopenaeus vannamei). Marine Biotechnology, 2004, 6(2): 157-164 |
Goyard E, Goarant C, Ansquer D, et al. Cross breeding of different domesticated lines as a simple way for genetic improvement in small aquaculture industries: heterosis and inbreeding effects on growth and survival rates of the Pacific blue shrimp Penaeus (Litopenaeus) stylirostris. Aquaculture, 2008, 278(1–4): 43-50 |
Liu P, Meng XH, He YY, et al. Genetic diversity in three wild populations of shrimp Fenneropenaeus chinensis in Yellow and Bohai Seas as revealed by microsatellite DNA. Oceanologia et Limnologia Sinica, 2004, 35(3): 252-257 |
刘萍, 孟宪红, 何玉英, 等. 中国对虾(Fenneropenaeus chinensis)黄、渤海3个野生地理群遗传多样性的微卫星DNA分析. 海洋与湖沼, 2004, 35(3): 252-257 DOI:10.3321/j.issn:0029-814X.2004.03.009 |
Luan S, Kong J, Wang QY. Genetic variation of wild and cultured populations of the Kuruma prawn Marsupenaeus japonicus (Bate 1888) using microsatellites. Aquaculture Research, 2006, 37(8): 785-792 DOI:10.1111/j.1365-2109.2006.01491.x |
Luo K, Kong J, Luan S, et al. Effect of inbreeding on survival, WSSV tolerance and growth at the postlarval stage of experimental full-sibling inbred populations of the Chinese shrimp Fenneropenaeus chinensis. Aquaculture, 2014, 420–421(3): 32-37 |
Meehan D, Xu Z, Zuniga G, et al. High frequency and large number of polymorphic microsatellites in cultured shrimp, Penaeus (Litopenaeus) vannamei (Crustacea: Decapoda). Marine Biotechnology (New York), 2003, 5(4): 311-330 DOI:10.1007/s10126-002-0092-z |
Meng XH, Kong J, Wang QY, et al. Study on seven geographic populations of prawn Fenneropenaeus chinensis based on microsatellite DNA. Marine Fisheries Research, 2008, 29(5): 1-10 |
孟宪红, 孔杰, 王清印, 等. 微卫星技术对黄、渤海海域7个不同地理群体中国对虾的遗传结构和遗传分化研究. 海洋水产研究, 2008, 29(5): 1-10 |
Moss DR, Arce SM, Otoshi CA, et al. Effects of inbreeding on survival and growth of Pacific white shrimp Penaeus (Litopenaeus) vannamei. Aquaculture, 2007, 272(S1): 30-37 |
Perez-Enriquez R, Hernández-Martínez F, Cruz P. Genetic diversity status of White shrimp Penaeus (Litopenaeus) vannamei broodstock in Mexico. Aquaculture, 2009, 297(1–4): 44-50 |
Rezaee S, Farahmand H, Nematollahi MA. Genetic diversity status of Pacific white shrimp (Litopenaeus vannamei) using SSR markers in Iran. Aquaculture International, 2016, 24(2): 479-489 DOI:10.1007/s10499-015-9939-y |
Rumisha C, Leermakers M, Elskens M, et al. Genetic diversity of the giant tiger prawn Penaeus monodon in relation to trace metal pollution at the Tanzanian coast. Marine Pollution Bulletin, 2017, 114(2): 759-767 |
Spielman D, Brook BW, Briscoe DA, et al. Does inbreeding and loss of genetic diversity decrease disease resistance. Conservation Genetics, 2004, 5(4): 439-448 DOI:10.1023/B:COGE.0000041030.76598.cd |
Valles-Jimenez R, Cruz P, Perez-Enriquez R. Population genetic structure of Pacific white shrimp (Litopenaeus vannamei) from Mexico to Panama: Microsatellite DNA variation. Marine Biotechnology, , 2004, 6(5): 475-484 DOI:10.1007/s10126-004-3138-6 |
Wang M, Wang W, Xiao G, et al. Genetic diversity analysis of spawner and recaptured populations of Chinese shrimp (Fenneropenaeus chinensis) during stock enhancement in the Bohai Bay based on an SSR marker. Acta Oceanologica Sinica, 2016, 35(8): 51-56 DOI:10.1007/s13131-016-0830-0 |
Wang J, Wang QY, Kong J, et al. SSR analysis on genetic diversity in breeding and wild populations of Fenneropenaeus chinensis. Progress in Fishery Sciences, 2018, 39(2): 104-111 |
王军, 王清印, 孔杰, 等. 中国明对虾人工选育群体与野生群体遗传多样性的SSR分析. 渔业科学进展, 2018, 39(2): 104-111 |
Xu Z, Primavera JH, de la Pena LD, et al. Genetic diversity of wild and cultured Black Tiger Shrimp (Penaeus monodon) in the Philippines using microsatellites. Aquaculture, 2001, 199(1): 13-40 |
Yu CF, Luan S, Zhang ZW, et al. Growth performance comparison of different inbreeding levels of families in Artemia sinica. Progress in Fishery Sciences, 2013, 34(6): 75-79 |
喻驰方, 栾生, 张志伟, 等. 中华卤虫不同近交水平家系生长性能比较. 渔业科学进展, 2013, 34(6): 75-79 DOI:10.3969/j.issn.1000-7075.2013.06.011 |
Zhao ZY, Liang LY, Bai LR. Analysis of genetic diversity among three wild populations of Penaeus monodon using microsatellite marker. Journal of Tropical Oceanography, 2018, 37(3): 65-72 |
赵志英, 梁丽运, 白丽蓉. 斑节对虾3个野生群体遗传多样性的微卫星标记分析. 热带海洋学报, 2018, 37(3): 65-72 |
Zhang K, Xiao G, Wang W, et al. Genetic variation analysis across six life periods in a natural population of the Chinese shrimp "Fenneropenaeus chinensis" in Bohai Bay using SSR markers. Russian Journal of Marine Biology, 2015, 41(1): 10-16 DOI:10.1134/S1063074015010113 |
Zhang L, Yang C, Zhang Y, et al. A genetic linkage map of Pacific white shrimp (Litopenaeus vannamei): Sex-linked microsatellite markers and high recombination rates. Genetica, 2007, 131(1): 37-49 |



