2. 上海海洋大学 水产科学国家级实验教学示范中心 上海 201306;
3. 上海海洋大学 中国科学技术部海洋生物科学国际联合研究中心 上海 201306
2. National Demonstration Center for Experimental Fisheries Science Education, Ministry of Science and Technology, Shanghai Ocean University, Shanghai 201306;
3. International Research Center for Marine Biosciences, Shanghai Ocean University, Shanghai 201306
真核生物基因组中存在大量的转座子元件,包含LINE1、Alu和SVA。迄今为止,3种逆转座子依旧具有活性:LINE1、Alu和SVA元素。LINE1逆转座子包含5'UTR、2个开放阅读框及具有PolyA尾的3'UTR (Dombroski et al, 1991)。LINE1属跳跃基因,占人类基因组约17%,为自主转座,通过RNA中间体自我传播;而Alu和SVA为非自主转座(Lander et al, 2001)。它们利用“复制-粘贴”机制,通过RNA中间体在整个基因组中繁殖,这一过程称为逆转录(Lu et al, 2016)。逆转座子的转座导致基因组改变,多数时候给基因组带来负面影响,如:改变基因组结构、影响基因表达、改变基因调控方式等(Gilbert et al, 2005; Tubio et al, 2014)。因此,宿主基因会通过启动子区域甲基化或者小RNA干扰等机制来抑制转座(Kinomoto et al, 2007; Levin et al, 2011)。但研究表明,转座元件与基因组是互益的(Faulkner et al, 2009),逆转座子的转座可导致新基因产生,这对于物种多样性和物种进化具有积极意义(Hamon et al, 2011; Volff et al, 2000)。
鱼类作为变温动物,水温变化在很大程度上影响其生理及行为(Perry et al, 2005)。南极大陆经历了漫长降温,水温常年处于0℃以下(Gordon, 2003),南极圈内与南极圈外同亚目鱼类相比较逆转座子的扩增倍率在8倍以上,有些甚至高达300倍(Chen et al, 2008)。这些基因可能参与了鱼类适应寒冷的过程,在漫长的历史进化中变化。同时,研究发现,在面对环境压力时,逆转座子LINE1的逆转座活性会受到环境因素影响(Butelli et al, 2012)。因此,推测寒冷可能会导致鱼类LINE1逆转座子扩增,并且可能与鱼类适应寒冷环境相关。
斑马鱼(Danio rerio)体型纤细,成体长为3~4 cm,对水质要求不高。孵出后约4个月达到性成熟,成熟鱼每隔几天可产卵一次,卵子体外受精,体外发育,胚胎发育速度快,胚体透明、子代多、遗传背景清楚,容易进行实验操作,易于在实验室内繁殖饲养,与人类基因87%相似(Watanabe et al, 2016),作为一种模式生物应用于生物学中(Howe et al, 2013; Sollars et al, 2003)。斑马鱼ZF4细胞来自孵化后1 d的斑马鱼胚胎(Driever et al, 1993),被广泛用于生物学实验中(Hu et al, 2015)。为研究低温对LINE1的影响,本研究检测了斑马鱼ZF4细胞18℃培养5 d和30 d、10℃培养3 h、6 h、1 d、3 d、5 d L1NE1 mRNA水平以及通过报告基因监测LINE1基因5'UTR在18℃ 3 d的活性变化,为研究寒冷环境下鱼类基因组中LINE1的表达变化奠定基础。
1 材料与方法 1.1 ZF4细胞和斑马鱼斑马鱼胚胎成纤维细胞ZF4购买于ATCC (American Type Culture Collection)。斑马鱼由实验室斑马鱼鱼房饲养,恒温循环水系统,水温为27℃~ 28℃,pH为6.8~7.8,用丰年虫(Artemia salina)饲养。
1.2 主要试剂和仪器胎牛血清和胰蛋白酶、DMEM︰F12液体培养基购于Gibco公司;T4连接酶和限制性内切酶购于New England;SYBR Green购于罗氏公司;Lipofectamine3000购于英潍捷基贸易有限公司;RT-qPCR反转录试剂盒购于宝生物工程(大连)有限公司;双荧光报告系统试剂盒购于Promega公司,引物由上海生工生物工程有限公司合成(表 1)。
|
|
表 1 PCR扩增引物 Tab.1 PCR amplification primers |
主要仪器:细胞低温培养箱(Galaxy170R, eppendorf)、LightCycler 480Ⅱ(罗氏);酶标仪(Bio-Rad公司);NanoDrop2000赛默飞世尔科技(中国)有限公司。
1.3 实时荧光定量PCR应用IDT引物设计软件(http://sg.idtdna.com/ sessionTimeout.aspx)设计RT-qPCR引物(LINE1-1, LINE1-1B, 表 1)。采用Trizol方法提取斑马鱼细胞和肌肉组织的RNA,使用TaKaRa反转录试剂盒(货号: RR047A)进行反转录实验,以反转录的cDNA为模板,进行SYBR Green荧光定量PCR实验。PCR条件:95℃预变性3 min;95℃变性30 s,58℃退火30 s,72℃延伸30 s,共35个循环;72℃延伸5 min。每个样品3个生物学重复,LINE1相对表达量的计算方法用2-ΔΔCt计算,β-actin作为内参。
1.4 载体构建及报告基因检测提取斑马鱼细胞基因组DNA,以基因组DNA为模板,上下游引物(L1-1,L1-1B, 表 1)分别引入酶切位点KpnⅠ和NheⅠ,利用PCR扩增基因5'UTR,KpnⅠ、NheⅠ酶切后用T4连接酶连接至载体PGL4.10中,将重组质粒转化到大肠杆菌内。
选择生长状态良好的ZF4细胞接种于6孔板中,次日细胞完全贴壁后弃除培养基,按Lipofectamine3000说明书进行转染。
采用Dual-Luciferase Reporter Assay System进行双荧光报告基因活性检测,按说明书进行测定。
1.5 统计分析方法统计学分析应用GraphPad Prism 5 (GraphPad software, 美国)软件分析。LINE1的表达数据和双荧光检测数据均来自3次独立重复实验,采用Student’s t-test方法分析统计学差异,P < 0.05表示具有显著性差异。
2 结果与分析 2.1 斑马鱼胚胎成纤维细胞(ZF4)和斑马鱼成鱼在低温处理下LINE1的表达变化有研究表明,长期生活在低温环境的南极鱼基因组与南极圈外同亚目鱼类的基因组相比较,LINE转座家族发生了8~300倍的扩增(Chen et al, 2008)。本研究选取LINE1的2个基因LINE1-1和LINE1-1B进行实时荧光定量PCR。在斑马鱼成鱼中,10℃为斑马鱼低温生存的临界温度,因此,选取10℃作为鱼类低温处理温度。与在常温(28℃)饲养的成鱼相比,在10℃低温处理3 h和6 h的成鱼肌肉组织中LINE1-1的mRNA水平(图 1A)有所下调,而在10℃低温处理1 d、3 d、5 d的成鱼肌肉组织中LINE1-1 mRNA水平显著上调,在成鱼中LINE1-1B的mRNA水平(图 1B)与LINE1-1的mRNA有相同趋势;在ZF4细胞中,选取18℃作为低温环境(Han et al, 2016),与常温(28℃)细胞对比,在18℃低温处理5 d的细胞中LINE1-1的mRNA水平有所下调,而在18℃低温处理30 d的细胞LINE1-1的mRNA水平显著上调(图 1C)。
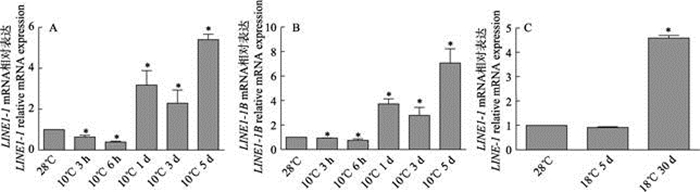
|
图 1 斑马鱼低温处理下LINE1表达的变化(*: P < 0.05.下同) Fig.1 Expression changes of LINE1 in zebrafish under low temperature treatment(*: P < 0.05. Same as below) |
实验室前期研究发现,低温环境会引起LINE1 CpG位点甲基化的改变(Han et al, 2016)。猜测低温环境会影响LINE1 5'UTR区域启动子的活性,以斑马鱼基因组DNA为模板,将2个LINE1基因的5'UTR区域进行PCR扩增(图 2)。
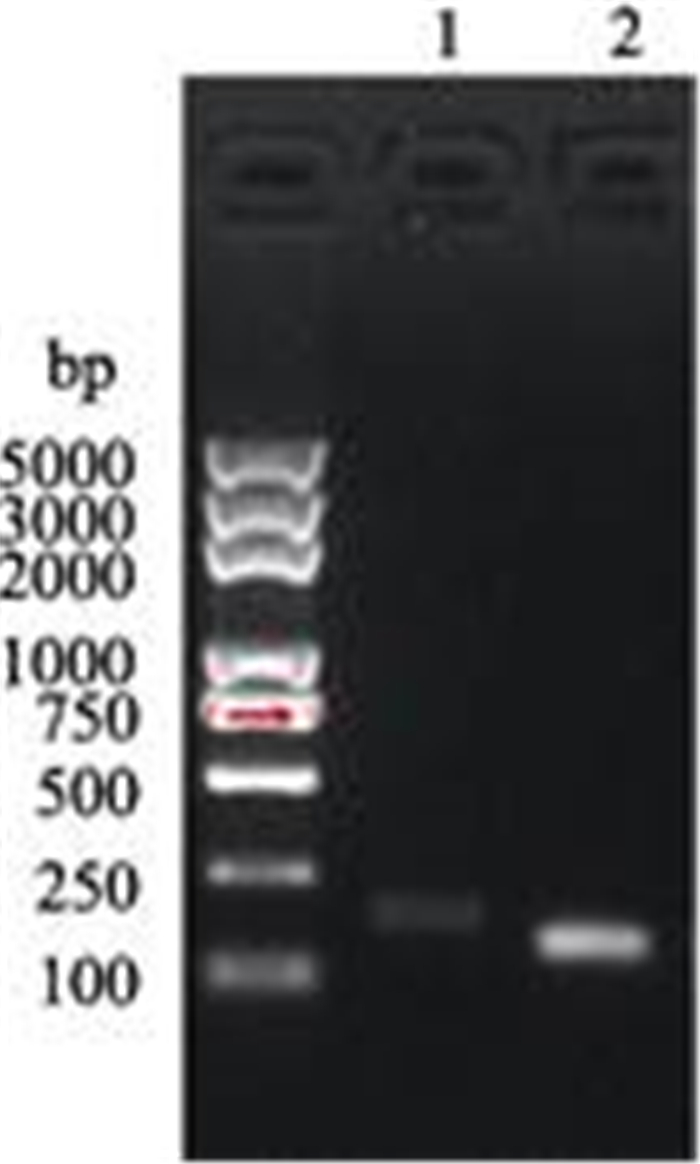
|
图 2 PCR产物琼脂糖凝胶电泳结果 Fig.2 The agarose gel electrophoresis results of PCR products 1: LINE1-1-promoter; 2: LINE1-1B-promoter |
载体PGL4.10-basic与PCR扩增片段经过KpnⅠ和NheⅠ双酶切后,在T4 DNA连接酶的作用下,将PCR扩增产物插入PGL4.10-basic载体中(图 3),将重组质粒送上海生工生物工程有限公司测序,测序结果与PCR扩增的基因序列相同,重组质粒构建成功。
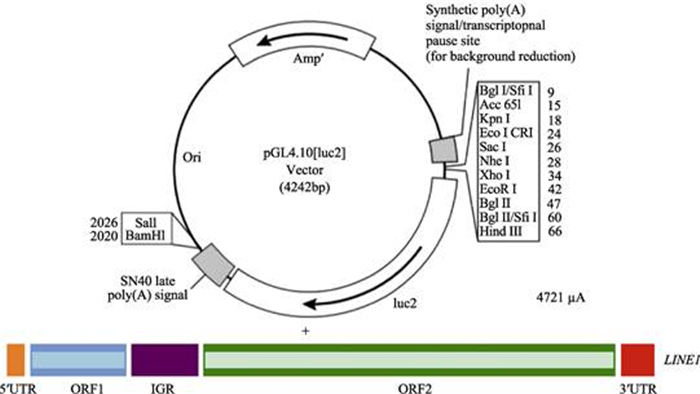
|
图 3 PGL4.10-LINE1 5'UTR质粒构建 Fig.3 Plasmid construction map of PGL4.10-LINE1 5'UTR |
通过检测相对荧光素酶活性,间接检测LINE1 5'UTR区域启动子活性,LINE1 5'UTR区域启动子活性越高,荧光素酶活性发光值越高。海肾荧光素作为检测系统内参,当LINE1 5'UTR区域启动子没有生物学活性时,不能检测到荧光素酶活性发光值。将构建的重组质粒转染进常温(28℃)培养ZF4细胞中,结果显示,与阴性对照PGL4.10-basic相比,重组质粒均能检测到荧光素酶活性发光值,而PGL4.10-basic不能检测到荧光素酶发光值,表明LINE1 5'UTR具有启动子活性(图 4A)。将重组质粒与PGL4.10-basic和PGL3- promoter转染到18℃低温处理3 d的ZF4细胞中,PGL4.10-basic为阴性对照,PGL3-promoter为阳性对照,结果显示,与常温(28℃)细胞相比,重组质粒在低温处理下荧光素酶活性发光值均有不同程度的下降,表明短期低温处理会影响LINE1基因5'UTR区域启动子活性(图 4B)。
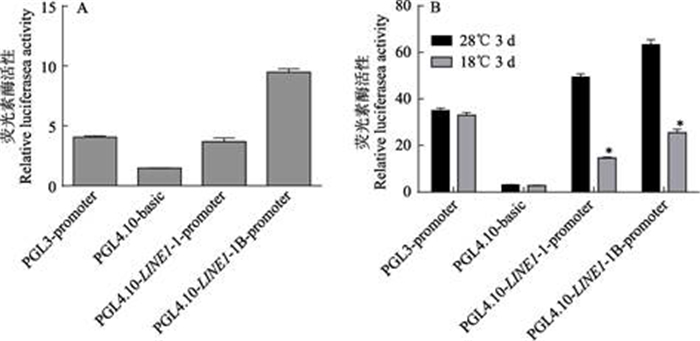
|
图 4 重组质粒PGL4.10-LINE1-promoter转染斑马鱼细胞后双荧光素酶相对活性分析 Fig.4 Analysis of relative activity of dual luciferase after transfection of zebrafish cells with recombinant plasmid PGL4.10-LINE1-promoter |
LINE1属于最丰富的一类自主转座因子。尽管多数LINE1被抑制活性,但依旧有很少一部分的LINE1具有活性,LINE1的插入虽与疾病相关,但LINE1同样影响基因组的进化(Beck et al, 2011)。当面对环境因素刺激时,基因组进行自身进化诱导转座子转座(McClintock, 1984),使生物对环境变化做出适应性改变。在之前研究中发现,南极圈内物种与南极圈外物种转座子扩增效率存在巨大差异。在西西里血橙中,寒冷诱导逆转座子的扩增(Butelli et al, 2012)。这提示LINE1的扩增可能与生物体对长期寒冷环境的适应相关。为了研究逆转座子在长期低温环境下的大量扩增是否与生物体对寒冷环境的适应相关,将ZF4细胞培养在18℃ 5 d,发现ZF4细胞生长明显停滞,而后20 d左右逐渐恢复生长状态,尽管相比28℃依旧缓慢。本研究发现,短期低温处理诱导斑马鱼成鱼及斑马鱼ZF4细胞LINE1的mRNA水平下调,长期低温处理诱导斑马鱼成鱼及斑马鱼ZF4细胞LINE1的mRNA水平显著上调,推测LINE1对生物体适应外界低温环境压力可能有积极作用。生物体在寒冷环境下转座子转座水平升高,可能增加生物体基因型的多样性,使其对环境压力产生适应性进化。
在自然界中遵循着优胜劣汰的法则,从生物的进化开始,生物体内有益于适应环境、使生物体更好生存下去的基因会保留,垃圾基因会逐渐被淘汰,那么在长期寒冷环境中,L1的显著扩增是否是生物体为了适应寒冷环境而选择的一种生物途径?在之前研究中发现,LINE1启动子甲基化水平在低温环境下有所改变:18℃低温处理ZF4细胞5 d,LINE1启动子甲基化水平升高,而低温处理30 d时,甲基化水平降低(Han et al, 2016)。生物体会通过甲基化水平来调节LINE1表达水平,LINE1与甲基化水平呈负相关关系(Hata et al, 1997),因此,甲基化水平升高时,LINE1被抑制,LINE1 mRNA水平呈下降趋势;当甲基化水平降低时,LINE1激活,LINE1 mRNA水平呈上升趋势。据此推测,低温环境会通过影响LINE1 5'UTR区域启动子活性,进而影响LINE1的扩增。构建重组质粒发现,低温确实能引起LINE1 5'UTR区域启动子活性改变,推测这种调节可能是生物体应对外界低温环境压力的方式之一,L1对生物体适应外界低温环境压力可能有积极作用。生物体在寒冷环境下转座子转座水平升高,可能增加生物体基因型的多样性,使其对环境压力产生适应性进化。
总之,本研究通过对斑马鱼细胞以及斑马鱼成鱼不同时间的低温处理,研究LINE1的表达变化,为更好了解LINE1在鱼类寒冷适应中的作用机制奠定了基础。
Beck CR, Garcia-Perez JL, Badge RM, et al. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics, 2011, 12: 187-215 DOI:10.1146/annurev-genom-082509-141802 |
Butelli E, Licciardello C, Zhang Y, et al. Retrotransposons control fruit-specific, cold-dependent accumulation of anthocyanins in blood oranges. Plant Cell, 2012, 24(3): 1242-1255 DOI:10.1105/tpc.111.095232 |
Chen Z, Cheng CH, Zhang J, et al. Transcriptomic and genomic evolution under constant cold in Antarctic notothenioid fish. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(35): 12944-12949 DOI:10.1073/pnas.0802432105 |
Dombroski BA, Mathias SL, Nanthakumar E, et al. Isolation of an active human transposable element. Science, 1991, 254(5039): 1805-1808 DOI:10.1126/science.1662412 |
Driever W, Rangini Z. Characterization of a cell line derived from zebrafish (Brachydanio rerio) embryos. In vitro Cellular & Developmental Biology. Animal, 1993, 29(9): 749-754 |
Faulkner GJ, Carninci P. Altruistic functions for selfish DNA. Cell Cycle (Georgetown, Tex.), 2009, 8(18): 2895-2900 DOI:10.4161/cc.8.18.9536 |
Gilbert N, Lutz S, Morrish TA, et al. Multiple fates of L1 retrotransposition intermediates in cultured human cells. Molecular and Cellular Biology, 2005, 25(17): 7780-7795 DOI:10.1128/MCB.25.17.7780-7795.2005 |
Gordon AL. Oceanography: The brawniest retroflection. Nature, 2003, 421(6926): 904-905 DOI:10.1038/421904a |
Hamon P, Duroy PO, Dubreuil-Tranchant C, et al. Two novel Ty1-copia retrotransposons isolated from coffee trees can effectively reveal evolutionary relationships in the Coffea genus (Rubiaceae). Molecular Genetics and Genomics: MGG, 2011, 285(6): 447-460 DOI:10.1007/s00438-011-0617-0 |
Han B, Li W, Chen Z, et al. Variation of DNA methylome of zebrafish cells under cold pressure. PLoS One, 2016, 11(8): e0160358 DOI:10.1371/journal.pone.0160358 |
Hata K, Sakaki Y. Identification of critical CpG sites for repression of L1 transcription by DNA methylation. Gene, 1997, 189(2): 227-234 DOI:10.1016/S0378-1119(96)00856-6 |
Howe K, Clark MD, Torroja CF, et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature, 2013, 496(7446): 498-503 DOI:10.1038/nature12111 |
Hu P, Liu M, Zhang D, et al. Global identification of the genetic networks and cis-regulatory elements of the cold response in zebrafish. Nucleic Acids Research, 2015, 43(19): 9198-213 DOI:10.1093/nar/gkv780 |
Kinomoto M, Kanno T, Shimura M, et al. All APOBEC3 family proteins differentially inhibit LINE-1 retrotransposition. Nucleic Acids Research, 2007, 35(9): 2955-2964 DOI:10.1093/nar/gkm181 |
Lander ES, Linton LM, Birren B, et al. Initial sequencing and analysis of the human genome. Nature, 2001, 409(6822): 860-921 DOI:10.1038/35057062 |
Levin HL, Moran JV. Dynamic interactions between transposable elements and their hosts. Nature Reviews. Genetics, 2011, 12(9): 615-627 DOI:10.1038/nrg3030 |
Lu XJ, Xue HY, Qi XL, et al. LINE-1 in cancer: Multifaceted functions and potential clinical implications. Genetics in Medicine, 2016, 18(5): 431-439 DOI:10.1038/gim.2015.119 |
McClintock B. The significance of responses of the genome to challenge. Science, 1984, 226(4676): 792-801 DOI:10.1126/science.15739260 |
Perry AL, Low PJ, Ellis JR, et al. Climate change and distribution shifts in marine fishes. Science, 2005, 308(5730): 1912-1915 DOI:10.1126/science.1111322 |
Sollars V, Lu X, Xiao L, et al. Evidence for an epigenetic mechanism by which Hsp90 acts as a capacitor for morphological evolution. Nature Genetics, 2003, 33(1): 70-74 DOI:10.1038/ng1067 |
Tubio JMC, Li Y, Ju YS, et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science, 2014, 345(6196): 1251343 |
Volff JN, Korting C, Schartl M. Multiple lineages of the non- LTR retrotransposon Rex1 with varying success in invading fish genomes. Molecular Biology and Evolution, 2000, 17(11): 1673-1684 DOI:10.1093/oxfordjournals.molbev.a026266 |
Watanabe E, Mano S, Nomoto M, et al. HSP90 stabilizes auxin-responsive phenotypes by masking a mutation in the auxin receptor TIR1. Plant & Cell Physiology, 2016, 57(11): 2245-2254 |



