2. 中国水产科学研究院黄海水产研究所 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
4. 青岛市西海岸新区渔业技术推广站 青岛 266400
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071;
4. Aquatic Technology Extension Station of Qingdao West Coast New Economic District, Qingdao 266400
随着环保风暴和民众食品质量安全意识的提高,对耐药菌及耐药基因的研究逐步成为热点。目前,国内外耐药菌和耐药基因的相关研究主要集中在畜禽类粪便(Mu et al, 2015; 张昊等, 2018; Brooks et al, 2014)和环境介质等方面(Lin et al, 2011; Ji et al, 2012),针对海水养殖环境下,养殖动物体内的耐药菌及耐药基因(Antimicrobial resistant genes, ARGs)方面的研究较少,而在刺参(Apostichopus japonicus)方面的相关研究还属于空白。近年来,刺参养殖在我国北方沿海地区发展迅速,并成为沿海地区重要的支柱性产业,为沿海渔业经济发展提供了重要途径。但是随着产业的快速发展,由于养殖过程中的不规范操作以及近岸环境污染等原因,导致疾病频发,病害成为海水养殖产业可持续发展的主要瓶颈。使用抗生素成为养殖者进行病害防控的重要手段,而抗生素的大量使用不仅严重影响养殖生物的质量和安全,也极大地破坏了邻近海区的生态环境,导致水质环境恶化、药物残留、细菌耐药以及微生态结构失衡(党宏月等, 2006; 赵小慧等, 2019; Niu et al, 2016; Zhang et al, 2012)。据统计,2012年我国抗生素年产量约21万t,其中,有46.1%的抗生素用于禽畜及水产养殖业;2013年这一比例上升至52%,且由于吸收和代谢不完全等释放到河流水体中的抗生素达到了5.38万t (Zhang et al, 2015)。此外,食品源动物体内残留的抗生素经食物链的富集作用也严重威胁人类的健康与安全。2016年刺参抗生素事件曾引发广泛的社会关注。除了对于耐药菌株的关注外,由于耐药基因具有持久性存在和可转移性的特性(Tamminen et al, 2011),耐药基因可通过质粒水平转移到其他非致病菌或条件致病菌中(张骞月等, 2015),对水产养殖业和人类健康也存在潜在的威胁(罗义等, 2008; 王瑞旋等, 2010),因此,亟需对养殖刺参环境及肠道的耐药菌现状进行监测。
刺参的养殖主要分为保苗期和池塘养成期2个时期,保苗期具有生长环境可控的特点,是刺参整个生长周期中的用药高峰期。目前,刺参苗种培育区主要包括辽宁和山东2个省区,其中,山东省苗种产量占全国苗种供应量的60%以上(中国渔业统计年鉴, 2018)。研究调查发现,目前刺参养殖常用抗生素包括四环素、萘啶酸、氟苯尼考、乙酰甲喹、庆大霉素和链霉素。鉴于此,本研究选取山东省3个主要的刺参主养区的6家育苗场,开展6个采样点刺参苗种肠道中耐药菌数量、种类及耐药基因丰度的检测,以期为解析刺参苗期抗生素耐药菌及耐药基因的现状和防控刺参养殖过程中耐药细菌的产生提供科学依据。
1 材料与方法 1.1 样品采集2018年1~4月,在山东省沿海3个刺参主养区(青岛、烟台和威海)选取6个育苗场采集10月龄的幼参,采样位点信息见图 1。每个采样点各随机采集50头刺参带回实验室,参苗规格为(12.35±5.60) g/头,活体解剖后,获取其肠道内容物,混匀后立即进行细菌培养,其余样品于–20℃保存。
|
图 1 采样点的地理位置 Fig.1 Geographical locations of samples collection |
取1 g肠道内容物样品加入1.5%的灭菌NaCl溶液制成10 ml悬液,然后10倍梯度稀释后分别涂布于灭菌的不含抗生素的2216E平板和分别含单种抗生素的2216E平板上,各种抗生素的浓度参考CLSI(2016)标准(Patel et al, 2016)和畜禽及水产动物耐药菌筛选的终浓度进行设定(李壹等, 2016; 谭爱萍等, 2019),分别为四环素(50 μg/ml)、萘啶酸(50 μg/ml)、乙酰甲喹(50 μg/ml)、氟苯尼考(50 μg/ml)、庆大霉素(32 μg/ml)和链霉素(50 μg/ml)。28℃培养24 h后,根据相应平板上的菌落数计算可培养细菌总量及耐药菌数量,按照以下公式计算相应抗生素耐药菌所占比例。
耐药细菌比例(%)=耐药细菌数/可培养细菌总数× 100%
1.3 抗生素耐药菌分离纯化及鉴定根据相应抗生素平板上菌落形态(菌落大小、边缘形状、颜色和透明度等)对所筛选得到的耐药菌进行分类和优势度统计后,用接种环挑取优势菌的单菌落,在2216E固体培养基进行2次纯化培养和保种。用细菌DNA提取试盒(TIANamp Bacteria DNA Kit)提取所分离耐药菌的细菌DNA,利用细菌16S rDNA通用引物(27F: 5′-AGAGTTTGATC(C/A)TGGCTCAG-3′, 1492R: 5′-GGTTACCTTGTTACGACTT-3′)扩增16S rDNA序列。PCR扩增反应体系为50 μl,扩增程序:94℃预变性5 min;30个循环:94℃变性60 s,55℃复性60 s,72℃延伸80 s;最后,72℃温育10 min。PCR产物用1%琼脂糖凝胶电泳检测合格后送交生工生物工程(上海)股份有限公司进行纯化、双向测序,测序结果在EzBioCloud数据库(http://www.ezbiocloud.net)中比对,确定相应的菌株分类地位。
1.4 肠道中耐药基因丰度检测取0.5 g肠道内容物,用E.Z.N.A.TM Soil DNA Kit (OMEGA, 美国)提取总DNA,操作步骤参照试剂盒说明书,DNA的完整性和纯度用1.0%琼脂糖凝胶电泳检测,利用Thermo Nanodrop 2000微量分光光度计(Thermo Scientific, 美国)测定浓度,检测合格的DNA用于荧光定量PCR检测。
针对四环素类、喹诺酮类、氯霉素类和氨基糖苷类这4类抗生素选取7种常见耐药基因进行定量分析,引物信息见表 1。利用PCR对目的片段进行扩增、纯化后,连接到pMD18-T vector (TaKaRa),之后转化到大肠杆菌DH5α感受态细胞进行克隆。利用Plasmid Mini Kit I Protocol (OMEGA, 美国)进行重组质粒的提取,以所提取的质粒为模板进行实时荧光定量PCR (qRT-PCR)扩增,构建相应基因扩增的标准曲线,利用Eppendrof实时荧光定量PCR仪自带的软件程序计算各基因扩增的Ct值、扩增效率(E)及其相关系数(R2)。采用qRT-PCR测定6个采样点样品中7种耐药基因和内参基因含量,每个样品设置3个平行。qRT-PCR反应体系如下:10 μl SYBR Premix Ex Taq TMⅡ(2×) (TaKaRa),上、下游引物(20 μmol/L)各0.8 μl,5 ng DNA模板(约5 ng),总体积为20 μl。qRT-PCR反应程序:95℃ 1min;95℃ 10 s,Tm 30 s,72℃ 30 s,40个循环。反应结束后,测定熔解曲线。根据标准曲线计算各基因拷贝数的绝对含量,并以各耐药基因的绝对拷贝数和16S rDNA绝对拷贝数的比值来计算各耐药基因的相对丰度。
|
|
表 1 耐药基因及16S rDNA引物信息 Tab.1 Primer sequences for antibiotic resistance genes and 16S rDNA |
利用SPSS 19.0软件对四环素、萘啶酸、氟苯尼考、庆大霉素和链霉素5种抗生素耐药菌所占比例与7种耐药基因的相对拷贝数进行线性回归分析,以P < 0.05作为差异显著水平,确定耐药菌所占比例和相关耐药基因相对拷贝数的相关性。
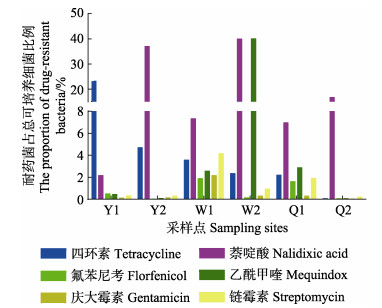
2 结果 2.1 可培养抗生素耐药菌的数量分析山东地区的3个刺参主养区6个采样点刺参苗种肠道中可培养细菌与耐药菌培养结果见表 2。由结果可知,6个采样点刺参苗种肠道中可培养细菌总数为1.37×105~6.21×106 CFU/g。6种抗生素耐药菌的数量分别为:四环素耐药菌的数量为(3.00±0.50)×103~(1.15± 0.30)×105 CFU/g;萘啶酸耐药菌的数量为(7.77±0.61)× 103~(1.04±0.21)×106 CFU/g;氟苯尼考耐药菌的数量为(2.33±0.59)×102~(5.17± 0.33)×104 CFU/g;乙酰甲喹耐药菌的数量为(1.63±0.29)×103~(4.31±0.23)×105 CFU/g;庆大霉素药菌的数量为(4.00±0.09)×102~(5.90±0.71)× 104 CFU/g;链霉素耐药菌的数量为(1.17±0.62)×103~ (1.13±0.20)×105 CFU/g。根据相应结果计算耐药菌在总可培养细菌中的占比(图 2)。由图 2可以看出,同一个采样点刺参肠道内容物中的不同抗生素耐药菌占比差异显著。Y1、Y2、Q2单个采样点均表现出单一种类抗生素耐药菌丰度高的特点,相应耐药菌种类分别为四环素耐药菌(23.15%)、萘啶酸耐药菌(37.04%)和萘啶酸耐药菌(16.76%)。W2采样点中萘啶酸耐药菌和乙酰甲喹耐药菌的占比均很高,分别达到39.94%和40.06%。W1和Q1采样点虽然有多种抗生素耐药菌的检出,但是占比均较低,检出率最高的萘啶酸耐药菌的比例也仅为7.32%和6.95%。所有采样点中氟苯尼考、庆大霉素和链霉素耐药菌的占比均不高,占比范围为0.01%~4.15%。
|
|
表 2 不同采样点可培养细菌及耐药细菌数量(CFU/g) Tab.2 The number of total cultured bacteria and drug-resistant bacteria at different sampling sites (CFU/g) |
|
图 2 各采样点刺参肠道中不同抗生素耐药菌占总可培养细菌比例 Fig.2 The proportion of drug-resistant bacteria in the intestinal of sea cucumber accounted to the total culturable bacteria |
根据含抗生素2216E培养基上形成的菌落形态、颜色、形状、透明度等表型形态指标,对筛选得到的耐药菌进行排重,选取目标菌落分离纯化,用于菌株保藏及分类地位鉴定,结果见表 3。由表 3可知,共获得98株耐药菌株,其中,四环素、萘啶酸、氟苯尼考、乙酰甲喹、庆大霉素和链霉素耐药菌株分别为18、22、10、14、11和23株。依据16S rDNA序列的EzBioCloud数据库比对结果,可以判断耐药菌分别分布在30个属。属水平统计结果显示,6个采样点所获的耐药菌株分别分布到6、7、13、6、14和8个属中;所分离到的98株耐药菌中,检出率最高的3个属为弧菌属(Vibrio)、芽孢杆菌属(Bacillus)和嗜冷杆菌属(Psychrobacter),分别检出15株、13株和12株,占耐药菌株总数的15.30%、13.27%和12.25%。
|
|
表 3 不同采样点抗生素培养基筛选到的耐药菌株数及属水平上的种类数 Tab.3 Number of strains and genera of drug-resistant bacteria screened by antibiotic culture medium at different sampling sites |
|
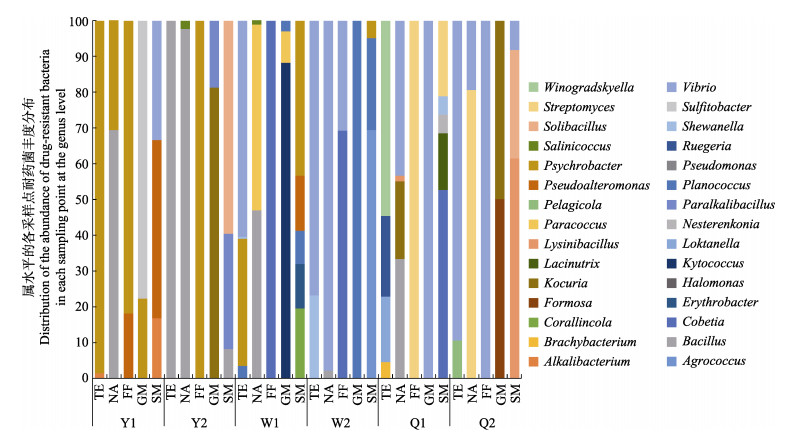
图 3 基于属水平的各采样点耐药菌丰度分布 Fig.3 Distribution of drug-resistant bacteria abundance at each sampling site based at genus level |
鉴于16S rDNA鉴定结果在属水平上的准确性高,根据相应菌株在属水平上耐药菌的分类地位和在抗生素平板上的优势度,统计属水平上各采样点不同抗生素耐药菌种类的丰度分布(图 3)。各采样点耐药菌种类差异较大,其中,Y1采样点的耐药菌种类丰度较高的菌为嗜冷杆菌属和假交替单胞菌属(Pseudoalteromonas),Y2采样点耐药菌种类主要为芽孢杆菌属和嗜冷杆菌属,W2采样点耐药菌种类主要为弧菌属和动性球菌属(Planococcus),Q2采样点耐药菌种类主要为弧菌属,以上4个采样点都存在同一个属的不同菌株耐多种抗生素的情况。W1和Q1这2个采样点耐药菌种类较多;在W1采样点同一种抗生素耐药菌株的多样性较高;在Q1采样点耐氟苯尼考、乙酰甲喹和庆大霉素3种抗生素的耐药菌株各集中于单一属的细菌中,而耐四环素、萘啶酸和链霉素的耐药菌株的多样性较高,分布在4~5个属中。
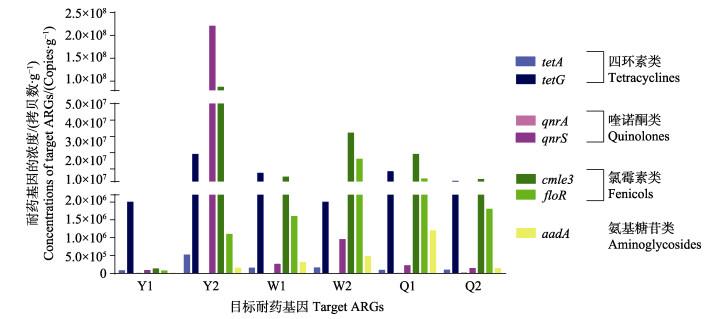
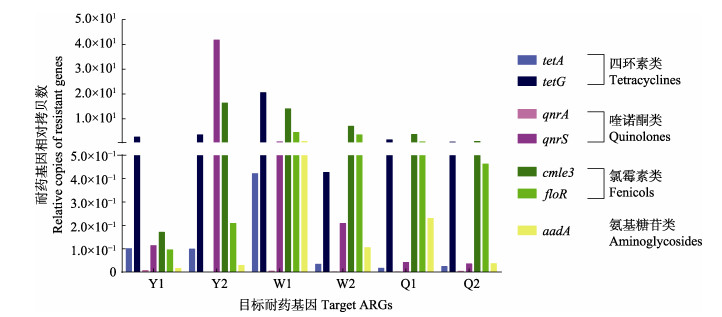
2.3 各样品中耐药基因丰度分析对刺参肠道中的四环素类、喹诺酮类、氯霉素类和氨基糖苷类共7种抗生素耐药基因的标准曲线公式见表 1,对7个基因的绝对含量和相对于16S rDNA的相对含量进行测算,结果见图 4和图 5。所检测的2个四环素耐药基因tetA和tetG拷贝数绝对含量分别为7.63×104~5.24×105和1.95×106~1.91×107 copies/g,相对16S rDNA的含量分别为1.69×10–2~1.01×10–1,同一采样点tetG拷贝数是tetA拷贝数的12~94倍。所检测的2个喹诺酮类耐药基因qnrA和qnrS的拷贝数绝对含量分别为1.71×103~1.51×104和8.57×104~2.21×108 copies/g,相对16S rDNA的含量分别为4.09×10–4~7.87×10–3和3.53× 10–2~4.18×101,同一采样点qnrS拷贝数是qnrA拷贝数的9~1.02×105倍。所检测的2个氯霉素类耐药基因cmle3和floR的拷贝数绝对含量分别为1.29×105~ 8.66×107和7.25× 104~1.64×107 copies/g,相对16S rDNA的含量分别为0.17~16.4和0.28~4.56,同一采样点cmle3拷贝数是floR拷贝数的1.77~78.7倍。氨基糖苷类耐药基因aadA的拷贝数绝对含量为1.18×104~1.20×106 copies/g,相对16S rDNA的含量为1.56×10–2~ 8.73×10–1。
|
图 4 刺参肠道中目标耐药基因的浓度 Fig.4 Concentrations of target ARGs in the intestinal of sea cucumber |
|
图 5 刺参肠道中目标耐药基因的相对拷贝数 Fig.5 The relative abundance of antibiotic resistance genes in the intestinal of sea cucumber |
针对四环素、萘啶酸、氟苯尼考、庆大霉素和链霉素5种药物作用下耐药菌所占比例与对应的7种耐药基因的相对拷贝数(基因绝对拷贝数与16S rDNA基因绝对拷贝数的比值)分别做线性回归分析(表 4)。结果显示,基因aadA相对拷贝数比例和链霉素以及庆大霉素耐药菌占比之间存在显著相关性(P < 0.05)。除此之外,tetA和tetC与四环素耐药菌占比、qnrA和qnrS与萘啶酸耐药菌占比、cmle3和floR与氟苯尼考耐药菌占比之间相关性不显著(P > 0.05)。
|
|
表 4 耐药菌占比与耐药基因相对含量的相关性分析 Tab.4 Analysis of correlation between the percentage of antibiotic-resistant bacteria and the relative abundance of antibiotic resistance genes |
水产养殖已经成为我国北方沿海重要产业,养殖过程的疾病频发导致了抗生素的广泛使用。养殖动物肠道微生物在抗生素的选择压力下,易产生耐药性,多项研究证实,抗生素耐药菌已普遍存在养殖环境中(张昊等, 2018;王岚等, 2017; 左志晗等, 2018)。本研究对山东地区3个刺参主产区的刺参苗种肠道中6种抗生素耐药菌进行了调查,不同采样点均获得了6种抗生素耐药菌,说明耐药现象在刺参苗期养殖中普遍存在。6种抗生素中,乙酰甲喹、萘啶酸和四环素耐药菌的数量较高,最高值分别达到了4.31×105、1.04×106和1.15×105 CFU/g,占可培养细菌总数的40.06%、39.94%和23.15%,而氟苯尼考、庆大霉素和链霉素耐药菌的占比相对较低。李壹等(2016)对山东地区海水养殖环境中的耐药菌进行了调查,发现在山东海水养殖区存在一定程度的抗生素耐药性菌,四环素耐药菌的比例为22.8%~49.0%,而氟苯尼考占比为0.1%~ 7.1%,说明养殖刺参肠道中的耐药菌与沿海海水环境中的耐药菌相似性较高,可能是由于养殖生物排泄物排入外海以及外海海水引入养殖系统再次使用的交互作用引起的。从3个地区6个采样点的耐药菌占比分布特点还可以看出,同一个地区不同采样点各抗生素耐药菌占比存在很大差异,如Y2中萘啶酸耐药菌丰度显著高于Y1,W2中萘啶酸和乙酰甲喹耐药菌的占比显著高于W1,相对于Q1耐药菌种类较多,Q2中仅有萘啶酸耐药菌是优势耐药菌,这些情况说明,刺参肠道中耐药菌的组成和分布除了受外海海水环境影响外,与不同养殖业者各自的养殖管理和用药习惯存在很大关系。
3.2 幼参肠道中耐药菌的分类特征据98株抗生素耐药菌的16S rDNA鉴定分类结果可以看出,耐药菌检出率排序:弧菌属 > 芽孢杆菌属 > 嗜冷杆菌属 > 假交替单胞菌属。这一结果与Dang等(2007)研究发现的弧菌是海水养殖环境最具优势的耐药菌属结论一致。弧菌和假交替单胞菌均是刺参主要的致病菌(王印庚等, 2006; 逄慧娟等, 2017),刺参养殖生产中大量使用的抗生素主要针对病原性弧菌和假交替单胞菌,药物的长期使用易导致其在抗生素选择的压力下耐药性的产生。芽孢杆菌属细菌作为水产养殖中重要的益生菌,被广泛应用于水环境和养殖动物肠道菌群调控(Pandiyan et al, 2013),本研究中,芽孢杆菌属细菌在耐药菌中占较高的优势,可能与养殖环境中大量使用芽孢杆菌属的益生菌,再利用抗生素进行疾病防控,导致芽孢杆菌属细菌产生耐药性有关。李研东(2004)利用体外耐药性诱导实验诱导出对氟喹诺酮类药物耐药的蜡样芽孢杆菌(Bacillus cereus),说明芽孢杆菌的耐药性与抗生素的诱导作用息息相关。因此,在养殖生产中益生菌和抗生素的使用应设置相应的时间差,以降低因抗生素使用带来的耐药益生菌产生风险。
3.3 幼参肠道耐药基因的分布特征鉴于耐药基因具有存在的持久性和可转移性,处于可移动元件上的耐药基因可转移到其他非致病菌或条件致病菌中,较多学者通过检测ARGs评估耐药风险。在智利三文鱼养殖场(Miranda et al, 2003)、泰国综合养鱼场(Agersø et al, 2007)、澳大利亚水产养殖场(Hoa et al, 2008)和日本金枪鱼养殖场(Furushita et al, 2003)均已经检测到多种ARGs。我国对水产养殖环境、养殖废水和养殖生物(Dang et al, 2006、2007; Su et al, 2017; Gao et al, 2012; Wen et al, 2016; Huang et al, 2017)中的耐药基因检测结果表明,水产养殖环境及养殖动物体内也存在多种ARGs。本研究所检测的7种抗生素耐药基因在刺参肠道中均被检出,进一步证明了相应耐药菌株的存在。但同一个采样点同一类抗生素的不同耐药基因的绝对含量却存在很大差异,如四环素耐药基因tetG的含量远高于tetA,喹诺酮类耐药基因qnrS的含量高于qnrA,这与李壹等(2016)对海水的检测结果相似,本研究对氯霉素类2个耐药基因cmle3和floR基因表达量检测也出现了类似的趋势,即cmle3的含量高于floR,这可能是由于不同基因在环境中的代谢速率差异以及基因在传递过程中的质粒或宿主局限造成的。从不同耐药基因相对含量与相应耐药菌占比的相关性分析可以看出,除基因addA相对丰度与链霉素和庆大霉素耐药菌占比极显著相关(P < 0.01)外,其他耐药基因相对丰度与相应抗生素耐药菌的占比均不存在显著相关性。李壹等(2016)对海水中耐药菌占比和相关耐药基因相对丰度的相关性分析也得出类似结论,即只有氯霉素相关耐药基因cmle1和cata1相对拷贝数比例和氯霉素耐药菌占比存在显著相关性,其他所有基因的相对丰度均与相应耐药菌占比不存在显著相关性。这可能是由两方面原因造成的:一是可培养细菌占所有细菌的比例很低,目前培养的耐药菌不能全部反映出总细菌中耐药菌的占比;二是因为不同耐药菌基因在不同细菌中的相对拷贝数和降解速率不同。
综合以上结果可以看出,目前,苗期刺参中存在一定的携带耐药菌和耐药基因的风险,其耐药菌的分布不仅受外海海水环境影响,也与不同养殖业者各自的养殖管理和用药习惯密切相关;弧菌属病原菌和芽孢杆菌属益生菌的耐药菌检出比例较高,提示了抗生素的使用已对致病性耐药菌和益生耐药菌的产生造成了一定的威胁;耐药基因丰度与耐药菌丰度相关性不显著,因此,后期应同时密切关注二者的分布特征,为评价耐药风险提供科学依据。此外,细菌耐药性在“肠道–环境–肠道”的循环中存在传递和放大的效应,随肠道排泄进入环境的耐药菌和耐药基因将极大的增加管控的难度和成本,因此,养殖过程中应密切关注肠道耐药细菌和肠道耐药基因的检测,建立抗生素使用的监测和预警机制,为科学选药提供依据,也为研究和推广降低耐药菌数量及耐药基因丰度的无害化处理过程提供科学参考。
Agersø Y, Petersen A. The tetracycline resistance determinant Tet 39 and the sulphonamide resistance gene sulII are common among resistant Acinetobacter spp.isolated from integrated fish farms in Thailand. Journal of Antimicrobial Chemotherapy, 2007, 59(1): 23-27 |
Brooks JP, Adeli A, McLaughlin MR. Microbial ecology, bacterial pathogens, and antibiotic resistant genes in swine manure wastewater as influenced by three swine management systems. Water Research, 2014, 57: 96-103 |
Dang HY, Song LS, Chen MN, et al. Concurrence of cat and tet genes in multiple antibiotic-resistant bacteria isolated from a sea cucumber and sea urchin mariculture farm in China. Microbial Ecology, 2006, 52(4): 634-643 |
Dang HY, Song LS, Zhang ZN. Harmfulness of bacterial antibiotic resistance from maricultural environments. Studia Marina Sinica, 2006, 47(1): 29-40 [党宏月, 宋林生, 张志南. 海水养殖环境细菌耐药性的危害. 海洋科学集刊, 2006, 47(1): 29-40] |
Dang H, Zhang X, Song L, et al. Molecular determination of oxytetracycline-resistant bacteria and their resistance genes from mariculture environments of China. Journal of Applied Microbiology, 2007, 103(6): 2580-2592 |
Furushita M, Shiba T, Maeda T, et al. Similarity of tetracycline resistance genes isolated from fish farm bacteria to those from clinical isolates. Applied and Environmental Microbiology, 2003, 69(9): 5336-5342 |
Gao PP, Mao DQ, Luo Y, et al. Occurrence of sulfonamide and tetracycline-resistant bacteria and resistance genes in aquaculture environment. Water Research, 2012, 46(7): 2355-2364 |
Hoa PTP, Nonaka L, Viet PH, et al. Detection of the sul1, sul2, and sul3 genes in sulfonamide—Resistant bacteria from wastewater and shrimp ponds of north Vietnam. Science of the Total Environment, 2008, 405(3): 337-384 |
Huang L, Xu YB, Xu JX, et al. Antibiotic resistance genes (ARGs) in duck and fish production ponds with integrated or non-integrated mode. Chemosphere, 2017, 168: 1107-1114 |
Ji XL, Shen QL, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai, China. Journal of Hazardous Materials, 2012, 235-236: 178-185 |
Li YD. Study on characteristics of drug susceptibility and mechanisms of quinolones resistance in Bacillus cereus. Masterxs Thesis of Jilin University, 2004 [李研东.蜡样芽孢杆菌的药敏特性及对氟喹诺酮类药物耐药机制的初步研究.吉林大学硕士研究生学位论文, 2004]
|
Li Y, Qu LY, Zhu PF, et al. Distribution characteristics of antibiotic resistance bacteria and related resistance genes in mariculture area of Shandong. Marine Environmental Science, 2016, 35(1): 55-62 [李壹, 曲凌云, 朱鹏飞, 等. 山东地区海水养殖区常见抗生素耐药菌及耐药基因分布特征. 海洋环境科学, 2016, 35(1): 55-62] |
Lin T, Hu LM, Guo ZG, et al. Sources of polycyclic aromatic hydrocarbons to sediments of the Bohai and Yellow Seas in East Asia. Journal of Geophysical Research, 2011, 116: D23305 |
Luo Y, Zhou QX. Antibiotic resistance genes (ARGs) as emerging pollutants. Acta Scientiae Circumstantiae, 2008, 28(8): 1499-1505 [罗义, 周启星. 抗生素抗性基因(ARGs)—一种新型环境污染物. 环境科学学报, 2008, 28(8): 1499-1505] |
Miranda CD, Kehrenberg C, Ulep C, et al. Diversity of tetracycline resistance genes in bacteria from Chilean salmon farms. Antimicrobial Agents and Chemotherapy, 2003, 47(3): 883-888 |
Mu QH, Li J, Sun YX, et al. Occurrence of sulfonamide-, tetracycline-, plasmid-mediated quinolone- and macrolide- resistance genes in livestock feedlots in Northern China. Environmental Science and Pollution Research, 2015, 22(9): 6932-6940 |
Niu ZG, Zhang K, Zhang Y. Occurrence and distribution of antibiotic resistance genes in the coastal area of the Bohai Bay, China. Marine Pollution Bulletin, 2016, 107(1): 245-250 |
Pang HJ, Liao MJ, Li B, et al. Etiology and treatment of enteritis disease in cultured junviles of Apostichopus japonicus. Progress in Fishery Sciences, 2017, 38(3): 188-197 [逄慧娟, 廖梅杰, 李彬, 等. 刺参(Apostichopus japonicus)保苗期"肠炎病"及其治疗方法. 渔业科学进展, 2017, 38(3): 188-197] |
Patel JB, Franklin R, George M, et al. Performance standards for antimicrobial susceptibility testing: Twenty-six informational supplement. Wayne: Clinical and Laboratory Standards Institute, 2016 |
Pandiyan P, Balaraman D, Thirunavukkarasu R, et al. Probiotics in aquaculture. Drug Invention Today, 2013, 5(1): 55-59 |
Su HC, Liu S, Hu XJ, et al. Occurrence and temporal variation of antibiotic resistance genes (ARGs) in shrimp aquaculture: ARGs dissemination from farming source to reared organisms. Science of the Total Environment, 2017, 607- 608: 357-366 |
Tamminen M, Karkman A, Lõhmus A, et al. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure. Environmental Science and Technology, 2011, 45(2): 386-391 |
Tan AP, Deng YT, Jiang L, et al. Correlation between PMQR genes/QRDR mutations and quinolones resistance in Aerominas from aquatic animals. Journal of China Agricultural University, 2019, 24(4): 102-111 [谭爱萍, 邓玉婷, 姜兰, 等. 水产动物源气单胞菌喹诺酮类耐药与PMQR基因、QRDR突变相关性分析. 中国农业大学学报, 2019, 24(4): 102-111] |
Van TTH, Chin J, Chapman T, et al. Safety of raw meat and shellfish in Vietnam: An analysis of Escherichia coli isolations for antibiotic resistance and virulence genes. International Journal of Food Microbiology, 2008, 124(3): 217-223 |
Wang HP, Yan H, Zhao JR, et al. Quantitative detection of six classes of antibiotic resistance and classⅠintegron genes in aquatic products. Modern Food Science and Technology, 2017, 33(5): 270-276 [王慧平, 闫鹤, 赵俊仁, 等. 水产品中6类抗生素抗性基因和Ⅰ类整合子的定量检测. 现代食品科技, 2017, 33(5): 270-276] |
Wang L, Wang YG, Zhang Z, et al. Diversity and drug resistance of bacterial pathogens isolated from bacterial ascetic disease in cultured turbot Scophthalmus maximus. Progress in Fishery Sciences, 2017, 38(4): 17-24 [王岚, 王印庚, 张正, 等. 养殖大菱鲆(Scophthalmus maximus)腹水病的病原多样性及其耐药性分析. 渔业科学进展, 2017, 38(4): 17-24] |
Wang RX, Feng J, Geng YJ, et al. Studies on the drug resistance of aquatic bacteria. Marine Environmental Science, 2010, 29(5): 770-777 [王瑞旋, 冯娟, 耿玉静, 等. 水产细菌耐药性的最新研究概况. 海洋环境科学, 2010, 29(5): 770-777] |
Wang YG, Fang B, Zhang CY, et al. Etiology of skin ulcer syndrome in cultured juveniles of Apostichopus japonicus and analysis of reservoir of the pathogens. Journal of Fishery Sciences of China, 2006, 13(4): 610-616 [王印庚, 方波, 张春云, 等. 养殖刺参保苗期重大疾病"腐皮综合征"病原及其感染源分析. 中国水产科学, 2006, 13(4): 610-616] |
Wen YP, Pu XY, Zheng W, et al. High prevalence of plasmid-mediated quinolone resistance and incQ plasmids carrying qnrS2 gene in bacteria from rivers near hospitals and aquaculture in China. PLoS One, 2016, 11(7): pone0159418 |
Zhang H, Wang PL, Yang QX, et al. Distribution of multidrug- resistant bacteria and antibiotic-resistant genes in livestock manures. Environmental Science, 2018, 39(1): 460-466 [张昊, 王盼亮, 杨清香, 等. 畜禽粪便中多重耐药细菌及耐药基因的分布特征. 环境科学, 2018, 39(1): 460-466] |
Zhang RJ, Zhang G, Zheng Q, et al. Occurrence and risks of antibiotics in the Laizhou Bay, China: Impacts of river discharge. Ecotoxicology and Environmental Safety, 2012, 80(1): 208-215 |
Zhang QY, Zhao WW, Wu W. Antibiotics resistance gene pollution and its research progress acheived in aquaculture environment. Journal of Agricultural Science and Technology, 2015, 17(6): 125-134 [张骞月, 赵婉婉, 吴伟. 水产养殖环境中抗生素抗性基因污染及其研究进展. 中国农业科技导报, 2015, 17(6): 125-134] |
Zhang QQ, Ying GG, Pan CG, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modeling, and linkage to bacterial resistance. Environmental Science and Technology, 2015, 49(11): 6772-6782 |
Zhao XH, Su J, Fan JF, et al. Research progress on bacterial resistance in marine environment. Chinese Journal of Antibiotics, 2019, 44(4): 406-412 [赵小慧, 苏洁, 樊景凤, 等. 海洋环境中细菌耐药性研究进展. 中国抗生素杂志, 2019, 44(4): 406-412] |
Zuo ZH, Li YH, Shao YC, et al. Isolation, identification and antibiotic resistance analysis of intestinal strains of aquaculture Litopenaeus vannamei in surrounding areas of Tianjin. Journal of Fisheries of China, 2018, 42(5): 797-807 [左志晗, 李艳红, 邵迎春, 等. 天津市周边地区养殖凡纳滨对虾肠道菌株的分离鉴定及耐药性分析. 水产学报, 2018, 42(5): 797-807] |



