2. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 浙江省近岸水域生物资源开发与保护重点实验室 温州 325005
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Laboratory of Marine Fisheries Science and Food Production Processes, Pilot National Laboratory of Marine Science and Technology (Qingdao), Qingdao 266071;
3. Zhejiang Key Laboratory of Exploitation and Preservation of Coastal Bio-Resource, Wenzhou 325005
凡纳滨对虾(Litopenaeus vannamei)原产地主要为中、南美的秘鲁北部至墨西哥太平洋沿岸,尤其以厄瓜多尔沿岸分布最为密集(王兴强等, 2004)。凡纳滨对虾为世界三大养殖对虾之一(张伟权, 1990),其产量约占全球对虾产量的70%(张龙等, 2019),具有成活率高、食性广、生长快、适应能力强等优点。凡纳滨对虾于1988年从美国引入,之后迅速在全国范围内普及(于洋, 2014; 唐扬等, 2018),成为我国海水养殖动物中养殖发展最快的一个种类(马春艳等, 2011)。目前,我国已成为世界上凡纳滨对虾养殖产量最高的国家(颉晓勇等, 2008; 童馨等, 2009)。
随着我国凡纳滨对虾市场需求量的增大,对虾苗种培育方式也出现了转变。目前,在集约化苗种培育技术已达峰值的背景下,有些育苗场为了进一步降低成本,种虾不经选育,长期近交繁殖,随着养殖产量的增大,随之而来的问题也越来越多。最大的问题莫过于养殖对虾遗传多样性下降,加之外来亲本得不到更新,国内养殖对虾出现了种质退化、病害暴发频繁的现象。从长远角度来看,查清我国凡纳滨对虾遗传背景,对进一步培育优良品种和促进可持续健康养殖具有重要指导意义。
分子标记目前已广泛运用于水产动物育种中(张琼等, 2011; 孙苗苗等, 2017; 王军等, 2018),其中,又以微卫星(Microsatellite或Simple Sequence Repeats)分子标记应用最为普遍。微卫星标记是2~6个碱基为核心的短串联重复序列,重复单位的重复次数在个体间呈高度变异性且数量丰富,分布于生物体整个基因组中(Tautz, 1989; Schlötterer et al, 1992)。微卫星标记具有多态性高、重复性好、操作简单、共显性遗传等优点,被广泛用于对虾群体遗传多样性分析(Bringmann et al, 1996; Postlethwait et al, 1998; 张天时等, 2005; 曾地刚等, 2008)。本研究利用8个扩增稳定的微卫星位点,对国内6个商业品牌凡纳滨对虾苗种进行遗传多样性分析,初步分析当前国内养殖凡纳滨对虾的遗传背景,为凡纳滨对虾优良品种的选育提供基础数据。
1 材料与方法 1.1 实验材料选用我国有代表性的6个品牌凡纳滨对虾商业苗种[平均体长为(49.73±1.53) mm,平均体重为(1.41± 0.11) g],分别命名为黄骅R、海南Z、海南S、广州Z、广州P和东营M。每个品牌取30尾个体进行肌肉组织DNA提取,共计180尾凡纳滨对虾个体。
1.2 实验方法采用天根海洋动物组织DNA提取试剂盒进行肌肉组织DNA提取,利用紫外分光光度计(BioImaging ystems, UVP)进行DNA浓度测量,并根据测量结果将DNA稀释至50 ng/μl。将稀释得到的DNA利用8个微卫星位点进行PCR扩增。PCR反应体系:总体积为20 μl,其中,模板DNA 1.5 μl,(Vazyme) 2 × Taq Master Mix (Dye Plus) 10 μl,正向和反向引物(10 mmol/L)各0.8 μl,灭菌超纯水6.9 μl。PCR反应程序:95℃预变性3 min;95℃变性30 s;72℃退火30 s,72℃延伸30 s,共30个循环;72℃延伸5 min;4℃保存。PCR产物微卫星分型于生工生物工程(上海)股份有限公司,利用ABI 3730XL测序仪完成,所用微卫星位点及其引物信息见表 1。
|
|
表 1 8个微卫星位点信息 Tab.1 The information of 8 microsatellites loci |
对6个品牌凡纳滨对虾商业苗种进行哈迪–温伯格平衡(Hardy-Weinberg equilibrium, HWE)检测和遗传多样性分析。利用GENEPOP version 3.4软件进行HWE检验,得到精确P值(PH-W)。通过Cervus 3.0计算各位点的等位基因数目(Number of alleles, Na),观测杂合度(Observed heterozygosities, Ho)、期望杂合度(Expected heterozygosities, He)、8个微卫星位点的多态信息含量(Polymorphism information content, PIC)和无效等位基因频率[Null alleles frequency, F(Null)]。利用GenAlEx 6.51软件计算6个品牌凡纳滨对虾在各位点的香农多样性指数(Shannon's diversity index)、8个位点每个品牌特有等位基因(Private alleles)平均值、各品牌间的遗传分化程度和基因流(包括总的FST值以及两两品牌间的FST值);分子变异方差分析(Analysis of molecular variance, AMOVA),估测遗传变异在品牌内和品牌间的分配情况;6个品牌间Nei’s无偏遗传距离、遗传相似性系数、品牌间遗传距离矩阵、个体间遗传距离矩阵;利用品牌间和个体间遗传距离矩阵进行主成分分析(Principal coordinate analysis, PCA)。利用NTSYSpc 2.1软件按照遗传一致度进行6个品牌凡纳滨对虾UPGMA聚类。
2 结果 2.1 6个商业品牌凡纳滨对虾苗种遗传多样性分析6个品牌凡纳滨对虾商业苗种的遗传多样性参数见表 2和图 1。在8个微卫星位点中,6个品牌凡纳滨对虾总等位基因数为36~76个,最大为黄骅R,最小为东营M。各品牌平均等位基因数为4.5~9.5个。黄骅R平均每个微卫星位点存在1.625个特有等位基因,在所有品牌中最多。东营M平均每个位点存在0.125个特有等位基因,为各品牌中最少。8个微卫星位点的多态信息含量(PIC)值为0.343~0.925,最高为TuMXLv7.56位点,最低为M1103位点,除M1103位点外,其他7个微卫星位点PIC都大于0.5。黄骅R、广州Z和海南Z的香农多样性指数较高,其他3个品牌较低。8个微卫星位点的无效等位基因频率范围为0.031~0.403,5个位点存在无效等位基因。8个微卫星位点分别对6个品牌凡纳滨对虾进行了48次HWE检验。其中,4.17%(2/48)的检测结果表现为显著的偏离(0.01 < P < 0.05),58.33%(28/48)表现为极显著偏离(P < 0.01),其余的37.5%(18/48)符合HWE (P > 0.05)。
|
|
表 2 6个品牌凡纳滨对虾在8个微卫星位点中的遗传多样性参数 Tab.2 Genetic diversity indices of 8 microsatellite loci of L. vannamei from 6 brands |

|
图 1 6个品牌凡纳滨对虾商业苗种遗传多样性参数均值(8个微卫星位点) Fig.1 Mean value of genetic diversity parameters of L. vannamei from 6 brands (8 microsatellite loci) |
分子变异方差分析(AMOVA)发现,仅有12%的遗传变异来自品牌间,24%来自品牌内个体间,其余64%的变异均来自所有品牌个体,这表明遗传变异主要存在于个体间(表 3)。
6个品牌凡纳滨对虾商业苗种的F统计量分别为0.359 (FIT),0.273 (FIS),0.118 (FST)。两两品牌间的值FST在0.034和0.111之间,均 < 0.15(表 4),且明显的分成两部分。其中,小于0.05的为4组,占全部15组的26.67%,没有出现遗传分化(FST < 0.05);介于0.05和0.15之间的为11组,占全部15组的73.33%,为中等程度分化(0.05 < FST < 0.15)。另外,从基因流分布情况来看,两两群体间Nm > 1,范围为1.806~7.027 (表 4)。
|
|
表 3 6个品牌凡纳滨对虾分子变异方差分析 Tab.3 Analysis of molecular variances (AMOVA) of microsatellites in 6 brands of L. vannamei |
|
|
表 4 6个品牌凡纳滨对虾FST值(下三角)与Nm值(上三角) Tab.4 Pair-wise FST (below the diagonal) and Nm (above the diagonal) between six brands of L. vannamei |
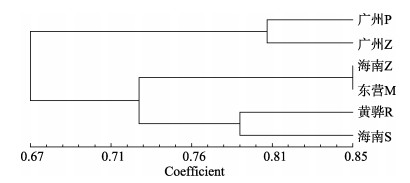
从表 5可以看出,东营M和海南Z品牌间的遗传距离最近(0.128),遗传相似性系数最大(0.880),亲缘关系最近;广州P和海南S品牌间的遗传距离最远(0.549),遗传相似性系数最小(0.578),亲缘关系最远。由图 2可以看出,6个品牌的凡纳滨对虾聚为2个明显的分支,广州P和广州Z聚为一支,东营M、黄骅R、海南Z和海南S聚为一支。
|
|
表 5 6个品牌凡纳滨对虾遗传相似性系数(下三角)与遗传距离(上三角) Tab.5 Genetic similarity coefficient (below the diagonal) and genetic distance (above the diagonal) of six brands' L. vannamei |
|
图 2 根据遗传一致度构建的UPGMA树 Fig.2 UPGMA dendrogram based on genetic identity of L. vannamei from 6 brands |
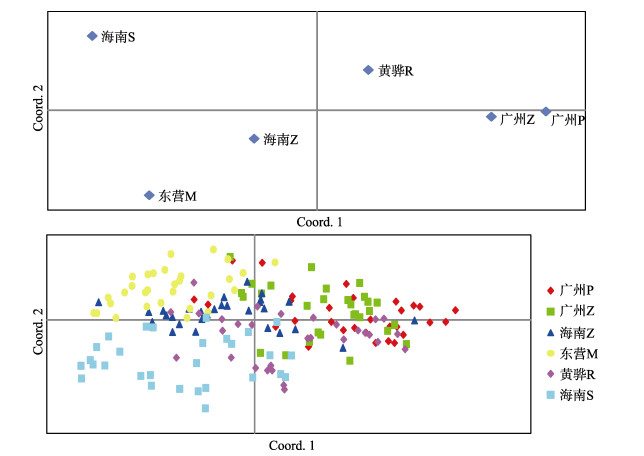
主成分分析(PCA)结果显示(图 3),6个品牌凡纳滨对虾商业苗种产生了明显的遗传歧化。其中,广州P和广州Z最为聚集,其次为东营M和海南Z,之后为黄骅R和海南S,主成分1 (Coord.1)和主成分2 (Coord.2)共解释了总遗传变异的71.22%。在个体水平上,主成分1 (Coord.1)和主成分2 (Coord.2)共解释了总遗传变异的20.05%。从图 3可知,6个品牌的180尾凡纳滨对虾个体聚集较为集中,其中,广州P、广州Z和黄骅R 3个品牌的凡纳滨对虾交集最大。同时,每个品牌的凡纳滨对虾个体无法单独聚为一类。
|
图 3 6个品牌凡纳滨对虾主成分分析 Fig.3 Principal coordinate analysis (PCA) for L. vannamei from six brands |
微卫星分子标记在种内有高度的遗传变异,是群体遗传分化分析的有效标记(孙效文等, 2008)。微卫星分子标记广泛分布于基因组中,数量众多,容易检测,在分子辅助育种和物种多样性检测等方面被广泛应用(张丽娟等, 2014)。群体的遗传多样性来源于物种适应复杂环境和生存进化(Li et al, 2016),其主要表现在等位基因数的丰富和均匀程度、遗传杂合度的大小、多态信息含量的高低3个方面(Beardmore et al, 1997; 王鹤等, 2016)。研究发现,等位基因越丰富,遗传杂合度数值越大,多态信息含量越高,则群体遗传变异更高,更能应对环境变化,也更能产生优质的种质资源(Eschenroeder et al, 2016)。
关于凡纳滨对虾微卫星标记开发和筛选的工作已有大量报道(Meehan, et al, 2003; 马宁等, 2013; 杨铭等, 2017) ADDIN EN.CITE。本研究从实验室发表过的文章(李东宇等, 2016)中筛选出8个扩增稳定的微卫星位点,分析了6个国内品牌的凡纳滨对虾商业苗种的遗传多样性、遗传结构和亲缘关系。结果显示,87.5% (7/8)的位点平均多态信息含量(PIC)都在0.5以上。根据Botstein等(1980)提出的衡量标准,当PIC > 0.5时,意味着该位点为高度多态位点,这也说明本研究所引用的微卫星标记的多态性较高。基因杂合度是衡量群体遗传变异水平的理想参数(王日芳等, 2017),8个微卫星位点中的观测杂合度(Ho)平均为0.486,期望杂合度(He)平均为0.745,观测杂合度小于期望杂合度,也从一个侧面说明了国内商业苗种凡纳滨对虾出现了杂合子缺失、纯合子过剩的情况。在进行微卫星分型的8个位点中,有5个(62.5%)位点显著偏离HWE (P < 0.05)。产生该结果的原因:一方面为实验对虾纯合子过剩、杂合子缺失;另一方面为水产动物生物结构较为简单,从而导致的遗传变异度也较高。因此,造成微卫星位点在同一物种不同个体中发生碱基形态和数目的变异,无法扩增正确的等位基因,扩增的无效等位基因会导致分型结果显著偏离HWE (舒妙安等, 2011; 宋忠魁等, 2013)。亲缘关系的远近可以用遗传距离来反映(陈子桂等, 2016; 刘洪涛等, 2018)。在本研究中,遗传距离最大的2个品牌是广州P和海南S,遗传距离最小的2个品牌是东营M和海南Z,并且针对个体的遗传关系主成分分析(PCA)表明,6个品牌的凡纳滨对虾无法依照每个品牌单独聚集,品牌间亲缘关系较为接近。这种结果表明,当前国内不同品牌的凡纳滨对虾来源存在一定的相似性,也可能是二代、三代苗种导致部分品牌对虾出现近交情况,使其亲缘关系逐渐接近。
种苗处于对虾产业链上游,其质量对养殖成败起关键作用(黄小帅等, 2019)。本研究所分析的6个品牌凡纳滨对虾商业苗种中,黄骅R与广州Z具有较高的遗传多样性,不同品牌的商业苗种间的遗传特征存在一定差异。在当前凡纳滨对虾养殖规模不断扩大的情况下,研究遗传因素与养殖生产性能之间的关联尤为重要,只有充分掌握对虾群体的遗传背景,才能合理利用杂交优势进行良种选育。
Beardmore JA, Mair GC, Lewis RI. Biodiversity in aquatic systems in relation to aquaculture. Aquaculture Research, 1997, 28(10): 829-839 DOI:10.1111/j.1365-2109.1997.tb01007.x |
Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314-331 |
Bringmann P, Rinke J, Appel B, et al. Microsatellite polymorphism and the population structure of Atlantic cod (Gadus morhua) in the northwest Atlantic. Canadian Journal of Fisheries and Aquatic Sciences, 1996, 53(4): 2706-2721 |
Chen ZG, Wang PP, Liao Y, et al. Genetic diversity analysis of F2 generation families of carp hybrid lines population using microsatellite. Fisheries Science and Technology Information, 2016, 43(2): 83-87 [陈子桂, 王培培, 廖愚, 等. 微卫星分析鲤鱼杂交系F2代家系群体遗传多样性. 水产科技情报, 2016, 43(2): 83-87] |
Eschenroeder JC, Roberts JH. Novel polymorphic microsatellite loci for distinguishing rock bass (Ambloplites rupestris), Roanoke bass (Ambloplites cavifrons), and their hybrids. Molecular Biology Reports, 2016, 43(10): 1035-1039 DOI:10.1007/s11033-016-4049-0 |
Huang XS, Xu Y, Hu XJ, et al. Genetic diversity analysis of first filial generation of seven introduced Litopenaeus vannamei populations using microsatellite DNA markers. South China Fisheries Science, 2019, 15(1): 54-62 [黄小帅, 徐煜, 胡晓娟, 等. 利用微卫星标记分析7个凡纳滨对虾引进群体子一代的遗传多样性. 南方水产科学, 2019, 15(1): 54-62] |
Jie XY, Su TF, Chen W, et al. Analysis on genetic diversity of six cultured stocks of Litopenaeus vannamei. South China Fisheries Science, 2008, 4(6): 42-49 [颉晓勇, 苏天凤, 陈文, 等. 凡纳滨对虾6个养殖群体遗传多样性的比较分析. 南方水产, 2008, 4(6): 42-49] |
Li DY, Kong J, Meng XH, et al. Development of multiplex PCR systems of microsatellite markers for Pacific white shrimp (Litopenaeus vannamei) and its application for parentage identification. Progress in Fishery Sciences, 2016, 37(3): 58-67 [李东宇, 孔杰, 孟宪红, 等. 凡纳滨对虾(Litopenaeus vannamei)微卫星多重PCR体系的建立及其在家系亲权鉴定中的应用. 渔业科学进展, 2016, 37(3): 58-67] |
Li Y, Zhao H, Peng Z, et al. Isolation and characterization of 21 polymorphic microsatellite markers for a new labeonine fish (Paraqianlabeo lineatus Zhao et al. 2014) using illumina paired-end sequencing. Journal of Applied Ichthyology, 2016, 32(1): 126-128 |
Liu HT, Yang MQ, He YG, et al. Microsatellite analysis of genetic diversity in eight geographical populations of Litopenaeus vannamei. Natural Science Journal of Hainan University, 2018, 36(2): 146-152 [刘洪涛, 杨明秋, 何玉贵, 等. 凡纳滨对虾八个地理群体遗传多样性的微卫星分析. 海南大学学报自然科学版, 2018, 36(2): 146-152] |
Ma CY, Ma HY, Ma LB, et al. Microsatellite analysis on genetic variation of imported Litopenaeus vannamei populanon and culture stocks. Marine Fisheries, 2011, 33(1): 1-8 [马春艳, 马洪雨, 马凌波, 等. 凡纳滨对虾引进群体和2个养殖群体遗传变异的微卫星分析. 海洋渔业, 2011, 33(1): 1-8] |
Ma N, Zeng DG. Isolation of microsatellite sequences from Litopenaeus vannamei. Southwest China Journal of Agricultural Sciences, 2013, 26(6): 2629-2633 [马宁, 曾地刚. 凡纳滨对虾微卫星序列的筛选. 西南农业学报, 2013, 26(6): 2629-2633] |
Meehan D, Xu Z, Zuniga G, et al. High frequency and large number of polymorphic microsatellites in cultured shrimp, Penaeus(Litopenaeus) vannamei [Crustacea: Decapoda]. Marine Biotechnology, 2003, 5(4): 311-330 DOI:10.1007/s10126-002-0092-z |
Postlethwait JH, Yan YL, Gates MA, et al. Vertebrate genome evolution and the zebrafish gene map. Nature Genetics, 1998, 18(4): 345-349 DOI:10.1038/ng0498-345 |
Schlötterer C, Tautz D. Slippage synthesis of simple sequence DNA. Nucleic Acids Research, 1992, 20(2): 211-215 DOI:10.1093/nar/20.2.211 |
Shu MA, Zhou YF, Zhu XY, et al. Microsatellite analysis on genetic diversity of seven wild populations of Scylla paramamosain in China. Journal of Fisheries of China, 2011, 35(7): 977-984 [舒妙安, 周宇芳, 朱晓宇, 等. 中国沿海拟穴青蟹群体遗传多样性的微卫星分析. 水产学报, 2011, 35(7): 977-984] |
Song ZK, Nie ZP, Wang YF. Screening for polymorphic primer pairs of (CA)n microsatellite from Scylla paramamosain (mud crab) genome. Marine Sciences, 2013, 37(10): 98-103 [宋忠魁, 聂振平, 王芳宇. 拟穴青蟹(CA)n微卫星DNA多态性引物筛选. 海洋科学, 2013, 37(10): 98-103] |
Sun MM, Yang QB, Chen BY, et al. Genetic diversity in four populations of Penaeus monodon assessed by AFLP makers. Marine Sciences, 2017, 41(3): 41-47 [孙苗苗, 杨其彬, 陈百尧, 等. 斑节对虾4个地理群体遗传多样性的AFLP分析. 海洋科学, 2017, 41(3): 41-47] |
Sun XW, Zhang XF, Zhao YY, et al. Development and application of microsatellite markers in aquatic species. Journal of Fishery Sciences of China, 2008, 15(4): 689-703 [孙效文, 张晓锋, 赵莹莹, 等. 水产生物微卫星标记技术研究进展及其应用. 中国水产科学, 2008, 15(4): 689-703] |
Tang Y, Meng XF, Shen RF, et al. Research and application of family selective breeding in culture of Pacific white shrimp Litopeneaus vannamei. Fisheries Science, 2018, 37(4): 555-563 [唐扬, 孟小菲, 沈瑞福, 等. 凡纳滨对虾家系选育的研究与应用. 水产科学, 2018, 37(4): 555-563] |
Tautz D. Hypervariabflity of simple sequences as a general source for polymorphic DNA markers. Nucleic Acids Research, 1989, 17(16): 6463-6471 DOI:10.1093/nar/17.16.6463 |
Tong X, Gong SY, Yu DH, et al. Genetic diversity of cultured Pacific white shrimp (Litopenaeus vannamei) stocks of different generations in China. Oceanologia et Limnologia Sinica, 2009, 40(2): 214-220 [童馨, 龚世圆, 喻达辉, 等. 凡纳滨对虾(Litopenaeus vannamei)不同世代养殖群体的遗传多样性分析. 海洋与湖沼, 2009, 40(2): 214-220] |
Wang H, Wang TT, Hu LP, et al. Microsatellite analysis of gentic characteristics in crosses between Korea population and China population of sea cucumber Apostichopus japonicus. Fisheries Science, 2016, 35(1): 60-66 [王鹤, 王田田, 胡丽萍, 等. 仿刺参中国群体与韩国群体杂交子代微卫星标记分析. 水产科学, 2016, 35(1): 60-66] |
Wang J, Wang QY, Kong J, et al. SSR analysis on genetic diversity in breeding and wild populations of Fenneropenaeus chinensis. Progress in Fishery Sciences, 2018, 39(2): 104-111 [王军, 王清印, 孔杰, 等. 中国明对虾人工选育群体与野生群体遗传多样性的SSR分析. 渔业科学进展, 2018, 39(2): 104-111] |
Wang RF, Li J, Li JT, et al. Genetic analysis of 33 microsatellite loci in 3 families of Exopalaemon carinicauda inbred line. Progress in Fishery Sciences, 2017, 38(4): 78-86 [王日芳, 李健, 李吉涛, 等. 脊尾白虾(Exopalaemon carinicauda)近交系3个家系33个微卫星座位的遗传分析. 渔业科学进展, 2017, 38(4): 78-86] |
Wang XQ, Ma S, Dong SL. Studies on the biology and cultural ecology of Litopenaeus vannamei: A review. Transactions of Oceanology and Limnology, 2004(4): 94-100 [王兴强, 马甡, 董双林. 凡纳滨对虾生物学及养殖生态学研究进展. 海洋湖沼通报, 2004(4): 94-100] |
Yang M, Yu Y, Zhang XJ, et al. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp (Litopenaus vannamei). Marine Sciences, 2017, 41(2): 96-102 [杨铭, 于洋, 张晓军, 等. 基于转录组数据的凡纳滨对虾微卫星标记开发. 海洋科学, 2017, 41(2): 96-102] |
Yu Y. Development of molecular markers and their applications in selective breeding of the Pacific white shrimp, Litopeneaus vannamei. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2014 [于洋.凡纳滨对虾分子标记的开发及其在遗传育种中的应用.中国科学院研究生院(海洋研究所)博士研究生学位论文, 2014]
|
Zeng DG, Chen XH, Li YM, et al. SSR Analysis of genetic diversity of Litopenaeus vannamei. Reservoir Fisheries, 2008, 28(1): 31-34 [曾地刚, 陈晓汉, 李咏梅, 等. 凡纳滨对虾遗传多样性的SSR分析. 水利渔业, 2008, 28(1): 31-34] |
Zhang L, Chen Z, Wang L, et al. Effect of stocking density on intermediate cultivation of Litopenaeus vannamei postlarvae. Progress in Fishery Sciences, 2019, 40(1): 76-83 [张龙, 陈钊, 汪鲁, 等. 放养密度对凡纳滨对虾苗种中间培育效果的影响. 渔业科学进展, 2019, 40(1): 76-83] |
Zhang LJ, Zhu F, Yin SW, et al. Microsatellite analysis of Odontobutis potamophila, O. yaluensis and their hybrid progeny. Freshwater Fisheries, 2014, 44(5): 9-14 [张丽娟, 祝斐, 尹绍武, 等. 河川沙塘鳢、鸭绿沙塘鳢及其杂交子代的微卫星分析. 淡水渔业, 2014, 44(5): 9-14] |
Zhang Q, Li XL, Xue SW, et al. Screening and polymorphism of EST-microsatellite markers in Litopenaeus vannamei. Acta Oceanologica Sinica, 2011, 33(1): 121-126 [张琼, 李喜莲, 薛淑雯, 等. 凡纳滨对虾EST-SSR标记的筛选及遗传多态性检测. 海洋学报, 2011, 33(1): 121-126] |
Zhang TS, Liu P, Li J, et al. Genetic diversity of cultured populations of Fenneropenaeus chinensis shrimp using microsatellites. Journal of Fisheries of China, 2005, 29(1): 6-12 [张天时, 刘萍, 李健, 等. 用微卫星DNA技术对中国对虾人工选育群体遗传多样性的研究. 水产学报, 2005, 29(1): 6-12] |
Zhang WQ. An important breeding species in the world-Biology of Litopenaeus vannamei. Marine Sciences, 1990, 14: 69-73 [张伟权. 世界重要养殖品种——南美白对虾生物学简介. 海洋科学, 1990, 14: 69-73] |



