太湖新银鱼(Neosalanx taihuensis Chen, 1956)属于鲑形目(Salmoniformes)、银鱼科(Salangidae)的新银鱼属,主要分布在黄河、淮河和长江中下游及其附属湖泊,是我国特有的银鱼种类,营养丰富,经济价值较高(解玉浩等, 1997; 倪勇等, 2006)。太湖新银鱼为一年生小型鱼类,生长速度快、世代离散,对环境变化比较敏感,种群易于波动(王忠锁等, 2002)。近年来,受围湖造田、过度捕捞、环境污染和生境破碎化等多种不利因素的影响,我国的银鱼天然资源急剧衰减,分化范围显著缩小,个别物种渐危,银鱼资源的可持续发展受到严重威胁(王忠锁等, 2002)。
遗传多样性是物种生存与进化的物质基础,开展鱼类遗传多样性研究可以为种质资源保护和遗传育种提供重要参考(Ward, 2000)。鱼类线粒体DNA (Mitochondrial DNA, mtDNA)具有分子小、母系遗传、进化速率快等特点,是鱼类分子系统学和群体遗传结构研究的理想分子标记(Xiao et al, 2000)。细胞色素b (Cytochrome b, Cyt b)基因是mtDNA的13个蛋白质编码基因之一,其结构和功能最为清晰,且其进化速度适中,被广泛运用于鱼类遗传多样性和遗传结构研究(张东亚等, 2009; 李大命等, 2015; 蓝昭军等, 2016; 张争世等, 2017; 周华兴等, 2019)。
目前,太湖新银鱼有关研究主要在营养(林伟信, 1992)、基础生物学(徐桂珍等, 1998)、种群生态(赵丽爽等, 2018)、生殖发育(杨战伟等, 2012)及移植引种(林炯等, 1998)等方面,而在分子生物学方面的研究较少。张际峰等(2008)测定了大银鱼(Protosalanx hyalocranius)和太湖新银鱼线粒体COⅡ及tRNA基因序列并分析了其亲缘关系。赵亮等(2010)研究了太湖新银鱼线粒体D-loop和Cyt b片段序列结构,并比较了二者的进化速率。王维维等(2013)对太湖新银鱼Cyt b基因PCR扩增条件进行了优化研究。另外,已有研究者利用多种分子标记技术研究太湖新银鱼遗传多样性,比如同工酶(张颖等, 2005)、RAPD(夏德全等, 2007)、AFLP (Kim et al, 2007)及线粒体Cyt b (Zhao et al, 2008; 罗宏伟等, 2009)和COⅠ(张迪等, 2012)等,但对太湖新银鱼种群遗传结构和地理分布格局的研究尚显不足。本研究采集江苏省太湖、高邮湖、洪泽湖和骆马湖4个湖泊太湖新银鱼样本,通过扩增和测定Cyt b基因序列,分析太湖新银鱼遗传多样性和遗传结构,以期为管理、保护和开发利用太湖新银鱼种质资源提供理论依据。
1 材料与方法 1.1 实验材料2018年8~12月采集太湖、高邮湖、洪泽湖和骆马湖的太湖新银鱼野生群体,其中,太湖群体32尾,高邮湖群体40尾,洪泽湖群体37尾,骆马湖群体35尾,共144尾样本。从每尾样本剪取肌肉组织,放入1.5 ml离心管中,加入适量的无水乙醇保存,带回实验室备用。
1.2 DNA提取、PCR扩增与测序采用TaKaRa公司的广谱性基因组DNA提取试剂盒,提取太湖新银鱼基因组DNA,将DNA溶于TE溶液中,操作步骤参照试剂盒说明书。采用1%琼脂糖凝胶电泳检测所提取DNA的完整性,用核酸蛋白定量仪检测其浓度。
扩增Cyt b序列的正向引物为L14321 (5′-CCA GTGACTTGAAAAACCACCG-3′),反向引物为H15634 (5′-CTTAGCTTTGGGAGTTAAGGGT-3′) (Zhang et al, 2007)。PCR扩增体系为50 μl:Premix Taq 25 μl,上下游引物各2 μl (10 μmol/L),DNA模板2 μl (40 ng/μl),用ddH2O补足至50 μl。PCR扩增条件:94℃预变性3 min;94℃变性30 s,55℃退火40 s,72℃延伸90 s,30个循环;最后,72℃延伸10 min。用1.5%的琼脂糖凝胶检测PCR产物,凝胶成像系统拍照。PCR产物送往生工生物工程(上海)股份有限公司进行双向测序,测序采用与PCR反应相同的引物。
1.3 数据分析采用BioEdit 7.0软件(Hall, 1999)和ClustalX 1.81软件(Thompson et al, 1997)对测序结果进行编辑和同源比对。使用DnaSP 5.0软件(Liorado et al, 2009)统计核苷酸变异位点、单倍型数目、单倍型多样性(Hd)和核苷酸多样性(π)。
利用MEGA 7.0软件(Kumar et al, 2016)统计序列的碱基组成,计算群体间的Kimura双参数模型(K2P)遗传距离,并构建单倍型建邻接(Neighbor-Joinning, NJ)进化树。同时采用Network 4.6.1.0软件(Bandelt et al, 1999)构建单倍型的简约中介(Reduced-Median, MJ)网络图,用以检测单倍型之间的进化关系。
使用Arlequin3.1软件(Excoffier et al, 2010)计算两两群体间的遗传分化指数Fst,采用AMOVA分析检验群体遗传结构,通过1000次重抽样来检验Fst值的显著性。通过Tajima’s D检验(Hickerson et al, 2007)、Fu’s Fs检验(Fu, 1997)和核苷酸不配对分布(Mismatch distributions)分析来检验太湖新银鱼群体的历史动态,以确定是否存在瓶颈效应或群体扩张。
2 结果与分析 2.1 Cyt b基因序列变异测序得到144尾太湖新银鱼的Cyt b基因全序列,序列长度为1141 bp。144条序列中共检测到29个变异位点,总变异率为2.54%。其中,单一信息位点有14个,简约信息位点15个。变异均为转换或颠换,转换与颠换的比值为4.6,无插入或缺失位点。4个群体所有个体序列中的A、C、T和G组成比例为21.1%、33.5%、28.0%和17.4%,其中,碱基G的含量明显低于其他3种碱基含量,表现出很强的碱基组成偏向性。
2.2 太湖新银鱼群体遗传多样性基于Cyt b的太湖新银鱼遗传多样性统计结果见表 1。144尾样本共定义了25个单倍型(H1~H25),高邮湖群体拥有最多的单倍型数(9个单倍型),骆马湖群体次之(7个单倍型),太湖和洪泽湖群体拥有的单倍型数相同(均有6个单倍型)。其中,单倍型H3分布于太湖、高邮湖和骆马湖群体,包含76个个体,是数量最多的单倍型。单倍型H7和H9由高邮湖和洪泽湖群体共享,分别包含2和14个个体。单倍型H4和H5为太湖群体特有,单倍型H1、H6、H12、H14、H21和H22为高邮湖群体特有,单倍型H2、H10和H13为洪泽湖群体特有,单倍型H16~H20和H24~H25为骆马湖群体特有。
|
|
表 1 太湖新银鱼群体的遗传多样性 Tab.1 Genetic diversity parameters of four N. taihuensis populations |
4个群体的单倍型多样性(Hd)为0.343±0.107~ 0.609±0.078,核苷酸多样性(π)为0.00057±0.00016~ 0.00094±0.00027。其中,高邮湖群体的遗传多样性最丰富,太湖群体的遗传多样性最低,但均表现出较低的遗传多样性水平。整体来看,太湖新银鱼群体总的单倍型多样性为0.682±0.037,核苷酸多样性为0.00231±0.00021。
2.3 太湖新银鱼群体遗传结构采用Mega 7.0软件计算太湖新银鱼群体内和群体间的遗传距离,结果见表 2。太湖、高邮湖和洪泽湖群体间的遗传距离均为0.001,与骆马湖群体间的遗传距离均为0.005。
|
|
表 2 太湖新银鱼Cyt b群体间的遗传分化指数(Fst)(对角线上方)和群体间的遗传距离(对角线下方) Tab.2 The fixation index (above diagonal) and genetic distance (below diagonal) among populations of N.taihuensis |
太湖新银鱼群体分子方差分析结果见表 3。结果显示,群体间分子变异占71.53%,群体内分子变异占28.47%,分子变异主要发生在群体间。群体间的遗传分化指数Fst=0.71531,且统计检验具有极显著性(P=0.0000),说明群体间出现了显著的遗传分化。进一步比较两两群体间的遗传分化指数,结果见表 2。太湖、高邮湖和洪泽湖群体间的遗传分化指数较小,且统计检验均不显著(P > 0.05),与骆马湖群体间的遗传分化指数较大,且统计检验均极显著(P < 0.01)。
|
|
表 3 太湖新银鱼群体分子方差分析结果 Tab.3 Results of AMOVA analysis of N. taihuensis populations |
为更好地了解太湖新银鱼各种群之间的亲缘关系,以近缘物种大银鱼为外类群,采用邻接法对25个单倍型进行系统发育分析,构建分子系统发育树(图 1)。从图 1可以看出,4个太湖新银鱼群体具有明显的地理遗传结构,形成2个分支:单倍型H1~H14和H21~H23聚为一支,由太湖、高邮湖和洪泽湖群体的个体组成;单倍型H15~H20和H24~H25聚为一支,由骆马湖群体的个体组成。

|
图 1 基于Cyt b基因构建的太湖新银鱼系统进化树 Fig.1 Phylogenetic tree for N. taihuensis based on mitochondrial Cyt b gene |
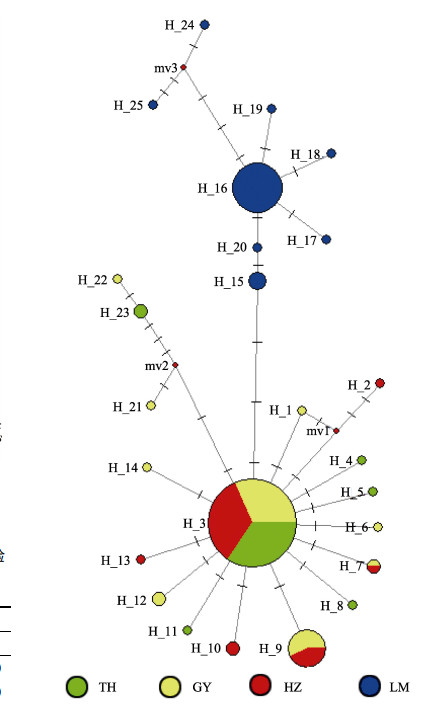
采用Network构建的单倍型最小网络进化图显示(图 2),整个进化图呈星状,单倍型H3位于网络结构图的中央。25个单倍型按其所处地理位置演化为两支,一支是由太湖、高邮湖和洪泽湖群体的单倍型组成,另一支由骆马湖群体独有的单倍型组成。网络图进一步支持了系统发育树的分析结果,4个湖泊太湖新银鱼群体形成2个进化单元,且2个单元间缺乏基因交流。
|
图 2 太湖新银鱼单倍型最小网络进化图 Fig.2 Haplotypes minimum spanning network for N. taihuensis |
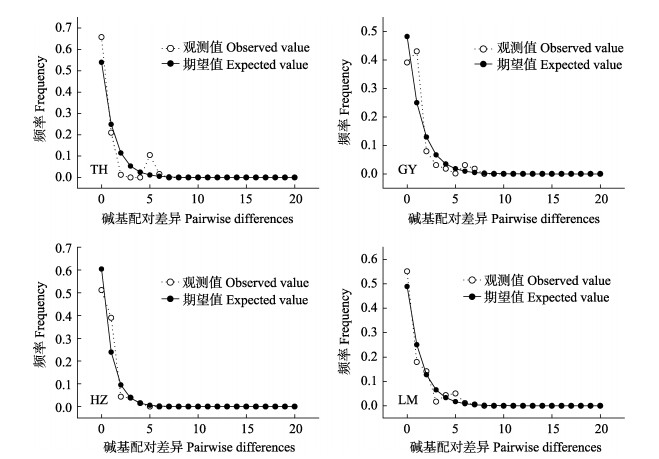
对4个太湖新银鱼所有个体进行中性检验(表 4)和歧点分布图分析(图 3)。结果显示,中性检测Fu’s Fs及Tajima’s D的值均为负值,且检验统计结果均具有显著性差异(P < 0.05),同时,歧点分布图呈单峰形,表明4个太湖新银鱼野生群体在进化过程中经历过种群扩张。
|
|
表 4 太湖新银鱼群体的Tajima’s D和Fu’s Fs中性检验 Tab.4 Neutral test of Fu's Fs and Tajima's D of N. taihuensis populations |
|
图 3 太湖新银鱼歧点分布 Fig.3 Mismatch distributions of N. taihuensis populations |
遗传多样性是指生物种内和种间遗传变异的总和,是生物进化和物种分化的基础,也是评价种群资源状况的重要依据。一个物种的遗传多样性水平越高,其进化潜力及适应环境能力也越强。单倍型多样性和核苷酸多样性是评价物种遗传多样性的两个重要参数。按照Grant等(1998)提出的标准,单倍型多样性以0.5为临界值,核苷酸多样性以0.005为临界值,二者的值越大,群体的多样性程度越高。本研究中,4个太湖新银鱼群体的单倍型多样性为0.343± 0.107~0.609±0.078,核苷酸多样为0.00057±0.00016~ 0.00094±0.00027。除高邮湖群体外,其他3个群体的单倍型多样性均低于0.5,且4个群体的核苷酸多样性均远小于0.005,表明太湖新银鱼遗传多样性处于较低水平,这也反映了银鱼资源趋于枯竭的现状,导致银鱼有效种群数量减少,遗传多样性水平较低。近年来,受过度捕捞、环境污染、栖息地破坏等多种因素的影响,江苏省湖泊银鱼野生资源严重衰退,已无法形成渔汛,失去捕捞价值(倪勇等, 2006),与本实验室多年调查结果一致(数据未给出)。从整体来看,4个群体总的单倍型多样性为0.682±0.037,核苷酸多样性为0.00231±0.00021,属于高单倍型多样性,低核苷酸多样性群体。已有文献报道,我国长江、淮河和珠江水系太湖新银鱼群体总的遗传多样性(Hd: 0.713±0.022; π: 0.0022±0.0001),其中,太湖群体的单倍型多样性和核苷酸多样性分别为0.798±0.039和0.0028±0.0002,洪泽湖群体的单倍型和核苷酸多样性分别为0.686±0.088和0.0021±0.0003)(Zhao et al, 2008)。比较发现,江苏省4个湖泊太湖新银鱼群体遗传多样性低于全国水平,且太湖和洪泽湖群体的遗传多样性下降明显,表明太湖新银鱼种质资源下降趋势尚无改变。另外,与其他银鱼种类遗传多样性相比,如三峡库区大银鱼(Hd: 0.804±0.032; π: 0.00146±0.00009) (罗宏伟等, 2009)、乔什新银鱼(Neosalanx jordani)(Hd: 0.590±0.047; π: 0.00088±0.00011)(赵亮等, 2010)、白肌银鱼(Leucosoma chinensis)(Hd: 0.888; π: 0.0990) (黄小彧等, 2012)、居氏银鱼(Salanx curvieri) (Hd: 0.967; π: 0.006)(司从利等, 2012)和黑龙江流域大银鱼(Hd: 0.827±0.038; π: 0.00202±0.00028)(Tang et al, 2008)等,江苏省4个湖泊太湖新银鱼遗传多样性明显低于大银鱼、白肌银鱼及居氏银鱼,仅高于乔氏新银鱼。因此,必须加强太湖新银鱼资源的保护力度,增加其种群数量,提高其遗传多样性水平,以避免太湖新银鱼种质资源的进一步衰退。
3.2 太湖新银鱼种群的历史动态鱼类遗传多样性与其进化历史密切相关,可以利用单倍型多样性和核苷酸多样性来估计种群的进化历史,当Hd≥0.5、π < 0.5%时,是受瓶颈效应后种群数量的迅速扩张导致;当Hd≥0.5、π≥0.5%时,表示种群稳定,具有比较悠久的进化历史;当Hd < 0.5、π≥0.5%时,种群经历了轻微的瓶颈效应,几乎没有影响到核苷酸变异;当Hd < 0.5、π < 0.5%时,表明种群近期经历了瓶颈效应(Grant et al, 1998)。本研究中,高邮湖群体的单倍型多样性 > 0.5,而核苷酸多样性 < 0.5%,属于高单倍型多样性、低核苷酸多样性的进化模式,与文献报道的我国珠江、淮河和长江流域太湖新银鱼遗传多样性模式一致(Zhao et al, 2008)。出现这种多样性模式,可能是群体受到瓶颈效应后种群迅速扩张所导致的结果。在群体扩张过程中,随着种群数量的急剧增加,导致单倍型多样性增加,而核苷酸的变异速率较低,没有足够的时间来积累核苷酸变异,因为核苷酸多样性的积累时间比单倍型多样性的积累时间要漫长得多(范启等, 2014; 蓝昭军等, 2016)。其他3个太湖新银鱼群体单倍型多样性和核苷酸多样性都较低,表明群体最近经历过瓶颈效应或受奠基者效应的影响所致。
利用中性检验和核苷酸歧点分布推测种群经过的历史。若Fu’s Fs和Tajima’s D呈负值,并且在统计学上有较显著的标准,则说明序列中含有比中性进化模型更多的核苷酸位点变化,可能预示着种群经历过扩张历史(Fu, 1997; Hickerson et al, 2007)。歧点分布曲线呈泊松状分布的单峰,说明种群趋于发生了瓶颈效应或者种群扩张(Barbosa et al, 2013)。中性检测结果显示(表 4),4个太湖新银鱼群体的Fu’s Fs和Tajima’s D的值为负值,且显著偏离中性检验;同时,歧点分布图均呈明显的单峰形(图 3),表明4个太湖新银鱼野生群体发生过历史扩张。
3.3 太湖新银鱼种群遗传结构研究群体遗传结构不仅可以用于评价物种群体的变异水平和不同地理群体之间的关系,还可以确定群体中的进化显著单元和管理单元,以及制定资源的保护和管理策略(O’Brien, 1994)。遗传分化系数(Fst)是反映不同地理种群间遗传分化的重要指标。Wright(1990)认为,若种群Fst为0~0.05,则表明其各种群间不存在分化;若Fst为0.05~0.15,则表明其各种群间存在中度分化;若Fst为0.15~0.25,则为高度分化。AMOVA结果显示,太湖新银鱼群体间变异占比为71.53%,群体内变异占比为28.47%,群体间变异是遗传变异的主要来源。4个群体间的遗传分化指数及其统计结果表明,太湖、高邮湖和洪泽湖群体间没有出现显著遗传分化,但均与骆马湖群体存在高度遗传分化,这说明4个太湖新银鱼群体分为2个进化类群,其中一个类群由太湖、高邮湖和洪泽湖群体组成,另一个类群由骆马湖群体组成,这与单倍型NJ进化树和最小网络进化图的分析结果相一致。从单倍型组成看,太湖、高邮湖和洪泽湖群体拥有共享单倍型,且该单倍型为占比最高的优势单倍型,表明3个群体间存在广泛的基因交流;而骆马湖群体则全部由独享单倍型个体组成,与其他3个群体缺乏基因交流,形成了明显的地理遗传结构。
一般来说,鱼类的遗传分化格局与其分布的水系格局吻合:流域之间存在明显的种群分化,同一流域内的种群通常分化不明显或没有遗传分化(Perdices et al, 2005; Hashiguchi et al, 2006; Xia et al, 2006)。从地理分布来看,太湖属于长江水系,高邮湖、洪泽湖和骆马湖则属于淮河水系。本研究结果说明,太湖新银鱼种群遗传分化格局与现有水系的分布格局明显不吻合,这与太湖新银鱼和乔什新银鱼的研究结果类似(Zhao et al, 2008; 赵亮等, 2010)。因此,太湖新银鱼群体遗传结构可能与其进化历史有密切关系(Zhao et al, 2008)。研究表明,银鱼最早可能起源于第三纪中期,地质史上的重大事件,尤其是第四纪冰期和间冰期气候的交替变化,导致银鱼栖息地数度隔离和连接,从而对银鱼的进化分化过程造成了重大影响。在冰期和间冰期的交替变化过程中,银鱼的生境不断扩展,生境异质性不断提高,逐渐分化、进化成现今的银鱼遗传格局(张广学, 1993; Zhao et al, 2008)。由单倍型最小进化网络图(图 2)可以看出,单倍型H3是太湖、高邮湖和洪泽湖群体的祖先单倍型,单倍型H16是骆马湖群体的祖先单倍型。随着最后一次冰期结束,气候变暖,海平面上升,太湖新银鱼的分布区扩大,种群开始扩张,逐渐产生不同的周单倍型,形成现今的遗传结构模式。
3.4 太湖新银鱼种质资源保护太湖新银鱼是我国特有的银鱼种类,属于典型的r-对策者,其种群易波动,对生态环境要求高(王忠锁等, 2002)。本研究对江苏省4个湖泊太湖新银鱼野生群体遗传多样性进行研究,结果表明,4个群体的遗传多样性处于较低水平,应当采取措施,加强太湖新银鱼各群体的种质资源保护力度。比如,严格控制捕捞强度,加强环境保护,延长禁渔期等,保证太湖新银鱼种群生殖繁衍和资源恢复。根据4个太湖新银鱼群体的遗传结构,建议将太湖、高邮湖和洪泽湖群体作为一个整体进行管理和保护,骆马湖群体单独进行管理和保护。本研究初步反映了太湖新银鱼的遗传背景、遗传结构及遗传变异水平,后续可以通过利用多种分子标记技术如SSR、SNP等联合分析,更全面地为太湖新银鱼资源的保护和合理开发提供理论依据。
致谢: 感谢江苏省太湖渔业管理委员会办公室、江苏省高宝-邵伯湖渔业管理委员会办公室、江苏省洪泽湖渔业管理委员会办公室和江苏省骆马湖渔业管理委员会办公室对样品采集提供的帮助。
Bandelt HJ, Forster P, Röhl AA. Median-joining network for inferring intraspecific phylogenies. Molecular Biology and Evolution, 1999, 16(1): 37-48 DOI:10.1093/oxfordjournals.molbev.a026036 |
Barbosa AM, Real R, Muñoz AR, et al. New measures for assessing model equilibrium and prediction mismatch in species distribution models. Diversity and Distributions, 2013, 19(10): 1333-1338 DOI:10.1111/ddi.12100 |
Excoffier L, Lischer HEL. Arlequin suite ver3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 2010, 10: 564-567 DOI:10.1111/j.1755-0998.2010.02847.x |
Fan Q, He SP. The pattern of upper and middle Yangze drainages shapes the genetic structure and diversity of Hemiculter leucisculus revealed by mitochondrial DNA locus. Acta Hydrobiologica Sinica, 2014, 38(4): 627-635 [范启, 何舜平. 长江流域种群遗传多样性和遗传结构分析.长江流域种群遗传多样性和遗传结构分析. 水生生物学报, 2014, 38(4): 627-635] |
Fu YX. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics, 1997, 147(2): 915-925 |
Grant WAS, Bowen BW. Shallow population histories in deep evolutionary lineages of marine fishes: Insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 1998, 89(5): 415-426 DOI:10.1093/jhered/89.5.415 |
Hall TA. BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series, 1999, 41: 95-98 |
Hashiguchi Y, Kado T, Kimura S, et al. Comparative phylogeography of two bitterlings, Tanakia lanceolata and T. limbata (Teleostei, Cyprinidae), in Kyushu and adjacent districts of western Japan, based on mitochondrial DNA analysis. Zoological Science, 2006, 23(4): 309-322 DOI:10.2108/zsj.23.309 |
Hickerson MJ, Meyer CP. Testing comparative phylogeographic models of marine vicariance and dispersal using a hierarchical Bayesian approach. BMC Evolutionary Biology, 2007, 8(3): 322-340 |
Huang XY, Zhang Q, Si CL, et al. Genetic diversity analysis of Leucosoma chinensis based on mtDNA Cytb gene sequences. Jiangsu Agricultural Science, 2012, 40(4): 45-48 [黄小彧, 章群, 司从利, 等. 基于细胞色素b基因的白肌银鱼遗传多样性. 江苏农业科学, 2012, 40(4): 45-48] |
Kim JK, Kai Y, Nakabo T. Genetic diversity of Salanx ariakensis (Salangidae) from Korea and Japan inferred from AFLP. Ichthyollgical Research, 2007, 54(4): 416-419 DOI:10.1007/s10228-007-0418-y |
Kumar S, Stecher G, Tamura K, et al. MEGA7: Molecular evolutionary genetics analysis version 7. 0 for bigger datasets. Molecular Biology and Evolution, 2016, 33(7): 1870-1874 DOI:10.1093/molbev/msw054 |
Lan SJ, Li Q, Shu H, et al. Genetic diversity and structure of Spiinibarbus holiandi based on nucleotide sequences of mtDNA cytochrome b gene. Oceanologia et Limnologia Sinica, 2016, 47(6): 1158-1165 [蓝昭军, 李强, 舒琥, 等. 基于线粒体细胞色素b基因的光倒刺鲃遗传多样性与遗传结构研究. 海洋与湖沼, 2016, 47(6): 1158-1165] |
Li DM, Zhang TQ, Tang SK, et al. Genetic polymorphism of mitochondrial DNA cytochrome b (cytb) sequences of Protosalanx chinensis in Lake Taihu. Jiangsu Journal of Agricultural of Sciences, 2015, 31(4): 840-845 [李大命, 张彤晴, 唐晟凯, 等. 太湖大银鱼(Protosalanx chinensis)细胞色素b基因序列多态性分析. 江苏农业学报, 2015, 31(4): 840-845] |
Lin J, Xu RW, Wu ZX, et al. On removal of food and distribution of Neosalanx taihuensis Chen in Qiandao Lake. Journal of Zhejiang College of Fisheries, 1998, 17(1): 59-62 [林炯, 徐如卫, 吴振兴, 等. 千岛湖中太湖新银鱼的移驯及其饵料与分布特性的初步研究. 浙江水产学院学报, 1998, 17(1): 59-62] |
Lin WX. Amino acid contents of tissues of Neosalanx taihuensis and N. oligodontis. Journal of Fisheries of China, 1992, 16(1): 71-74 [林信伟. 太湖新银鱼和寡齿新银鱼组织内氨基酸的含量. 水产学报, 1992, 16(1): 71-74] |
Liorado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451-1452 DOI:10.1093/bioinformatics/btp187 |
Luo HW, Duan XB, Wang K, et al. Genetic polymorphism analysis of mitochondrial DNA cytochrome b sequences of three species of salangids in the Three Gorges Reservior. Freshwater Fisheries, 2009, 39(6): 16-21 [罗宏伟, 段辛斌, 王珂, 等. 三峡库区3种银鱼线粒体DNA细胞色素b基因序列多态性分析. 淡水渔业, 2009, 39(6): 16-21] |
Ni Y, Wu HL. Fishes of Jiangsu Province. Beijing: China Agriculture Press, 2006: 32-38 [倪勇, 伍汉霖. 江苏鱼类志. 北京: 中国农业出版社, 2006: 32-38]
|
O'Brien SJ. A role for molecular genetics in biological conservation. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(13): 5748-5755 DOI:10.1073/pnas.91.13.5748 |
Perdices A, Sayanda D, Coelho MM. Mitochondrial diversity of Opsariichthys bidens (Teleostei, Cyprinidae) in three Chinese drainages. Molecular Phylogenetics and Evolution, 2005, 37(3): 920-927 |
Si CL, Zhang Q, Huang XY, et al. Genetic diversity of Salanx curvieri in south China inferred from mtDNA cytb sequences. Marine Fisheries, 2012, 34(1): 1-6 [司从利, 章群, 黄小彧, 等. 基于细胞色素b基因序列分析的华南居氏银鱼遗传多样性研究. 海洋渔业, 2012, 34(1): 1-6] |
Tang FJ, Li DM, Liu W, et al. Evolutionary tendency of clearhead icefish Protosalanx hyalocranius inferring mitochondrial DNA variation analyses in Amur (Heilongjiang) River catchment, China. International Journal of Agriculture and Biological, 2008, 20(10): 2329-2334 |
Thompson JD, Gibson TJ, Plewniak F, et al. The ClustalX windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 1997, 25(24): 4876-4882 DOI:10.1093/nar/25.24.4876 |
Wang WW, Zhao L, Wei XS. Optimization of PCR amplification conditions of mitochondrial Cytb gene of Neosalanx taihuensis. Journal of Suzhou University, 2013, 28(1): 71-73 [王维维, 赵亮, 位晓三. 太湖新银鱼线粒体Cytb基因PCR扩增条件的优化. 宿州学院学报, 2013, 28(1): 71-73] |
Wang ZS, Fu CZ, Lei GC. Biodiversity of Chinese icefishes (Salangidae) and their conserving strategies. Biodiversity Science, 2002, 10(4): 416-424 [王忠锁, 傅萃长, 雷光春. 中国银鱼的多样性及其保护对策. 生物多样性, 2002, 10(4): 416-424] |
Ward RD. Genetics in fisheries management. Hydrobiologia, 2000, 420(1): 191-201 DOI:10.1023/A:1003928327503 |
Wright S. Evolution in Mendelian populations. Bulletin of Mathematical Biology, 1990, 52(1-2): 241-295 DOI:10.1016/S0092-8240(05)80011-4 |
Xia DQ, Cao Y, Wu TT. Study on lineages of Protosalanx chinensis, Neosalanx taihuensis and N. oligodont is in Taihu Lake with RAPD technique. Journal of Fishery Sciences of China, 2007, 14(1): 12-15 [夏德全, 曹莹, 吴婷婷. 用RAPD方法分析太湖大银鱼、太湖新银鱼和寡齿的亲缘关系. 中国水产科学, 2007, 14(1): 12-15] |
Xia YZ, Chen YY, Sheng Y. Phylogeographic structure of lenok (Brachymystax lenok Pallas) (Salmoninae, Salmonidae) populations in water systems of eastern China, inferred from mitochondrial DNA sequences. Zoological Studies, 2006, 45(2): 190-200 |
Xiao WH, Zhang YP. Genetics and evolution of mitochondrial DNA in fish. Acta Hydrobiologica Sinica, 2000, 24(4): 384-391 |
Xie YH, Xie H. Classification, distribution, and population ecology of Salangidae fishes. Chinese Journal of Fisheries, 1997, 10(2): 11-19 [解玉浩, 解涵. 银鱼科鱼类的分类分布和种群生态. 水产学杂志, 1997, 10(2): 11-19] |
Xu GZ, Hu B, Bao CH, et al. The experiment in biological habit of Salangid in Dongpu Reservoir and its multiplication. Journal of Anhui Agricultural Sciences, 1998, 11061106(4): 383-385 [徐桂珍, 胡波, 鲍传和, 等. 董铺水库太湖新银鱼生物学及其增殖试验. 安徽农业科学, 1998, 11061106(4): 383-385] |
Yang ZW, Li ZJ, Liu JS, et al. A comparative study on reproductive characteristics of different spawning stocks of the icefish (Neosalanx taihuensis) in the Danjiangkou Reservoir. Freshwater Fisheries, 2012, 42(5): 58-62 [杨战伟, 李钟杰, 刘家寿, 等. 丹江口水库太湖新银鱼不同繁殖群体的繁殖特征比较. 淡水渔业, 2012, 42(5): 58-62] |
Zhang D, Lei GC, Gong C, et al. Genetic diversity of Neosalanx taihuensis based on mitochondrial COⅠ sequences. Journal of Lake Sciences, 2012, 24(2): 299-306 [张迪, 雷光春, 龚成, 等. 基于COⅠ基因序列的太湖新银鱼遗传多样性. 湖泊科学, 2012, 24(2): 299-306] |
Zhang DY, Wang CQ, Liu SP, et al. Population genetic structure analysis of endangered species Placocheilus cryptonemus in the Nujiang River based on Cyt b sequences of mtDNA. Journal of Fishery Sciences of China, 2009, 16(4): 477-486 [张东亚, 汪登强, 刘绍平, 等. 怒江濒危鱼类缺须盆唇鱼基于线粒体Cytb序列的群体遗传结构分析. 中国水产科学, 2009, 16(4): 477-486] |
Zhang GX. Scientific treatise on systematic and evolutionary zoology (Vol 2). Beijing: China Science and Technology Press, 1993: 65-67 [张广学. 系统进化动物学论文集(第二集). 北京: 中国科学技术出版社, 1993: 65-67]
|
Zhang J, Li M, Xu MQ, et al. Molecular phylogeny of icefish Salangidae based on complete mtDNA cytochrome b sequences, with comments on estuarine fish evolution. Biological Journal of the Linnean Society, 2007, 91(2): 325-340 DOI:10.1111/j.1095-8312.2007.00785.x |
Zhang JF, Hao PY, Nie LW, et al. Studies on the analysis of sequences of COⅡ and its connected tRNA genes of mitochondrial genome from 2 species of salangidae and their phylogenetic relationship. Periodical of Ocean University of China, 2008, 38(3): 424-428 [张际峰, 郝培应, 聂刘旺, 等. 2种银鱼线粒体COⅡ及侧翼tRNA基因的测定分析及其亲缘关系研究. 中国海洋大学学报, 2008, 38(3): 424-428] |
Zhang Y, Dong S, Wang Q, et al. The isozyme genetic structures in large icefish (Protosalanx hyalocranius) and Taihu Lake icefish (Neosalanx taihuensis). Journal of Dalian Fisheries University, 2005, 20(2): 111-115 [张颖, 董仕, 王茜, 等. 大银鱼和太湖新银鱼同工酶遗传组成的研究. 大连水产学院学报, 2005, 20(2): 111-115] |
Zhang ZS, Hu JB, Ye XY, et al. Genetic diversity of the pregnant's schizothoracin (Schizothorax prenati) based on partial mtDNA cytb sequences. Acta Hydrobiologia Sinica, 2017, 41(3): 609-616 [张争世, 胡冰洁, 叶祥益, 等. 基于mtDNA Cyt b序列分析齐口裂腹鱼群体遗传多样性. 水生生物学报, 2017, 41(3): 609-616] |
Zhao L, Xie BG, Liu ZJ, et al. Molecular structure and DNA substitution rate of the mitochondrial control region and cytochrome b in Taihu salangid, Neosalanx taihuensis. Chinese Journal of Zoology, 2010, 45(2): 27-38 [赵亮, 谢本贵, 刘志瑾, 等. 太湖新银鱼线粒体D-loop和Cyt b片段序列结构与进化速率比较. 动物学杂志, 2010, 45(2): 27-38] |
Zhao L, Zhang J, Liu ZJ, et al. Complex population genetic and demographic history of the Salangid, Neosalanxtaihuensis, based on cytochrome b sequences. BMC Evolutionary Biology, 2008, 8(5): 201 |
Zhao L, Zhang J, Liu ZJ, et al. Population genetic structure and demographic history of Neosalanx jordani based on cytochrome b sequences. Biodiversity Science, 2010, 18(3): 251-26 [赵亮, 张洁, 刘志瑾, 等. 乔氏新银鱼基于细胞色素b序列的种群遗传结构和种群历史. 生物多样性, 2010, 18(3): 251-261] |
Zhao LS, Cheng F, Zhang L, et al. Growth, mortality and evaluation of resource utilization for two salangidae, Protosalanx hyalocranius and Neosalanx taihuensis in Hongze Lake. Acta Hydrobiologia Sinica, 2018, 42(2): 240-249 [赵丽爽, 程飞, 张磊, 等. 洪泽湖大银鱼和太湖新银鱼的生长、死亡参数及资源利用状况. 水生生物学报, 2018, 42(2): 240-249] |
Zhou HX, Hu YT, Duan GQ, et al. Population genetic analyses of Acrossocheilus wenchowensis in Xin'an Basin based on the mitochondrial DNA cytochrome b gene. Progress in Fishery Sciences, 2019, 40(2): 43-50 [周华兴, 胡玉婷, 段国庆, 等. 基于线粒体细胞色素b基因序列的新安江流域温州光唇鱼群体遗传研究. 渔业科学进展, 2019, 40(2): 43-50] |



