2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;
3. 南京农业大学无锡渔业学院 无锡 214081;
4. 中国海洋大学海洋生命学院 青岛 266003;
5. 上海海洋大学水产与生命学院 上海 201306
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071;
3. Wuxi Fisheries College, Nanjing Agricultural University, Wuxi 214081;
4. College of Marine Life Sciences, Ocean University of China, Qingdao 266003;
5. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
金乌贼(Sepia esculenta Hoyle, 1885)俗称墨鱼或乌鱼,主要分布于中国沿海海域、日本北海道以南、朝鲜西南海域及菲律宾群岛海域(Okutani, 1995)。金乌贼是一种具有较高经济价值的优良品种,曾是我国渤、黄海重要捕捞对象。然而,20世纪80年代以来,由于过度捕捞、栖息地破坏等原因,金乌贼资源急剧衰退(郝振林等, 2007)。金乌贼具有营养丰富、生长迅速、个体大(均重700 g)、生命周期短(约1年)、世代更新快和洄游规律性强等特点,为保护和恢复金乌贼资源,我国学者在金乌贼基础生物学、增养殖技术及种群遗传学等方面开展了系列研究(郑小东等, 2002、2003; 赵厚钧等, 2004; 郝振林等, 2008; 韩松等, 2010; Zheng et al, 2007、2009),并于2010年突破了金乌贼人工繁育和增殖放流技术(佚名, 2010 )。为更好地开展金乌贼繁育及增殖养护工作,有必要从分子层面精准评估现有种群的种质状况和遗传多样性。
简单重复序列(Simple sequence repeat, SSR)分子标记技术,也称为微卫星DNA序列标记,其串联重复的核心序列为1~6个核苷酸(Powell et al, 1996; Toth et al, 2000)。作为一种遗传标记,SSR广泛存在于原核和真核生物基因中,具有重复性好、共显性高、稳定性强、多态性丰富等特点(Tuler et al, 2015)。按照SSR的来源,可分为表达序列标签SSR (EST-SSR)和基因组SSR(g-SSR)。近年来,相对于传统的EST- SSR开发,通过转录组测序平台开发SSR标记已在多种水产动物中得到应用。本研究在金乌贼Pacific Biosciences单分子实时(Single-molecule real-time, SMRT)测序技术和Illumina RNA-Seq技术转录组测序获得生物学数据信息的基础上,利用Micro Satellite (MISA)软件筛选SSR位点并分析其分布及组成特征,以期为后续的金乌贼群体遗传结构分析、遗传图谱构建、重要性状QTL定位和良种选育等研究提供基础数据。
1 材料与方法 1.1 动物材料实验用金乌贼于2017年1月~7月取自青岛金沙滩水产开发有限公司,挑选体形完整,无伤害的个体。分别从生长(约6月龄)、产卵(10~11月龄)和产后濒死(11~12月龄)三个关键时期随机选择3个雌性金乌贼亲体。从选择的雌性个体解剖脑亚脚叶/嗅叶、视腺、卵巢和缠卵腺组织样品,浸没在RNAlater(天根, 中国)中,并于–80℃储存。
1.2 转录组数据来源提取金乌贼不同时期的脑亚脚叶/嗅叶、视腺、卵巢和缠卵腺组织样本的总RNA,送天津诺禾致源生物信息科技有限公司,采用PacBio SMRT测序技术和Illumina RNA-Seq技术进行转录组测序,通过整合PacBio SMRT和Illumina测序数据,生成高质量的金乌贼转录组,包含177, 951条全长转录本(Zhang et al, 2019),以此作为分析数据。
1.3 金乌贼EST-SSR的筛选为检测金乌贼的SSR位点,利用MISA(v1.0) (http://pgrc.ipk-gatersleben.de/misa)对其Unigene序列进行分析。SSR位点包含了单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸以及六核苷酸重复。判断的标准为单核苷酸重复至少12次;二核苷酸重复至少6次;三核苷酸重复至少5次、四核苷酸重复至少5次、五核苷酸重复至少4次、六核苷酸重复至少4次。
2 结果与分析 2.1 金乌贼转录组SSR位点数量与分布金乌贼转录组测序共获得177, 951条全长转录本,序列总长度为198, 175, 926 bp。检索Unigene序列,共发现161, 327个SSR位点,分布在64, 933条Unigene中,发生频率(含SSR的Unigene数与Unigene总数之比)为36.49%,出现频率(SSR数目与Unigene总数之比)为90.66%,平均每1, 228 bp含有1个SSR位点。此外,有33, 136条Unigene含超过1个SSR位点,33, 260条Unigene含有复合型SSR(表 1)。
|
|
表 1 金乌贼转录组中的SSR搜索结果 Tab.1 Searching results of SSR in transcriptome of S. esculenta |
金乌贼EST-SSR类型丰富,从表 2可以发现,单核苷酸到四核苷酸重复最多,占总SSR位点数量的99.55%,其中,单核苷酸和二核苷酸所占的比例较大,分别占总数的46.00%和39.93%;其次为三核苷酸和四核苷酸,分别为9.48%和4.14%。五、六核苷酸重复率总计0.448%,数量很少。
|
|
表 2 金乌贼EST-SSR不同重复基元分布情况 Tab.2 Distribution of different repeat motifs in S. esculenta |
金乌贼SSR中,单核苷酸重复以A/T重复基序最多,占总SSR的44.79%;二核苷酸重复以AT/AT和AC/GT重复基序为主要类型,共占SSR总数的34.73%;三核苷酸重复则以AAT/ATT和ATC/ATG出现频率最高,共占4.71%;四核苷酸重复则以AAAG/CTTT居多,AGAT/ATCT次之。其他五核苷酸和六核苷酸重复基元类型较多,数量非常少,出现频率很低,见表 3。
|
|
表 3 金乌贼EST-SSR重复基元的类型 Tab.3 Type of EST-SSR repeat motifs in S. esculenta |
重复片段的长度会影响SSR多态性,而SSR重复次数可以决定重复片段的长度。金乌贼SSR的碱基重复次数很广泛,整体波动在5~100次范围内,多集中于5~29次。单核苷酸重复12~102次;二核苷酸重复6~50次;三核苷酸重复5~24次;四核苷酸重复6~19次;五核苷酸重复6~19次;六核苷酸重复6~12次。重复12次的频率最高,共有18, 289条,占总SSR数的11.34%,其次是重复6次(10.98%)、13次(7.73%)、7次(6.82%)、14次(6.28%),30次以上(含30)占3.40%。总体上,金乌贼转录组SSR的重复次数以11~20次最多,占总SSR比例为47.92%,4~10次次之,为35.03%;重复次数21~30次为14.24%;重复次数大于30次为2.80% (图 1)。
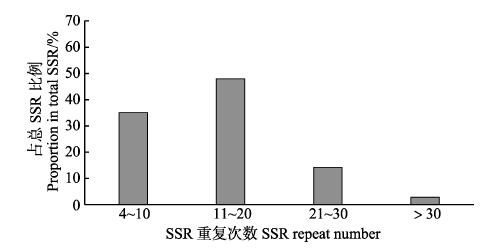
|
图 1 金乌贼转录组EST-SSR不同重复基元的重复次数 Fig.1 Repeat numbers of EST-SSR with different motifs in transcriptome of S. esculenta |
SSR位点中重复基元的长度是影响其多态性高低的重要因素,当SSR≥20 bp时,其多态性高;重复基元长度12≤SSR<20 bp时,多态性中等;而低于12 bp时,多态性极低。本研究中去除了12 bp以下的SSR,长度≥12 bp的SSR共有161, 327个。从金乌贼转录组中SSR基元长度分布情况可以看出,其中,重复基元长度在12~19 bp的SSR位点最多,共有95, 323个,占SSR总数的59.09%;其次为20~30 bp,共有43, 977个,占SSR总数的27.26%;长度在31~40 bp的,共10, 931个,占SSR总数的6.78%;长度在41~ 50 bp的,共5, 588个,占SSR总数的3.46%;基元长度大于50 bp的数量最少,共5, 508(3.41%)。根据该结果,可推测该研究中Ⅱ型SSR(长度为12~20 bp)的SSR的比例为59.09%,具有中等多态性,有较高的潜在可用性。而具有高度多态性的Ⅰ型SSR(长度≥20 bp)中,含有低级基元(二、三核苷酸重复)SSR位点数量占优,这部分SSR位点具有丰富的多态性(图 2)。
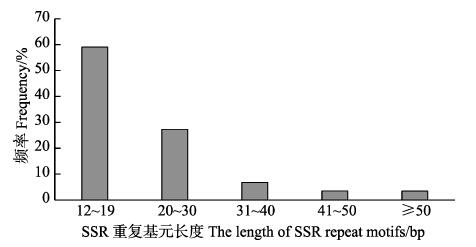
|
图 2 金乌贼转录组SSR位点重复基元序列长度分布频率 Fig.2 Frequency of SSR repeat sequence length distribution in transcriptome of S. esculenta |
本研究以金乌贼转录组信息对其EST-SSR进行开发。结果显示,金乌贼转录组中含有丰富的SSR位点。从金乌贼转录组的177, 951条Unigene中,共搜索出161, 327个SSR,分布在64, 933条Unigene中,SSR的出现频率为90.66%。与其他水生动物SSR的出现频率相比,明显高于扁玉螺(Neverita didyma) (86.53%) (卢玮筱等, 2018)、黄姑鱼(Nibea albiflora) (39.30%) (龚诗琦等, 2016)、翘嘴鳜(Siniperca chuatsi) (27.51%) (袁文成等, 2015)、罗氏沼虾(Macrobrachium rosenbergii) (45.91%) (王传聪等, 2018)、无针乌贼(Sepiella japonica) (48.70%) (吕振明等, 2017; 管奥等, 2018)、马氏珠母贝(Pinctada martensii) (13.34%) (王忠良等, 2015)、栉江珧(Atrina pectinata) (8.2%) (李东明等, 2017)、缢蛏(Sinonovacula constricta) (8.89%) (刘博等, 2012)、泥蚶(Tegillarca granosa) (14.83%) (周小龙等, 2013) (表 5)。
|
|
表 5 目前已获得EST-SSRs出现频率的部分水生动物 Tab.5 Some aquatic animals that have obtained the frequency of EST-SSRs |
SSR检索标准、数据库丰富程度以及物种差异,都会影响SSR的出现频率(Varshney et al, 2005)。如张秀英等(2012)采用不同检索条件从栉孔扇贝BES文库筛选SSR,表明SSR出现频率随检索标准的提高而降低。数据库丰富程度也会影响EST-SSR位点出现频率,石耀华等(2008)和王忠良等(2015)先后从马氏珠母贝6, 979和74, 007条EST序列中筛选得到243和9, 872个EST-SSR位点,位点出现频率分别为3.48%和13.34%。Franklin等(2005)和王艳红等(2011)对凡纳滨对虾(Litopenaeus vannamei)不同数量的EST序列进行EST-SSR筛选,其出现频率分别为4.9%和7.8%。
大量研究表明,在已开发的EST-SSR标记中,二核苷酸和三核苷酸重复基元类型最为丰富。虾夷扇贝和栉江珧的EST-SSR均以二核苷酸重复基元所占比例最高,分别占40.54%和77.08% (李云峰等, 2010; 李东明等, 2017);缢蛏SSR以三核苷酸重复SSR比例最高,为37.13% (刘博等, 2012)。本研究发现,金乌贼EST-SSR重复类型中,以单核苷酸重复基元最多,比例达到46.00%,其次为二核苷酸,为39.93%。这和无针乌贼(吕振明等, 2017)、马氏珠母贝(王忠良等, 2015)、罗氏沼虾(王传聪等, 2018)和扁玉螺(卢玮筱等, 2018)中单核苷酸重复基元是主导类型的研究结果相似。同时,本研究发现,金乌贼转录组中,SSR位点最多的是单核苷酸的重复基元A/T,占总SSR的44.79%;其次为二核苷酸的AT/AT和AC/GT,共占SSR总数的34.73%。此外,金乌贼转录组SSR位点重复基元序列长度在20 bp以上的SSR位点共有66, 004个,占SSR总数的40.91%,并且其中含有低级基元(二、三核苷酸重复) SSR位点数量占优,这部分SSR可能具有较高的利用价值。
4 结论整体而言,金乌贼转录组SSR出现频率较高且类型丰富,共检测出161, 327个SSR位点,分布在64, 933条Unigene中,SSR位点发生频率为36.49%,出现频率高达90.66%。这些SSR在提高物种遗传多样性潜能方面发挥着重要的作用。研究结果为下一阶段开发金乌贼多态性SSR分子标记提供了基础数据,也为开展金乌贼种质资源保护利用、遗传多样性评价和未来分子标记辅助育种等研究奠定基础。
Franklin P, Juan O, Mariuxi Z, et al. Development of EST-SSR markers by data mining in three species of shrimp: Litopenaeus vannamei, Litopenaeus stylirostris, and Trachypenaeus birdy. Marin Biotechnology, 2005, 7(5): 554-569 DOI:10.1007/s10126-004-5099-1 |
Gong SQ, Wang ZY, Xiao SJ, et al. Development and verification of SSR based on transcriptome of yellow drum, Nibea albiflora. Journal of Jimei University (Natural Science), 2016, 21(4): 241-246 |
龚诗琦, 王志勇, 肖世俊, 等. 黄姑鱼转录组SSR的开发与验证. 集美大学学报(自然科学版), 2016, 21(4): 241-246 |
Guan A, Wu YT, Chen Y, et al. Deep sequence-based transcriptome analysis of microsatellites in the cuttlefish (Sepiella japonica). Progress in Fishery Sciences, 2018, 39(3): 144-151 |
管奥, 毋玉婷, 陈宇, 等. 曼氏无针乌贼转录组微卫星特征分析. 渔业科学进展, 2018, 39(3): 144-151 |
Han S, Zheng XD, Li Q, et al. Ultrastructure of Sepia esculenta (Cephalopoda: Sepiida) spermatozoa and its damage by low temperature. Oceanologia et Limnologia Sinica, 2010, 41(4): 483-488 |
韩松, 郑小东, 李琪, 等. 金乌贼(Sepia esculenta)精子超微结构和低温损伤电镜观察. 海洋与湖沼, 2010, 41(4): 483-488 |
Hao ZL, Zhang XM, Zhang PD, et al. Fluorescent marking method in golden cuttlefish Sepia esculenta (Cephalopoda: Sepiidae). Journal of Fisheries of China, 2008, 32(4): 577-583 |
郝振林, 张秀梅, 张沛东, 等. 金乌贼荧光标志方法的研究. 水产学报, 2008, 32(4): 577-583 |
Hao ZL, Zhang XM, Zhang PD. Biological characteristics and multiplication techniques of Sepia esculenta. Chinese Journal of Ecology, 2007, 26(4): 601-606 |
郝振林, 张秀梅, 张沛东. 金乌贼的生物学特性及增殖技术. 生态学杂志, 2007, 26(4): 601-606 |
Li DM, Yang AG, Wu B, et al. Development and application of the EST-SSR markers in Atrina pectinate. Progress in Fishery Sciences, 2017, 38(2): 137-142 |
李东明, 杨爱国, 吴彪, 等. 栉江珧(Atrina pectinata) EST-SSR标记的开发与应用. 渔业科学进展, 2017, 38(2): 137-142 |
Li YF, Liu WD, Gao XG, et al. Construction of cDNA libraries from mantle and kidney of Japanese scallop (Mizuhopecten yessoensis) and ESTs analysis. Journal of Fishery Sciences of China, 2010, 17(3): 578-585 |
李云峰, 刘卫东, 高祥刚, 等. 虾夷扇贝外套膜和肾脏组织cDNA文库构建以及EST的初步分析. 中国水产科学, 2010, 17(3): 578-585 |
Liu B, Shao YQ, Teng SS, et al. Characterization, development and utilization of EST-derived microsatellites in Sinonovacula constricta. Oceanologia et Limnologia Sinica, 2012, 43(1): 132-137 |
刘博, 邵艳卿, 滕爽爽, 等. 缢蛏(Sinonovacula constricta) EST-SSR分布特征及引物开发利用. 海洋与湖沼, 2012, 43(1): 132-137 |
Liu HT, Liu JY, Yang MQ, et al. SSR and SNP polymorphic feature analysis based on Cheilinus undulates transcriptome. Genomics and Applied Biology, 2020, 1-28 |
刘洪涛, 刘金叶, 杨明秋, 等. 基于转录组测序的波纹唇鱼SSR和SNP多态特征分析. 基因组学与应用生物学, 2020, 1-28 |
Lu WX, Chen YX, Qi PZ. Analysis of SSR information in the transcriptional group of Neverita didyma. Progress in Fishery Sciences, 2018, 37(3): 215-220 |
卢玮筱, 陈永霞, 祁鹏志. 扁玉螺转录组SSR信息分析. 渔业科学进展, 2018, 37(3): 215-220 |
Lü ZM, Hou L, Gong L, et al. Isolation and analysis on EST microsatellites of Sepiella japonica by de novo high- throughput transcriptome sequencing. Oceanologia et Limnologia Sinica, 2017, 48(4): 877-883 |
吕振明, 侯龙, 龚理, 等. 基于de novo高通量测序的曼氏无针乌贼(Sepiella japonica) ESTs中微卫星位点筛选与特征分析. 海洋与湖沼, 2017, 48(4): 877-883 |
Okutani T. Cuttlefish and squids of the world in color. Tokyo: National Cooperative Association of Squid Processors, 1995, 1–185
|
Powell W, Machray GC, Provan J. Polymorphism revealed by simple sequence repeats. Trends in Plant Science, 1996, 1(7): 215-222 DOI:10.1016/S1360-1385(96)86898-0 |
Rizhao Fisheries Research Institute. The first success of the artificial seedling rearing of Sepia esculenta Hoyle in our country. Fishery Information And Strategy, 2010, 25(4): 37 |
日照市水产研究所. 金乌贼全人工育苗在我国首获成功. 渔业信息与战略, 2010, 25(4): 37 |
Shi YH, Hong K, Guo XM, et al. Microsatellite markers screening from EST sequences of Pinctada martensii Dunker. Journal of Fisheries of China, 2008, 32(2): 174-181 |
石耀华, 洪葵, 郭希明, 等. 马氏珠母贝EST微卫星的筛选. 水产学报, 2008, 32(2): 174-181 |
Tan J, Yao GY, Liu ZG, et al. SSR information analysis and unigene functional annotation of transcriptome from Argopecten irradians "Bohaihong" and Argopecten irradians concentricus. Genomics and Applied Biology, 2018, 37(12): 5242-5250 |
谭杰, 姚高友, 刘志刚, 等. 扇贝"渤海红"与墨西哥湾扇贝转录组SSR信息分析及unigene功能注释. 基因组学与应用生物学, 2018, 37(12): 5242-5250 |
Toth G, Gaspari ZJ. Microsatellites in different eukaryotic genomes survey and analysis. Genome Research, 2000, 10(7): 967-981 DOI:10.1101/gr.10.7.967 |
Tuler AC, Carrijo TT, Nóia LR, et al. SSR markers: A tool for species identification in Psidium (Myrtaceae). Molecular Biology Reports, 2015, 42(11): 1501-1513 DOI:10.1007/s11033-015-3927-1 |
Varshney RK, Graner A, Sorrells ME. Genic microsatellite markers in plants: Features and applications. Trends in Biotechnology, 2005, 23(1): 48-55 DOI:10.1016/j.tibtech.2004.11.005 |
Wang CC, Tang XY, Xiang J, et al. Analysis of SSR marker information in the transcriptome of Macrobrachium rosenbergii. Jiangsu Agricultural Sciences, 2018, 46(22): 56-59 |
王传聪, 唐修阳, 项杰, 等. 罗氏沼虾转录组SSR标记信息分析. 江苏农业科学, 2018, 46(22): 56-59 |
Wang QL, Liu Y, Song HM, et al. Development and primer selection of EST-SSR molecular markers based on transcriptome sequencing of Osteoglossum bicirrhosum. Freshwater Fisheries, 2016, 46(6): 8-13 |
王且鲁, 刘奕, 宋红梅, 等. 双须骨舌鱼转录组EST-SSR标记开发与引物筛选. 淡水渔业, 2016, 46(6): 8-13 |
Wang YH, Hu CQ, Zhang LP, et al. A preliminary study on microsatellite markers screening from EST sequences of Litopenaeus vannamei. Journal of Fisheries of China, 2011, 35(7): 969-976 |
王艳红, 胡超群, 张吕平, 等. 凡纳滨对虾EST微卫星标记初步筛选. 水产学报, 2011, 35(7): 969-976 |
Wang ZL, Ding Y, Xu YH, et al. Polymorphism of EST-SSRs from Pinctada martensii based on transcriptome datasets. Oceanologia et Limnologia Sinica, 2015, 46(3): 687-693 |
王忠良, 丁燏, 许尤厚, 等. 基于转录组数据的马氏珠母贝EST-SSR位点的信息分析及其多态性检测. 海洋与湖沼, 2015, 46(3): 687-693 |
Yuan WC, Huang HZ, Li WL, et al. Analysis of EST-SSRs information in Siniperca chuatsi transcriptome and detection of polymorphism. Oceanologia et Limnologia Sinica, 2015, 46(2): 403-409 |
袁文成, 黄鹤忠, 李文龙, 等. 翘嘴鳜(Siniperca chuatsi)转录组EST-SSR位点的信息分析及其多态性检测. 海洋与湖沼, 2015, 46(2): 403-409 |
Zhang JY, Liu CL, He MC, et al. A full-length transcriptome of Sepia esculenta using a combination of single-molecule long-read (SMRT) and Illumina sequencing. Marine Genomics, 2019, 43: 54-57 DOI:10.1016/j.margen.2018.08.008 |
Zhang XY, Zhang XJ, Zhao C, et al. The development of BAC-end sequence-based microsatellite markers and analysis on population genetic diversity in Zhikong scallop (Chlamys farreri). Journal of Fisheries of China, 2012, 36(6): 815-824 DOI:10.3724/SP.J.1231.2012.27808 |
张秀英, 张晓军, 赵翠, 等. 栉孔扇贝BES-SSR的开发及遗传多样性分析. 水产学报, 2012, 36(6): 815-824 |
Zhao HJ, Wei BF, Hu M, et al. Preliminary study on Sepia esculenta oosperm hatching and effects of different adhesion substrates. Transactions of Oceanology and Limnology, 2004, 11(3): 64-68 |
赵厚钧, 魏邦福, 胡明, 等. 金乌贼受精卵孵化及不同材料附着基附卵效果的初步研究. 海洋湖沼通报, 2004, 11(3): 64-68 |
Zheng XD, Ikeda M, Kong LF, et al. Genetic diversity and population structure of the golden cuttlefish, Sepia esculenta (Cephalopoda: Sepiidae) indicated by microsatellite DNA variations. Marine Ecology, 2009, 30(4): 448-454 DOI:10.1111/j.1439-0485.2009.00294.x |
Zheng XD, Ikeda M, Barinova A, et al. Isolation and characterization of microsatellite DNA loci from the golden cuttlefish, Sepia esculenta Hoyle (Cephalopoda). Molecular Ecology Resources, 2007, 7(1): 40-42 |
Zheng XD, Wang RC. Morphological study on radula of nine cephalopods in the coastal waters of China. Journal of Fisheries of China, 2002, 26(5): 417-422 |
郑小东, 王如才. 中国沿海九种头足类齿舌的形态学. 水产学报, 2002, 26(5): 417-422 |
Zheng XD, Yang JM, Wang HY, et al. Analysis and evaluation of the nutritive composition of the ink of the golden cuttlefish Sepia esculenta. Chinese Journal of Zoology, 2003, 38(4): 32-35 |
郑小东, 杨建敏, 王海艳, 等. 金乌贼墨汁营养成分分析及评价. 动物学杂志, 2003, 38(4): 32-35 |
Zhou XL, Zhu JH, Dong YH, et al. Development and comparative study of genomic-SSR and EST-SSR in Tegillarca granosa. Oceanologia et Limnolgia Sinica, 2013, 44(2): 467-475 |
周小龙, 朱靖华, 董迎辉, 等. 泥蚶(Tegillarca granosa)基因组SSR和EST-SSR的开发及比较研究. 海洋与湖沼, 2013, 44(2): 467-475 |



