of Enterocytozoon hepatopenaei
虾肝肠胞虫(Enterocytozoon hepatopenaei, EHP)是一种严格细胞内寄生的微孢子虫,成熟的孢子呈椭圆形,大小为(1.4±0.3)×(0.8±0.1) μm,后端有1个空泡,胞内含1个细胞核,5~6圈极丝,1个与极丝相连的锚定盘和1层电子致密的孢子壁(乔毅等, 2018)。EHP于2009年首次从斑节对虾(Penaeus monodon)中发现并命名(Tourtip et al, 2009),感染EHP的对虾往往正常进食,但生长缓慢甚至停滞,直接影响到养殖产量,给养殖户带来严重的经济损失(刘珍等, 2016; 宋增磊等, 2019),如何预防和治疗EHP已经成为对虾养殖产业急需解决的科学问题。明确EHP的入侵机理是切断EHP的传播途径和针对性用药的基础。EHP的一些生物合成途径和三羧酸循环的基因缺失表明其对宿主有很强的依赖性(Katinka et al, 2001),需要从宿主吸收营养完成生活史发育,因此,在进化过程中微孢子虫进化出一种极其复杂和独特的感染机制以抵抗宿主免疫系统的攻击。
分泌蛋白(Secreted protein)是在细胞内合成后分泌到细胞外起作用的蛋白质,已有研究表明,其是介导真核病原微生物与宿主之间相互作用的重要因子(McKerrow et al, 1993; Gupta et al, 2012)。在其他微孢子虫中已有研究表明,一些分泌蛋白在虫体入侵宿主过程中扮演着重要角色(李田等, 2013; Wang et al, 2015)。因此,查找和分析EHP的分泌蛋白对于揭示入侵机理和虫体的免疫逃避将具有重要意义。目前,对EHP在虾体内的感染机制了解甚少,尤其是在宿主–寄生虫相互作用水平上。本研究在基因组测序数据的基础上,利用生物信息学方法在全基因组范围内预测EHP的分泌蛋白,同时,对所得分泌蛋白的功能进行注释,并对其序列特征和基序进行分析,研究结果可为筛选EHP致病相关因子和探究其侵染机理提供参考。
1 材料与方法 1.1 基因组数据来源和主要分析软件虾肝肠胞虫基因组数据来源于GenBank数据库(http://www.ncbi.nlm.nih.gov),其NCBI登录号为MNPJ00000000.1。
用到的主要分析软件和网站如下:蛋白质跨膜结构域预测软件Tmhmm-2.0c(Krogh et al, 2001),线粒体蛋白预测软件MitoProtⅡ(Claros et al, 1996),细胞核蛋白预测软件Nucpred-1.1(Brameier et al, 2007)、NLStradamus.1.7(Nguyen Ba et al, 2009)和PredictNLS (https://rostlab.org/owiki/index.php/PredictNLS),蛋白质信号肽预测软件SignalP-4.0(Petersen et al, 2011),蛋白质GPI锚定位点预测软件Kohgpi-1.5(Fankhauser et al, 2005),蛋白质亚细胞定位预测软件TargetP-1.1 (Emanuelsson et al, 2000)和WoLF PSORT(Horton et al, 2007),真核生物分泌蛋白预测程序EuSecPred2.0 (https://silkpathdb.swu.edu.cn/eusecpred),氨基酸组成分析软件WebLogo(http://weblogo.threeplusone.com/create.cgi) (Crooks et al, 2004),蛋白质序列基序分析程序为MEME(http://meme-suite.org/tools/meme) (Bailey et al, 2009)。
1.2 分泌蛋白的预测针对EHP全基因组的蛋白质编码序列,运行EuSecPred 2.0在线流程筛选分泌蛋白(Druzhinina et al, 2012)。由于本程序的局限性,只能预测到经典途径分泌的蛋白质,可能会漏掉部分非经典途径分泌的蛋白质。运行Tmhmm程序去除跨膜蛋白,运行Nucpred、NLStradamus.pl和PredictNLS程序剔去其中的细胞核蛋白质,然后,通过SignalP-4.0过滤掉其中无信号肽序列的蛋白质,通过MitoProt程序过滤掉定位于线粒体的蛋白,利用TargetP和WoLF PSORT对具有信号肽的蛋白质进行亚细胞定位预测,保留定位于细胞膜外的蛋白质,最后,利用Kohgpi程序剔除具有GPI锚定位点的蛋白质,最终得到EHP的分泌蛋白集合。
1.3 分泌蛋白的功能分析利用BLASTP程序,将预测获得的EHP分泌蛋白序列分别与数据库Nr和Swiss-Prot数据库进行比对,将比对结果中得分最高的同源序列的功能作为对应分泌蛋白的功能参考(李田等, 2013; 罕园园等, 2014)。
1.4 分泌蛋白的序列特征分析根据SingalP-4.0信号肽预测的结果,截取所有分泌蛋白的信号肽序列,并统计分泌蛋白和信号肽序列的长度以及各类氨基酸组成,同时,截取信号肽剪切位点前后各3个氨基酸,并利用WebLogo对其组成进行统计,然后,利用MEME程序预测分泌蛋白和信号肽中的基序。
2 结果与分析 2.1 预测的虾肝肠胞虫分泌蛋白利用EuSecPred流程对获得的2548个虾肝肠胞虫蛋白质序列进行预测,发现具有跨膜域的蛋白有2018个,1741个蛋白质具有细胞核定位信号,144个蛋白质具有信号肽序列,126个蛋白具有线粒体定位信号,119个蛋白具有亚细胞定位信号,筛选后最终获得了109个分泌蛋白(表 1)。
|
|
表 1 预测的虾肝肠胞虫分泌蛋白基因及序列分析 Tab.1 Coding genes and sequence information of the predicted EHP secreted proteins |
预测获得109个分泌蛋白,对其进行功能注释得出,其中60个蛋白质(占预测所得分泌蛋白的54.05%)为无明确功能信息的蛋白质,另外49个分泌蛋白在数据库中检索到了同源蛋白,其中酶类数目最多,包括具有调控宿主细胞增殖和免疫系统功能的酶类:多肽N-乙酰氨基半乳糖转移酶和泛素羧基末端水解酶;同时,还预测到1个孢壁蛋白:SWP7,孢壁蛋白被认为在粘附、侵染以及致病等方面扮演着重要角色;另外,预测到2种蛋白抑制因子:α-胰蛋白酶抑制剂重链H1、亮氨酸拉链假定肿瘤抑制因子2,这2种蛋白抑制因子可能会抑制宿主免疫系统、调控细胞增殖;还预测到2种糖蛋白(图 1)。
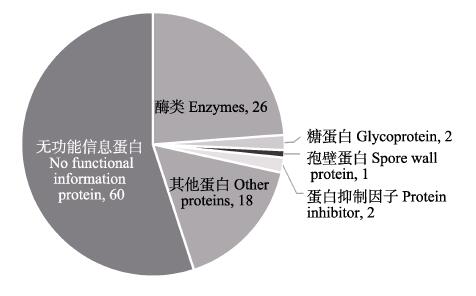
|
图 1 虾肝肠胞虫分泌蛋白的功能分类 Fig.1 Functional classification of EHP secreted proteins |
虾肝肠胞虫109个分泌蛋白序列长度范围为30~700 aa,主要集中在30~400 aa,平均长度为219.34 aa,中值为181.00 aa(图 2);信号肽长度范围在9~32 aa,主要长度集中在15~20 aa,平均长度为18.09 aa,中值为18.00 aa (图 3)。
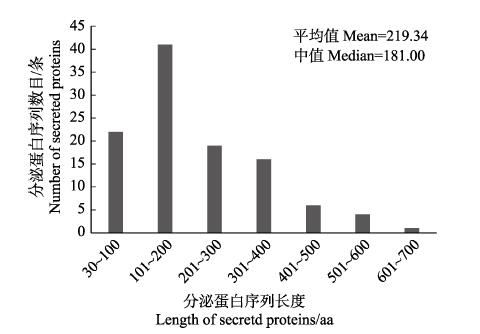
|
图 2 虾肝肠胞虫分泌蛋白氨基酸长度分析 Fig.2 Analysis of the lengths of EHP scecreted proteins |

|
图 3 虾肝肠胞虫分泌蛋白信号肽长度分析 Fig.3 Analysis of the signal peptides length of secreted proteins in EHP |
对分泌蛋白和信号肽的氨基酸组成统计分析发现,分泌蛋白和信号肽均以疏水性氨基酸为主(图 4)。分泌蛋白序列中疏水性氨基酸占比为42.57%,其次是亲水性氨基酸,占比为29.30%。而信号肽同样以疏水性氨基酸为主,占比高达68.93%,而亲水性氨基酸比例仅为22.42%。
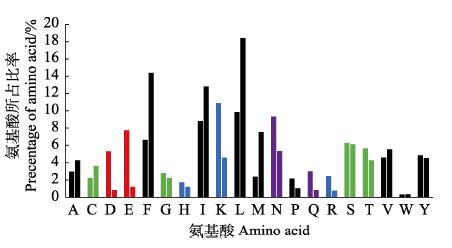
|
图 4 虾肝肠胞虫分泌蛋白的氨基酸组成 Fig.4 Amino acid composition of EHP secreted proteins A:丙氨酸Ala;C:半胱氨酸Cys;D:天冬氨酸Asp;E:谷氨酸Glu;F:苯丙氨酸Phe;G:甘氨酸Gly;H:组氨酸His;I:异亮氨酸Ile;K:赖氨酸Lys;L:亮氨酸Leu;M:甲硫氨酸Met;N:天冬酰胺Asn;P:脯氨酸Pro;Q:谷氨酰胺Gln;R:精氨酸Arg;S:丝氨酸Ser;T:苏氨酸Thr;V:缬氨酸Val;W:色氨酸Trp;Y:酪氨酸Tyr。每种氨基酸上方左侧柱为在所有分泌蛋白序列中占有的比率,右侧柱为在所有信号肽序列中占有的比率。黑色为疏水性氨基酸,绿色为亲水性氨基酸,红色为酸性氨基酸,蓝色为碱性氨基酸,紫色为中性氨基酸。下同 Left column above each amino acid is the ratio in all secreted protein sequences. Right column above each amino acid is the ratio in all signal peptide sequences.Black column: Hydrophobic amino acid; Green column: Hydrophilic amino acid; Red column: Acid amino acid; Blue column: Basic amino acid. The same as below |
统计信号肽剪切位点前后各3个氨基酸的组成(图 5),分析发现,与整个信号肽氨基酸组成类似,信号肽剪切位点氨基酸组成同样以疏水性氨基酸为主,其占比为47.90%,其中,异亮氨酸(I)和丙氨酸(A)含量最丰富;亲水性氨基酸占比为34.95%,其中,丝氨酸(S)和天冬酰胺(N)所占比例较高。从各个位置的氨基酸组成分析,上游的–3位主要为异亮氨酸(I),–2位主要为苯丙氨酸(F),紧邻信号肽剪切位点的–1和1位主要为丙氨酸(A),下游的3位同样主要为异亮氨酸(I) (图 6)。
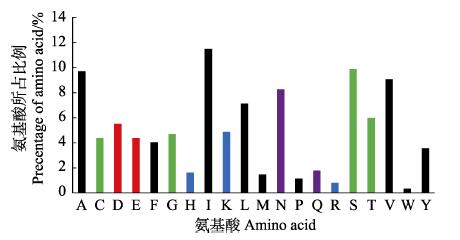
|
图 5 虾肝肠胞虫分泌蛋白信号肽剪切位点处氨基酸的组成 Fig.5 The composition of amino acids flanking the signal peptide splice site in the secretory proteins of EHP |
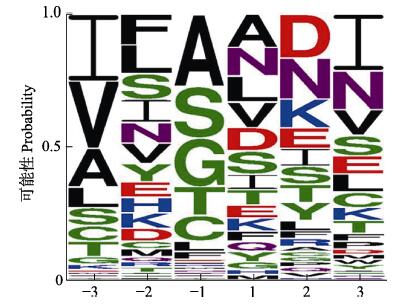
|
图 6 虾肝肠胞虫分泌蛋白信号肽剪切位点处氨基酸组成 Fig.6 Composition patterns of amino acids flanking the signal peptide splice site in the secreted proteins of EHP |
对虾肝肠胞虫信号肽区域的基序分析,发现了1种氨基酸组成模式:NV[VT][IK]CA[ED][SA](图 7)。对其非信号肽区分析发现3种基序:VAYDMFLSRCILHMLDVMMLYVE NESFMDQVAEVFCFNWTATEFYNSIIR,[QM]L[RK] [FA]QN[TR][ND]G[SC][NK]D[NE][KE]I[SK][QE]L[KF] [IE]KIK[TE][MI]C[NK][RK]I[ED][LA][IF][VI][NF][MK] [VI][VE]Q[QA][QM][TQ][YN][QE][LI]K[ME]DD[PH]和SCFGKFSFPISNRSAEYFKTVYDQWNELTVKIPV KIYRTTL(图 8)。

|
图 7 虾肝肠胞虫分泌蛋白信号肽基序 Fig.7 Motif in signal peptide of EHP secreted proteins |

|
图 8 虾肝肠胞虫非信号肽区基序 Fig.8 Motif in non-signal peptide of EHP secreted proteins |
对于专性胞内寄生的微孢子虫来说,目前,仅有一些关于孢壁蛋白分离方法的报道(吴正理等, 2007),尚没有分泌蛋白的分离方法。生物信息学算法的快速发展为成功预测微孢子虫的分泌蛋白提供了可能。本研究基于虾肝肠胞虫基因组数据库,运行EuSecPred 2.0在线流程在全基因组2548条蛋白质中进行筛选和预测,最终获得了109条分泌蛋白,这些分泌蛋白组成以疏水性氨基酸为主,这与其信号肽和剪切位点前后各3个氨基酸组成相一致。
本研究预测获得的109条分泌蛋白,其中,60个是无功能注释的蛋白质,这部分蛋白质可能是虾肝肠胞虫分泌的特有蛋白质。在有明确功能注释的49个分泌蛋白中,有些是与微孢子虫黏附和入侵相关的蛋白,如孢壁蛋白(Spore wall protein,SWP)、钙粘蛋白(Cadherin)和多肽N-乙酰氨基半乳糖转移酶12 (Polypeptide N-acetylgalactosam inyltransferase 12,GALNT12)。孢壁(Spore wall)是微孢子虫最外层的结构,在侵染过程中,孢壁应最先与宿主细胞接触。孢壁蛋白作为孢壁的主要成分,对维持孢壁稳固结构和保护胞内原生质起着重要作用,同时直接参与侵染过程中与宿主细胞的特异性识别过程。Jaroenlak等(2018)鉴定并分离了EHP的第1个孢壁蛋白(EhSWP1),在其N末端发现了3个肝素结合基序,进一步的体外结合实验、竞争实验和诱变研究表明,EhSWP1能够和肝素结合进而引发入侵。同样,Yang等(2018)研究证明,家蚕微孢子虫(Nosema bombycis)的部分孢壁蛋白在粘附和侵染过程中起关键作用,本研究中筛选的SWP7也可能与EHP的入侵相关。钙粘蛋白是1种Ca依赖的细胞粘着糖蛋白,可能与宿主细胞的靶向信号识别并粘附;同时,Frixione等(1994)研究表明,Ca2+会以某种方式触发孢子萌发。GALNT12是合成O-糖链的起始酶,而粘蛋白主要由O-糖链组成,参与很多与粘附相关的过程,O-糖链还可调控细胞表面受体的表达,进而影响细胞的生长和凋亡(刘可人等, 2006)。因此,推测EHP侵染过程中分泌的GALNT1可能参与到宿主细胞的识别和粘附。
在这些分泌蛋白中还有一些抑制宿主免疫功能的分泌蛋白。α-胰蛋白酶抑制剂重链H1(Inter-alpha- trypsin inhibitor heavy chain H1, ITIH1)可调控细胞外基质稳定并抑制C5转化酶活性(Zhuo et al, 2008),进而抑制宿主免疫系统。泛素羧基末端水解酶(Ubiquitin carboxyl-terminal hydrolase,UCH)可水解被泛素(Ubiquitin)标记的需要被分解的蛋白质,也可以标记跨膜蛋白,然后通过酶促反应相互连接,进而介导靶蛋白的降解或跨膜运输。Ciechanover等(2000)研究表明,泛素蛋白水解系统在广泛的细胞基本过程中发挥重要作用。其中,包括调节细胞周期、调节免疫和炎症反应、控制信号转导途径、发育和分化、DNA修复等。这些复杂的过程是通过蛋白质的单个或子集的特异性降解来控制的。虾肝肠胞虫UCH7在侵染过程中的信号传递及调控宿主免疫方面行使何种功能尚需研究进行明确。
另外,还有调控细胞增殖的蛋白,比如亮氨酸拉链假定肿瘤抑制因子2 (Leucine zipper putative tumor suppressor, LZTS2),拥有特定的亮氨酸拉链结构域(LZ),参与Wnt/β-catenin信号通路调控β-catenin的表达及细胞内分布,与NF-κB拥有广泛的相互作用,从而调控细胞增殖和凋亡(Peng et al, 2011; 王枭雄等, 2014)。在微管系统中,LZTS2也有重要的调节作用,可抑制细胞的有丝分裂和迁移(张轶等, 2016)。EHP分泌的LZTS2是否能抑制宿主细胞的增殖和迁移从而抑制宿主细胞行使其功能尚需进一步研究。
分泌蛋白在多种寄生虫系统中介导粘附和侵袭,对于明确胞内寄生虫的感染机制至关重要。本研究初步筛选了109个分泌蛋白,接下来进一步对这些分泌蛋白进行功能研究对于理解虾肝肠胞虫的入侵机理具有重要意义。
Nguyen Ba A, Pogoutse A, Provart N, et al. NLStradamus: A simple Hidden Markov Model for nuclear localization signal prediction. BMC bioinformatics, 2009, 10(1): 202 |
Bailey TL, Boden M, Buske FA, et al. MEME SUITE: Tools for motif discovery and searching. Nucleic Acids Research, 2009, 37(S2): W202-W208 |
Brameier M, Krings A, MacCallum RM. NucPred—Predicting nuclear localization of proteins. Bioinformatics, 2007, 23(9): 1159-1160 DOI:10.1093/bioinformatics/btm066 |
Ciechanover A, Orian A, Schwartz AL. Ubiquitin-mediated proteolysis: Biological regulation via destruction. BioEssays, 2000, 22(5): 442-451 DOI:10.1002/(SICI)1521-1878(200005)22:5<442::AID-BIES6>3.0.CO;2-Q |
Claros MG, Vincens P. Computational method to predict mitochondrially imported proteins and their targeting sequences. European Journal of Biochemistry, 1996, 241(3): 779-786 |
Crooks GE, Hon G, Chandonia JM, et al. WebLogo: A sequence logo generator. Genome research, 2004, 14(6): 1188-1190 DOI:10.1101/gr.849004 |
Druzhinina IS, Shelest E, Kubicek CP. Novel traits of Trichoderma predicted through the analysis of its secretome. FEMS Microbiology Letters, 2012, 337(1): 1-9 DOI:10.1111/j.1574-6968.2012.02665.x |
Emanuelsson O, Nielsen H, Brunak S, et al. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence. Journal of Molecular Biology, 2000, 300(4): 1005-1016 DOI:10.1006/jmbi.2000.3903 |
Fankhauser N, Mäser P. Identification of GPI anchor attachment signals by a Kohonen self-organizing map. Bioinformatics, 2005, 21(9): 1846-1852 DOI:10.1093/bioinformatics/bti299 |
Frixione E, Ruiz L, Undeen AH. Monovalent cations induce microsporidian spore germination in vitro. Journal of Eukaryotic Microbiology, 1994, 41(5): 464-468 DOI:10.1111/j.1550-7408.1994.tb06043.x |
Gupta N, Hartmann A, Lucius R, et al. The obligate intracellular parasite Toxoplasma gondii secretes a soluble phosphatidylserine decarboxylase. Journal of Biological Chemistry, 2012, 287(27): 22938-22947 DOI:10.1074/jbc.M112.373639 |
Han YY, Ma KL. A genome-wide prediction and analysis of secreted proteins of Tupaia chinensis. Chinese Journal of Comparative Medicine, 2014, 24(4): 33-37 |
罕园园, 马开利. 树鼩全基因组分泌蛋白的预测分析. 中国比较医学杂志, 2014, 24(4): 33-37 |
Horton P, Park KJ, Obayashi T, et al. WoLF PSORT: Protein localization predictor. Nucleic acids research, 2007, 35(S2): W585-W587 |
Jaroenlak P, Boakye DW, Vanichviriyakit R, et al. Identification, characterization and heparin binding capacity of a spore- wall, virulence protein from the shrimp microsporidian, Enterocytozoon hepatopenaei (EHP). Parasites and Vectors, 2018, 11(1): 177 DOI:10.1186/s13071-018-2758-z |
Katinka MD, Duprat S, Cornillot E, et al. Genome sequence and gene compaction of the eukaryote parasite Encephalitozoon cuniculi. Nature, 2001, 414(6862): 450-453 DOI:10.1038/35106579 |
Krogh A, Larsson B, von Heijne G, et al. Predicting transmembrane protein topology with a hidden markov model: Application to complete genomes. Journal of Molecular Biology, 2001, 305(3): 567-580 DOI:10.1006/jmbi.2000.4315 |
Li T, Liu XL, Han B, et al. A genome-wide prediction and analysis on secreted proteins of Nosema bombycis. Science of Sericulture, 2013, 39(2): 295-301 |
李田, 刘显林, 韩冰, 等. 家蚕微孢子虫全基因组分泌蛋白的预测分析. 蚕业科学, 2013, 39(2): 295-301 |
Liu KR, Jin MF, Wu SL. Prediction of O-glycosylation site and catalytic characteristics of glycosylation enzyme. Chemistry of Life, 2006, 26(1): 25-27 |
刘可人, 金美芳, 吴士良. O-糖基化位点预测及糖基化酶催化特点. 生命的化学, 2006, 26(1): 25-27 |
Liu Z, Zhang QL, Wan XY, et al. Development of real-time PCR assay for detecting microsporidian Enterocytozoon hepatopenaei and the application in shrimp samples with different growth rates. Progress in Fishery Sciences, 2016, 37(2): 119-126 |
刘珍, 张庆利, 万晓媛, 等. 虾肝肠胞虫(Enterocytozoon hepatopenaei)实时荧光定量PCR检测方法的建立及对虾样品的检测. 渔业科学进展, 2016, 37(2): 119-126 |
McKerrow JH, Sun E, Rosenthal PJ, et al. The proteases and pathogenicity of parasitic protozoa. Annual Review of Microbiology, 1993, 47(1): 821-853 |
Peng Y, Clark C, Luong R, et al. The leucine zipper putative tumor suppressor 2 protein LZTS2 regulates kidney development. Journal of Biological Chemistry, 2011, 286(46): 40331-40342 |
Petersen TN, Brunak S, Heijn GV, et al. SignalP 4. 0: Discriminating signal peptides from transmembrane regions. Nature Methods, 2011, 8(10): 785-786 |
Qiao Y, Shen H, Wan XH, et al. Preliminary isolation and morphological observation of Enterocytozoon hepatopenaei infecting the shrimp Penaeus vannamei. Journal of Fishery Sciences of China, 2018, 25(5): 1051-1058 DOI:10.3724/SP.J.1118.2018.17430 |
乔毅, 沈辉, 万夕和, 等. 南美白对虾肝肠胞虫的分离及形态学观察. 中国水产科学, 2018, 25(5): 1051-1058 |
Song ZL, Dong X, Zhao RH, et al. Evaluation on the detection of Enterocytozoon hepatopenaei in pooled DNA samples of Litopenaeus vannamei based on TaqMan qPCR. Progress in Fishery Sciences, 2019, 40(3): 122-132 |
宋增磊, 董宣, 赵若恒, 等. 基于TaqMan qPCR检测凡纳滨对虾样品中虾肝肠胞虫并样检测方法的评价. 渔业科学进展, 2019, 40(3): 122-132 |
Tourtip S, Wongtripop S, Stentiford GD, et al. Enterocytozoon hepatopenaei sp. nov. (Microsporida: Enterocytozoonidae), a parasite of the black tiger shrimp Penaeus monodon (Decapoda: Penaeidae): Fine structure and phylogenetic relationships. Journal of Invertebrate Pathology, 2009, 102(1): 21-29 |
Wang XX, Yang G, Zhang DM, et al. LZTS2 tumor suppressor gene and advancements in tumor research. Chinese Journal of Clinical Oncology, 2014, 41(20): 1328-1332 |
王枭雄, 杨光, 张大明, 等. LZTS2抑癌基因与肿瘤相关性的研究进展. 中国肿瘤临床, 2014, 41(20): 1328-1332 |
Wang Y, Dang X, Ma Q, et al. Characterization of a novel spore wall protein NbSWP16 with proline-rich tandem repeats from Nosema bombycis (microsporidia). Parasitology, 2015, 142(4): 534-542 DOI:10.1017/S0031182014001565 |
Wu ZL, Tan XH, Pan GQ, et al. Extraction of spore wall proteins of Nosema bombycis with improved methods. Science of Sericulture, 2007, 33(1): 62-66 |
吴正理, 谭小辉, 潘国庆, 等. 家蚕微孢子虫(Nosema bombycis)孢壁蛋白提取方法的优化研究. 蚕业科学, 2007, 33(1): 62-66 |
Yang D, Pan L, Chen Z, et al. The roles of microsporidia spore wall proteins in the spore wall formation and polar tube anchorage to spore wall during development and infection processes. Experimental Parasitology, 2018, 187: 93-100 DOI:10.1016/j.exppara.2018.03.007 |
Zhang Y, Yu WW, Tao RY, et al. LZTS2: A novel tumor suppressor play an important role in tumor. Progress in Modern Biomedicine, 2016, 16(12): 2372-2375 |
张轶, 余稳稳, 陶瑞雨, 等. LZTS2抑癌基因在肿瘤中的作用机制. 现代生物医学进展, 2016, 16(12): 2372-2375 |
Zhuo L, Kimata K. Structure and function of inter-α-trypsin inhibitor heavy chains. Connective Tissue Research, 2008, 49(5): 311-320 DOI:10.1080/03008200802325458 |



