2. 上海海洋大学水产与生命学院 上海 201306
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306
虾血细胞虹彩病毒(Shrimp hemocyte iridescent virus, SHIV)和红螯螯虾虹彩病毒(Cherax quadricari- natus iridovirus, CQIV)是近年报道的、虹彩病毒科中可感染甲壳类的2种新发病毒(Xu et al, 2016; Qiu et al, 2017),对对虾养殖业构成了威胁。2014年,SHIV首次在浙江一养殖场的凡纳滨对虾(Litopenaeus vannamei)中被发现。随后几年的流行病学调查显示,我国沿海包括河北、山东、江苏、浙江和福建等省部分区域养殖凡纳滨对虾中均有SHIV检出(Qiu et al, 2017、2018)。Xu等(2016)研究显示,红螯螯虾(Cherax quadricarinatus)中分离到的一种虹彩病毒(CQIV)对凡纳滨对虾也具有较强致病性。
SHIV基因组为双链DNA,长度约为166 kb,与CQIV的基因组同源性为99.97%。2019年,国际病毒分类委员会将二者作为2个病毒株,统一命名为十足目虹彩病毒1(Decapod iridescent virus, DIV1),并将其归入虹彩病毒科的一个新属——十足目虹彩病毒属(Decapodiridovirus)(Xu et al, 2016; Qiu et al, 2017、2019; 陈形, 2019)。目前,虹彩病毒科共有6个属,其中,蛙病毒属(Ranavirus)、肿大细胞病毒属(Megalocytivirus)和淋巴囊肿病毒属(Lymphocystivirus)均可感染鱼类等脊椎动物(Chinchar et al, 2011; Kurita et al, 2012)。例如,大鲵虹彩病毒(Giant seaperch iridovirus, GSIV)感染会导致大鲵(Andrias davidanus)全身水肿、多脏器受损,该病毒的流行使大鲵养殖业遭受了重大打击(孟彦, 2013);而新加坡石斑鱼虹彩病毒(Singapore grouper iridovirus, SGIV)则会引起石斑鱼全身性疾病,威胁石斑鱼养殖业的发展(Teng et al, 2008; Li et al, 2014)。研究发现,DIV1可感染凡纳滨对虾、红螯螯虾、罗氏沼虾(Macrobrachium rosenbergii)、克氏原螯虾(Procambarus clarki)和脊尾白虾(Exopalaemon carinicauda)的造血组织和血细胞,被感染细胞出现核固缩,细胞质内可见嗜碱性包涵体(Qiu et al, 2017、2019)。感染DIV1后的对虾出现肝胰腺颜色变浅、空肠空胃、停止摄食、活力下降等症状(Qiu et al, 2017、2019)。
目前,DIV1的检测主要依赖Qiu等(2018)基于ATPase基因(GenBank登录号:KY681040)建立的TaqMan探针实时荧光定量PCR和套式PCR检测方法(陈蒙蒙, 2017; 邱亮, 2018)。由于DIV1实时荧光定量PCR方法对检测仪器设备要求较高,检测时间长,无法实现在取样现场对患病对虾进行快速检测。而环介导等温扩增(Loop-mediated isothermal amplification, LAMP)具有扩增效率高、扩增速度快、恒温扩增无需复杂仪器设备等特点,非常适用于现场检测(Priyanka et al, 2019; Jiang et al, 2012; Shirato, 2019),目前已被广泛应用于人类、动物卫生和食品相关病原微生物的检测(Notomi et al, 2015; Wong et al, 2018; Silva et al, 2020)。因此,本研究基于DIV1的主要衣壳蛋白基因建立了其LAMP检测技术,以期为DIV1病原的实验室和现场检测及防控提供新的技术支持。
1 材料与方法 1.1 实验材料参考Qiu等(2017)的方法,对购自山东日照的健康凡纳滨对虾进行DIV1人工感染实验,并使用基因组DNA提取试剂盒(天根, 北京, 中国)提取健康个体和患病个体的基因组DNA。利用NanoDrop 2000c测定所提取DNA的质量和浓度(Thermo Scientific, Waltham, 美国),然后,将DNA保存于–80℃冰箱备用。研究中用到的虾肝肠胞虫(EHP)、致急性肝胰腺坏死病副溶血弧菌(VpAHPND)、对虾偷死野田村病毒(CMNV)、传染性皮下及造血组织坏死病病毒(IHHNV)、白斑综合征病毒(WSSV)、桃拉综合征病毒(TSV)和黄头病毒(YHV)等病原阳性核酸均来源于作者实验室。
1.2 DIV1-LAMP引物设计本研究利用Primer Explorer V5.0软件,根据DIV1的衣壳蛋白基因序列(GenBank登录号为ATE87157.1),设计了3对LAMP扩增引物(表 1),包括2条内引物、2条外引物以及2条环引物。由上海生工生物技术有限公司合成该引物。
|
|
表 1 DIV1-LAMP反应引物序列 Tab.1 Primer sequences for DIV1-LAMP assay |
以DIV1阳性核酸为模板,25 μl LAMP初始反应体系中包含2.5 μl 10×Isothermal amplification buffer、4 mmol/L MgSO4、1.2 mol/L Betaine、1.2 mmol/L dNTPs、1.6 μmol/L DIV1-FIP/BIP、0.2 μmol/L DIV1- F3/B3、0.8 μmol/L LF/LB、6.4 U Bst 2.0 WarmStart® DNA聚合酶、0.8 μmol/L EvaGreen®及适量的水,于实时荧光定量PCR仪(Bio-Rad, California, 美国)中设置8个温度梯度(58.0℃~68.0℃),反应50 min (50个循环,每个循环1 min);MgSO4终浓度按照3.2、4.0、4.8、5.6 mmol/L,dNTPs终浓度按照0.8、1.0、1.2、1.4 mmol/L进行反应体系正交实验优化;Bst 2.0 WarmStart® DNA聚合酶用量按照3.2、4.8、6.4、8.0、9.6 U进行添加;核酸染料EvaGreen®终浓度分别按照0.4、0.8、1.2、1.6、2.0 μmol/L的添加量进行优化。每个待优化指标的单个变量设置3个重复,依据扩增Ct值和SD值选出最佳反应条件。
1.4 DIV1的质粒标准品制备以DIV1阳性核酸为模板,通过DIV1-F3/B3引物进行PCR扩增,获取DIV1靶基因核酸目的片段。将纯化的目的片段接入pMD18-T载体(TaKaRa, 大连, 中国)进行克隆和测序验证,对测序验证正确的克隆扩大培养。随后,用质粒提取试剂盒(Omega Inc., Norcross, 美国)提取pMD18-DIV1质粒,利用NanoDrop 2000c测定质粒浓度。用TE缓冲液将pMD18-DIV1质粒10倍梯度稀释成质粒标准品,置于–80℃保存备用。
1.5 DIV1-LAMP分析灵敏度检验和标准曲线的建立使用梯度稀释的pMD18T-DIV1质粒(3.54×109~ 100拷贝/反应)和无RNA酶H2O作为模板,获得DIV1的LAMP扩增曲线以确定该方法的检测灵敏度,使用BioRad CFX96软件(版本6.0.14),以扩增的Ct值(y)相对于pMD18-DIV1质粒浓度的对数值(x)生成标准曲线。每个浓度设置3个重复。
1.6 DIV1-LAMP分析特异性检验分别以EHP、VpAHPND、CMNV、IHHNV、WSSV、TSV和YHV等病原的阳性DNA或cDNA为模板,检验本研究所建立的LAMP方法的特异性。反应以3.54×105拷贝/反应pMD18-DIV1质粒为阳性对照,以未感染DIV1的对虾组织DNA为阴性对照,以无RNA酶的水为空白对照。
1.7 DIV1-LAMP方法可重复性和可重现性检验按照世界动物卫生组织(OIE)《水生动物疫病诊断手册》中检测方法确认章节的要求,对新建立的DIV1-LAMP检测方法进行可重复性和可重现性分析。LAMP检测方法组内可重复性测试,采用10倍浓度梯度稀释的质粒为模板,对每个浓度的模板设置3次平行测试,利用平行测试产生的Ct平均值和标准偏差(SD)来计算组内变异系数CV。可重现性则以不同实验室环境条件下LAMP检测方法对质粒标准品扩增曲线的稳定性来评定,并利用SPSS软件单因素方差分析(One-way ANOVA)对扩增Ct值进行差异显著性分析。
1.8 DIV1-LAMP方法对实际样品的检验利用本研究建立的LAMP方法对人工感染DIV1的凡纳滨对虾样品进行定性分析,同时,利用TaqMan荧光定量PCR对上述样品进行定性和定量分析,并对比DIV1-LAMP检测方法和TaqMan荧光定量PCR的分析结果。
1.9 基于FTA卡和LAMP的DIV1现场快速检测方法利用1.2 μl GeneFinder®替换DIV1-LAMP反应体系中的EvaGreen®,并将其通过粘附剂预先封闭于200 μl PCR小管管盖内,PCR管内添加30 μl DIV1 LAMP反应预混液(不含模板和荧光染料EvaGreen®);按照Whatman FTA卡说明书,利用其提取4尾人工感染DIV1的凡纳滨对虾组织核酸及2尾健康对虾组织核酸,将核酸分别加入上述含有30 μl DIV1 LAMP反应预混液的PCR小管内,同时,设置阳性对照和阴性对照;在64.4℃条件下,保温50 min,然后,通过在90℃条件下保温3 min使预置在管盖内的GeneFinder®染料脱落进入LAMP反应预混液,完成扩增产物染色。PCR小管内有扩增产物生成,则显示为荧光绿色,表明样品为DIV1阳性;PCR小管内无扩增产物生成,则显示为橙黄色,表明样品为DIV1阴性。
2 结果 2.1 DIV1的LAMP反应体系条件的优化LAMP优化结果显示,扩增反应温度在64.4℃时,Ct值(15.44)和重复间Ct值标准偏差(SD值)(0.09)最小(图 1a)。在64.4℃扩增温度下,综合考虑扩增Ct均值及重复测试Ct值标准偏差最小、成本经济和方法稳定等因素,确立优化后的扩增反应预混体系中dNTPs和MgSO4的浓度分别为1.2和4.0 mmol/L (图 1b),Bst 2.0 WarmStart® DNA聚合酶的用量为6.4 U(图 1c),EvaGreen®的用量为0.8 μmol/L (图 1d)。
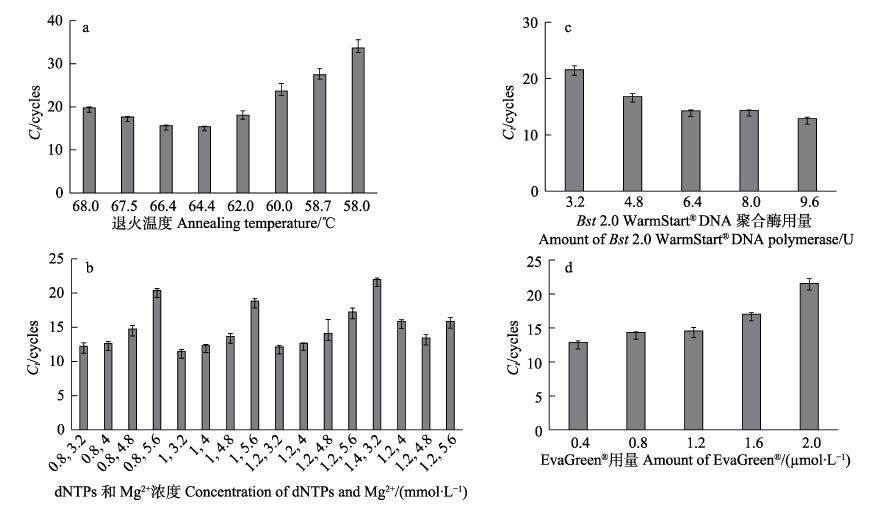
|
图 1 十足目虹彩病毒(DIV1) LAMP反应体系的优化 Fig.1 Optimization of DIV1-LAMP reaction |
使用优化后的LAMP反应体系对10倍梯度稀释的pMD18-DIV1质粒标准品进行检测,结果显示,质粒标准品在3.54×109~3.54×102拷贝/反应范围内均有较强的荧光信号(图 2a),说明本研究建立的DIV1的LAMP检测方法对质粒模板的检测下限可低至3.54× 102拷贝/反应。在3.54×109~3.54×103拷贝/反应的起始模板浓度范围内,pMD18-DIV1起始模板浓度对数值与扩增反应Ct值之间具有良好的线性关系(图 2b)。其关系曲线为Ct=–2.918 Lg(Sq)+39.331,R2=0.996。
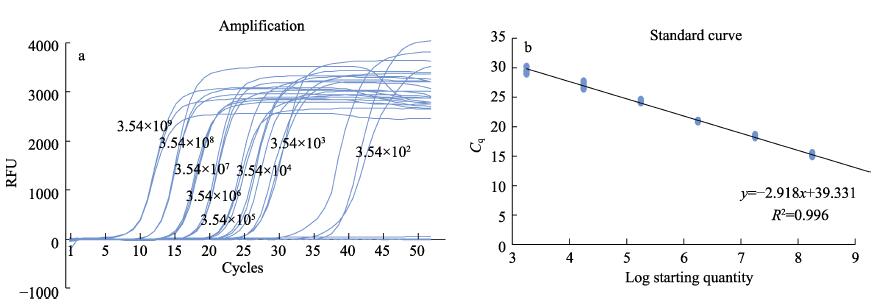
|
图 2 十足目虹彩病毒(DIV1) LAMP方法以pMD18-DIV1质粒标准品为模板产生的扩增曲线(a)和标准曲线(b) Fig.2 Amplification curve (a) and standard curve (b) of DIV1-LAMP detection method by using pMD18-DIV1 plasmid standard as templates a:以梯度稀释的pMD18-DIV1质粒标准品为模板的扩增曲线,pMD18-DIV1质粒的拷贝数:3.54×109, 3.54×108, 3.54×107, 3.54×106, 3.54×105, 3.54×104, 3.54×103, 3.54×102拷贝(从左到右);b:反应起始模板(pMD18-DIV1)质粒浓度对数值和Ct值之间的标准曲线方程 a: LAMP amplification curve of serially diluted 10-fold pMD18-DIV1 plasmid. Copies of pMD18-DIV1 plasmids: 3.54×109, 3.54×108, 3.54×107, 3.54×106, 3.54×105, 3.54×104, 3.54×103, 3.54×102 (from left to right); b: Standard curve equation between log concentration of pMD18-DIV1 plasmid and Ct value. |
以DIV1、EHP、VpAHPND、CMNV、IHHNV、WSSV、TSV和YHV等主要虾类病原DNA或cDNA为模板进行DIV1-LAMP检测的特异性分析,结果显示,只有当检测样品含有DIV1阳性核酸时,扩增反应才产生“S”形扩增曲线,其他病原的核酸均无阳性扩增曲线(图 3)。该结果说明,本研究建立的DIV1- LAMP方法与上述其他常见的对虾病原无交叉反应,可特异性检测DIV1。
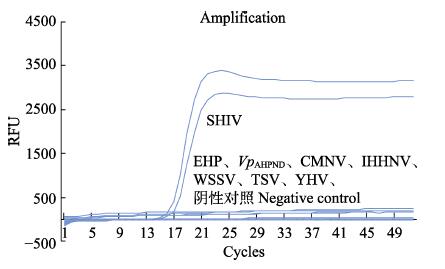
|
图 3 十足目虹彩病毒(DIV1) LAMP方法的分析特异性 Fig.3 Analytical specificity of shrimp hemocyte iridescent virus (DIV1) LAMP assay |
以3.54×109~3.54×103拷贝/反应的pMD18-DIV1质粒标准品为模板,对DIV1-LAMP检测方法的可重复性进行评估。结果显示,在起始模板浓度为3.54×109~3.54×103拷贝/反应的范围内,DIV1-LAMP检测方法组内和组间检测的变异系数CV分别小于1.89%和1.63% (表 2),说明在此起始模板浓度范围内该方法稳定性较好。
|
|
表 2 十足目虹彩病毒(DIV1) LAMP检测组内和组间重复性 Tab.2 Intra-assay and inter-assay variability of DIV1-LAMP assay |
以3.54×109~3.54×103拷贝/反应的质粒标准品为模板,在3个不同实验室环境下进行DIV1可重现性分析,结果显示,在上述模板浓度范围内DIV1-LAMP反应的组间Ct值的P值均大于0.05 (表 3),说明不同环境下,该方法扩增标准曲线与即时检测的标准品曲线差异不显著。说明本研究建立的DIV1-LAMP方法在不同实验室环境具有良好的可重现性。
|
|
表 3 DIV1-LAMP分析法的组间变异性 Tab.3 Analysis of variance of inter-assay variability of DIV1-LAMP assay |
本研究对人工感染DIV1 3 d后的40尾凡纳滨对虾样品(编号20191114001~20191114040)分别进行了LAMP检测和TaqMan荧光定量PCR检测,结果显示,2种方法对病毒载量超过102 copies/μl的样品定性结果基本一致;而对病毒载量低于102 copies/μl的部分样品,LAMP方法检测结果与TaqMan荧光定量PCR检测结果不一致(表 4),说明本方法不适用于分析DIV1载量低于102 copies/μl的样品。
|
|
表 4 利用DIV1-LAMP和TaqMan荧光定量PCR方法对人工感染DIV1凡纳滨对虾的检测结果 Tab.4 Detection results of DIV1-LAMP and TaqMan qPCR for L. vannamei samples artificially infected with DIV1 |
取4尾人工感染DIV1的凡纳滨对虾及2尾健康凡纳滨对虾,利用Whatman FTA卡现场制备核酸,将核酸加入内置GeneFinder®、含有DIV1-LAMP扩增反应预混体系的PCR小管内进行等温扩增反应,50 min后进行显色,结果显示,阳性对照和4尾人工感染DIV1的凡纳滨对虾样品对应的PCR管内呈荧光绿色,为DIV1阳性,而阴性对照及2尾健康凡纳滨对虾样品对应的PCR管内呈橙红色,为DIV1阴性(图 4)。该结果说明,利用Whatman FTA卡进行核酸现场制备,结合核酸染料GeneFinder®预制于扩增反应体系小管内的方法,可以实现对感染DIV1对虾的现场快速检测。

|
图 4 利用FTA卡和DIV1-LAMP方法现场检测凡纳滨对虾样品中DIV1的显色图 Fig.4 Results of on-site detection of DIV1 in L. vannamei by using FTA card and DIV1-LAMP assay P:阳性对照;N:阴性对照;S1~S6:凡纳滨对虾样品 P: Positive control; N: Negative control; S1~S6: Samples of L. vannamei |
本研究基于DIV1主要衣壳蛋白基因设计DIV1的LAMP扩增引物,在优化LAMP反应温度、各试剂组分浓度和用量的基础上建立了DIV1-LAMP实时荧光检测技术。本着成本经济和体系稳定的原则,以求得扩增反应最小Ct值和SD值为目标,确定优化后的DIV1-LAMP扩增温度和反应体系,反应温度在64.4℃时最佳;优化后的反应体系中的MgSO4和dNTPs浓度分别为4.0和1.2 mmol/L,Bst 2.0 WarmStart® DNA聚合酶和EvaGreen®的用量分别为6.4 U和0.8 μmol/L。该技术通过Bst 2.0 WarmStart® DNA聚合酶进行等温扩增,利用EvaGreen®染料指示反应过程,能够实现对DIV1靶基因的实时荧光快速检测。
以10倍浓度梯度稀释的pMD18-DIV1质粒为模板扩增并构建的标准扩增曲线结果显示,在3.54×109~3.54×103拷贝/反应的范围内,起始模板浓度对数值与扩增Ct值间呈良好的线性关系(R2=0.996),利用扩增Ct值可准确评估起始模板拷贝数,这表明在该浓度范围内,本研究所建立的LAMP方法适合作为DIV1定量检测的有效工具。前期多项有关LAMP实时荧光定量方法的研究显示,由于LAMP反应本身的特性,当起始模板浓度 < 1000拷贝时,起始模板浓度对数值与扩增Ct值的相关系数明显降低(Mori et al, 2004; Suzuki et al, 2011; Wei et al, 2013; Zhang et al, 2017)。本研究中,DIV1-LAMP在低拷贝起始模板条件下的定量分析结果与此前的相关报道一致,这也说明DIV1-LAMP方法不适于对低拷贝(< 1000)模板进行准确定量。Qiu等(2018)基于DIV1基因组中ATPase基因(ORF114R)所设计的引物和探针,建立了DIV1的TaqMan探针荧光定量PCR方法,该方法于实验室条件下可在2.5 h内完成检测,检测限低至4拷贝/反应,可用于DIV1感染早期的检测预警,适用于对对虾苗种、出入境对虾样品等进行DIV1病原的筛查。与已报道的DIV1 TaqMan荧光定量PCR检测方法相比,本研究建立的DIV1-LAMP检测方法在实验室条件下可在50 min完成反应,检测时间大大缩短,且成本低廉,可用于发病对虾(一般携带较高拷贝病原)样品的快速检测和辅助诊断,适用于水产技术推广部门、研究单位进行DIV1流行病学调查的大规模定性或定量检测。
作者实验室此前比较了基于DIV1 ATPase基因和衣壳蛋白基因开发的2种TaqMan探针荧光定量PCR检测方法,发现上述2个目标基因对检测方法灵敏度和特异性的影响很小。本研究对人工感染DIV1的凡纳滨对虾样品分别进行基于ATPase基因的TaqMan荧光定量PCR方法和基于衣壳蛋白基因的LAMP方法检测,结果显示,2种方法对病毒载量低于102 copies/μl的部分样品的检测结果不一致,推测其原因可能是由于本研究中所建立的DIV1-LAMP方法不适用于分析病毒载量低于102 copies/μl的样品所致。
目前,LAMP技术在病原检测领域的应用已十分广泛,该技术也被用于多种水生动物相关致病细菌、病毒和真菌的检测中。如Arunrut等(2016)报道一种以胶体金为探针的LAMP-AuNP方法,可快速检测VpAHPND的PirA基因,检出限为100 CFU,具有很高的实用价值。Zhang等(2017)和李小平(2018、2019)分别利用RT-LAMP技术研发出对虾偷死野田村病毒和行动障碍野田村病毒(Movement disorder nodavirus, MDNV)的定量检测方法,并通过将核酸染料添加到反应体系中的方法,使LAMP检测结果可视化。Kumar等(2018)利用LAMP方法与Whatman FTA卡相结合的方法,用于检测引起对虾生长缓慢的虾肝肠胞虫(Enterocytozoon hepatopenaei, EHP)的SSU rRNA基因,可实现对EHP的现场快速检测。基于对虾养殖者、养殖企业、水产技术推广部门和研究单位等开展DIV1现场快速检测的需求,本研究在建立DIV1-LAMP荧光定量方法的基础上,通过结合Whatman FTA卡现场制备对虾组织核酸和显色染料预置的方法,实现了对DIV1的现场快速检测。整个检测无需PCR仪等复杂或昂贵的仪器设备,只需温度计和保温杯提供恒温水浴即可完成检测,检测过程不超过1 h,单个检测反应成本相当于TaqMan荧光定量PCR检测方法的1/10 ~ 1/5。
综上所述,本研究建立并优化了一种检测DIV1的LAMP实时荧光定量检测方法,其特异性强,速度快,成本低,适用于在实验室内对患病对虾进行DIV1的快速检测或辅助诊断。同时,本研究还将Whatman FTA卡核酸现场制备方法与DIV1-LAMP方法相结合,进一步开发了DIV1的现场快速高效检测技术,适用于开展养殖对虾的DIV1现场快速检测。本研究为DIV1这一新发虾类病原提供了可靠的检测方法,对实现低成本、大规模、快速DIV1检疫,以及控制该病毒性疫病的传播和流行具有重要意义。
致谢: 中国水产科学研究院黄海水产研究所养殖生物疾病控制与分子病理学研究室研究生协助DIV1感染对虾取样和核酸提取,谨致谢忱。
Arunrut N, Kampeera J, Sirithammajak S, et al. Sensitive visual detection of AHPND bacteria using loop-mediated isothermal amplification combined with DNA-functionalized gold nanoparticles as probes. PLoS One, 2016, 11(3): e0151769 DOI:10.1371/journal.pone.0151769 | |
Chen MM. Application of real-time PCR in detection of pathogens VPAHPND and SHIV of Litopenaeus vannamei. Masterxs Thesis of Shanghai Ocean University, 2017
| |
陈蒙蒙.实时荧光定量PCR在凡纳滨对虾病原VpAHPND及SHIV检测中的应用.上海海洋大学硕士研究生学位论文, 2017
| |
Chen X. Susceptible host survey of Decapod iridescent virus 1(DIV1). Masterxs Thesis of Shanghai Ocean University, 2019
| |
陈形.十足目虹彩病毒1(DIV1)的易感宿主调查.上海海洋大学硕士研究生学位论文, 2019
| |
Chinchar VG, Yu KH, Jancovich JK. The molecular biology of frog virus 3 and other iridoviruses infecting cold-blooded vertebrates. Viruses, 2011, 3(10): 1959-1985 DOI:10.3390/v3101959 | |
Jiang T, Liu J, Deng YQ, et al. Development of RT-LAMP and real-time RT-PCR assays for the rapid detection of the new duck Tembusu-like BYD virus. Archives of Virology, 2012, 157(12): 2273-2280 DOI:10.1007/s00705-012-1431-7 | |
Kumar TS, Krishnan AN, Rajan JJS, et al. Visual loop-mediated isothermal amplification (LAMP) for the rapid diagnosis of Enterocytozoon hepatopenaei (EHP) infection. Parasitology Research, 2018, 117(5): 1485-1493 DOI:10.1007/s00436-018-5828-4 | |
Kurita J, Nakajima K. Megalocytiviruses. Viruses, 2012, 4(4): 521-538 DOI:10.3390/v4040521 | |
Li P, Yan Y, Wei S, et al. Isolation and characterization of a new class of DNA aptamers specific binding to Singapore grouper iridovirus (SGIV) with antiviral activities. Virus Research, 2014, 188: 146-154 DOI:10.1016/j.virusres.2014.04.010 | |
Li XP. Establishment and application of nucleic acid detection techniques for two emerging shrimp viruses. Masterxs Thesis of Shanghai Ocean University, 2018 | |
李小平.两种对虾新发病毒核酸检测技术的建立及应用.上海海洋大学硕士研究生学位论文, 2018
| |
Li XP, Wan XY, Zhang QL, et al. Molecular epidemiological survey of covert mortality nodavirus (CMNV) in cultured crustaceans in China in 2016~2017. Progress in Fishery Sciences, 2019, 40(2): 65-73 | |
李小平, 万晓媛, 张庆利, 等. 2016~2017年中国沿海省市虾类偷死野田村病毒(CMNV)分子流行病学调查. 渔业科学进展, 2019, 40(2): 65-73 | |
Meng Y. Isolation, identification, genome sequencing of a novel Ranavirus isolate in Chinese giant salamander (Andrias davidianus) and genetic diversity of Chinese giant salamander. Doctoral Dissertation of Huazhong Agricultural University, 2013
| |
孟彦.大鲵虹彩病毒病病原的分离、鉴定、基因组测序及大鲵群体遗传多样性分析.华中农业大学博士研究生学位论文, 2013
| |
Mori Y, Kitao M, Tomita N, et al. Real-time turbidimetry of LAMP reaction for quantifying template DNA. Journal of Biochemical and Biophysical Methods, 2004, 59(2): 145-457 DOI:10.1016/j.jbbm.2003.12.005 | |
Notomi T, Mori Y, Tomita N, et al. Loop-mediated isothermal amplification (LAMP): Principle, features, and future prospects. Journal of Microbiology, 2015, 53(1): 1-5 | |
Priyanka N, Siddhant J, Prasad S. An integrated one-step assay combining thermal lysis and loop-mediated isothermal DNA amplification (LAMP) in 30 min from E. coli and M. smegmatis cells on a paper substrate.. Sensors and Actuators B: Chemical, 2019, 291(15): 74-80 | |
Qiu L, Chen MM, Wan XY, et al. Characterization of a new member of Iridoviridae, shrimp hemocyte iridescent virus (SHIV), found in white leg shrimp (Litopenaeus vannamei). Scientific Reports, 2017, 7(1): 11834 DOI:10.1038/s41598-017-10738-8 | |
Qiu L, Chen MM, Wan XY, et al. Detection and quantification of shrimp hemocyte iridescent virus by TaqMan probe based real-time PCR. Journal of Invertebrate Pathology, 2018, 154: 95-101 DOI:10.1016/j.jip.2018.04.005 | |
Qiu L, Chen X, Zhao R, et al. Description of a natural infection with Decapod iridescent virus 1 in farmed giant freshwater prawn, Macrobrachium rosenbergii. Viruses, 2019, 11(4): 354 DOI:10.3390/v11040354 | |
Qiu L. Viral metagenomics analysis of farmed shrimp and molecular epidemiological study of shrimp hemocyte iridescent virus (SHIV). Doctoral Dissertation of Shanghai Ocean University, 2018
| |
邱亮.养殖对虾的病毒宏基因组分析及虾血细胞虹彩病毒(Shrimp hemocyte iridescent virus, SHIV)的分子流行病学研究.上海海洋大学博士研究生学位论文, 2018
| |
Shirato K. Detecting amplicons of loop-mediated isothermal amplification (LAMP). Microbiology and Immunology, 2019, 63(10): 407-412 DOI:10.1111/1348-0421.12734 | |
Silva SJRD, Pardee K, Pena L. Loop-mediated isothermal amplification (LAMP) for the diagnosis of Zika virus: A review. Viruses, 2020, 12(1): 193-203 | |
Suzuki Y, Narimatsu S, Furukawa T, et al. Comparison of real-time reverse transcription loop-mediated isothermal amplification and real-time reverse transcription polymerase chain reaction for detection of noroviruses in municipal wastewater. Journal of Bioscience and Bioengineering, 2011, 112(4): 369-372 DOI:10.1016/j.jbiosc.2011.06.012 | |
Teng Y, Hou Z, Gong J, et al. Whole-genome transcriptional profiles of a novel marine fish iridovirus, Singapore grouper iridovirus (SGIV) in virus-infected grouper spleen cell cultures and in orange-spotted grouper, Epinephulus coioides. Virology, 2008, 377(1): 39-48 | |
Wei H, Zeng J, Deng C, et al. A novel method of real-time reverse transcription loop-mediated isothermal amplification developed for rapid and quantitative detection of human astrovirus. Journal of Virological Methods, 2013, 188(1–2): 126-131 | |
Wong YP, Othman S, Lau YL, et al. Loop-mediated isothermal amplification (LAMP): A versatile technique for detection of micro-organisms. Journal of Applied Microbiology, 2018, 124(3): 626-643 DOI:10.1111/jam.13647 | |
Xu L, Wang T, Li F, et al. Isolation and preliminary characterization of a new pathogenic iridovirus from redclaw crayfish Cherax quadricarinatus. Diseases of Aquatic Organisms, 2016, 120(1): 17-26 DOI:10.3354/dao03007 | |
Zhang QL, Liu S, Yang HL, et al. Reverse transcription loop-mediated isothermal amplification for rapid and quantitative assay of covert mortality nodavirus in shrimp. Journal of Invertebrate Pathology, 2017, 150: 130-135 DOI:10.1016/j.jip.2015.09.001 |




