2. 青岛瑞滋集团有限公司 青岛 266400
2. Qingdao Ruizi Company, Qingdao 266400
刺参又名仿刺参(Apostichopus japonicus),广泛分布于太平洋西部的中国、俄罗斯、韩国与日本沿海,具有很高的药用和营养保健价值,是我国海水养殖中重要的经济品种(廖玉麟, 1997; FAO, 2012)。近年来,由于过度捕捞和环境污染的加剧,野生刺参自然资源趋于枯竭,刺参已被世界自然保护联盟收录到濒危物种红色名录的濒危(EN)等级(Purcell et al, 2013)。随着刺参养殖产业和海洋牧场的快速发展,不同地区人工繁育的苗种在各养殖主产区和养殖海域跨地域使用,在自然条件下与原种群相互交配也会造成种质渗透,因此,对刺参原地理种群的种质鉴别日益迫切。
近年来,同工酶(张涛等, 2017)、微卫星DNA标记(苗贵东等, 2011)、线粒体DNA (Sun et al, 2010; 郝君等, 2013)等标记技术在水产种质研究领域被广泛应用。其中,微卫星DNA标记又称SSR标记,因具有多态性高、遵循孟德尔遗传规律、可重复性强、共显性、容易获得等特点,被广泛应用于遗传多样性研究中。另外,还可充分利用其高多态性的特点构建指纹图谱,用于品种之间乃至个体之间基因组序列差异解析,用于亲缘关系很近的品种或个体的准确辨别(McConnell et al, 1995; Scribner et al, 1996; Dewoody et al, 2000)。本研究利用SSR标记技术对采自中国、韩国和俄罗斯的8个不同地理种群刺参进行遗传结构分析及指纹图谱构建,以期为刺参种质资源现状评估和保护提供基础数据,同时,为后续种质资源鉴定、开发利用和良种选育提供依据。
1 材料与方法 1.1 实验材料本研究所用野生刺参分别采集于中国青岛、烟台,俄罗斯符拉迪沃斯托克,韩国浦项、木浦和群山,共6个海域,其中,韩国刺参的获取是在中国水产科学研究院基本科研业务费“中韩渔业联合研究与合作交流”项目支持下,与韩国海参产业协会合作,完成不同地理群体刺参采集,并由韩方完成DNA提取;俄罗斯符拉迪沃斯托克地区刺参群体是通过海关相关手续引入。根据所采集刺参体色的差异,韩国浦项采集的刺参又分为红参、黄参和黑参,合计采集6个海域8个不同地理群体刺参225头。不同群体的采样地点及信息见图 1和表 1。

|
图 1 刺参采样地点 Fig.1 Sampling sites for A. japonicus populations |
|
|
表 1 刺参采样信息 Tab.1 Sampling information for A. japonicus populations |
利用OMEGA Mollusc DNA Kit提取刺参基因组DNA,利用NanoDrop 1000及1%琼脂糖凝胶电泳检测DNA纯度及完整性后,稀释到50 ng/μl,置于-20℃冰箱保存备用。利用刺参13个微卫星位点的引物进行PCR扩增,对PCR产物直接进行位点扫描。13对引物序列见表 2,引物由本实验室设计并验证,由青岛擎科生物公司合成。PCR反应总体积为20 μl,包含2×TSINGKE Master Mix 10 μl、10 μmol/L正反向引物各1 μl、模板DNA 1 μl,加ddH2O补足至20 μl。PCR反应扩增程序:94℃预变性5 min、94℃ 30 s、56℃ 30 s、72℃ 30 s,经30个循环后,72℃延伸15 min,4℃保存。PCR产物经1.5%的琼脂糖凝胶电泳检测后,送青岛擎科生物公司利用ABI3130进行扩增片段分型和等位基因分析。
|
|
表 2 引物序列信息 Tab.2 Information of primer sequences |
利用Popgene软件统计整个群体及各群体的等位基因数(A)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Nei's标准遗传距离(Ds)、群体间基因流(Nm),χ2检验估计群体Hardy-Weinberg平衡偏离(Yeh et al, 1997)。利用Cervus软件计算各位点多态信息含量(PIC)(Jo et al, 2017),利用Arlequin 3.5计算群体间遗传分化系数(Fst)(Excoffie et al, 2010)。采用Excel绘制刺参8个地理群体的指纹模式图,并根据位点特异性,构建数字化指纹图谱。使用Structure 2.3中贝叶斯聚类法对所有个体进行聚类分析,K值设为10,首先根据个体基因型计算各可能自由交配群数K的概率L(K),再基于相邻K概率计算第一级概率变化L′(K)=L(K)-L(K-l)和第二级概率变化L′′(K)=L′(K+l)- L′(K),获得L′′(K)绝对值,最后用L′′(K)绝对值除以L(K)标准差得到ΔK(Evanno et al, 2005)。
2 结果与分析 2.1 不同地理种群刺参的遗传多样性分析利用13对引物对采集的8个地理种群进行遗传多样性分析,结果见表 3。13对引物全部扩增稳定并具有多态性,13个位点在8个群体中共检测出252个等位基因,单个位点的观测等位基因数(A)为10(AJ06)~34(AJ07),平均等位基因数为19.4。各位点的有效等位基因共83.8个,各位点的有效等位基因数的范围为1.7(AJ06)~11.8(AJ09),平均有效等位基因数为6.5。多个位点上的等位基因分布不均匀,表现为低频等位基因较多,等位基因数和有效等位基因数的差别较大。样本总的观测杂合度和期望杂合度分别为0.20(AJ05)~0.83(AJ09)和0.42(AJ06)~0.92 (AJ09),平均值分别为0.47和0.80。13个位点的多态信息含量为0.465(AJ06)~0.909(AJ09),除AJ06为中度多态性位点(0.25 < PIC < 0.5)外,其余12个位点均为高度多态性位点(PIC>0.5)。
|
|
表 3 各刺参群体在13个SSR位点的多样性指数 Tab.3 The genetic diversity indices of 13 SSR markers for different populations of A. japonicus |
不同地理群体分析结果显示,各群体平均观测等位基因数为5.5(RU)~10.2(SK-QS-B),平均有效等位基因数为3.8(RU)~4.9(SK-QS-B)。不同地理群体的观测杂合度为0.40(RU和SK-PX-R)~0.54(SK-PX-B),期望杂合度为0.68(SK-PX-R和SK-MP-B)~0.76(QD),8个群体的多态信息含量(PIC)为0.6392(SK-MP-B)~ 0.7122(QD),各群体均显示为高度多态性(PIC>0.5),说明这几个群体均具有较高的遗传多样性。对8个群体共104个位点进行Hardy-Weinberg平衡检测,结果显示,有73个位点显著偏离Hardy-Weinberg平衡(P < 0.05),8个群体中均有大量位点表现为杂合子缺失状态(Fis>0)。
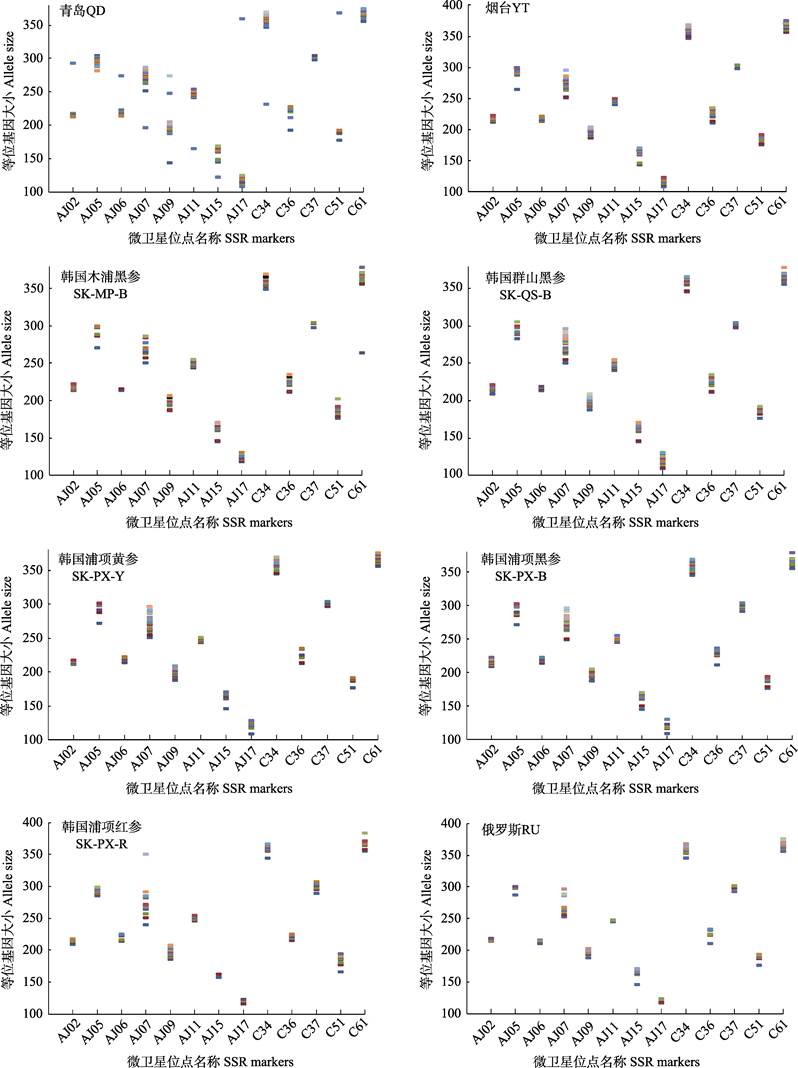
2.2 不同地理种群刺参的指纹图谱构建利用13个微卫星位点在各群体中的扩增结果绘制相应群体的DNA指纹模式图。从图 2可以看出,13个微卫星位点均具有较高的特异性,每一位点都具有特异性等位基因,相应特异等位基因可用于不同地理群体刺参的鉴别。将这些位点所含有的228个特异性等位基因进行计算机数字化处理,以“1”和“0”分别表示相应等位基因“有”和“无”,将这13个微卫星标记产生的特异性多态位点依次排列构建成数字化指纹图谱(表 4),可为不同地理种群刺参的种质鉴定提供参照。
|
图 2 8个群体刺参的微卫星DNA指纹模式 Fig.2 DNA fingerprints of eight populations of A. japonicus |
|
|
表 4 8个地理群体刺参的数字化DNA指纹 Tab.4 The numbered SSR DNA fingerprint of eight populations of A. japonicus |
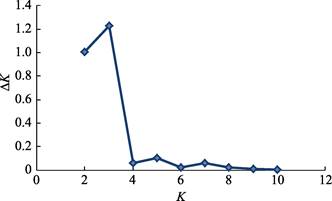
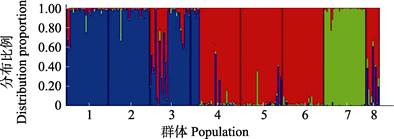
利用Structure软件进行群体结构分析的集群K与统计量ΔK的关系中,ΔK峰值出现在K=3(图 3),表明8个刺参群体的预测自由交配组数为3,因此,后续分群选择K=3。基于分子标记的个体分配模式显示(表 5),中国青岛群体(95.1%)、烟台群体(96.9%)样本中90%以上个体被分配于3号自由交配群;韩国的5个群体在1、2、3号自由交配群中皆有分布,其中,韩国木浦群体主要分布于3号自由群(67.2%),其次分布于1号自由群(31.7%);韩国群山群体则主要分布于1号自由群(76.6%),其次分布于3号自由群(22.1%);韩国浦项黄参群体(94.6%)和韩国浦项黑参群体(97.8%)几乎全部分布于1号自由群;韩国浦项红参群体(97.2%)几乎全部分布于2号自由群;俄罗斯群体在3个自由群中皆有分布,在1号自由群中含78.2%,2号自由群有4.8%,3号自由群有16.9%。当K=3时,贝叶斯分析法的聚类结果见图 4。从图 4可以看出,3个不同颜色分别代表不同的自由群,红色代表 1号自由群,绿色为2号自由群,蓝色为3号自由群。图 4中不同颜色分布与表 5中的分配比例相一致。自由分配模式表明,不同地理群体刺参遗传结构与其所在地理位置具有一定的相关性,但又受其他因素影响,导致这种分布模式与地理位置不完全一致。
|
图 3 集群K与统计量ΔK的关系 Fig.3 Scatter plot of possible number of cluster |
|
|
表 5 分配模式检验得到的8个地理群体刺参在3个自由交配群中的分布比例 Tab.5 Distribution proportion of eight geographical populations of A. japonicus in three inferred clusters tested by distribution pattern |
|
图 4 K=3时贝叶斯分析法的聚类结果 Fig.4 Histogram of the Bayesian analysis with K=3 1:青岛;2:烟台;3:韩国木浦黑参;4:韩国群山黑参;5:韩国浦项黄参;6:韩国浦项黑参;7:韩国浦项红参;8:俄罗斯 1: QD; 2: YT; 3: SK-MP-B; 4: SK-QS-B; 5: SK-PX-Y; 6: SK-PX-B; 7: SK-PX-R; 8: RU |
不同群体之间的遗传分化系数及基因流的统计结果见表 6。各群体之间存在不同程度的遗传分化,青岛群体与烟台群体、青岛群体与韩国木浦黑参群体、韩国木浦黑参群体与韩国群山黑参群体、韩国群山黑参群体与韩国浦项黄参及黑参群体、韩国浦项黄参与黑参群体的群体间遗传分化系数较小(0.05> Fst>0),说明以上群体间存在轻度遗传分化;韩国浦项红参群体与其他群体间的遗传分化系数较大(Fst> 0.15),说明韩国浦项红参群体与7个刺参群体间存在高度遗传分化;青岛群体与韩国群山黑参群体、韩国浦项黄参和黑参群体、俄罗斯群体,烟台群体与韩国群山群体等存在中度遗传分化(0.15>Fst>0.05)。
|
|
表 6 不同群体间遗传分化系数值及基因流 Tab.6 Genetic differentiation and gene flow between different populations |
不同群体间的遗传距离和遗传相似指数结果见表 7,韩国浦项黑参群体与韩国群山黑参群体的遗传相似指数最高(Is=0.9279),遗传距离最近(DS=0.0749),而韩国浦项红参和烟台群体间的遗传距离最远(DS=1.3573),遗传相似度最小(Is=0.2574)。聚类分析显示(图 5),烟台群体、青岛群体与韩国木浦黑参群体聚为一支,韩国浦项黄参群体、韩国群山黑参群体和韩国浦项黑参群体聚为一支,而韩国浦项红参群体作为外群,单独聚为一支。
|
|
表 7 不同群体间的遗传距离和相似性指数 Tab.7 Nei's genetic identity and genetic distance in different populations |

|
图 5 不同地理群体的聚类分析 Fig.5 UPGMA tree of the eight populations constructed by Nei's genetic distance |
近年来,SSR标记被广泛应用于水产动物养殖群体遗传变异的监测(赵哲霞等, 2014; 程珂等, 2018)、家系鉴别(李雪燕等, 2013)、地理种群遗传多样性(傅建军等, 2015)等研究中。谭杰等(2007)利用微卫星技术分析发现,蓬莱养殖刺参群体遗传多样性低于烟台崆峒岛野生刺参群体。高悦勉等(2004)研究发现,大连大孤山、旅顺塔河湾、长海县大长山、海洋岛及瓦房店复州湾海域地区野生刺参与大连金州杏树屯养殖群体遗传多样性基本无差别,这可能与该研究采样样本的地理区域过于集中有关。本研究利用13个微卫星位点对8个不同地理群体225个刺参个体的遗传多样性进行分析。结果显示,13个位点在8个群体中均具有较高的遗传多样性,但不同位点又具有一定的差异性。对于不同位点,等位基因数目不等,其中,位点AJ07在8个群体中的等位基因数最多,有34个,而位点C37的等位基因数最少,有10个。不同位点样本的观测杂合度和期望杂合度也具有一定的差异性,分别为0.20~0.83和0.42~0.92。这种不同位点等位基因数、观测杂合度、期望杂合度存在较大差异的现象在刺参微卫星研究中广泛存在(Zhan et al, 2007; 卢超等, 2010)。13个位点的多态信息含量为0.465~0.909,除AJ06为中度多态性(0.25 < PIC < 0.5)外,其余12个位点均为高度多态性(PIC>0.5),说明本研究所使用的13个微卫星位点具有良好的多态性,利用这些位点可更客观地检测出不同群体的遗传差异性。不同地理群体间的平均等位基因数也存在显著差异,韩国群山黑参平均等位基因数目(10.2)最多,俄罗斯群体最少(5.5),这可能与其采样个体较少有关。8个刺参群体观测杂合度(Ho)范围为0.40~0.54,期望杂合度范围为0.68~0.76,8个群体的多态信息含量范围为0.6392~0.7122,各群体均显示为高度多态性(PIC>0.5),说明几个群体均具有较高的遗传多样性。本研究使用的ABI 3730XL测序仪对等位基因进行分型分析,与以往电泳法读带相比,获得的等位基因大小更精确,因此,每个位点所检测到的等位基因数目更多,所测定出的群体的遗传多样性也更高。
DNA指纹图谱技术以其高灵敏度常用于近缘物种鉴别。在水产领域,已应用于罗非鱼(Oreochromis)、剑尾鱼(Xiphophorus hellerii)、鳊鲂鱼(Parabramis, Megalobrama)、大口黑鲈(Micropterus salmoides)的群体或品系的鉴定(宋红梅等, 2009; 全迎春等, 2011; 张倩倩等, 2014; 樊佳佳等, 2012)。而在刺参遗传多样性研究方面,指纹图谱应用较少。本研究利用13个微卫星位点,构建了8个刺参群体的DNA指纹数据库,并根据其等位基因片段大小制成了微卫星DNA指纹模式图谱,显示这13个微卫星位点均具有特异性,可用于不同群体刺参的群体鉴定,可为杂交种和亲权鉴定提供参考。
对8个刺参群体进行遗传结构分析发现,韩国浦项黑参群体与韩国群山黑参群体的遗传相似指数最高(0.9279),遗传距离最近(0.0749),而韩国浦项红参和烟台群体间的遗传距离最远(1.3573),遗传相似度最小(0.2574)。聚类分析结果显示,烟台群体、青岛群体与韩国木浦黑参群体聚为一支,而韩国浦项黄参群体、韩国群山黑参群体和韩国浦项黑参群体聚为一支,而韩国浦项红参群体作为外群,单独聚为一支,聚类分析与分群结果相一致。Kim等(2008)利用9个微卫星位点研究了韩国5个地理种群的海参遗传进化关系,显示韩国南海海域2个群体聚为一支,然后与西海海域群体聚在一起,最外侧是韩国东海群体,与本研究结果一致。Chang等(2010)利用微卫星构建系统发育树发现,日本、韩国及中国大连刺参聚为一支,日本红参与俄罗斯海参聚为一支;潘传燕等(2012)研究发现,中国群体与韩国东海岸群体聚为一支,而韩国西海岸群体、俄罗斯群体和日本群体聚为另外一支。这些研究都表明,影响不同地理群体的刺参遗传分化的因素很多,除了地理位置外,还受海流、季风、气候、苗种逃逸等影响(谭杰等, 2007a、b)。对韩国浦项地区3个不同颜色刺参的分析表明,韩国浦项红参群体的遗传分化及遗传结构与其他群体差异较大,说明体色与遗传关系具有相关性。Kanno等(2006)利用11个微卫星位点对日本3个颜色刺参种群进行物种差异分析,结果显示,红色刺参与绿色、黑色刺参之间存在较大遗传差异,且在聚类分析中,红色刺参单独聚为一支,绿色和黑色刺参聚为一支,与本研究结果一致。
本研究利用13对微卫星引物对6个海域8个地理群体的研究发现,各群体均具有较高的遗传多样性,并且不同群体刺参遗传分化结果与其所处的地理位置及其体色具有一定的相关性,是生态环境、海流、地理阻隔相互作用的结果,构建微卫星指纹图谱可以将8个群体区分开,相关结果将为刺参种质资源鉴定和挖掘利用提供数据支撑。
Chang YQ, Feng ZG, Yu JP, et al. Genetic variability analysis in five populations of the sea cucumber Stichopus(Apostichopus japonicus) from China, Russia, South Korea and Japan as revealed by microsatellite markers. Marine Ecology, 2010, 30(4): 455-461 |
Cheng K, Chen C, Shi Y, et al. Genetic diversiy monitoring of selected generations of Chinese soft-shelled turtle (Pelodiscus sinensis) based on microsatellites. Genomics and Applied Biology, 2018, 37(9): 3774-3781 [程珂, 陈辰, 史燕, 等. 基于微卫星的中华鳖(Pelodiscus sinensis)选育世代遗传多样性监测. 基因组学与应用生物学, 2018, 37(9): 3774-3781] |
Dewoody JA, Fletcher DE, Wilkins SD, et al. Genetic monogamy and biparental care in an externally fertilizing fish, the largemouth bass (Micropterus salmoides). Proceedings of the Royal Society B:Biological Sciences, 2000, 267(1460): 2431-2437 DOI:10.1098/rspb.2000.1302 |
Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software structure:A simulation study. Molecular Ecology, 2005, 14(8): 2611-2620 DOI:10.1111/j.1365-294X.2005.02553.x |
Excoffier L, Lischer HEL. Arlequin suite Ver 3.5:A new series of programs to perform population genetics analyses under Linux and Windows. Molecular Ecology Resources, 2010, 10: 564-567 DOI:10.1111/j.1755-0998.2010.02847.x |
Fan JJ, Bai JJ, Li SJ, et al. Establishment of DNA fingerprinting and analysis on genetic structure of largemouth bass with microsatellite. Acta Hydrobiologica Sinica, 2012, 36(4): 600-609 [樊佳佳, 白俊杰, 李胜杰, 等. 大口黑鲈微卫星DNA指纹图谱的构建和遗传结构分析. 水生生物学报, 2012, 36(4): 600-609] |
FAO.Sea cucumbers: Commerically important sea cucumbers of the world.//FAO Fisheries and Aquaculture Technical Paper, Rome: FAO, 2012, 6: 1-150
|
Fu JJ, Xu RW, Xue T, et al. Genetic analysis of three stocks of loach with microsatellite markers and D-Loop partial sequences. Journal of Fisheries of China, 2015, 39(4): 465-474 [傅建军, 徐如卫, 薛婷, 等. 3种泥鳅微卫星标记和D-Loop部分序列遗传变异分析. 水产学报, 2015, 39(4): 465-474] |
Gao YM, Sun JB. The biochemical genetics of Stichopus japonicus Selenka population. Journal of Dalian Fisheries, 2004, 19(1): 30-34 [高悦勉, 孙静波. 刺参种群同工酶的生化遗传分析. 大连水产学院学报, 2004, 19(1): 30-34] |
Hao J, Yang Q, Bao D, et al. The sequence comparison of mtDNA D-loop and adjacent regions in six fish species. Journal of Dalian Ocean University, 2013, 28(2): 160-165 [郝君, 杨蔷, 鲍迪, 等. 6种鱼mtDNA D-loop及其邻近区段的序列比较分析. 大连海洋大学学报, 2013, 28(2): 160-165] |
Jo J, Oh J, Lee H, et al. Draft genome of the sea cucumber Apostichopus japonicus and genetic polymorphism among color variants. GigaScience, 2017, 6(1): 1-6 |
Kanno M, Suyama Y, Li Q, et al. Microsatellite analysis of Japanese sea cucumber, Stichopus(Apostichopus japonicus), supports reproductive isolation in color variants. Marine Biotechnology, 2006, 8(6): 672-685 |
Kim M, Choi T, An HS. Population genetic structure of sea cucumber, Stichopus japonicus in Korea using microsatellite markers. Aquaculture Research, 2008, 39(10): 1038-1045 |
Li XY, Sun GH, Yang JM, et al. Parentage identification using microsatellite DNA in artificial breeding of Apostichopus japonicus.. Oceanologia et Limnologia Sinica, 2013, 44(5): 1263-1269 [李雪燕, 孙国华, 杨建敏, 等. 微卫星标记在刺参(Apostichopus japonicus)人工繁育中亲本识别的应用. 海洋与湖沼, 2013, 44(5): 1263-1269] |
Liao YL. Fauna Sinica Echinodermata Holothuroidea. Beijing:Science Press, 1997, 148-150 [廖玉麟. 中国动物志棘皮动物门海参纲. 北京:科学出版社, 1997, 148-150] |
Lu C, Bao ZM, Peng W, et al. Isolation and characterization of microsatellite markers for sea cucumber (Apostichopus japonicus) by screening the SSR-enriched library. Periodical of Ocean University of China, 2010, 40(S1): 137-141 [卢超, 包振民, 彭薇, 等. 富集文库-菌落原位杂交法筛选仿刺参微卫星标记. 中国海洋大学学报, 2010, 40(S1): 137-141] |
McConnell SK, O'Reilly P, Hamilton L, et al. Polymorphic microsatellite loci from Atlantic salmon (Salmon salar):Genetic differentiation of North American and European populations. Canadian Journal of Fisheries and Aquatic Sciences, 1995, 52(9): 1863-1872 |
Miao GD, Du M, Yang JF, et al. Development of two microsatellite multiplex PCR systems for turbot (Scophthalmu smaximus) and its application for family management. Periodical of Ocean University of China, 2011, 41(1): 97-106 [苗贵东, 杜民, 杨景峰, 等. 大菱鲆亲子鉴定的微卫星多重PCR技术建立及应用. 中国海洋大学学报, 2011, 41(1): 97-106] |
Pan CY, Zang YP, Liao MJ, et al. Development of microsatellite DNA markers of sea cucumber Apostichpus japonicus and their application in population genetic variation and structure analysis. Progress in Fishery Sciences, 2012, 33(4): 72-82 [潘传燕, 臧云鹏, 廖梅杰, 等. 仿刺参微卫星标记的筛选及群体遗传结构分析. 渔业科学进展, 2012, 33(4): 72-82] |
Purcell S W, Mercier A, Conand C, et al. Sea cucumber fisheries:Global analysis of stocks, management measures and drivers of overfishing. Fish and Fisheries, 2013, 14(1): 34-59 |
Quan YC, Liu YF, Bai JJ. DNA fingerprint structure of aquatic experimental animal Swrodtail, Xiphophorus hellerii. Chinese Journal of Comparative Medicine, 2011, 21(6): 30-33 [全迎春, 刘宇飞, 白俊杰. 水生实验动物剑尾鱼的DNA指纹图谱构建. 中国比较医学杂志, 2011, 21(6): 30-33] |
Scribner KT, Gust JR, Fields RL. Isolation and characterization of novel salmon microsatellite loci:Cross-species amplification and population genetic applications. Canadian Journal of Fisheries and Aquatic Sciences, 1996, 53(4): 833-841 |
Song HM, Bai JJ, Ye X, et al. Establishment of DNA fingerprinting of tilapia with microsatellite. Journal of Fisheries of China, 2009, 33(3): 357-363 [宋红梅, 白俊杰, 叶星, 等. 罗非鱼微卫星DNA指纹图谱的构建. 水产学报, 2009, 33(3): 357-363] |
Sun X, Li Q, Kong L. Comparative mitochondrial genomics within sea cucumber (Apostichopus japonicus):Provide new insights into relationships among color variants. Aquaculture, 2010, 309(1-4): 280-285 |
Tan J, Sun HL, Liu P, et al. Genetic diversity of three geographical populations of Apostichopus japonicus revealed by microsatellite DNA. Journal of Fisheries of China, 2007a, 31(4): 437-442 [谭杰, 孙慧玲, 刘萍, 等. 3个仿刺参地理种群遗传变异的微卫星DNA分析. 水产学报, 2007a, 31(4): 437-442] |
Tan J, Sun HL, Liu P, et al. Microsatellites analysis of genetic variation between natural and culture populations of Apostichopus japonicus. Marine fisheries research, 2007b, 28(3): 38-43 [谭杰, 孙慧玲, 刘萍, 等. 仿刺参自然群体和养殖群体间遗传变异的微卫星标记研究. 海洋水产研究, 2007b, 28(3): 38-43] |
Yeh FC, Boyle TJ. Population genetic analysis of co-dominant and dominant markers and quantitative traits. Belgian Journal of Botany, 1997, 129(2): 157 |
Zhan AB, Bao ZM, Lu W, et al. Development and characterization of 45 novel microsatellite markers for sea cucumber (Apostichopus japonicus). Molecular Ecology Notes, 2007, 7(6): 1345-1348 |
Zhang QQ, Chen J, Jiang XY, et al. Establishment of DNA fingerprinting and analysis on genetic structure of different Parabramis and Megalobrama populations with microsatellite. Journal of Fisheries of China, 2014, 38(1): 15-22 [张倩倩, 陈杰, 蒋霞云, 等. 不同鳊鲂鱼类群体微卫星DNA指纹图谱的构建和遗传结构分析. 水产学报, 2014, 38(1): 15-22] |
Zhang T, Chen JW, Zhang L, et al. Morphological characteristics of Squaliobarbus curriculus and tissue specificity analysis for two isozymes. Journal of Southern Agriculture, 2017, 48(12): 2281-2287 [张涛, 陈建武, 张林, 等. 赤眼鳟形态特征及其两种同工酶的组织特异性分析. 南方农业学报, 2017, 48(12): 2281-2287] |
Zhao ZX, Jiang S, Wang BH, et al. Identification and genetic diversity analysis of Pelteobagrus basedon SSR molecular marker. Journal of Nanchang University (Natural Science), 2014, 38(5): 498-501 [赵哲霞, 蒋珊, 王滨花, 等. 黄颡鱼属SSR分子鉴定及其遗传多样性. 南昌大学学报(理科版), 2014, 38(5): 498-501] |



