2. 中国水产科学研究院生物技术研究中心 北京 100141
2. Research Centre for Aquatic Biotechnology, Chinese Academy of Fishery Sciences, Beijing 100141
大菱鲆(Scophthalmus maximus),俗称“欧洲比目鱼”或“多宝鱼”,是中国北部沿海地区重要的海水养殖经济鱼类。大菱鲆具有生长速度快、肉质鲜美以及抗逆性强等诸多优点,其养殖区域迅速扩展、产量产值不断攀升(雷霁霖等, 2002、2005)。然而,随着工厂化养殖规模和集约化程度的扩大,特别是种质资源的退化,成活率低、病害严重和生产缓慢等问题逐渐显现(李杰等, 2019; 王岚等, 2017)。因此,开展大菱鲆相关经济性状的选育研究,培育具有强抗逆性和高生长性能的新品种对大菱鲆产业发展具有重要意义。传统的育种方法是基于目标性状的表型值来开展选育工作(Hulata, 2001),对于高生长性能品种选育,通常挑选符合标准的个体进行群体混养或是建立多个家系并选择优良的家系作为亲本用于进一步选育。然而,分子生物学和基因组学的快速发展则促使鱼类育种研究由传统选育和杂交育种向基于基因组信息的分子育种转变(Xu et al, 2015),如以基因定位和数量性状位点(Quantitative trait locus, QTL)作图为基础的分子标记辅助选择(Marker-assisted selection, MAS)育种(Wang et al, 2015; 刘晓菲等, 2019)。利用目标性状与分子标记间的显著关联性,依据标记对后代个体进行全基因组选择或是综合标记与表型信息构建选择指数筛选个体,加速育种过程,提高选育效率。
随着测序成本的下降与高通量基因分型技术的迅猛发展,基于海量单核苷酸多态性(Single-nucleotide polymorphisms, SNP)标记的全基因组关联分析(Genome-wide association studies, GWAS)已取代利用稀疏遗传标记的QTL定位,成为鉴定与目标性状显著关联的遗传变异的主要方法。早期GWAS研究主要针对单一性状表型(Yu et al, 2006; Burton et al, 2007),而在进行复杂性状的遗传解析且记录有多个相关性状表型值时,考虑多个性状联合信息的多性状GWAS策略通常要优于逐个性状与遗传位点间的关联分析(Kim et al, 2009; Bolormaa et al, 2010)。相较单一性状GWAS分析,表型具有潜在相关性的多性状GWAS分析更具优势,主要是因为:当不同性状间存在遗传相关时,因考虑了单一性状分析中忽略的性状间协方差,多性状分析提高了检验功效(Stephens, 2013)和参数估计的精度(Zhu et al, 2009);在多性状GWAS分析过程中,数量性状核苷酸(Quantitative trait nucleotides, QTNs)的统计推断只进行一次统计检验,与逐个性状单独分析相比,降低了多重检验造成的误差(Klei et al, 2008);存在基因多效性时,单个遗传变异与多个性状相关联,使得多性状GWAS分析更具生物学意义(Chavali et al, 2010);Porter等(2017)综合比较了当下流行的多性状GWAS分析方法,发现大多数现存的多性状GWAS分析方法具有明显相似的统计检验功效,与单一性状GWAS相比,能够大幅增加显著遗传变异的检测效率。
本研究采用实验成本较低的简化基因组(2b- RAD)测序技术进行SNP分型,基于多性状线性混合模型对体重–体长和体长–体宽–尾柄宽共2种多性状相关表型组合进行全基因组关联分析,关联定位控制这些性状的一因多效QTNs,为大菱鲆的体重和体尺性状改良提供理论基础。此外,对逐个性状进行了单一性状线性混合模型GWAS分析,并将其结果与多性状GWAS分析结果进行比较。
1 材料与方法 1.1 实验群体及表型测量实验用鱼共585条,取自29个全同胞家系组成的大菱鲆育种群体,4月龄时,对群体中所有大菱鲆个体注射电子标记,并提取鳍条组织DNA用于后续的2b-RAD测序。随后,将标记后的大菱鲆随机混养在2个6 m×6 m×1.5 m具有循环水养殖系统的水泥池中,控制水温在5℃~24℃,每天定时投喂商用饵料2次至饱食。实验用鱼按孵化日期至表型测量时的生长天数为275~1001 d不等。对实验用鱼进行11次不定期表型测量,每次测量前利用50 mg/ml MS-222鱼用安定剂麻醉待测大菱鲆个体,避免应激反应造成的鱼体损伤。利用电子秤称量每个个体的体重(Body weight, g, BM)性状,同时在统一的参考标尺下,用数码相机由固定高度向下垂直拍摄相应个体的体尺性状。根据拍摄图形,利用ImageJ软件标定每条大菱鲆的体长(Body length, cm, BL)、体宽(Body width, cm, BW)和尾柄宽(Caudal peduncle width, cm, CPW)共3个体尺性状的表型值。采用简化基因组2b-RAD高通量标记分型技术对具有表型测量记录的个体进行SNP分型,共获得30049个多态SNP分子标记,参考大菱鲆基因组(ASM318616v1)建立多态标记物理图谱。挑选生长周期为473 d左右(前后皆不超过5 d)大菱鲆个体的表型观测值作为多性状GWAS分析的表型值,使用PLINK v1.9 (http://www.cog-genomics.org/plink2)对相应样本个体的基因型数据进行质量控制。剔除低于90%最小检出率的个体以及最小检出频率低于95%、最小哈代温伯格平衡为1.0×10–6、最小等位基因频率小于3%和方差变异大于0.05的SNPs,最终得到441个样本的23988个SNP标记用于全基因组关联分析。
1.2 关联统计分析本研究使用GEMMA软件(Zhou et al, 2014)中的多性状线性混合模型(Multivariate linear mixed model, mvLMM)进行多个性状表型值和SNPs标记的全基因组关联分析,所用模型为:
| $ Y = WA + x\beta + G + E $ |
式中,Y为n×t维表型矩阵,n是样本个数,t是分析性状个数;W为n×c维协变量(非遗传固定效应,如性别、年龄等)矩阵,A为c维相应系数行向量,c是包含截距项在内的协变量个数;x为n维当前检验SNP的基因型指示变量列向量,β为t维当前检验SNP的加性遗传效应行向量;G为n×t维不包括当前检验SNP的剩余多基因效应矩阵,G~MN(0, Vg
最终,全基因组上每个SNP都能得到1个一因多效Wald统计量和相对应的统计概率P值。此外,还利用因子谱分解线性混合模型(Factored spectrally transformed linear mixed model, FaST-LMM) (Lippert et al, 2011)逐个性状进行单一性状的GWAS分析,检测与各个性状相关联的QTNs。使用R语言绘制曼哈顿和Quantile-Quantile (QQ)图,同时,统计用于判别群体分层影响大小的膨胀系数或称基因组控制(Genomic control, GC)值,在实际研究中GC值被定义为所有SNP标记的卡方统计量均值(Price et al, 2010)。
1.3 遗传参数估计和基因注释将质量控制后的基因型数据用PLINK软件(Chang et al, 2015)处理为分析所需的格式,再利用单性状约束最大法估计法(Restricted maximum likelihood, REML)逐个估计各性状的遗传参数。查找关联分析得到的显著SNP位点在全基因组上所处的物理位置,于大菱鲆全基因组(GCA_003186165.1)上选择距离其最近的候选基因进行注释分析。
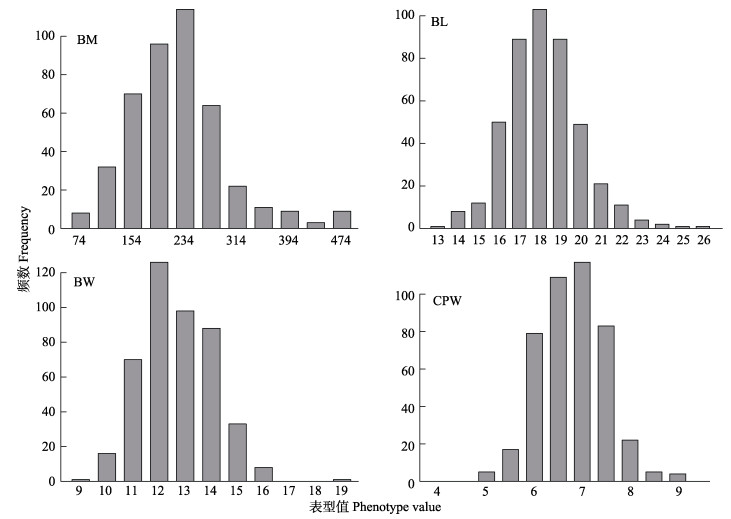
2 结果 2.1 表型值的描述性统计选取生长天数为473 d左右的441条大菱鲆的体重、体长、体宽和尾柄宽4个性状进行联合GWAS分析。4个性状的表型频数分布见图 1。从图 1可以看出,各个性状的表型值基本都符合正态分布,具有一定的可靠性,适合后续的GWAS分析。各性状的原始表型的统计分析和遗传力估计值见表 1。此外,还对原始以及校正了性别、池子和测定日期等非遗传固定效应后的表型值进行了性状间的相关分析,发现各性状表型值间具有较强的相关性(表 2)。
|
图 1 4个性状的频数分布 Fig.1 Frequency distribution histograms of four traits |
|
|
表 1 大菱鲆体重和体尺性状的描述性统计 Tab.1 Descriptive statistics of body weight and morphological traits in turbot |
|
|
表 2 大菱鲆体重和体尺性状相关程度 Tab.2 Degree of correlation for body mass and morphological traits in turbot |
根据质量控制结果,441条大菱鲆的23988个SNPs被用于体重和体尺性状的联合GWAS分析,分别进行了BM-BL和BL-BW-CPW两个性状组合的多性状线性混合模型以及逐个性状的单一性状线性混合模型GWAS分析。对BM-BL两个性状的多性状GWAS分析后(图 2和表 3)发现,超过基因组显著水平(5% Bonferroni校正阈值,即2.084×10–6)的QTN位点共9个,其中,各有1个显著QTN位于1、10和20号染色体上,即SNP_1_31495825、SNP_10_ 6893888和SNP_20_18773114,分别有2个显著QTNs位于3 (SNP_3_21601589与SNP_3_24007357)、5(SNP_5_25635891与SNP_5_26888833)和22(SNP_22_ 6190492与SNP_22_6380029)号染色体上。而在相同显著水准下,BL-BW-CPW 3个体尺性状的GWAS分析则在10号染色体上检测到2个显著的QTN位点SNP_10_2724296和SNP_10_6893888。2组多性状GWAS分析的GC值分别为BM-BL(1.035)和BL- BW-CPW(1.044)。

|
图 2 大菱鲆体重与体尺多性状GWAS曼哈顿和QQ图 Fig.2 Manhattan and Q-Q plots of multi-trait GWAS on body mass and morphological traits in turbot |
|
|
表 3 大菱鲆体重和体尺性状显著关联的QTNs信息 Tab.3 Information of QTNs significantly associated with body mass and morphological traits in turbot |
对体重和体尺的逐个性状单一性状GWAS分析结果(图 3和表 3)显示,在BM性状中,于3和5号染色体上各检测到1个显著的QTN位点,即SNP_3_21601589和SNP_5_26888833,10号染色体上检测到2个显著的QTN位点SNP_10_2724296和SNP_10_6893888。BL和BW性状分别在62和8号染色体上检测到1个显著的QTN位点SNP_6_ 6047375和SNP_8_3870447。而在CPW性状中则没有检测到显著的遗传变异位点。各性状单一性状GWAS分析的GC值分别为1.026 (BM)、1.021 (BL)、1.034 (BW)和1.031 (CPW)。

|
图 3 大菱鲆体重与体尺单一性状GWAS曼哈顿和QQ图 Fig.3 Manhattan and Q-Q plots of single-trait GWAS on body mass and morphological traits in turbot |
以大菱鲆多性状与单一性状GWAS获得的显著SNP位点为探针,根据其在大菱鲆全基因组的位置,向上下游寻找距离最近的候选基因。在本研究检测到的17个QTNs附近共找到12个候选基因(表 3),分别为GRIK2 (Glutamate receptor, ionotropic, kainate 2)、cyp21a2 (Cytochrome P450, family 21, subfamily A, polypeptide 2)、tshz3b (Teashirt zinc finger homeobox 3b)、adamts17 (ADAM metallopeptidase with thrombo- spondin type 1 motif, 17)、SYT8 (Synaptotagmin 8)、myt1la (Myelin transcription factor 1-like, a)、psmd4a (Proteasome 26S subunit, non-ATPase 4a)、grm4 (Glutamate receptor, metabotropic 4)、BFAR (Bifunctional apoptosis regulator)和3个新基因。对这些候选基因进行GO注释分析发现,细胞组分主要涉及核膜、细胞质膜及细胞核等;分子功能主要涉及离子通道活性、激素活性、氧化还原酶活性、肽酶活性、核苷酸结合、蛋白结合及G蛋白偶联受体活性等;而生物学进程则主要涉及离子转运、糖皮质激素生物合成过程、氧化还原过程、细胞信号转导、基因表达调控、蛋白酶解、胞外分泌、转录调控和G蛋白偶联受体信号通路等。其中,3个新基因在大菱鲆基因组中未有相关注释信息。
3 讨论GWAS作为一种定位影响重要经济性状分子标记的有效方法,是以全基因组SNPs直接关联复杂性状表型,广泛应用于动植物分子标记开发和辅助选择育种等方面(Santana et al, 2015; He et al, 2017)。随着诸多水产动物基因组测序工作相继完成以及测序成本的下降,水产动物生长和抗病等复杂性状的GWAS分析多有研究报道。Gutierrez等(2015)对大西洋鲑(Salmo salar)在性成熟时的生长速率性状进行GWAS分析,检测到多个在大西洋鲑代谢和生长发育过程中发挥重要作用的候选基因。Zhou等(2019)利用GWAS揭示了半滑舌鳎(Cynoglossus semilaevis)抗病的遗传机制,共定位了与抗病性状显著关联的33个SNP位点,结合基因表达和甲基化分析,检测到数个影响半滑舌鳎抗病力的候选基因。Jiang等(2019)则对罗非鱼(Tilapia)的耐盐性状进行GWAS和QTL定位研究,为罗非鱼耐盐性遗传机理揭示及进一步的功能分析奠定了基础。目前,大菱鲆生长和体尺相关性状的GWAS分析鲜有应用,但QTL定位研究已在大菱鲆生长(Enrique et al, 2011)和气单胞菌(Aeromonas salmonicida)耐药性(Silvia et al, 2011)等相关性状中开展。
现有的绝大多数GWAS研究都是关联全基因组上SNP位点与单个性状表型值,即使当多个相关表型性状存在时,往往也会选择逐个性状分别进行单一性状GWAS分析(Stephens, 2013)。在多个相关表型性状条件下,单一性状关联定位方法忽略了QTNs对多个表型性状的共同影响。相较于单一性状关联分析方法,多性状GWAS方法具有更高的统计功效和参数估计及定位精确度。通过比较大菱鲆体重和体尺多性状GWAS与各个性状逐个进行单一性状GWAS的定位结果发现,在相同的显著水准下,前者检测到的QTN数多于后者(表 3)。在BM-BL性状组合的多性状GWAS分析中,共检测到9个QTNs,而在相对应的BM和BL单一性状GWAS分析中,BM中检测到4个QTNs,BL中仅定位到1个QTN,且BM中检测到的4个QTNs中有3个与多性状GWAS分析得到的QTNs重复。同样,BL-BW-CPW性状组合多性状GWAS分析检测到的QTNs也多于3个性状各自进行单一性状GWAS分析得到的QTNs。除了能够检测到更多的QTNs,由于多性状GWAS分析检测到的QTN是一因多效QTN,相较于单一性状GWAS分析检测到的QTN在生物学和实际应用层面更有意义。
在大菱鲆全基因组上寻找距离每个QTN最近的基因,共找到12个候选基因,其中,有9个已知的功能基因及3个新基因。这些影响大菱鲆体重和体尺性状的候选基因在全基因组分布较为分散,没有明显集中区域。本研究为首次对大菱鲆体重和体尺性状进行全基因组关联分析,检测到在大菱鲆相关研究中尚未见报道的显著的已知功能基因。相关研究表明,GRIK2为离子型受体,它偶联离子通道并形成受体通道复合物,且与细胞缺血缺氧损伤有关(张冬梅等, 2010)。Eachus等(2017)和Weger等(2018)的研究均表明,cyp21a2在斑马鱼(Brachydanio rerio var)幼鱼的糖皮质激素生物合成中发挥着重要作用。Erickson等(2011)以斑马鱼为实验动物模型,发现tshz3b可能调控后脑中的Hox功能,而Hox功能则被认为与形态发生和器官形成有关联(Gair et al, 2003)。由此可见,该基因可能参与大菱鲆体形态发育过程。Myt1la可能在有丝分裂结束阶段发挥重要作用(Nakajima et al, 2008),BFAR则能与p75NTR的蛋白相互作用并抑制p75NTR信号转导,而p75NTR可与高亲和力受体TrkA协同作用促进细胞增殖或与细胞内配体结合诱导细胞凋亡(李红梅等, 2011)。本研究找到的候选基因可能都是大菱鲆体重和体尺性状的重要候选功能基因,它们在大菱鲆生长发育过程中的影响有待进一步的功能实验验证。
Allison DB, Thiel B, St Jean P, et al. Multiple phenotype modeling in gene-mapping studies of quantitative traits: Power advantages. American Journal of Human Genetics, 1998, 63(4): 1190-1201 DOI:10.1086/302038 |
Bolormaa S, Pryce JE, Hayes BJ, et al. Multivariate analysis of a genome-wide association study in dairy cattle. Journal of Dairy Science, 2010, 93(8): 3818-3833 DOI:10.3168/jds.2009-2980 |
Burton PR, Clayton DG, Cardon LR, et al. Genome-wide association study of 14, 000 cases of seven common diseases and 3, 000 shared controls. Nature, 2007, 447(7145): 661-678 DOI:10.1038/nature05911 |
Chang CC, Chow CC, Tellier LC, et al. Second-generation PLINK: Rising to the challenge of larger and richer datasets. GigaScience, 2015, 4(7): 1-16 |
Chavali S, Barrenas F, Kanduri K, et al. Network properties of human disease genes with pleiotropic effects. BMC Systems Biology, 2010, 4(78): 1-11 |
Eachus H, Zaucker A, Oakes JA, et al. Genetic disruption of 21-hydroxylase in zebrafish causes interrenal hyperplasia. Endocrinology, 2017, 158(12): 4165-4173 DOI:10.1210/en.2017-00549 |
Enrique SM, Alex C, Miguel AT, et al. Detection of growth- related QTL in turbot (Scophthalmus maximus). BMC Genomics, 2011, 12(1): 473 DOI:10.1186/1471-2164-12-473 |
Erickson T, Pillay LM, Waskiewicz AJ. Zebrafish Tshz3b negatively regulates hox function in the developing hindbrain. Genesis, 2011, 49(9): 725-742 DOI:10.1002/dvg.20781 |
Gair HJC, Lovegrove B. Beyond homeosis-HOX function in morphogenesis and organogenesis. Differentiation, 2003, 71(8): 461-476 DOI:10.1046/j.1432-0436.2003.7108004.x |
Gutierrez AP, José MY, Fukui S, et al. Genome-wide association study (GWAS) for growth rate and age at sexual maturation in Atlantic salmon (Salmo salar). PLoS One, 2015, 10(3): e0119730 DOI:10.1371/journal.pone.0119730 |
He JB, Meng S, Zhao TJ, et al. An innovative procedure of genome-wide association analysis fits studies on germplasm population and plant breeding. Theoretical and Applied Genetics, 2017, 130(11): 2327-2343 DOI:10.1007/s00122-017-2962-9 |
Hulata G. Genetic manipulations in aquaculture: A review of stock improvement by classical and modern technologies. Genetica, 2001, 111(1-3): 155-173 |
Jiang DL, Gu XH, Li BJ, et al. Identifying a long QTL cluster across chrLG18 associated with salt tolerance in Tilapia using GWAS and QTL-seq. Marine Biotechnology, 2019, 21(2): 250-261 DOI:10.1007/s10126-019-09877-y |
Kim S, Sohn KA, Xing EP. A multivariate regression approach to association analysis of a quantitative trait network. Bioinformatics, 2009, 25(12): 204-212 DOI:10.1093/bioinformatics/btp218 |
Klei L, Luca D, Devlin B, et al. Pleiotropy and principal components of heritability combine to increase power for association analysis. Genetic Epidemiology, 2008, 32(1): 9-19 DOI:10.1002/gepi.20257 |
Lei JL, Ma AJ, Chen C, et al. The present status and sustainable development of turbot (Scophthalmus maximus L. ) culture in China. Engineering Sciences, 2005(5): 34-38 [雷霁霖, 马爱军, 陈超, 等. 大菱鲆(Scophthalmus maximus L.)养殖现状与可持续发展. 中国工程科学, 2005(5): 34-38] |
Lei JL, Men Q, Wang YG, et al. The factory farming mode of Dalingyu "greenhouse shed + deep well seawater". Marine Fisheries Research, 2002, 23(4): 1-7 [雷霁霖, 门强, 王印庚, 等. 大菱鲆"温室大棚+深井海水"工厂化养殖模式. 海洋水产研究, 2002, 23(4): 1-7] |
Li HM, Shi HL, Huo KK. p75NTR signal transduction suppressed by BFAR and p75NTR interactions. Scientia Sinica Vitae, 2011, 41(12): 1140-1147 [李红梅, 施慧莉, 霍克克. BFAR与p75NTR的蛋白相互作用抑制p75NTR信号转导. 中国科学: 生命科学, 2011, 41(12): 1140-1147] |
Li J, Liu YK, Bai L, et al. Isolation and identification of Mycobacterium marinum associated with splenic and renal granuloma disease of cultured turbot (Scophthalmus maximus). Progress in Fishery Sciences, 2019, 40(5): 195-199 [李杰, 刘耀宽, 白露, 等. 大菱鲆脾肾结节病病原菌的分离和鉴定. 渔业科学进展, 2019, 40(5): 195-199] |
Lippert C, Listgarten J, Liu Y, et al. FaST linear mixed models for genome-wide association studies. Nature Methods, 2011, 8(10): 833-835 DOI:10.1038/nmeth.1681 |
Liu XF, Ma AJ, Huang ZH, et al. Expression characteristics analysis of major QTL candidate genes in response to high temperature stress in turbot (Scophthalmus maximus). Journal of Fisheries of China, 2019, 43(6): 1407-1409 [刘晓菲, 马爱军, 黄智慧, 等. 大菱鲆高温胁迫应答主效QTL候选基因的表达特性分析. 水产学报, 2019, 43(6): 1407-1409] |
Nakajima H, Yonemura S, Murata M, et al. Myt1 protein kinase is essential for Golgi and ER assembly during mitotic exit. Journal of Cell Biology, 2008, 181(1): 89-103 DOI:10.1083/jcb.200708176 |
Porter HF, O'Reilly PF. Multivariate simulation framework reveals performance of multi-trait GWAS methods. Scientific Reports, 2017, 7: 38837 DOI:10.1038/srep38837 |
Price AL, Zaitlen NA, Reich D, et al. New approaches to population stratification in genome-wide association studies. Nature Reviews Genetics, 2010, 11(7): 459-463 DOI:10.1038/nrg2813 |
Santana MHA, Ventura RV, Utsunomiya YT, et al. A genomewide association mapping study using ultrasound-scanned information identifies potential genomic regions and candidate genes affecting carcass traits in Nellore cattle. Journal of Animal Breeding and Genetics, 2015, 132(6): 420-427 DOI:10.1111/jbg.12167 |
Silvia TRR, Toro MA, Bouza C, et al. QTL detection for Aeromonas salmonicida resistance related traits in turbot (Scophthalmus maximus). BMC Genomics, 2011, 12(1): 541 DOI:10.1186/1471-2164-12-541 |
Stephens M. A unified framework for association analysis with multiple related phenotypes. PLoS One, 2013, 8(7): e65245 DOI:10.1371/journal.pone.0065245 |
Wang L, Wang YG, Zhang Z, et al. Diversity and drug resistance of bacterial pathogens isolated from bacterial ascetic disease in cultured turbot Scophthalmus maximus. Progress in Fishery Sciences, 2017, 38(4): 17-24 [王岚, 王印庚, 张正, 等. 养殖大菱鲆(Scophthalmus maximus)腹水病的病原多样性及其耐药性分析. 渔业科学进展, 2017, 38(4): 17-24] |
Wang WJ, Hu YL, Ma Y, et al. High-density genetic linkage mapping in turbot (Scophthalmus maximus L. ) based on SNP markers and major sex- and growth-related regions detection. PLoS One, 2015, 10(3): e0120410 DOI:10.1371/journal.pone.0120410 |
Weger M, Diotel N, Weger BD, et al. Expression and activity profiling of the steroidogenic enzymes of glucocorticoid biosynthesis and the FDX1 co-factors in zebrafish. Journal of Neuroendocrinology, 2018, 30(4): e12586 DOI:10.1111/jne.12586 |
Xu K, Duan W, Xiao J, et al. Development and application of biological technologies in fish genetic breeding. Science China, Life Sciences, 2015, 58(2): 187-201 DOI:10.1007/s11427-015-4798-3 |
Yu JM, Pressoir G, Briggs WH, et al. A unified mixed-model method for association mapping that accounts for multiple levels of relatedness. Nature Genetics, 2006, 38(2): 203-208 DOI:10.1038/ng1702 |
Zhang DM, Wu AM, Lou LX, et al. Alteration of ion channel gene expression profile in rat model of post-myocardial infarction heart failure. Chinese Journal of Integrated Traditional and West Medicine, 2010, 30(1): 53-57 [张冬梅, 吴爱明, 娄利霞, 等. 心肌梗死后心力衰竭模型大鼠离子通道基因表达谱的变化. 中国中西医结合杂志, 2010, 30(1): 53-57] |
Zhou Q, Su ZC, Li YZ, et al. Genome-wide association mapping and gene expression analyses reveal genetic mechanisms of disease resistance variations in Cynoglossus semilaevis. Frontiers in Genetics, 2019, 10: 1167 DOI:10.3389/fgene.2019.01167 |
Zhou X, Stephens M. Efficient multivariate linear mixed model algorithms for genome-wide association studies. Nature Methods, 2014, 11(4): 407-409 DOI:10.1038/nmeth.2848 |
Zhu W, Zhang H. Why do we test multiple traits in genetic association studies?. Journal of Korean Statistics Society, 2009, 38(1): 25-27 DOI:10.1016/j.jkss.2008.10.007 |



