2. 上海海洋大学 上海水产养殖工程技术研究中心 上海 201306;
3. 鲁东大学农学院 烟台 264025;
4. 山东省农业农村厅 济南 250013;
5. 山东省渔业技术推广站 济南 250013;
6. 烟台海育海洋科技有限公司 烟台 264001
2. Shanghai Ocean University, Shanghai Engineering Research Center of Aquaculture, Shanghai 201306;
3. Ludong University, College of Agriculture, Yantai 264025;
4. Department of Agriculture and Rural Affairs of Shandong Province, Jinan 250013;
5. Shandong Fishery Technology Extension Station, Jinan 250013;
6. Yantai Haiyu Ocean Technology Co., Ltd, Yantai 264001
刺参(Apostichopus japonicus),又称仿刺参,生活在岩礁底的浅海中,广泛分布于西太平洋沿岸浅海。随着刺参养殖规模的迅速扩大,刺参产业发展面临的种质退化、病害频发、极端气候灾害等严峻问题日益突出(高凯伦, 2018; 张春云等, 2004)。目前,刺参苗种产业中亲参的来源主要是遗传背景不清楚的“野生”刺参以及累代繁育的人工养殖刺参,由此构成的苗种繁育的种质基础群体,其后代往往出现生长缓慢、个体差异大、应激性差、抗逆抗病性弱等现象,严重影响了刺参养殖产业的健康发展(侯西坦, 2016; 曲洪霞等, 2016)。与此同时,近年来气候变化带来的极端高温天气造成山东、辽宁等大面积养殖池塘刺参大量死亡,对沿海刺参养殖产业带来沉重打击。因此,培育刺参优良抗逆新品种是解决刺参优良种质匮乏和安全度夏瓶颈问题的重要出路,刺参种业关乎刺参产业乃至整个水产养殖业的提质增效和稳定发展。针对目前对优良种质的需求,全国多团队进行了不同表型特征刺参的新品种创制和研发,本课题组经过连续4代多年的高温驯化和胁迫选育,培育出一个高温耐受性能提高的刺参新品系,在养殖生产试验中表现出度夏成活率高、产量提高等优势。
近20年以来,表观遗传学研究取得了长足进展,已有研究表明,遗传表型改变不仅来源于DNA序列的改变,也存在不依赖序列变化的表观遗传修饰。表观遗传变异研究主要有DNA的甲基化修饰、核小体中组蛋白翻译后修饰、非编码RNA调控、组蛋白变体及受催化的染色质重塑等方面(Wolffe et al, 1999; 王淑妍等, 2016; 康静婷等, 2013)。DNA甲基化是表观遗传修饰的一个重要方式,也是目前在机制方面研究相对最多、最深入的表观遗传过程(邱峥艳等, 2012)。环境条件变化能够诱导DNA甲基化发生变异,并且环境条件变化能够诱导具有持续性和可遗传性的DNA甲基化和表观变异(李佳蓉等, 2018)。某些DNA甲基化在多种表观遗传机制中具有中心协调的作用,可以在后代进行稳定传递,DNA甲基化的这些特性决定其在动植物育种中,特别是抗逆性状的遗传改良方面具有非常大的应用潜力。在我们的刺参耐高温品系的选育过程中,其抗逆耐高温性状是在胁迫环境条件下驯化和筛选获得,这种优良性状是否具有表观遗传层面的遗传基础,需要进行探究。
甲基化敏感扩增多态性(Methylation-sensitive amplification polymorphisms, MSAP)技术是DNA甲基化模式研究最有效且经济实用的应用技术(Angers et al, 2010; Guarino et al, 2019; Zhao et al, 2015),由于采用的限制酶的位点特性,可以同时进行甲基化敏感位点表观遗传多样性分析,也可以进行非甲基化多态位点的遗传多样性分析(吴彪等, 2015; Zhao et al, 2015)。本研究运用MSAP技术分析了刺参育种过程中的选育基础群体和2个选育群体(选育F1代和选育F4代) 3个刺参群体的DNA甲基化多态性和模式,揭示不同刺参群体的表观遗传多样性及其遗传结构,就选育活动对基因组DNA甲基化的影响进行探讨,以期从表观遗传学角度来阐释选育过程中的遗传基础的改变。
1 材料与方法 1.1 实验材料实验所用群体为刺参耐高温新品系选育过程中的选育基础群体F、选育F1代和选育F4代。其中,基础群体为4个种质保护区野生刺参群体繁殖获得,选育F1代和F4代为选育过程代表群体,是采用群体选育技术经过浮游幼体期27℃热驯化120 min和16月龄期32℃高温胁迫72 h淘汰选择,并辅以生长速度筛选获得。在选育生产过程中,由于亲本较为珍贵,F2和F3亲本较少,没有保留用作分析样本,F1和F4作为选育初和选育完成群体可以代表选育过程且能体现选育的遗传特征。3个刺参群体分别记作F、F1和F4。从3个群体中分别随机选取18月龄、体重为200 g左右的15头刺参保存待分析。
1.2 实验方法 1.2.1 基因组DNA的提取剖取待测刺参的肌肉附带内膜组织,用于全基因组DNA提取,采用改进的十六烷基三甲基溴化铵(CTAB)法进行(全志星等, 2017)。取组织块约50 mg,置于预冷的研钵中,在液氮中充分碾磨后,加入65℃预热的CTAB裂解液[2% CTAB (W/V),0.2 mmol/L Tris-HCl (pH=8.0),0.05 mmol/L EDTA(pH 8.0),1.4 mmol/L NaCl,2% β-巯基乙醇(V/V) 1 ml,1% PVP-40(W/V)],等体积氯仿抽提上清液后,用3 mmol/L醋酸钾和异丙醇沉淀DNA,最后经无水乙醇漂洗,加入1 mmol/L NaCl溶解DNA,通过1.5%琼脂糖电泳检测DNA是否降解,检测OD260 nm/280 nm以确定DNA的纯度,存于–20℃中备用。
1.2.2 MSAP实验流程参考郭婷婷等(2013)的方法并进行优化,所用引物和接头序列见表 1。
|
|
表 1 MSAP引物和接头序列 Tab.1 MSAP primer and linker sequence |
选用EcoRⅠ、MspⅠ和HpaⅡ进行酶切反应,分别以2种酶切组合进行样品DNA的酶切,即EcoRⅠ+HpaⅡ和EcoRⅠ+MspⅠ。10 μmol/L的EcoRⅠ-A和100 μmol/L的M-H-A单链等体积混合制备接头,0.5 μl接头与上述酶切产物15 μl、175 U的T4连接酶在25 μl连接体系下,16℃水浴过夜,获得连接产物。以引物E00-A和MH00-T进行预扩增,PCR反应条件:94℃预变性2 min,94℃ 30 s,56℃ 30 s,72℃ 1 min,30个循环,72℃延长5 min,最后4℃保存。以选择性扩增引物的不同组合进行选择性扩增,PCR反应条件:94℃预变性2 min,94℃ 30 s,65℃ (每个循环递减1℃) 30 s,72℃ 1 min,共进行10个递减循环;94℃ 30 s、56℃ 30 s、72℃ 1 min,20个循环,72℃保温延长5 min,最后4℃保存。
1.2.3 MSAP数据分析通过优化MSAP实验条件,从不同引物组合中筛选多态性好、重复性高、条带清晰的10对引物组合进行实验分析。利用Quantity One软件标记出5%聚丙烯酰胺凝胶电泳图谱中100~ 500 bp区间扩增出来的条带。根据扩增产物在2个泳道内有无的情况,可将条带类型分为4种,Ⅰ型2个泳道均有条带;Ⅱ型H泳道有带,M泳道无带,代表半甲基化位点;Ⅲ型H泳道无带,M泳道有带;Ⅳ型2个泳道均没有带。根据Cervera等(2002)提出的方法,把MSAP二维矩阵数据分为甲基化敏感位点(Methylation-susceptible loci, MSL)和非甲基化位点(Non-methylated loci, NML)两大类。MSL指只能够被HpaⅡ或MspⅠ中的一种酶切开,为(1, 0)或(0, 1)情况,即甲基化模式(Ⅱ、Ⅲ型)记为1,其余模式(Ⅰ、Ⅳ型)记为0。NML指同时能被HpaⅡ和MspⅠ酶切开,为(1, 1)情况,即甲基化模式Ⅰ型记为1,其余模式(Ⅱ、Ⅲ、Ⅳ型)都记为0。
利用Popgene软件对等位基因数、杂合度、香农多态性指数等种群遗传学参数进行分析和计算。通过NTsys软件将0/1矩阵转化成相应的MSP(甲基化敏感位点)和MISP(甲基化不敏感位点)聚类图谱,基于甲基化敏感位点的表观遗传距离和基于非甲基化位点的遗传距离进行Mantel检验。
2 结果与分析 2.1 刺参群体MSAP多态性及遗传多样性刺参群体遗传多样性的各项指标显示(表 2),10对引物获得的806个位点中,多态性位点数量为698个,多态性位点的百分比为86.60%,表明本研究所用引物组合适用于刺参群体表观遗传多样性的研究。10对引物观察等位基因数(Na)平均值为1.8767,有效等位基因数(Ne)平均值为1.4809,平均杂合度(H)为0.2897,平均香农多态性指数(I)为0.4439。引物组合E3M3多态性位点百分比高达95.51%,产生的Na、Ne、H和I高于其他所有引物组合,是遗传多样性贡献率最大的引物对;引物组合E1M1的Ne、H和I均为最低值,是遗传多样性贡献率最低的引物对。
|
|
表 2 引物组合的遗传多样性信息 Tab.2 Genetic diversity information of primer combinations |
分别对3个刺参群体进行甲基化敏感位点(MSL)和非甲基化位点(NML)的多态性统计与分析(表 3),NML百分比在47.7%~50.1%范围内;香农多态性指数I为0.3981~0.4662,Neixs基因多样度H为0.2264~ 0.2475,选育基础群体F的种群多态性指数最高。MSL百分比在63%~65%范围内;I为0.5873~0.6030,H为0.2598~0.2892。无论是以MSL还是NML计算的遗传多态性指数,选育F1代和F4代均小于选育基础群体F,表明选育降低了群体的遗传多样性。选育F1代和F4代的NML百分比分别为50.1%和47.7%;I分别为0.4337和0.3981;H分别为0.2460和0.2264。选育F1代和F4代的MSL百分比分别为66%和65%;I分别为0.5995和0.5873;H分别为0.2654和0.2598。无论是以MSL还是NML计算的遗传多态性指数,选育F1代均高于F4代,说明随着群体选育代数的累加,群体遗传多样性具有逐渐降低的趋势。同时,基于MSL计算都大于基于NML得到的遗传多样性,表明表观变异出现频率高于基因序列遗传变异。
|
|
表 3 3个刺参群体MSAP多态性及遗传多样性 Tab.3 MSAP polymorphism and genetic diversity of three A. japonicus populations |
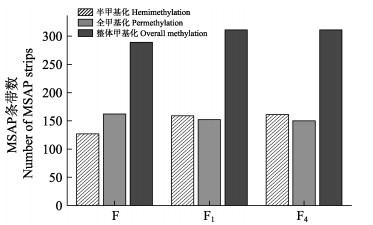
根据MSAP获得的4种带型进行不同甲基化模式的统计,表 4为3个刺参群体甲基化类型统计结果,类型Ⅰ条带数百分比为48.39%~51.86%,选育基础群体获得的类型Ⅰ条带数最多,为418条;类型Ⅳ条带数最少,占比12.28%~13.03%。类型Ⅱ条带数百分比为15.76%~19.98%,选育F1和F4代获得的类型Ⅱ条带数较选育基础群体有所增高,其中,F4代最多,为161条。选育F1和F4代的类型Ⅲ和类型Ⅳ条带数相近。半甲基化和全甲基化总体称整体甲基化,将整体甲基化、半甲基化和全甲基化模式比较分析发现(图 1),选育基础群体半甲基化低于全甲基化,2个选育群体半甲基化位点升高,整体甲基化水平高于选育基础群体,选育F1和F4代呈现相同的改变趋势。
|
|
表 4 3个刺参群体DNA甲基化类型统计 Tab.4 Statistics of DNA methylation types of three A. japonicus populations |
|
图 1 3个刺参群体甲基化状态比较 Fig.1 Comparison of the methylation status of three A. japonicus populations |
对刺参3个群体基于甲基化敏感位点和非甲基化位点的遗传距离进行Mantel检验,结果显示,相关系数r值为0.305~0.480,平均值为0.373,种群的表观遗传距离与序列遗传距离的相关性均达到显著水平(P < 0.01),说明基于甲基化敏感位点计算的表观遗传变异一定程度上依赖于序列遗传变异(表 5)。
|
|
表 5 3个刺参群体种群遗传距离与表观遗传距离的Mantel检测 Tab.5 Mantel detection of population genetic distance and epigenetic distance of three A. japonicus populations |
生物体通过基因表达的改变来实现表型上的改变,借助表型上的变化来不断进行生理及行为上的调节,进而适应环境变化,在基因表达调控方式中,作为表观遗传重要内容的DNA甲基化是重要途径之一(王忠华等, 2013)。目前,很多研究已经证明环境胁迫刺激能够使生物体通过改变DNA甲基化状态从而调控基因的表达以适应新环境(甄艳等, 2018; 高凯伦, 2018)。生物外界生存环境条件的改变,不仅可通过DNA甲基化修饰对生物体本身产生表观遗传上的变异,有些DNA甲基化修饰还可以经遗传传递给后代(Kakutani et al, 1999),这些环境胁迫诱导的DNA甲基化修饰通常认为具有可逆或可丢失性,但越来越多的研究证明,有些DNA甲基化比较稳定并可以延续若干个世代(Kakutani, 2002),这种现象称为跨代遗传,这一特点为DNA甲基化等表观遗传变异在动植物育种上的应用提供了可能。DNA甲基化在优良性状新品种的育种工作中,尤其是抗逆性状的遗传改良方面具有巨大的应用潜力(Richardson et al, 2006; Schmitz et al, 2013; Ruizgarcia et al, 2005)。
环境胁迫对生物体基因组DNA甲基化的影响是复杂多向的,DNA甲基化的变化不仅可以产生有利的表型变异,也可能产生有害的表型变异,而目前进行有益性状表型的育种还不能通过有目的地改变DNA甲基化状态来实现。在本研究刺参的耐高温品系的育种过程中,对选育群体进行了多次的高温胁迫淘汰,这种环境胁迫是否通过DNA甲基化的修饰改变获得了有利的表型,增强了刺参选育系的耐高温抗逆性状,需要探究。现阶段利用DNA甲基化的检测方法来追踪检测刺参育种过程中表观遗传的改变,可以为后期更深入利用表观遗传修饰来进行抗逆品种的培育和遗传改良提供理论依据。
3.2 选育群体的遗传多样性MSAP检测到条带分为甲基化敏感位点和非甲基化位点。甲基化敏感位点的多态性是由CCGG位点的胞嘧啶甲基化变化引起,一般认为,甲基化变异易受环境的诱导(Richards et al, 2010; Schmid et al, 2018; 郭万里, 2006)。本研究中,基于甲基化敏感位点的Shannon指数,即能够表示表观遗传多样性的参数,在基础群体和选育群体中有所改变,可以推测选育群体在高温胁迫淘汰选育环境下发生了甲基化变异。非甲基化位点变异主要由基因组序列变异引起,可以利用这些序列变异评估群体遗传多样性。本研究结果显示,随着选育世代的增加,基于非甲基化位点的Shannon多样性指数逐渐降低,F > F1 > F4,说明选育的筛选措施降低了种群的遗传多样性,种群的非甲基化位点变异出现随选育增强的渐变特征。另外,基于甲基化敏感位点计算得到的表观遗传多样性都大于基于非甲基化位点变异得到的遗传多样性,在3个选育相关种群中,表观变异出现频率高于传统遗传变异,表明在3个刺参群体中由DNA甲基化引起的表观变异更易受外界环境影响。
一般认为,表观变异比遗传变异更易受环境影响,环境诱导促进表型变异,从而适应环境(Angers et al, 2010; Guarino et al, 2019; Vaughn et al, 2007),快速而直接,且体现一定的独立性,同时,表观遗传变异可促进遗传变异的产生,有助于生物对环境的适应及促进表型上的多样性(Lewis et al, 2007; 燕雪飞, 2014)。本研究中,3个刺参群体基于甲基化敏感位点和基于非甲基化位点的遗传距离相关性分析显示,二者在3个种群相关性显著,说明在刺参优良品系的选育过程中,表观变异在一定程度上与遗传变异相互依赖和影响。
3.3 选育对甲基化状态和模式的影响从刺参选育基础群体F和选育F1代、F4代甲基化模式分析结果来看,3个种群间甲基化模式有较大的不同,其中,F1代与F4代的半甲基化位点和整体甲基化位点比较相近,且二者都表现为半甲基化位点高于各自全甲基化位点。张鹏远(2012)研究表明,不同的地域环境确实对烟草甲基化状态产生了影响;周艳华等(2015)研究也表明,茶树在抗寒响应中出现DNA甲基化现象,因此,不同刺参群体甲基化模式存在显著差异,推测是由于选育逆境胁迫适应所致。选育F4代获得的类型Ⅱ条带数最多为161条,明显高于未选育刺参,这种改变可认为是选育群体由于选育活动获得的表观遗传上的改变。本研究中,不同刺参群体在选育过程中经历不同的生存环境可能是导致甲基化模式差异的原因。
甲基化状态和模式数据说明,选育改变了DNA甲基化水平,并对模式产生了影响,刺参选育F1与F4代经过选育后获得一些甲基化位点的改变,甲基化状态变化趋势相近,说明温度胁迫选育改变了刺参群体的基因组的甲基化状态。这可能是由于基因组原来的甲基化水平、模式等甲基化状态受到选育压力的影响,开启某些功能基因的转录表达等生理代谢过程,致使选育对象产生了适应机制来应对环境胁迫的影响,并且在选育过程中将这种机制传递给了后代。在刺参的抗逆耐高温选育期间,选育材料在胁迫环境响应和适应中,表观遗传修饰可能起到很大作用。
4 结论与展望本研究利用10对MSAP引物对3个刺参选育群体的遗传多样性及表观遗传多样性进行分析,从遗传物质基础的角度揭示选育群体的遗传改变与进展,为抗逆新品种选育中的表观遗传研究提供了参考。DNA甲基化在水产动物遗传育种中的研究刚刚起步,但随着DNA甲基化分析技术的进步,尤其是随着新一代大规模测序技术的迅速发展,DNA甲基化相关研究在群体遗传关系分析、分子育种、性状分析及分子标记辅助选择等方面将发挥重要作用。
Angers B, Castonguay E, Massicotte R. Environmentally induced phenotypes and DNA methylation: How to deal with unpredictable conditions until the next generation and after. Molecular Ecology, 2010, 19(7): 1283-1295 DOI:10.1111/j.1365-294X.2010.04580.x |
Cervera MT, Ruiz GL, Martinez ZJ. Analysis of DNA methylation in Arabidopsis thaliana based on methylation-sensitive AFLP markers. Molecular Genetics and Genomics, 2002, 268(4): 543-552 DOI:10.1007/s00438-002-0772-4 |
Gao KL. The expression of growth-related genes and transcriptome analysis of individual size differences in Apostichopus japonicus. Masterxs Thesis of Dalian Ocean University, 2018 [高凯伦. 刺参(Apostichopus japonicus)生长相关基因的表达及个体大小差异的转录组分析. 大连海洋大学硕士研究生学位论文, 2018]
|
Guarino F, Cicatelli A, Brundu G, et al. The use of MSAP reveals epigenetic diversity of the invasive clonal populations of Arundo donax L. PLoS One, 2019, 14(4): e0215096 DOI:10.1371/journal.pone.0215096 |
Guo TT, Sun GH, Yang JM, et al. MSAP analysis of genome DNA methylation in different tissues of Apostichopus japonicus. Oceanologia et Limnologia Sinica, 2013, 44(1): 77-82 [郭婷婷, 孙国华, 杨建敏, 等. 刺参(Apostichopus japonicus)不同组织基因组甲基化状态MSAP分析. 海洋与湖沼, 2013, 44(1): 77-82] |
Guo WL. Molecular genetic and epigenetic diversity and genetic structure of wild barley (Hordeum brevisubulatum (Trin. ) Link. ) natural populations endemic to Songnen Plain, China. Doctoral Dissertation of Northeast Normal University, 2006, 22-26 [郭万里. 中国松嫩平原短芒野大麦天然种群分子遗传与表观遗传多样性及其种群遗传结构的研究. 东北师范大学博士研究生学位论文, 2006, 22-26]
|
Hou XT. Assessment of important economic traits and genetic diversity of four selective strains of sea cucumber Apostichopus japonicus. Masterxs Thesis of Tianjin Normal University, 2016 [侯西坦. 刺参(Apostichopus japonicus)不同选育品系的重要经济性状评价及遗传差异分析. 天津师范大学硕士研究生学位论文, 2016]
|
Kakutani T, Munakata K, Richards EJ, et al. Meiotically and mitotically stable inheritance of DNA hypomethylation induced by ddm1 mutation of Arabidopsis thaliana. Genetics, 1999, 151(2): 831-838 DOI:10.1093/genetics/151.2.831 |
Kakutani T. Epi-alleles in plants: Inheritance of epigenetic information over generations. Plant Cell Physiology, 2002, 43(10): 1106-1111 DOI:10.1093/pcp/pcf131 |
Kang JT, Liang QJ, Liang C, et al. Overview on epigenetics and its progress. Science and Technology Review, 2013(19): 66-74 [康静婷, 梁前进, 梁辰, 等. 表观遗传学研究进展. 科技导报, 2013(19): 66-74] |
Lewis NL, Zhan S. The plant genomexs methylation status and response to stress: Implications for plant improvement. Current Opinion in Plant Biology, 2007, 10: 317-322 DOI:10.1016/j.pbi.2007.04.012 |
Li JR, Jiang HZ. Application of epigenetics for genetic breeding of domestic animals. Journal of Economic Animal, 2018(3): 132-138 [李佳蓉, 姜怀志. 表观遗传学在家养动物遗传育种中的应用. 经济动物学报, 2018(3): 132-138] |
Qiu ZY, Guo J, Yang C, et al. Research progress of DNA methylation in animal genetics and breeding. China Animal Husbandry & Veterinary Medicine, 2012, 39(7): 173-178 [邱峥艳, 郭将, 杨春, 等. DNA甲基化在动物遗传育种中的研究进展. 中国畜牧兽医, 2012, 39(7): 173-178] |
Qu HX, Liu XT, Yang YN. Problems and countermeasures in the development of sea cucumber industry. Agriculture and Technology, 2016, 36(22): 138-138 [曲洪霞, 刘心田, 杨燕妮. 刺参产业发展存在的问题及应对措施. 农业与技术, 2016, 36(22): 138-138] |
Quan ZX, Tian JY, Zhang SY, et al. Studies on epigenetic diversity variation characteristics of Flaveria bidentis genome DNA of four different geographical distributions. Journal of Agro- Environment Science, 2017, 36 [全志星, 田佳源, 张思宇, 等. 不同入侵地区黄顶菊DNA表观遗传多样性变化特征. 农业环境科学学报, 2017, 36(4): 625-634] |
Richards CL, Bossdorf O, Verhoeven KJ. Understanding natural epigenetic variation. New Phytologist, 2010, 187(3): 562-564 |
Richardson DM, Pysek P. Plant invasions: Merging the concepts of species invasiveness and community invasibility. Progress in Physical Geography, 2006, 30(2): 409-431 |
Ruizgarcia L, Cervera MT, Martinezzapater JM, et al. DNA methylation increases throughout Arabidopsis development. Planta, 2005, 222(2): 301-306 |
Schmid MW, Heichinger C, Schmid DC, et al. Contribution of epigenetic variation to adaptation in Arabidopsis. Nature Communications, 2018, 9(1): 4446 |
Schmitz RJ, Schultz MD, Urich MA, et al. Patterns of population epigenomic diversity. Nature, 2013, 495(7440): 193-198 |
Vaughn MW, Tanurdzic M, Lippman Z, et al. Epigenetic natural variation in Arabidopsis thaliana. PLoS Biology, 2007, 5: 1617-1629 |
Wang SY, Guo JF, Liu XT, et al. Research progress of abiotic stress induced epigenetic variation in plants. Acta Botanica Boreali-Occidentalia Sinica, 2016(3): 631-640 [王淑妍, 郭九峰, 刘晓婷, 等. 非生物胁迫下植物表观遗传变异的研究进展. 西北植物学报, 2016(3): 631-640] |
Wang ZH, Wang D. Application of DNA methylation analysis in genetics and breeding of plants and animals. Chinese Bulletin of Life Sciences, 2013, 25(4): 435-441 [王忠华, 王迪. DNA甲基化分析技术在动植物遗传育种中的应用. 生命科学, 2013, 25(4): 435-441] |
Wolffe AP, Matzke MA. Epigenetics: Regulation through repression. Science, 1999, 286(5439): 481-486 |
Wu B, Yang AG, Dong YH, et al. MSAP analysis on genome- wide DNA methylation in selected and wild Japanese scallop Patinopecten yessoensis. Oceanologia et Limnologia Sinica., 2015, 46(4): 976-982 [吴彪, 杨爱国, 董迎辉, 等. 虾夷扇贝选育群体与野生群体基因组DNA甲基化的MSAP分析. 海洋与湖沼, 2015, 46(4): 976-982] |
Yan XF. Genetic diversity and differentiation of the wild soybean (Glycine soja Sieb. et Zucc. ) in China. Doctoral Dissertation of Shenyang Agricultural University, 2014 [燕雪飞. 中国野生大豆遗传多样性及其分化研究. 沈阳农业大学博士研究生学位论文, 2014]
|
Zhang CY, Wang YG, Rong XJ, et al. Natural resources culture and problems of sea cucumber worldwide. Progress in Fishery Sciences, 2004, 25(3): 89-97 [张春云, 王印庚, 荣小军, 等. 国内外仿刺参自然资源、养殖状况及存在问题. 渔业科学进展, 2004, 25(3): 89-97] |
Zhang PY. The MSAP analysis for genomic methylation of tobacco planting in different environments of Dali in Yunnan Province. Masterxs Thesis of Yunnan University, 2012 [张鹏远. 云南大理地区不同地域环境下烟草基因组甲基化的MSAP分析. 云南大学硕士研究生学位论文, 2012]
|
Zhao Y, Chen M, Storey KB, et al. DNA methylation levels analysis in four tissues of sea cucumber Apostichopus japonicus based on fluorescence-labeled methylation-sensitive amplified polymorphism (F-MSAP) during aestivation. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2015, 181: 26-32 |
Zhen Y, Zheng XH, Shi JS. Epigenetic mechanism of plant under stress. Genomics and Applied Biology, 2018, 37(2): 924-927 [甄艳, 郑秀化, 施季森. 表观遗传调控植物逆境胁迫的适应机制. 基因组学与应用生物学, 2018, 37(2): 924-927] |
Zhou YH, Cao HL, Yue C, et al. Changes of DNA methylation levels and patterns in tea plant (Camellia sinensis) during cold acclimation. Acta Agronomica Sinica, 2015, 41(7): 1047-1055 [周艳华, 曹红利, 岳川, 等. 冷驯化不同阶段茶树DNA甲基化模式的变化. 作物学报, 2015, 41(7): 1047-1055] |



