2. 上海海洋大学 上海水产养殖工程技术研究中心 上海 201306;
3. 鲁东大学农学院 烟台 264025;
4. 山东省农业农村厅 济南 250013;
5. 烟台海育海洋科技有限公司 烟台 264001
2. Shanghai Ocean University, Shanghai Engineering Research Center of Aquaculture, Shanghai 201306;
3. Ludong University, College of Agriculture, Yantai 264025;
4. Department of Agriculture and Rural Affairs of Shandong Province, Jinan 250013;
5. Yantai Haiyu Ocean Technology Co., Ltd, Yantai 264001
刺参(Apostichopus japonicus)是我国重要的水产养殖物种之一,自然分布在35~44°N的西北太平洋沿岸,包括中国黄渤海海域、俄罗斯东部沿岸、日本和韩国沿岸。刺参是典型的温带种类,水温变化对刺参的摄食、代谢和生殖发育等各种生理活动有重要影响(曹学顺, 2014)。温度过高或过低都不适宜刺参的生长,尤其是在高温逆境环境中,刺参的活动和摄食降低,甚至体内自由基代谢紊乱、病变死亡(谢兆文等, 2016; 高杨, 2017)。温度变化对刺参生理变化、生长发育和遗传变化的影响研究一直受到广泛关注。
表观遗传学是一种不改变DNA序列的可遗传变异,它的调节机制主要包括DNA甲基化修饰、组蛋白修饰、染色体重塑、非编码RNA调控等,这些修饰方式之间相互作用、共同调节基因组的功能(Crabtree, 2020; Kulis et al, 2010),且这些表观遗传修饰极易受到环境的诱导(康静婷等, 2013)。DNA甲基化是基因组DNA的一种重要修饰方式,通过影响核酸空间构象、稳定性及其与蛋白质相互作用方式参与基因表达调控,进而参与细胞的生长发育、逆境胁迫应答等过程(钟焱等, 2019)。在水产动物中,环境通过影响DNA甲基化水平进而影响基因表达,从而使生物的生理活动及表型发生变化以适应环境(McGhee et al, 2014)。Navarro-Martin等(2011)研究发现,DNA甲基化介导了温度影响欧洲鲈鱼(Dicentrarchus labrax)性别变化的生物学过程;吴彪等(2016)研究表明,虾夷扇贝(Patinopecten yessoensis)受到急性升温胁迫处理后,基因组DNA总甲基化率下降;Li等(2017)发现,盐胁迫处理半滑舌鳎(Cynoglossus semilaevis)肝脏后,生长相关基因igf1的外显子甲基化水平与其表达水平呈负相关。在刺参应对逆境响应方面,也有研究发现,DNA甲基化在其中发挥重要的调控作用。高杉等(2017)采用甲基化敏感扩增多态性技术(MSAP)分析了健康和“化皮病”刺参体壁组织的甲基化水平差异。李尚俊等(2017)研究发现,在高温环境下,刺参的表观修饰相关基因(DNMT1、HDAC3和MLL5)的表达量发生显著变化。赵业(2015)研究发现,刺参夏眠期的甲基化水平高于非夏眠期。邹荣婕等(2014)发现,经啉类药物处理的刺参组织DNA甲基化水平低于对照组,说明DNA甲基化在刺参应对外界刺激的重要作用。
本研究采用全基因组重亚硫酸盐测序技术(WGBS),对不同温度处理的刺参的消化道组织的全基因组C位点甲基化水平进行检测,探讨温度变化对刺参基因组DNA甲基化的影响。同时,以刺参的体壁、消化道、纵肌和呼吸树为实验材料,通过甲基化酶联免疫吸附法(ELISA)检测,分析温度对刺参不同组织的甲基化程度的影响,探究刺参DNA甲基化调控与组织特异性的相关性,以期从DNA甲基化水平解释刺参响应温度变化的生理过程,为深入研究环境与表观遗传变异的关系及逆境响应机理提供依据。
1 材料与方法 1.1 实验材料实验用2龄刺参由山东省东营市华春渔业有限公司提供,体质量为(130.3±5.5) g,从养殖池塘养殖群体中随机采集,定时投喂饲料,每日换水量为1/3,养殖期间持续充气。
1.2 养殖实验与样品采集实验用刺参在水温为(20.0±0.5)℃的实验室条件下暂养5 d,暂养结束后,分组进行不同温度养殖实验,对照组(T20)刺参在(20.0±0.5)℃水温下持续养殖至取样;实验A组(T26)从水温(20.0±0.5)℃以0.6℃/d的速度在10 d内提高至(26.0±0.5)℃后,维持养殖至取样;实验B组(T32)从水温(20.0±0.5)℃以0.6℃/d的速度10 d内提高至(26.0±0.5)℃后,再以2℃/d的速度在3 d内提高至(32.0±0.5)℃,保持2 d。每个实验组设置3个平行,所有实验组刺参样品15 d养殖实验后取样,每个平行组取10个刺参,分别剖取呼吸树、纵肌、消化道和体壁组织,并用灭菌生理盐水(1.5% NaCl)冲洗,置于2 ml冻存管后迅速放入液氮罐中,送回实验室,保存于-80℃。
1.3 基因组DNA的提取DNA提取采用天根生化科技(北京)有限公司的海洋动物组织基因组DNA提取试剂盒,1%琼脂糖凝胶电泳检测DNA完整性,使用紫外分光光度计检测DNA的浓度和纯度,将各组样品的浓度调整为100~ 200 ng/μl,于–20℃保存备用。
1.4 高温处理后消化道的全基因组DNA甲基化测序重亚硫酸盐WGBS实验步骤参考郭添福等(2018)的高分辨率甲基化分析方法,进一步对样品进行DNA甲基化定量。主要构建流程包括:检测合格的刺参消化道基因组DNA样品,首先,用超声波打成平均大小为200~300 bp的片段,对打断后的DNA片段进行末端修复、腺苷酸化,并添加接头;随后,进行Bisulfite处理(EZ DNA Methylation GoldTM Kit, Zymo Research),经处理后,未发生甲基化的C变成U,而甲基化的C保持不变;最后,对单链DNA片段进行PCR扩增(KAPA HiFi HotStart Uracil + ReadyMix)得到DNA文库,库检合格后,利用Illumina HiSeq进行高通量测序,测序读长PE150,构建Bisulfite全基因组甲基化图谱,测序在北京诺禾致源有限公司完成。
1.5 ELISA测定不同组织DNA甲基化水平提取刺参纵肌、呼吸树、消化道和体壁4个组织的基因组DNA,利用Zymo Research公司的5-mC DNA ELISA试剂盒检测相应样品基因组DNA甲基化水平。具体实验步骤:待测样品基因组DNA和标准品(100 ng)均稀释成100 μl反应液,于PCR仪98℃变性5 min,冰浴10 min,移至96孔板,37℃孵育1 h,缓冲液洗板3次,37℃孵育30 min。最后,加入甲基化胞嘧啶抗体,37℃孵育1 h,洗板同上,并进行显色反应,酶标仪检测样品吸光度。
1.6 数据统计与分析测序结果的甲基化水平计算:ML=mC/(mC+ umC)。其中,ML为甲基化水平,mC和umC分别代表覆盖区域甲基化C位点和未甲基化C位点的read数目。所有实验结果均来自至少3次独立实验,结果为均值±标准差(x±SD)。运用SPSS 19.0软件对各个组的甲基化水平进行ANOVA分析和Duncan多重比较,获得两两之间的统计学差异(P < 0.05)。
ELISA测定DNA甲基化水平,通过制备阴性对照(100 ng/μl)和阳性对照(100 ng/μl)的混合物来制备已知5-mC不同百分比的(5%、10%、25%、50%、75%和100%)标准品,根据标准品吸光度值所绘制的标准曲线,即可得到各个样品的整体甲基化水平。
2 结果 2.1 不同处理下刺参消化道DNA甲基化模式分析由全基因组DNA甲基化测序(WGBS)结果(表 1)可知,刺参消化道组织的常温对照组T20基因组的甲基化位点数为5050974个,T26组的基因组甲基化位点数为5315261,T32组基因组的甲基化位点数为4714193个。在所有的处理组和对照组中,CpG(简写CG)类型甲基化位点数目为4563209~5134931,也有一定数量的甲基化胞嘧啶发生在CHH位点(116612~141870),而CHG类型甲基化位点相对较少(34372~42059)。
|
|
表 1 甲基化位点数目及类型 Tab.1 Number and type statistics of methylation sites |
T20、T26和T32温度组的全基因组总甲基化水平分别为(1.70±0.01)%、(1.79±0.11)%和(1.59±0.04%) (表 2),T26组基因组的总甲基化水平表现为上升,T32组基因组的总甲基化水平表现为下降,且低于T20组,方差分析表明,T26组的总甲基化水平和T32组存在显著差异(P < 0.05)。对不同类型的甲基化来说,C碱基甲基化水平表现出与总甲基化水平一致的趋势,T26组CG类型C碱基甲基化水平较T20升高,而T32组较T26组降低,且差异显著(P < 0.05)。在2种非CG类型的甲基化模式中,CHG类型T20组与T32组存在显著差异(P < 0.05),CHH类型中3组差异不大,且都呈现低甲基化的变异模式。大部分DNA甲基化发生在CG类型的C碱基上,CG类型的C碱基DNA甲基化程度为13.32%~14.99%,显示中等的DNA甲基化水平。
|
|
表 2 不同温度下刺参全基因组甲基化水平 Tab.2 The methylation levels of C bases of Apostichopus japonicus at different temperature (%) |
对3个温度下CG、CHH和CHG位点甲基化与总体甲基化占比情况进行统计,结果见图 1。mCG类型甲基化百分比均在96%以上,mCHH类型甲基化百分比在T20、T26和T26组样品分别为2.79%、2.42%和2.57%,mCHG类型甲基化百分比最少,均在1%以下。
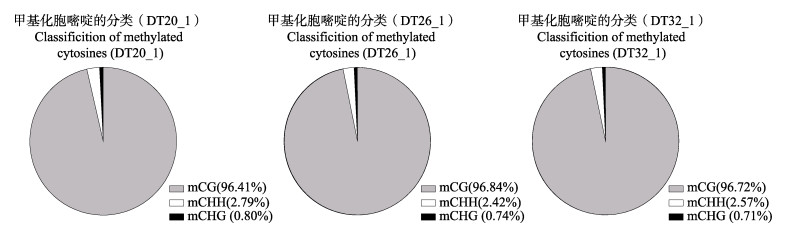
|
图 1 不同温度条件下甲基化C位点的数量及其占总甲基化C位点的比例 Fig.1 The number of methylated C sites and their proportion to the total methylated C sites in different sequence environments |
根据测得的甲基化比例,统计不同甲基化水平的甲基化位点中3种类型占各自类型的百分比可以看出(图 2),在甲基化水平为30%时,处于这一水平的CHG和CHH甲基化位点分别占本类型的30%左右,为最高点,且显著高于CG类型,之后随着位点甲基化水平的增高,其所占百分比下降;而随着甲基化位点甲基化水平的增高,CG类型的百分比总体呈明显的增高趋势,在甲基化水平为100%时,占比达到该类型的最高点。
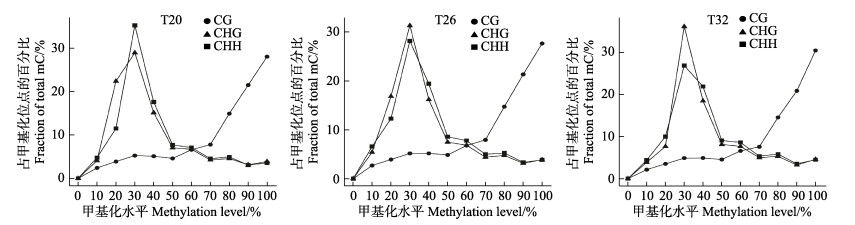
|
图 2 所有样本甲基化位点水平分布 Fig.2 The level distribution of methylation sites in all samples 图中每个数据来自3个生物学重复 Each data in the figure comes from 3 biological replicates |
采用5-mC DNA ELISA Kit试剂盒检测不同温度条件下刺参的呼吸树、消化道、纵肌和体壁4种组织的基因组的甲基化水平,并进行不同组间方差分析。数据显示,在3种不同温度下,刺参呼吸树和消化道组织的甲基化水平范围为(2.68±0.10)%~(3.29±0.06)%,均高于纵肌和体壁组织,其中,体壁的甲基化水平在(2.05±0.15)%~(2.11±0.11)%之间,纵肌的甲基化水平最低,为(1.68±0.07)%~(1.76±0.03)%。各组织的总甲基化水平从高到低依次为呼吸树 > 消化道 > 体壁 > 纵肌(图 3)。不同温度对刺参组织的甲基化水平有所改变,T26组刺参样品消化道和呼吸树组织的甲基化水平显著高于T20对照组(P < 0.05),T32组刺参样品消化道和呼吸树组织的甲基化水平又显著低于T26对照组(P < 0.05)。呼吸树和消化道组织的甲基化水平总体呈一致趋势,在温度从20℃升至26℃时,甲基化水平升高,而温度继续升至32℃时,甲基化水平下降;而纵肌和体壁组在3个温度下的甲基化水平各组间均无显著差异。

|
图 3 不同温度下5-mC甲基化试剂盒检测各组织DNA甲基化水平变化 Fig.3 Using 5-mC methylation kit to detect tissues DNA methylation levels under different experimental conditions 不同小写字母为差异显著(P < 0.05) Different letters represent significant difference (P < 0.05) |
研究结果表明,DNA甲基化参与了海洋动物环境胁迫适应性机制,诸如盐度(环朋朋, 2018)、重金属(周新文等, 2001)、温度(侯艳雯等, 2019; 王艺雅, 2015)等环境因子的逆境胁迫下,甲基化会有明显差异。Flores等(2013)提出,环境诱导影响DNA甲基化的变异,可能通过增加基因组被标记区域的突变性,改变生物的表型,使其适应自然选择。温度一直是影响水产动物生存的重要因素,在不同温度条件下,基因组内的甲基化变异能够通过改变某些特定功能基因DNA甲基化状态来影响基因的转录,进而调节生物体内胁迫响应机制等来提高动物的耐寒性或耐热性。朱华平等(2013)分析了耐寒罗非鱼(Oreochroms mossambcus)与正常组基因组DNA甲基化的差别,结果表明,低温诱导DNA总甲基化下降,同时主要发生去甲基化过程。吴彪等(2016)研究发现,虾夷扇贝受到急性升温胁迫处理后,基因组DNA总甲基化率下降。王翠丽(2019)研究发现,高温胁迫过程中,近江牡蛎(Ostrea rivularis)的核心启动子甲基化水平与Hsp90基因的表达呈负相关。孔宁(2016)研究发现,在温度、盐度胁迫下,皱纹盘鲍(Haliotis discus)的表观遗传结构发生改变,调控相关抗逆基因表达。这些研究结果均表明,温度变化可诱导水产动物基因或组织的甲基化状态发生改变。
本研究通过WGBS和ELISA对刺参消化道等组织甲基化水平进行检测发现,不同温度下刺参组织中的甲基化水平有所改变,刺参消化道组织和呼吸树组织的T26组甲基化水平最高。刺参在26℃养殖环境下处于夏眠或生理代谢活动较弱的状态,这期间刺参消化、呼吸等生理代谢活动调节减弱(Gao et al, 2008)。处于休眠状态的T26组刺参基因组甲基化水平的增高,一定程度上说明刺参这些组织中的一些功能基因受到甲基化调节而表达水平下降,相应地,减弱其参与调控的生理代谢活动来维持刺参的休眠状态。刺参消化道组织和呼吸树组织的T32组甲基化水平较T26组降低。32℃是对刺参生命活动产生危害的胁迫温度,在这个温度下,刺参会发生系列应激反应以自我保护,生理反应和活动变化意味着刺参体内需要启动一系列转录表达和调控,而甲基化水平的降低调节可能起重要作用。
3.2 刺参基因组甲基化模式DNA甲基化作为一种重要的表观遗传修饰,主要有3种类型:CG、CHG和CHH。在动物中,胞嘧啶的甲基化多发生于CpG二核苷酸序列中,CG是主要甲基化修饰类型,非CG类型的序列(CHH和CHG)在基因中十分少见(Cokus et al, 2008; Zhang et al, 2015)。脊椎动物通常存在于基因间区和富含重复序列的区域,而在无脊椎动物中,DNA甲基化主要发生于基因内部,基因间区大部分未被甲基化(Schaefer et al, 2010)。在胚胎干细胞研究(Lister et al, 2009)中,CHG和CHH型的甲基化位点分别占17.3%和7.2%,Sun等(2014)检测到栉孔扇贝(Chlamys farreri)基因组中,分别有14.9%~16.5%的CpG类型和5.1%~6.3%的CHG类型的甲基化位点。不同温度条件下的刺参消化道DNA样品经过重亚硫酸盐处理后,进行WGBS测序分析,甲基化位点类型分析显示,在所有甲基化胞嘧啶(mCs)中,有超过96%的CpGs,且CG类型的C碱基呈现中等甲基化水平(24.11%~24.48%),CHG和CHH类型的C碱基呈现低甲基化水平(0.49%~0.56%)。
在不同温度刺参甲基化模式的对比分析中,3个温度组消化道的总甲基化率和CG类型C碱基甲基化水平之间存在显著差异(P < 0.05),T26组消化道高于T20和T32组,CHG和CHH类型的C碱基甲基化水平在2组之间差异不显著(P > 0.05)。已研究的大多数无脊椎动物表现出镶嵌式的甲基化模式,无论是在无脊椎动物,还是脊椎动物,基因内CpG甲基化的功能都是保守的,它能够抑制基因内部异常转录的起始(Suzuki et al, 2007)。目前,非CG类型的甲基化与基因表达的关系尚不清楚,值得更深入的研究。
3.3 组织间的甲基化差异分析不同组织间的基因组甲基化水平的差异是生物界普遍存在的现象。在水产动物的不同组织间甲基化研究中,杜盈等(2013)利用MSAP技术分析野生组中国明对虾(Fenneropenaeus chinensis)和选育品种“黄海1号”的甲基化水平时发现,野生组中国明对虾的纵肌和鳃组织甲基化水平差异极显著(P < 0.05),而二者与血液组织的甲基化水平差异极显著(P < 0.01);“黄海1号”的鳃组织和血液组织甲基化水平相近,都极显著低于纵肌组织(P < 0.01)。罗少杰(2017)运用甲基化检测技术对马氏珠母贝(Phyllostachys mollissima)的边缘膜区、套膜区和中央膜区的基因组甲基化水平进行了研究,发现各组织甲基化水平具有显著性差异,其中,中央膜区的甲基化水平最高[(19.04±2.55)%]。这些结果显示,无脊椎动物不同组织间甲基化水平存在差异,可能是由于DNA甲基化在细胞分化以及生长发育调控中发挥重要作用(Sun et al, 2014; 何暮春等, 2018)。
果蝇(Drosophilid)、水蚤(Daphnia magna)、牡蛎(C. gigas)等部分无脊椎动物的甲基化研究发现,其基因组甲基化处于较低水平(Regev et al, 1998; Riviere et al, 2013; Hearn et al, 2019)。本研究结果表明,刺参在不同温度水体中,不同组织DNA总甲基化水平在1%~4%之间,并呈现呼吸树、消化道、体壁、纵肌的甲基化程度依次降低的趋势。由于技术原因,通过WGBS和ELISA方法测得的消化道甲基化水平存在差异,但总体属于基因组甲基化水平较低的无脊椎动物类型。在同一温度下,刺参的不同组织间甲基化水平存在明显差异,且随着温度的升高,纵肌和体壁组织甲基化水平基本不变,而呼吸树和消化道组织的甲基化水平总体趋势一致,在温度从20℃升至26℃时,甲基化水平显著升高,而在温度继续升至32℃时,甲基化水平显著下降。曹哲明等(2009)认为,DNA甲基化是基因表达调控的方式,甲基化程度高的组织相对来说基因表达水平较低。在升温过程中,消化道的基因表达程度最低,说明不同组织对温度变化的敏感性不同,这与之前的研究结果相符。刺参经历高温休眠时期,呼吸代谢和摄食效率发生改变,作为这一过程主要的功能器官——消化道和呼吸树受到的影响最大,同时会发生不同程度的退化和萎缩(Zhao et al, 2015)。
4 总结本研究对高温胁迫下刺参基因组DNA甲基化的变化情况进行了探究。相较于生活在正常水温的个体,经历不同温度处理的刺参的表观基因组发生显著性改变,使得相关基因表达或抑制。目前,动物在环境温度胁迫下,全基因组范围内的去甲基化过程和甲基化的协调机制,以及动物表型的形成和环境适应性的表观遗传机制,都值得关注。未来研究应该更多关注全基因组表观变化启动的分子机制,例如,DNA甲基化对基因表达抑制的调控作用;以及关注动物中表观遗传变异与物种表型差异、环境适应如何相互联系。表观遗传学在环境胁迫中的作用和机制尚有诸多问题需进一步探讨。
Cao XS. Study on the effect of temperature on the growth, physiology and nutritional components of Apostichopus japonicus. Masterxs Thesis of Dalian Ocean University, 2014 [曹学顺. 温度对刺参的生长、生理及营养成分的影响研究. 大连海洋大学硕士研究生学位论文, 2014]
|
Cao ZM, Yang J. Analysis of the methylation in genome DNA from different tissues of Anodonta woodiana. Ecology and Environment, 2009, 18(6): 2011-2016 [曹哲明, 杨健. 背角无齿蚌不同组织的基因组DNA甲基化分析. 生态环境学报, 2009, 18(6): 2011-2016 DOI:10.3969/j.issn.1674-5906.2009.06.001] |
Crabtree JS. Fundamentals of epigenetics. Clinical Precision Medicine. Academic Press, 2020: 27-37
|
Cokus SJ, Feng SH, Zhang XY, et al. Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning. Nature, 2008, 452(7184): 215-219 DOI:10.1038/nature06745 |
Du Y, He YY, Li J, et al. MSAP analysis of genomic DNA in different tissues of wild and "Yellow Sea No.1" Fenneropenaeus chinensis. Journal of Fishery Sciences of China, 2013, 20(3): 536-543 [杜盈, 何玉英, 李健, 等. 野生和"黄海1号"中国明对虾不同组织基因组DNA的MSAP分析. 中国水产科学, 2013, 20(3): 536-543] |
Flores KB, Wolschin F, Amdam GV. The role of methylation of DNA in environmental adaptation. Integrative and Comparative Biology, 2013, 53(2): 359-372 DOI:10.1093/icb/ict019 |
Gao F, Yang H, Xu Q, et al. Phenotypic plasticity of gut structure and function during periods of inactivity in Apostichopus japonicus. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2008, 150(3): 255-262 DOI:10.1016/j.cbpb.2008.03.011 |
Gao S, Yang AF, Dong Y, et al. MSAP analysis of DNA methylation in the body wall of Apostichopus japonicus. Acta Hydrobiologica Sinica, 2017, 41(3): 637-642 [高杉, 杨爱馥, 董颖, 等. 仿刺参"化皮病"体壁组织DNA甲基化的MSAP分析. 水生生物学报, 2017, 41(3): 637-642] |
Gao Y. Effects of variable temperature on immunity and disease resistance of Apostichopus japonicus. Hebei Fisheries, 2017(9): 1-4, 41 [高杨. 变温对仿刺参(Apostichopus japonicus)免疫力及抗病力的影响. 河北渔业, 2017(9): 1-4, 41 DOI:10.3969/j.issn.1004-6755.2017.09.001] |
Guo TF, Zhang ZY, Chen D, et al. Difference analysis of high resolution single base methylation in muscle genome of large white pigs of different genders. Acta Veterinaria et Zootechnica Sinica, 2018, 49(11): 2326-2339 [郭添福, 张志燕, 陈冬, 等. 不同性别大白猪纵肌全基因组高分辨率单碱基甲基化差异分析. 畜牧兽医学报, 2018, 49(11): 2326-2339 DOI:10.11843/j.issn.0366-6964.2018.11.004] |
He MC, Zhang JY, Liu CL, et al. DNA methylation level and pattern in growing Sepia esculenta Hoyle. Progress in Fishery Sciences, 2018, 39(4): 46-55 [何暮春, 张金勇, 刘长琳, 等. 金乌贼生长期DNA甲基化水平和模式. 渔业科学进展, 2018, 39(4): 46-55] |
Hearn J, Pearson M, Blaxter M, et al. Genome-wide methylation is modified by caloric restriction in Daphnia magna. BMC Genomics, 2019, 20(1): 197 DOI:10.1186/s12864-019-5578-4 |
Hou YW, Liu W, Jiang PL, et al. Effects of low temperature on DNA methylation of zebrafish ZF4 cells. Journal of Fishery Sciences of China, 2019, 26(2): 271-279 [侯艳雯, 刘玮, 姜蓬垒, 等. 低温对斑马鱼ZF4细胞基因组DNA甲基化水平的影响. 中国水产科学, 2019, 26(2): 271-279] |
Huan PP. Function of DNA methylation in salinity adaptation of Portunus trituberculatus. Masterxs Thesis of Shanghai Ocean University, 2018 [环朋朋. DNA甲基化在三疣梭子蟹盐度适应中的功能研究. 上海海洋大学硕士研究生学位论文, 2018]
|
Kang JT, Liang QJ, Liang C, et al. Overview on epigenetics and its progress. Science and Technology Review, 2013, 31(19): 66-74 [康静婷, 梁前进, 梁辰, 等. 表观遗传学研究进展. 科技导报, 2013, 31(19): 66-74 DOI:10.3981/j.issn.1000-7857.2013.19.011] |
Kong N. Effects of temperature and salinity on growth and development of Haliotis discus hannai "97"population. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2016 [孔宁. 温度、盐度对皱纹盘鲍"97"选群生长发育的影响. 中国科学院研究生院(海洋研究所)博士研究生学位论文, 2016]
|
Kulis M, Esteller M. DNA methylation and cancer. Advances in genetics. Academic Press, 2010, 70: 27-56
|
Li S, He F, Wen H, et al. Low salinity affects cellularity, DNA methylation, and mRNA expression of igf1 in the liver of half smooth tongue sole (Cynoglossus semilaevis). Fish Physiology and Biochemistry, 2017, 43(6): 1587-1602 DOI:10.1007/s10695-017-0395-7 |
Li SJ, Sun GH, Li XY, et al. Expression characteristics of epigenetic regulation related genes in Apostichopus japonicus under high temperature stress. Journal of Fishery Sciences of China, 2017, 24(3): 470-476 [李尚俊, 孙国华, 李雪燕, 等. 高温胁迫下仿刺参表观遗传调控相关基因的表达特征. 中国水产科学, 2017, 24(3): 470-476] |
Lister R, Pelizzola M, Dowen RH, et al. Human DNA methylomes at base resolution show widespread epigenomic differences. Nature, 2009, 462(7271): 315 DOI:10.1038/nature08514 |
Luo SJ. Study on DNA methylation difference and function in different regions of mantle of Phyllostachys mollissima. Masterxs Thesis of Guangdong Ocean University, 2017 [罗少杰. 马氏珠母贝外套膜不同区域的DNA甲基化差异及功能研究. 广东海洋大学硕士研究生学位论文, 2017]
|
McGhee KE, Bell AM. Paternal care in a fish: Epigenetics and fitness enhancing effects on off spring anxiety. Proceedings of the Royal Society B: Biological Sciences, 2014, 281(1794): 20141146 DOI:10.1098/rspb.2014.1146 |
Navarro-Martin L, Viñas J, Ribas L, et al. DNA methylation of the gonadal aromatase (cyp19a) promoter is involved in temperature-dependent sex ratio shifts in the European sea bass. PLoS Genetics, 2011, 7: e1002447 DOI:10.1371/journal.pgen.1002447 |
Regev A, Lamb MJ, Jablonka E. The role of DNA methylation in invertebrates: Developmental regulation or genome defense?. Molecular Biology and Evolution, 1998, 15(7): 880 DOI:10.1093/oxfordjournals.molbev.a025992 |
Riviere G, Wu GC, Fellous A, et al. DNA methylation is crucial for the early development in the oyster C. gigas. Marine Biotechnology, 2013, 15: 739-753 DOI:10.1007/s10126-013-9523-2 |
Schaefer M, Lyko F. Lack of evidence for DNA methylation of Invader4 retroelements in Drosophila and implications for Dnmt2-mediated epigenetic regulation. Nature Genetics, 2010, 42(11): 920-921 DOI:10.1038/ng1110-920 |
Sun Y, Hou R, Fu X, et al. Genome-wide analysis of DNA methylation in five tissues of Zhikong scallop, Chlamys farreri. PLoS One, 2014, 9(1): e86232 DOI:10.1371/journal.pone.0086232 |
Suzuki MM, Kerr ARW, de Sousa D, et al. CpG methylation is targeted to transcription units in an invertebrate genome. Genome Research, 2007, 17(5): 625-631 DOI:10.1101/gr.6163007 |
Wang CL. Effects of high temperature stress on Hsp90 expression and CpG methylation in Ostrea rivularis Gould. Guangxi Sciences, 2019, 26(4): 424-429 [王翠丽. 高温胁迫对近江牡蛎Hsp90表达及CpG甲基化的影响. 广西科学, 2019, 26(4): 424-429 DOI:10.3969/j.issn.1005-9164.2019.04.011] |
Wang YY. Effects of high temperature on the methylation level of Oreochroms mossambcus genome. Masterxs Thesis of Shandong Agricultural University, 2015 [王艺雅. 高温诱导对罗非鱼全基因组甲基化水平的影响研究. 山东农业大学硕士研究生学位论文, 2015]
|
Wu B, Yang AG, Sun XJ, et al. Effects of acute temperature stress on genomic DNA methylation of Patinopecten yessoensis. Progress in Fishery Sciences, 2016, 37(5): 140-146 [吴彪, 杨爱国, 孙秀俊, 等. 急性温度胁迫对虾夷扇贝(Patinopecten yessoensis)基因组DNA甲基化的影响. 渔业科学进展, 2016, 37(5): 140-146] |
Xie ZW, Wang SK, Lin X, et al. Effects of high temperature stress on antioxidant enzymes of Apostichopus japonicus. Fisheries Science, 2016, 35(4): 327-333 [谢兆文, 王寿昆, 林旋, 等. 高温胁迫对仿刺参抗氧化酶等指标的影响. 水产科学, 2016, 35(4): 327-333] |
Zhang Z, Liu G, Zhou Y, et al. Genome-wide and single-base resolution DNA methylomes of the sea lamprey (Petromyzon marinus) reveal gradual transition of the genomic methylation pattern in early vertebrates. BioRxiv, 2015: 033233
|
Zhao Y. Study on gene expression patterns and basic characteristics of DNA methylation in Apostichopus japonicus (Selenka) during aestivation. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2015 [赵业. 刺参Apostichopus japonicas (Selenka)夏眠期间基因表达模式及DNA甲基化基础特征研究. 中国科学院研究生院(海洋研究所)博士研究生学位论文, 2015]
|
Zhao Y, Chen MY, Storey KB, et al. DNA methylation levels analysis in four tissues of sea cucumber Apostichopus japonicus based on fluorescence-labeled methylation-sensitive amplified polymorphism (F-MSAP) during aestivation. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2015, 181: 26-32 DOI:10.1016/j.cbpb.2014.11.001 |
Zhong Y, Xu H, Peng FL. Significance and research progress of DNA methylation in gene expression regulation. China Medical Herald, 2019, 16(14): 33-36 [钟焱, 徐慧, 彭凤兰. DNA甲基化在基因表达调控中的意义及研究进展. 中国医药导报, 2019, 16(14): 33-36] |
Zhou XW, Zhu GN, Jilisa M, et al. Influence of Cu, Zn, Pb, Cd and their heavy metal ion mixture on the DNA methylation level of Carassius auratus. China Environmental Science, 2001, 21(6): 549-552 [周新文, 朱国念, Jilisa M, 等. Cu、Zn、Pb、Cd及其混合重金属离子对鲫鱼(Carassius auratus) DNA甲基化水平的影响. 中国环境科学, 2001, 21(6): 549-552 DOI:10.3321/j.issn:1000-6923.2001.06.017] |
Zhu HP, Lu MX, Huang ZH, et al. Effects of low temperature on DNA methylation of Oreochroms mossambcus. Journal of Fisheries of China, 2013, 37(10): 1460-1467 [朱华平, 卢迈新, 黄樟翰, 等. 低温对罗非鱼基因组DNA甲基化的影响. 水产学报, 2013, 37(10): 1460-1467] |
Zou RJ, Jiang XY, Xu YJ, et al. Analysis of global DNA methylation impacted by quinoxaline in Apostichopus japonicus tissue using high performance liquid chromatography. Journal of Instrumental Analysis, 2014, 33(7): 780-785 [邹荣婕, 姜向阳, 徐英江, 等. 基于高效液相色谱的喹(噁)啉类药物对刺参DNA甲基化水平的影响研究. 分析测试学报, 2014, 33(7): 780-785 DOI:10.3969/j.issn.1004-4957.2014.07.007] |



