2. 临沂大学生命科学学院 山东 临沂 276000
2. College of Life Sciences, Linyi University, Linyi, Shandong 276005, China
虫草属(Cordyceps)真菌是一大类昆虫病原真菌,分布于不同的地理区域,其在不同生长时期都能侵染节肢动物和昆虫(Sung et al, 2007)。据估计,全球共有蛹虫草(Cordyceps militaris)、中华虫草(C. sinensis)、细虫草(C. gracilis)等500余种虫草属真菌,寄主范围广泛且多样性丰富(Zheng et al, 2011)。近几十年里,对虫草属物种进行了大量研究,涉及到虫草种类的区域探索、人工培育、微生物群落组成和活性成分的代谢机制等(Shrestha et al, 2005; Lee et al, 2010; Kramer et al, 2017; Xia et al, 2019)。一些变形虫草物种,如拟青霉属(Paecilomyces)、绿僵菌属(Metarhizium)和白僵菌属(Beauveria)在昆虫的生物防治中也发挥了重要作用(Zimmermann, 1993; Meyling et al, 2007)。Cordyceps confragosa是虫草属的一种,具有广泛地理分布,其变形阶段蜡蚧轮枝菌(Verticillium lecanii)在农业实践中被用作昆虫的生物防治剂(Doug et al, 2012)。实验室在新疆昌吉地区某养殖场内,从患病河鲈(Perca fluviavilis)病灶处分离纯化到1株C.confragosa菌株CHL02。经柯赫氏法则验证,CHL02菌株为患病河鲈的病原菌,ITS rDNA分子方法鉴定为C. confragosa。该菌使健康河鲈出现游动迟缓、食欲不振等症状,在河鲈体表形成溃疡,覆有白色微黄的菌斑,直至河鲈衰歇死亡(魏冬梅, 2018)。
全基因组测序(whole genome sequencing, WGS)已被广泛应用于流行病学、疫苗开发、微生物进化等领域,为微生物特异性生物学的研究提供分子生物学基础,如致病机制、共生机制、独特的代谢机制等,还为病原-宿主相互作用的发病机制提供深刻见解(Yu et al, 2017; Rantsiou et al, 2017)。迄今为止,一些虫草种类已经被测序并发表在Ensembl真菌数据库中,包括蛹虫草、布氏虫草(C. brongniartii)、广东虫草(C. guangdongensis) (Zhang et al, 2018)等,以便探索虫草属的种间进化关系及其生物活性物质的生物合成机制和代谢途径(Vongsangnak et al, 2017)。2016年,Shang等(2016)报道了昆虫源C. confragosa RCEF 1005的全基因组测序,并结合其他动植物病原真菌探究真菌致病性的趋同进化。
本研究首次报道了河鲈源C. confragosa CHL02的全基因组测序,并进行了比较基因组分析。基于组装水平,通过基因预测获得C.confragosa基因组组成,利用一系列功能数据库进行基因功能注释、次级代谢产物基因簇预测,并进行病原宿主互作以及比较基因组学分析,旨在为C. confragosa的基因组成和功能研究奠定一定基础,也为了解该病原菌感染河鲈的发生机制提供理论依据。
1 材料与方法 1.1 材料与试剂C.confragosa CHL02菌株来自实验室前期保存菌株(魏冬梅, 2018),用于全基因组测序分析。沙堡氏液体(蛋白胨10 g/L、葡萄糖40 g/L)培养基,马铃薯葡萄糖琼脂(PDA)培养基,购于青岛海博生物技术公司;抗生素青霉素和链霉素购于北京索莱宝科技有限公司。
1.2 菌株培养及DNA提取从实验室低温(4℃)保藏的PDA斜面培养基上刮取适量待测菌株,接种于200 mL沙堡氏液体培养基,25℃培养72 h,离心收集菌体。采用SDS法提取基因组DNA,所得DNA经1%琼脂糖凝胶电泳检测,QubitⓇ2.0荧光仪(Thermo Scientific)定量,检测合格的DNA样品送北京康普森生物公司进行后续测序分析。
1.3 基因组测序与组装随机打断DNA样品,构建小片段测序文库(350 bp),基于Illumina PE150平台进行双末端测序。对测序得到的原始数据进行低质量过滤处理,分别去除超过40 bp低质量碱基(质量值≤38)、N碱基达到10 bp、与连接物之间overlap超过15 bp以及样品宿主的reads,然后去除duplication污染,得到有效数据,进行碱基含量和质量分布分析。基因组组装前,采用K-mer统计分析来估计基因组大小,用SOAP de novo (V.2.04)软件组装有效数据(Li et al, 2008),并使用CISA软件进行整合(Lin et al, 2013)。原始reads与组装好的基因组序列进行比对,通过统计组装序列的GC含量和reads覆盖深度,总结基因组的GC偏向性和重复序列情况,判断组装结果是否正常。
1.4 基因组组分分析通过基因预测软件Augustus (V.2.7)(Stanke et al, 2008)对组装结果进行基因de novo预测,同时,运用GeneMark软件(V.4.17) (Besemer et al, 2001)进行比较,获得基因组蛋白编码基因,用于后续的基因功能注释分析。通过TRF (Tandem Repeats Finder, V.4.07)和RepeatMasker (V.4.0.5)软件分别预测串联重复序列和转座子TE (Benson, 1999; Saha et al, 2008);采用tRNAscan-SE (V.1.3.1)和rRNAmmer软件分别进行tRNA和rRNA的预测(Lowe et al, 1997; Lagesen et al, 2007)。
1.5 基因功能注释将CHL02菌株的基因序列分别与已知的各功能数据库进行比对(e-value≤1×10-5),对于每一条序列的比对结果,选取score最高的比对结果(identity≥40%,coverage≥40%)进行基因功能注释。主要参考Nr (non-redundant protein database,非冗余的蛋白质数据库)(Li et al, 2002)、GO (gene ontology,基因本体论)(Ashburner et al, 2000)、KOG (cluster of orthologous groups of proteins, 蛋白相邻类的聚簇)(Tatusov et al, 2000)和KEGG (Kyoto encyclopedia of genes and genomes,京都基因和基因组百科全书)(Kanehisa et al, 2000)功能数据库。
1.6 专有数据库注释通过碳水化合物相关酶数据库CAZy (carbohydrate- active enzymes database)进行碳水化合物酶分类注释(Cantarel et al, 2000);采用antiSMASH软件(v.2.0.2)预测次级代谢产物基因簇(Medema et al, 2011);PHI (pathogen host interaction, 病原与宿主互作数据库)进行相关致病基因的注释分析(Martin et al, 2015)。
1.7 比较基因组分析从GenBank数据库中获得参考菌株蛹虫草(C. militaris CM01)、粗糙虫草菌(C. confragosa RCEF 1005)、文森虫草菌(C. fumosorosea)、爪哇虫草菌(C. javanica)和球孢白僵菌(Beauveria bassiana)的基因组信息,与菌株CHL02的基因组序列直接进行比较分析,统计其基本特征。利用OrthoVenn2在线工具(https://orthovenn2.bioinfotoolkits.net/task/create)对测序和参考菌株基因组的蛋白序列进行直系同源聚类分析(默认参数:e-value≤1×10-5,inflation value≤1.5) (Wang et al, 2010),通过CVTree3在线工具(http://tlife.fudan.edu.cn/cvtree/cvtree/)对这些菌株的全基因组构建组分矢量(氨基酸短串K值取6),利用neighbor- joining系统发育树进行系统发育分析(Wang et al, 2010)。
2 结果与分析 2.1 基因组数据概况基于Illumina PE150测序平台,CHL02菌株获得原始数据6.982 Gb。过滤低质量的序列片段后,得到6.188 Gb的有效数据,有效数据的GC含量为52.96%,Q20和Q30值分别为97.51%和93.25%,测序质量较好。有效数据的碱基分析图显示(图 1),碱基A与T曲线重合,G与C曲线重合,碱基所在reads位置上的平均错误率百分比低,说明碱基组成平衡,质量良好。

|
图 1 碱基含量(a)和质量分析(b) Fig.1 Analysis of base content(a) and quality(b) |
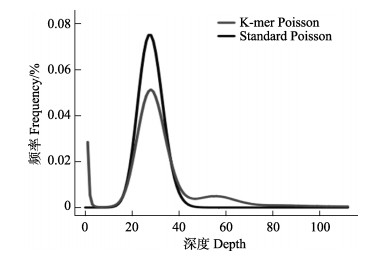
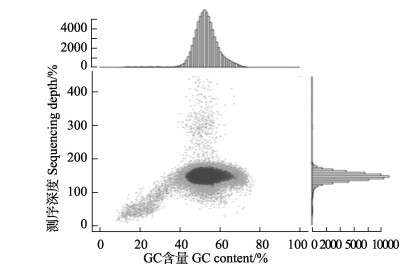
组装前,通过K-mer分析判断样品的基因组大小、杂合情况和重复序列信息,如图 2所示,15-mer统计图显示只有1个主峰出现,且主峰后面无明显的拖尾现象,表明基因组无明显的重复序列,CHL02基因组大小约38.09 Mb,覆盖深度为27.33 X。通过组装构建,获得521个contig,407个scaffold,组装后的基因组大小为36.17 Mb,GC含量为53.09% (GenBank登录号: WHIX00000000.1)。GC-Depth分析显示,菌株呈泊松分布,GC无明显偏向性,在GC含量为20%~30%处存在1处散点区域,可能是受线粒体DNA影响(图 3)。
|
图 2 15-mer分析统计分布 Fig.2 15-mer depth-frequency distribution |
|
图 3 GC含量与测序深度关联分布 Fig.3 Distribution of GC content and sequence depth |
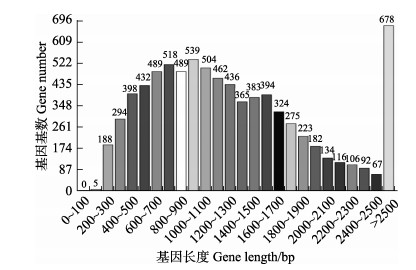
通过不同软件预测,获得CHL02菌株的基因组组分。预测到8093个编码基因,基因长度分布集中在400~1700 bp及大于2500 bp,其中,大于2500 bp的基因分布最多,达到678个,约占编码基因的8.4% (图 4)。还预测到1618个TEs (1140个逆转录转座子、461个DNA转座子以及17个未知TEs)、4571个串联重复序列、114个tRNA、25个rRNA、2个sRNA以及16个snRNA。
|
图 4 C. confragosa CHL02基因长度分布 Fig.4 Gene length distribution of C. confragosa CHL02 |
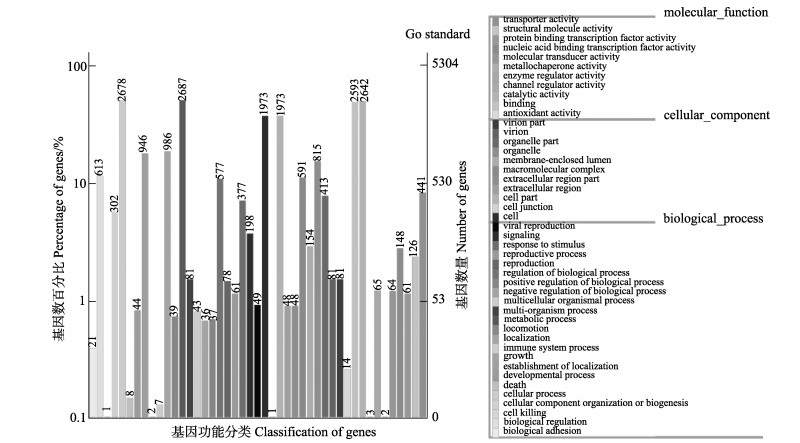
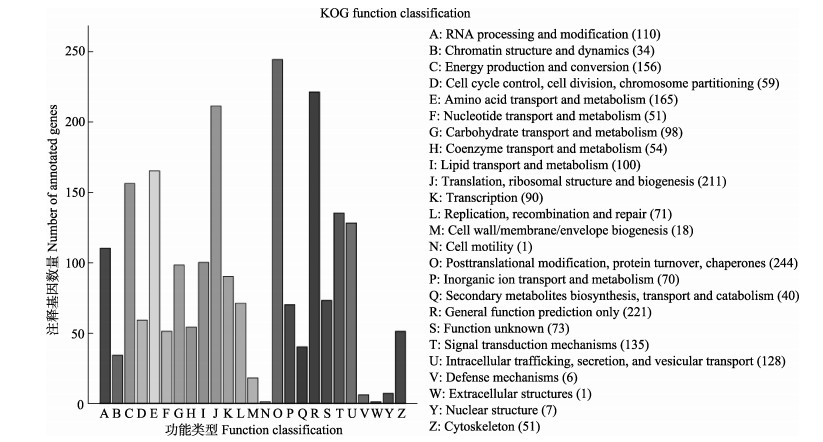
Nr库基因功能注释显示,8093个编码基因中,有7724个匹配到已知功能蛋白,与CHL02菌株最相关的物种为C. confragosa。共有5303个基因获得GO功能注释(图 5),包括3个功能方面的内容,分子功能主要与催化活性(catalytic activity)和结合(binding)有关,细胞组分主要与细胞(cell)和细胞部分(cell part)有关,而生物过程主要与代谢过程(metabolic process)和细胞进程(cellular process)有关。1985个基因成功注释到KOG数据库(图 6),其中,翻译后修饰,蛋白质转换,伴侣(posttranslational modification, protein turnover, chaperones, O)、一般功能预测(general function prediction only, R)、翻译核糖体结构和生物合成(translation, ribosomal structure and biogenesis, J)占据比例较大。KEGG富集分析(图 7)显示,预测的5057个编码蛋白共涉及378条代谢途径,主要集中在萜类化合物和聚酮类化合物的代谢、碳水化合物代谢、氨基酸代谢、运输和分解代谢以及转录途径中。
|
图 5 C. confragosa CHL02蛋白质GO功能聚类分析 Fig.5 GO cluster analysis of C. confragosa CHL02 proteins |
|
图 6 C. confragosa CHL02蛋白质KOG聚类分析 Fig.6 KOG cluster analysis of C. confragosa CHL02 A:RNA加工和修饰;B:染色质结构和动力学;C:能量生产与转换;D:细胞周期调控,细胞分裂,染色体划分;E:氨基酸运输与代谢;F:核苷酸转运和代谢;G:碳水化合物转移和代谢;H:辅酶转运和代谢;I:脂质运输和代谢;J:翻译,核糖体结构和生物起源;K:转录;L:复制,重组和修复;M:细胞壁/膜/被膜生物合成;N:细胞运动;O:翻译后修饰,蛋白质转换,分子伴侣;P:无机离子运输和代谢;Q:次级代谢产物的生物合成,转运和代谢;R:一般功能预测;S:功能未知;T:信号转导机制;U:胞内运输,分泌和囊泡运输;V:防御机制;W:胞外结构;Y:核心结构;Z:细胞骨架 |

|
图 7 C. confragosa CHL02 KEGG代谢通路分类 Fig.7 KEGG classification of metabolic pathways map of C. confragosa CHL02 |
CAZy是能催化碳水化合物降解、修饰以及生物合成的相关酶系家族。共预测到392个潜在的编码碳水化合物活性酶相关的基因,分布于不同的酶家族,占编码基因的4.84%。其中含量最高的是糖苷水解酶家族(glycoside hydrolase, GH) (226),占全部碳水化合物酶的57.65%,其余依次为糖苷转移酶(glycosyl transferases, GT) (87)、碳水化合物结合模块(carbohydrate-binding modules, CBM) (51)、氧化还原酶(auxiliary activities, AA) (34)、碳水化合物酯酶(carbohydrate esterases, CE) (22)以及多糖裂解酶家族(polysaccharide lyases, PL) (3),所占比例分别为22.19%、13.01%、8.67%、5.61%和0.76%。识别数目最多是GH家族的第3大组群GH18,为25个,它含有许多真菌的几丁质酶,主要负责和其他的降解酶共同改造和回收真菌自身细胞壁。
2.6 次级代谢产物合成基因簇分析次级代谢产物的编码基因通常成簇存在于基因组中,编码具有多种功能的复合酶。共预测到22个次级代谢产物合成基因簇,涉及到萜烯类(terpene)、Type 1聚酮合酶(t1PKS)、非核糖体肽合成酶(NRPS)、t1PKS-NRPK、羊毛硫肽类化合物(lantipeptide)、Terpene-NRPS以及其他化合物的合成,表明CHL02菌株具有合成多种化合物的能力。其中,涉及到NRPS合成以及未知化合物的基因簇数目最多都为6个。与已知次级代谢产物进行Blast比对发现,CHL02菌株通过部分NRPS途径、t1PKS-NRPS混合代谢途径以及t1pks途径可以产生白僵菌素(beauvericin)、镰刀菌素(fusarin)及胞外嗜铁素(epichloenin)等。
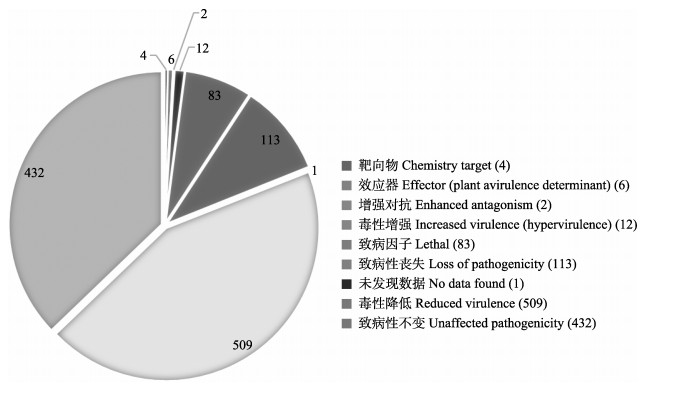
2.7 病原宿主互作相关基因病原宿主互作基因数据库,包括微生物致病菌对不同类寄主的致病相关基因,对寻找药物干预的靶基因研究有重要作用。通过基因注释,菌株CHL02共有1162个PHI相关基因,主要分布在毒性降低(reduced virulence)、致病性不变(unaffected pathogenicity)、致病性丧失(loss of pathogenicity)、混合功能(mixed outcome)、致病因子(lethal)、毒性增强(increased virulence (hypervirulence)、效应器(effector) (plant avirulence determinant)和靶向物(chemistry target)等模块中(图 8)。大部分的基因表达减弱导致病原菌对相应寄主致病能力降低,并在病原菌与寄主互作过程中发挥不同的作用,包括防御机制、转录因子、碳水化合物运输和代谢、细胞内转运、无机离子运输和代谢、分泌和膜泡运输等。83个致病因子基因主要来自于禾谷镰刀菌(Fusarium graminearum)、烟曲霉菌(Aspergillus fumigatus)以及球孢白僵菌,尤其是球孢白僵菌中几丁质酶基因BbCHIT1过表达导致致病性增强。与毒性增强相关的致病基因Ops3和Ohmm基因也源自球孢白僵菌,分别具有转录因子和跨膜蛋白作用。而效应器中的Blys2和Blys5基因,同样存在于球孢白僵菌中,作为效应分子介导真菌-昆虫的相互作用。
|
图 8 C. confragosa CHL02在PHI数据库中的匹配结果 Fig.8 The match result of C. confragosa in PHI database |
CHL02菌株与5株参考菌株的基因组信息进行直接比较分析,其基本特征见表 1。结果显示,CHL02菌株组装的Scaffold数目仅次于文森虫草菌,远多于其他参考菌株,可能是因为存在测序gaps;基因组最大,与其较接近的是昆虫源粗糙虫草菌RCEF 1005;CHL02菌株的GC含量为53.09%,高于参考菌株蛹虫草CM01、爪哇虫草菌和球孢白僵菌;编码蛋白基因数量最少,虽然具有虫草菌的基本基因特征,但在进化上可能存在一定差异。
|
|
表 1 C. confragosa CHL02与参考菌株的全基因组特征比较 Tab.1 Comparison of genomic features of the C. confragosa CHL02 genome and with reference strains |
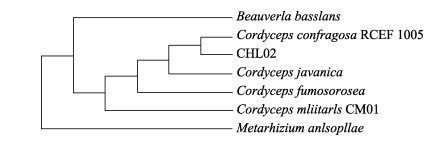
OrthoVenn2直系同源聚类分析发现,共得到10, 829个基因clusters,5309个直系同源基因clusters(至少含有2个物种)以及5520个单拷贝基因clusters。6株菌的共有基因簇(核心基因组)有5742个,占总基因的53.02%,CHL02菌株的特有基因簇最少,为7个,而其他参考菌株的特有基因簇分别是球孢白僵菌51个,文森虫草菌24个,粗糙虫草菌RCEF 1005 23个,爪哇虫草菌18个,蛹虫草CM01 12个。此外,CHL02菌株与粗糙虫草菌RCEF 1005的共有基因簇最多,为7679个,基因组成对比较相似矩阵热图也显示二者具有较高的聚类(图 9)。系统发育树(图 10)显示,CHL02菌株与粗糙虫草菌RCEF 1005的遗传距离最近,其次是爪哇虫草菌和文森虫草菌。

|
图 9 菌株直系同源聚类分析 Fig.9 Cluster analysis of strains direct homology a为菌株间基因集合的韦恩图,b为各菌株对应的基因数目,c为成对比较热图 Fig.a is the Venn diagram of gene clusters among strains, Fig.b is the number of gene clusters corresponding to each strain, and Fig.c is the pairwise heatmap |
|
图 10 菌株CVTree系统进化分析树 Fig.10 Phylogenetic analysis tree of strain by CVTree |
生物信息学和基因组测序的发展,有利于理解微生物的多样性、进化以及物种间相互作用等(吴明林等, 2018; 吴欢欢等, 2019)。全基因组测序技术已广泛用于动植物病原菌的鉴定与分析,可在分子水平上系统研究致病菌的遗传进化信息、致病机制和与寄主的互作机制等,为病原菌的鉴别分类、耐药性的检测、防病策略的制定及疫苗开发提供有价值的参考数据(孙静等, 2019; Yu et al, 2017)。除实验室前期从新疆昌吉地区某养殖场内患病河鲈分离得到的病原菌C.confragosa CHL02分离株外,未见其他鱼源C. confragosa的相关报道。因此,本研究旨在通过全基因组测序和比较基因组分析,了解河鲈源C. confragosa CHL02菌株的基因组序列信息,为深入研究其致病机制提供一定的理论基础。
CHL02菌株基因组测序结果显示,组装后的基因组大小为36.17 Mb,预测到8093个编码基因,有7724个匹配到已知功能蛋白,涉及调控、转运、环境适应和次级代谢活动等。GO注释表明CHL02菌株的蛋白功能主要集中在生物学过程,参与大量的代谢过程,以及对自身或外界环境的催化活性,为蛋白质的分泌过程和功能发挥奠定分子基础。KOG分类具有局限性,有很多基因没有在KOG里分类或功能未知,更深层次的基因功能信息还有待进一步研究。KEGG注释表明,菌株具有丰富完整的代谢途径,使生物体不断进行物质和能量交换,保持它们自身活动需要物质和对外界环境及时做出反应,增强病原菌的环境适应性。涉及菌株次生代谢产物生物合成的相关基因也比较丰富,这些基因代谢途径与基因在细胞内发挥的作用有着密切关系。
CHL02菌株的逆转录转座子包含一些常见的转座子亚群Gypsy、R1、Copia、Jockey、ERV1、Pao和DIRS等,这些亚群也是真菌中比较丰富的转座子类型(Kempken et al, 1998)。DNA转座子中Hat、CMC-EnSpm、TcMar和MULE-MuDR在真菌中较为丰富,而Maverick、Novosib、PIF-Harb和piggyBac的分布较为罕见,仅存在于少数真菌类群中,P、Ginger和Merlin转座子也仅在少数物种中被识别(Muszewska et al, 2017)。转座子具有影响基因编码能力、破坏基因功能的作用,对CHL02菌株转座子分类注释有助于研究C. confragosa基因组的大小和进化(Wessler, 2006),也为发现与其致病性及进化相关基因研究提供了思路(McCue et al, 2012)。
CHL02菌株CAZymes基因的分布与蛹虫草不同,表示虫草属菌不同种在基因组水平上存在进化差异和环境适应性,可为进一步研究CHL02菌株的遗传和分子机制奠定基础(Zheng et al, 2011)。随着水产养殖业越来越多地使用豆粕、各种谷类等植物源性饲料,鱼类更易受到真菌感染(Matejova et al, 2016)。次级代谢产物合成基因簇预测结果显示,预测的22个次级代谢产物合成基因簇涉及多种化合物合成途径,其中,PKS、NRPS和PKS-NRPS混合代谢途径是大多数真菌毒素合成的关键代谢途径,CHL02菌株能够合成白僵菌素和镰刀菌素等真菌毒素。KEGG富集分析中有868个基因参与了CHL02菌株的萜类和聚酮类化合物的代谢,而萜类化合物的代谢过程中也会产生多种真菌毒素(Yun et al, 2015)。此外,存在6个未知化合物的基因簇,可为后续发掘未知的具有特殊作用化合物的研究奠定基础。几乎所有与疾病相关的基因都在KEGG代谢通路中被注释,这些基因在病原体与宿主的相互作用中发挥着不同的作用,如防御机制、复制、重组与修复、信号转导、碳水化合物运输与代谢、无机离子运输与代谢等。CHL02菌株和其他病原菌一样,通过释放致病因子(胞外酶、激素和毒素)等来完成对寄主的侵染,而CoRAS1基因、转录因子、泛素蛋白、蛋白磷酸酶、蛋白激酶直接或间接参与致病过程(柳凤等, 2018),这将有助于进一步研究CHL02菌株的发病机制。
Nr库同源注释发现,CHL02菌株与粗糙虫草菌 RCEF 1005具有很高的同源性(91.6%),基因组直接比较发现,二者在基因组组装和编码蛋白上具有一定的差异,这可能是因为相同物种的不同来源分离株的进化和环境适应性不同。同样,与参考菌株基因组相比,CHL02菌株的基因组越大,其包含的遗传信息中涉及代谢相关基因和压力耐受基因的可能性就越大,这为适应不同的生存环境提供了特有的基因组代谢机制。基因聚类分析和系统发育树均显示,CHL02菌株与粗糙虫草菌RCEF 1005具有很高的同源性,亲缘关系较近,但病原宿主互作分析结果显示,CHL02菌株的致病基因BbCHIT1、Ops3、Ohmm、Blys2和Blys5同样存在于球孢白僵菌而不是粗糙虫草菌RCEF 1005,所涉及的致病机制还有待进一步研究。
综上所述,本研究通过全基因组测序和比较基因组分析,初步掌握了河鲈源致病菌C. confragosa CHL02菌株基因组的整体分布、基因组成、蛋白功能、致病因子和进化地位等,为后续该菌株侵染河鲈的作用机制和防治等相关研究提供了分子水平上的参考依据。
ASHBURNER M, BALL C A, BLAKE J A, et al. Gene ontology: Tool for the unification of biology. Nature Genetics, 2000, 25(1): 25-29 DOI:10.1038/75556 |
BENSON G. Tandem repeats finder: A program to analyze DNA sequences. Nucleic Acids Research, 1999, 27(2): 573-580 DOI:10.1093/nar/27.2.573 |
BESEMER J, LAMSADZE A, BORODOVSKY M. GeneMarkS: A self- training method for prediction of gene starts in microbial genomes: Implications for finding sequence motifs in regulatory regions. Nucleic Acids Research, 2001, 29(12): 2607-2618 DOI:10.1093/nar/29.12.2607 |
CANTAREL B L, COUTINHO P M, RANCUREL C, et al. The carbohydrate-active enzymes database (CAZy): An expert resource for glycogenomics. Nucleic Acids Research, 2009, 37(suppl 1): D233-D238 |
DOUG J, SKILLMAN J, VANDERMEER J. Indirect biological control of the coffee leaf rust, Hemileia vastatrix, by the entomogenous fungus Lecanicillium lecanii in a complex coffee agroecosystem. Biological Control, 2012, 61(1): 89-97 DOI:10.1016/j.biocontrol.2012.01.004 |
KANEHISA M, GOTO S, KAWASHIMA S, et al. The KEGG resource for deciphering the genome. Nucleic Acids Research, 2004, 32(suppl 1): D277-D280 |
KEMPKEN F, ULRICH K. Transposons in filamentous fungi: Facts and perspectives. BioEssays, 1998, 20(8): 652-659 DOI:10.1002/(SICI)1521-1878(199808)20:8<652::AID-BIES8>3.0.CO;2-K |
KRAMER GJ, NODWELL JR. Chromosome level assembly and secondary metabolite potential of the parasitic fungus Cordyceps militaris. BMC Genomics, 2017, 18(1): 912 DOI:10.1186/s12864-017-4307-0 |
LAGESEN K, HALLIN P, RØDL E A, et al. RNAmmer: Consistent and rapid annotation of ribosomal RNA genes. Nucleic Acids Research, 2007, 35(9): 3100-3108 DOI:10.1093/nar/gkm160 |
LEE J O, SHRESTHA B, SUNG G H, et al. Cultural characteristics and fruiting body production in Cordyceps bassiana. Mycobiology, 2010, 38(2): 118-121 DOI:10.4489/MYCO.2010.38.2.118 |
LI R Q, LI Y R, KRISTIANSEN K, et al. SOAP: Short oligonucleotide alignment program. Bioinformatics, 2008, 24(5): 713-714 DOI:10.1093/bioinformatics/btn025 |
LI W, JAROSZEWSKI L, GODZIK A. Tolerating some redundancy significantly speeds up clustering of large protein databases. Bioinformatics, 2002, 18(1): 77-82 DOI:10.1093/bioinformatics/18.1.77 |
LIN S H, LIAO Y C. CISA: Contig integrator for sequence assembly of bacterial genomes. PLoS One, 2013, 8(3): e60843 DOI:10.1371/journal.pone.0060843 |
LIU F, OU X C, ZHAN R L. Complete genome sequencing of Xanthomonas citri pv. mangiferaeindicae XC01 in mango. Journal of Fruit Science, 2018, 35(10): 86-94 [柳凤, 欧雄常, 詹儒林. 杧果细菌性角斑病病原菌XC01菌株的全基因组测序及序列分析. 果树学报, 2018, 35(10): 86-94] |
LOWE T M, EDDY S R. tRNAscan-SE: A program for improved detection of transfer RNA genes in genomic sequence. Nucleic Acids Research, 1997, 25(5): 955-964 DOI:10.1093/nar/25.5.955 |
MARTIN U, RASHMI P, ARATHI R, et al. The pathogen-host interactions database (PHI-base): Additions and future developments. Nucleic Acids Research, 2015, 43: 645-655 DOI:10.1093/nar/gku1287 |
MATEJOVA I, SVOBODOVA Z, VAKULA J, et al. Impact of mycotoxins on aquaculture fish species: A review. Journal of the World Aquaculture Society, 2016, 48(2): 186-200 |
MCCUE A D, SLOTKIN R K. Transposable element small RNAs as regulators of gene expression. Trends in Genetics, 2012, 28(12): 616-623 DOI:10.1016/j.tig.2012.09.001 |
MEDEMA M H, BLIN K, CIMERMANCIC P, et al. AntiSMASH: Rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research, 2011, 39(suppl 2): W339-W346 |
MEYLING N V, EILENBERG J. Ecology of the entomopathogenic fungi Beauveria bassiana and Metarhizium anisopliae in temperate agroecosystems: Potential for conservation biological control. Biological Control, 2007, 43(2): 145-155 DOI:10.1016/j.biocontrol.2007.07.007 |
MUSZEWSKA A, STEPNIEWSKA-DZIUBINSKA M, GINALSKI K. Cut-and-paste transposons in fungi with diverse lifestyles. Genome Biology and Evolution, 2017, 9(12): 3463-3477 DOI:10.1093/gbe/evx261 |
RANTSIOU K, KATHARIOU S, WINKLER A, et al. Next generation microbiological risk assessment opportunities of whole genome sequencing (WGS) for foodborne pathogen surveillance, source tracking and risk assessment. International Journal of Food Microbiology, 2017, 287: 3-9 |
SAHA S, BRIDGES S, MAGBANUA Z V, et al. Empirical comparison of ab initio repeat finding programs. Nucleic Acids Research, 2008, 36(7): 2284-2294 DOI:10.1093/nar/gkn064 |
SHANG Y, XIAO G, PENG Z, et al. Divergent and convergent evolution of fungal pathogenicity. Genome Biology and Evolution, 2016, 8(5): 1374-1387 DOI:10.1093/gbe/evw082 |
SHRESTHA B, SUNG J M. Notes on cordyceps species collected from the central region of Nepal. Mycobiology, 2005, 33(4): 235-239 DOI:10.4489/MYCO.2005.33.4.235 |
STANKE M, DIEKHANS M, BAERTSCH R D. Using native and syntenically mapped cDNA alignments to improve de novo gene finding. Bioinformatics, 2008, 24(5): 637-644 DOI:10.1093/bioinformatics/btn013 |
SUN J, WAN X Y, YANG Q, et al. Case studies: Pathogenic agent and microbiome analysis for zoea of Litopenaeus vannamei suffering from an unknown disease. Progress in Fishery Sciences, 2019, 40(5): 134-144 [孙静, 万晓媛, 杨倩, 等. 病例研究: 未知病因的凡纳滨对虾溞状幼体的病原和微生物组分析. 渔业科学进展, 2019, 40(5): 134-144] |
SUNG G H, HYWEL-JONES N L, SUNG J M, et al. Phylogenetic classification of Cordyceps and the clavicipitaceous fungi. Studies in Mycology, 2007, 57(1): 5-59 |
TATUSOV R L, FEDOROVA N D, JACKSON J D, et al. The COG database: An updated version includes eukaryotes. BMC Bioinformatics, 2003, 4(1): 41-54 DOI:10.1186/1471-2105-4-41 |
VONGSANGNAK W, RAETHONG N, MUJCHARIYAKUL W, et al. Genome-scale metabolic network of Cordyceps militaris useful for comparative analysis of entomopathogenic fungi. Gene, 2017, 626: 132-139 DOI:10.1016/j.gene.2017.05.027 |
WANG H, XU Z, GAO L, et al. A fungal phylogeny based on 82 complete genomes using the composition vector method. BMC Evolutionary Biology, 2009, 9(1): 195 DOI:10.1186/1471-2148-9-195 |
WANG Y, COLEMAN-DERR D, CHEN G, et al. OrthoVenn: A web server for genome wide comparison and annotation of orthologous clusters across multiple species. Nucleic Acids Research, 2015, 43(W1): 78-84 DOI:10.1093/nar/gkv487 |
WEI D M. The research progress on the diversity of fish saprolegniasis pathogens. Masterxs Thesis of Xinjiang University, 2018, 1-113 [魏冬梅. 新疆鱼类水霉病病原菌多样性研究. 新疆大学硕士研究生学位论文, 2018, 1-113] |
WESSLER S R. Transposable elements and the evolution of eukaryotic genomes. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(47): 17600-17601 DOI:10.1073/pnas.0607612103 |
WU H H, WANG W J, LÜ D, et al. Turbot (Scophthalmus maximus) biodiversity assessment using high-throughput Illumina sequencing to analyze juvenile turbot intestines and their bacterial cultures. Progress in Fishery Sciences, 2019, 40(4): 84-94 [吴欢欢, 王伟继, 吕丁, 等. 应用高通量测序技术分析大菱鲆幼鱼肠道及其养殖环境的微生物群落结构. 渔业科学进展, 2019, 40(4): 84-94] |
WU M L, LI H Y, JIANG H, et al. Genomic characterization and phylogenetic analysis of grass carp reovirus AH528 strain. Progress in Fishery Sciences, 2018, 39(5): 36-43 [吴明林, 李海洋, 江河, 等. 草鱼呼肠孤病毒AH528株全基因组特征及进化分析. 渔业科学进展, 2018, 39(5): 36-43] |
XIA F, ZHOU X, LIU Y, et al. Composition and predictive functional analysis of bacterial communities inhabiting Chinese cordyceps insight into conserved core microbiome. BMC Microbiology, 2019, 19: 105 DOI:10.1186/s12866-019-1472-0 |
YU Z H, GENG Y, WANG K Y, et al. Complete genome sequence of Vibrio mimicus strain SCCF01 with potential application in fish vaccine development. Virulence, 2017, 8(6): 1028-1030 DOI:10.1080/21505594.2016.1250996 |
YUN C S, MOTOYAMA T, OSADA H. Biosynthesis of the mycotoxin tenuazonic acid by a fungal NRPS-PKS hybrid enzyme. Nature Communications, 2015, 6: 8758 DOI:10.1038/ncomms9758 |
ZHANG C H, DENG W Q, YAN W J, et al. Whole genome sequence of an edible and potential medicinal fungus, Cordyceps guangdongensis. G3 Genesgenetics, 2018, 8(6): 1863-1870 DOI:10.1534/g3.118.200287 |
ZHENG P, XIA Y, XIAO G, et al. Genome sequence of the insect pathogenic fungus Cordyceps militaris, a valued traditional Chinese medicine. Genome Biology, 2011, 12(11): R116 DOI:10.1186/gb-2011-12-11-r116 |
ZIMMERMANN G. The entomopathogenic fungus Metarhizium anisopliae and its potential as a biocontrol agent. Pest Management Science, 1993, 37(4): 375-379 DOI:10.1002/ps.2780370410 |



