叶尔羌高原鳅(Triplophysa yarkandensis) (Day, 1877),又叫叶尔羌条鳅(郭焱等, 2012),俗称狗头鱼,主要分布于塔里木河水系(叶尔羌河、喀什噶尔河、木札提河、阿克苏河和车尔臣河等),是塔里木河水系特有鱼类(何长才, 1996; 郭焱, 2012)。目前,关于叶尔羌高原鳅的研究多集中在形态学(宋勇等, 2018; 陈生熬等, 2017)、摄食生长(陈生熬等, 2014; 王帅等, 2017)、病理学(姚娜等, 2018; 雷曼红等, 2010)等方面。近年来,随着分子生物学的发展,部分学者(Chen et al, 2016; González et al, 2013; 魏玉众等, 2019)对叶尔羌高原鳅基因组学进行研究,但关于叶尔羌高原鳅种群遗传学的研究尚未见报道。
微卫星(microsatellite)又称为简单序列重复(simple sequence repeats, SSRs),是由1~6 bp的重复单位组成的串联重复DNA序列(Ibrahim et al, 2015),具有多态性高、变异性强、数据易统计分析等突出优点(Dewoody, 2000)。鱼类微卫星标记开发中常以二碱基重复类型为主(翟云等, 2020; Castoe et al, 2012),但也有学者认为,三碱基重复较二碱基重复具有更高的筛选效率和多态性(李文升等, 2011; Thiel et al, 2013)。随着人们对分子遗传学的深入研究,DNA分子标记技术得到越来越广泛的应用,它能够直接反映生物个体间或者种群间基因的差异。近年来,微卫星也被广泛应用于水产养殖业中,史君洁等(2018)、马秀英等(2016)、李景芬等(2016)和毛守康等(2016)分别对鲤鱼(Cyprinus carpio)、黄河鲤(C. carpio)、黄颡鱼(Pelteobagrus fulvidraco)和梭鱼(Liza haematocheila)等水产动物遗传多样性进行研究,找出了具有多态性的SSR标记。本研究基于高通量测序平台对叶尔羌高原鳅基因组进行随机测序,并挑选出100个含二碱基重复、三碱基重复的微卫星序列,并依据其侧翼序列合成引物,最终筛选出15对多态性较高的微卫星引物,对塔里木河5个河段叶尔羌高原鳅群体的遗传多样性、种群分化及遗传结构进行分析,旨在为塔里木河特有鱼类的保护提供技术支持。
1 材料与方法 1.1 实验材料叶尔羌高原鳅样品于2015-2019年每年4-7月采集于塔里木河流域。其中,阿尔干(AEG) 25尾,台特玛湖(TTMH) 15尾,车尔臣河(CECH) 27尾,阿克苏河(AKSH) 17尾,台南河(TNH) 18尾,共计102尾,用酒精和福尔马林浸泡保存在塔里木大学水产养殖实验基地,采样点分布见图 1。

|
图 1 塔里木河流域采样地点 Fig.1 Sampling sites in Tarim River |
取20 mg除去福尔马林的肌肉组织,按照常规的苯酚–氯仿抽提基因组织DNA(刘忠浩, 2019)。采用核酸蛋白检测仪(DS-11, DENOVIX)检测基因组DNA的浓度及纯度,用1%的琼脂糖电泳检测(电泳仪Bio- Rad)质量,将纯度较好的DNA于–20℃保存备用。
1.3 微卫星引物的设计与合成叶尔羌高原鳅微卫星引物的获取参考吕振明等(2017)的方法。将样品送至上海生工生物工程股份有限公司,采用Illumina Hiseq PE150高通量测序平台进行全基因组从头测序,原始数据通过de novo拼接和组装获取拼接序列,使用MISA对拼接序列进行微卫星搜索,得到的微卫星最小间距为200 bp。随机挑选100个二碱基重复、三碱基重复且重复次数在10次以上的微卫星序列进行引物开发和合成。
1.4 微卫星引物的筛选随机选取8尾叶尔羌高原鳅个体的基因组DNA为模板,对合成的引物进行筛选并优化反应条件。PCR反应体系总体积为30 μL:PCR Mix 15 μL (天根, 北京),DNA模板3 μL,上游引物1.5 μL,下游引物1.5 μL,剩余体积双蒸水补足。反应条件:95℃预变性3 min;94℃变性30 s,退火30 s (温度见表 1),72℃延伸40 s,以上程序循环34次;最后72℃继续延伸10 min,反应产物采用8%非变性聚丙烯酰胺凝胶电泳检测,经0.1%硝酸银染色后,在化学发光成像分析系统(Bio-Rad)下拍照保存。
|
|
表 1 叶尔羌高原鳅微卫星位点及PCR引物信息 Tab.1 Information on polymorphic microsatellite markers of T. yarkandensis |
选取15对多态性较高引物,对塔里木河5个河段的叶尔羌高原鳅群体样本基因组DNA进行扩增,反应体系和程序同1.4,将产物送至北京华世百奥生物技术有限公司,基于Fragment Analyzer 5200毛细电泳平台分析微卫星数据。
1.6 数据处理与分析利用Cervus 3.03软件计算出5个群体叶尔羌高原鳅的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)和多态信息含量(PIC)。利用PopGen32分析群体内近交系数(Fis)、群体遗传分化指数(Fst)、Shannon多样性指数(H)、基因多样性指数、Hardy-Weinberg平衡检测及遗传距离(GD)与遗传相似度(GS)。基于群体间的Nei´s遗传距离使用MEGA 7.0软件构建UPGMA系统进化树。
2 结果 2.1 高通量测序结果及微卫星引物信息高通量测序共获得27, 802条存在微卫星位点的序列,在检测出的微卫星序列中,二碱基重复占优势,占全部微卫星序列的34.34%;三碱基重复占5.22%,四碱基重复占2.53%,五碱基重复占2.86%,六碱基重复占12.46%。根据微卫星筛选要求和引物设计原则,共设计100对引物,其中,79对引物成功扩增,但只有39对引物具有多态性(表 1)。部分引物(Y7、Y11和Y45)经硝酸银染色后显示出多态性(图 2)。
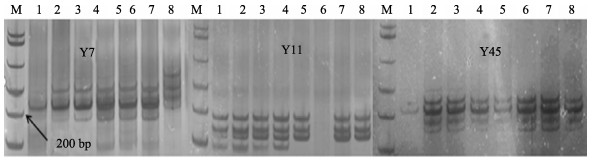
|
图 2 叶尔羌高原鳅SSR引物聚丙烯酰胺凝胶电泳 Fig.2 T. yarkandensis SSR primer polyacrylamide gel electrophoresis M:Marker;泳道1~8:8个不同样品PCR产物 M: Marker; Lane 1~8: Eight different samples of PCR product |
15对多态性较高的引物在5个叶尔羌高原鳅群体中进行扩增,分析扩增结果,得到种群遗传多样性信息(表 2)。叶尔羌高原鳅的等位基因数介于30~52之间,有效等位基因数为6.593~30.626,平均等位基因数和有效等位基因数分别为48.467和15.181,其中,Y39位点的等位基因数最少,为30,位点Y7、Y11、Y17、Y28、Y42、Y44、Y54和Y80的等位基因数最多,为52。各位点的Shannon多样性指数为2.447~3.699,观测杂合度为0.043~0.826,多态信息含量为0.853~0.975。种群间遗传分化系数为0.043~ 0.295,平均值为0.102,群体内近交系数为–0.096~ 0.760,多数大于零,对位点进行Hardy-Weinberg平衡检测发现,15个位点均符合Hardy-Weinberg平衡(P > 0.05),说明群体受外来因素干扰小,群体遗传结构处于稳定状态。
|
|
表 2 叶尔羌高原鳅15个微卫星位点的遗传多样性 Tab.2 Genetic diversity information of 15 microsatellites in T. yarkandensis |
5个叶尔羌高原鳅群体遗传多样性结果见表 3。从表 3可以看出,叶尔羌高原鳅的等位基因数为10.429~18.143,其中,车尔臣河群体最多,阿克苏河群体最少;平均有效等位基因数为7.969~10.572,其中,车尔臣河群体最多,台特玛湖群体最少;平均观测杂合度为0.517~0.706,其中,阿克苏河群体最高,台特玛湖群体最低;平均期望杂合度为0.887~0.908,其中,阿尔干群体最高,台南河群体最低;5个群体Shannon多样性指数为2.077~2.516,其中,车尔臣河群体最高,阿克苏河群体最低;多态信息含量为0.760~0.877,其中,阿尔干群体最多,阿克苏河群体最少,且多态信息含量均P > 0.5。
|
|
表 3 叶尔羌高原鳅5个群体的遗传多样性 Tab.3 Characteristics of 15 microsatellite loci in 5 populations of T. yarkandensis |
根据15对引物的扩增结果,对5个群体间的遗传距离和遗传相似度进行分析计算(表 4)。5个群体间的遗传距离为0.606~1.901,其中,阿克苏河与台南河群体的遗传距离最小(0.606),阿尔干与台南河群体遗传距离最大(1.901)。各群体间遗传相似度为0.149~ 0.545,其中,阿尔干群体与台南河群体遗传相似度最小(0.149),阿克苏河与台南河群体的遗传相似度最大(0.545)。基于群体间Nei´s遗传距离构建UPGMA系统进化树如图 3所示。结果显示,5个叶尔羌高原鳅群体聚为3类,车尔臣河与台特玛湖群体聚为一类、阿克苏河与台南河群体聚为一类、阿尔干群体单独聚为一类。
|
|
表 4 叶尔羌高原鳅5个群体的遗传距离和遗传相似度 Tab.4 Genetic distance and genetic similarity of 5 populations in T. yarkandensis |
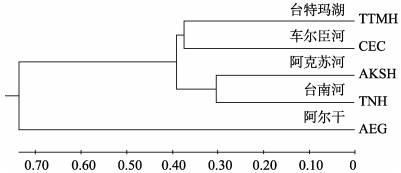
|
图 3 基于Nei's遗传距离构建的5个叶尔羌高原鳅群体UPGMA聚类树 Fig.3 UPGMA tree among 5 populations of T. yarkandensis constructed based on Nei's genetic distance |
多态信息含量是评价微卫星位点的重要参考标准,PIC > 0.5表明位点具有较高的多态性,0.25 < PIC < 0.5表明该位点具有中度多态性,PIC < 0.25表明位点具有低度多态性(丁海燕等, 2015)。张智等(2019)利用高通量测序法对西昌华吸鳅(Sinogastromyzon sichangensis)基因组进行测序并筛选出29对具有多态性的引物,平均等位基因数为14.5,观测杂合度和期望杂合度分别为0.620和0.882,多态信息含量为0.859。李薇等(2017)基于高通量测序成功筛选出达氏鲟(Acipenser dabryanus) 25个微卫星位点,每个位点的等位基因数为7.2,观测杂合度为0.744,期望杂合度为0.727,除了1个位点以外,其余位点的多态信息含量均 > 0.5,其多态性较好。本研究基于高通量测序技术开发的二碱基重复、三碱基重复的100对微卫星引物中39对微卫星位点表现出扩增多态性,选取多态性较高的15对引物用于叶尔羌高原鳅群体的遗传多样性分析,发现其等位基因数为30~52,且8个位点都具有较高的等位基因数(52),各位点的平均多态信息含量为0.853~0.975,均 > 0.7,说明这15个微卫星位点均具有高度多态性(PIC > 0.5),群体遗传多样性丰富,表明利用高通量测序技术能够较好地开发多态性良好的微卫星标记,可用于后续分子实验。
3.2 叶尔羌高原鳅群体的遗传多样性水平生物群体的遗传多样性是种质资源的重要组成部分,它是物种适应多变的环境条件、维持长期生存和进化的基础(颜元杰等, 2019)。关于鱼类遗传多样性的研究有很多报道,丁海燕等(2015)用6个微卫星标记分析了安徽、甘肃、广东、广西、黑龙江、江苏和重庆7个泥鳅(Misgurnus anguillicaudatus)群体的遗传多样性,发现7个泥鳅群体遗传多样性丰富,泥鳅群体存在较明显的近交或斑块化分布,我国泥鳅呈现东西向梯度分布并在南北向可能存在扰动。刘洁等(2019)对鄱阳湖地区及珠江流域的7个泥鳅群体进行研究发现,目前,我国泥鳅群体的遗传多样性处于中等水平,泥鳅群体间的遗传分化受自身迁移能力和地理隔离等因素的影响。本研究中,5个叶尔羌高原鳅群体的有效等位基因数、diversity index on多样性指数大小的顺序均为车尔臣河 > 阿尔干 > 台南河 > 台特玛湖 > 阿克苏河群体,平均多态信息含量均在0.7以上,大小顺序为阿尔干 > 台南河 > 车尔臣河 > 台特玛湖 > 阿克苏河,说明各群体的遗传多样性水平都较高,其中,车尔臣河群体、阿尔干群体具有较高的等位基因丰富度和较高的遗传多样性水平,阿克苏河群体具有较低的等位基因丰富度和相对较低的遗传多样性水平,5个群体的种质资源良好,具有种群稳定性。造成以上现象的原因可能是阿克苏河、台南河位于塔里木河中上游,阿尔干、车尔臣河、台特玛湖位于塔里木河下游,上游叶尔羌高原鳅随水流到达下游,造成基因交流,导致下游群体的等位基因数增加,遗传多样性水平较高,说明叶尔羌高原鳅群体间的等位基因丰富度和遗传多样性水平受自身迁移能力和地理分布等因素的影响,与上述学者的研究结果一致。
3.3 叶尔羌高原鳅群体的遗传结构种群的遗传结构受地理隔离、生存环境、种群瓶颈、基因流、选择等多种因素的影响,其中任一因素的变化都可能会造成该种群的遗传结构改变(武世雄等, 2018)。遗传距离是衡量群体间遗传关系的指标,遗传距离越小,说明两群体间亲缘关系越近;反之,亲缘关系越远(王林龙等, 2019)。李大命等(2019)研究了江苏省4个太湖新银鱼(Neosalanx taihuensis)的种群遗传结构,将其分为2个进化类群,并发现其遗传分化与水系的分布不吻合,可能与其进化历史有密切关系。李旭鹏等(2020)研究发现,6个凡纳滨对虾(Litopenaeus vannamei)群体内近交系数Fis > 0,说明其群体内存在杂合度降低的现象,综合Ho < He,表明其存在一定程度的近交现象。本研究中,5个群体的遗传分化系数Fst为0.043~0.295,大多数在0.05~0.15之间,说明叶尔羌高原鳅群体间属于中等程度的遗传分化;群体内除引物Y24、Y85在群体内近交系数Fis < 0外,其余引物在群体内近交系数均 > 0,说明群体内出现了近交现象;5个群体间的遗传距离和遗传相似度分别为0.606~1.901和0.149~0.545,其中,阿克苏河与台南河群体的遗传距离最小、相似度最大,阿尔干与台南河群体遗传距离最大、相似度最小。Nei´s遗传距离聚类树将5个群体聚为3类:台特玛湖与车尔臣河群体、阿克苏河与台南河群体、阿尔干群体,说明台特玛湖与车尔臣河群体、阿克苏河与台南河群体的遗传距离较近,相似度较高,与群体地理位置的分布相吻合。上述结果表明,塔里木河各个河段叶尔羌高原鳅之间虽然有一定的差异,但仍然有基因交流的现象,可能是从2001年起实施了“塔里木河流域近期综合治理工程”,到2013年已完成了14次生态输水,水源多次到达台特玛湖(阿布都米吉提等, 2016),部分叶尔羌高原鳅顺水而下而产生的。
ABLEKIM A, KASIMU A, KURBAN A, et al. Evolution of small lakes in lower reaches of Tarim River based on multi- source spatial data. Geographical Research, 2016, 35(11): 2071-2090 [阿布都米吉提·阿布力克木, 阿里木江·卡斯木, 艾里西尔·库尔班, 等. 基于多源空间数据的塔里木河下游湖泊变化研究. 地理研究, 2016, 35(11): 2071-2090] |
CASTOE T A, STREICHER J W, MEIK J M, et al. Thousands of microsatellite loci from the venomous coralsnake Micrurus fulvius and variability of select loci across populations and related species. Molecular Ecology Resources, 2012, 12(6): 1105-1113 DOI:10.1111/1755-0998.12000 |
CHEN S A, YAO N, SONG Y, et al. Effect of morphological traits on body mass of Triplophysa (Hedinichthgs) yarkandensis (Day). Xinjiang Agricultural Sciences, 2017, 4(3): 546-556 [陈生熬, 姚娜, 宋勇, 等. 塔里木河叶尔羌高原鳅形态性状对体质量的影响. 新疆农业科学, 2017, 4(3): 546-556] |
CHEN S A, YAO N, WANG Z C, et al. Study on feeding habits and growth of Triplophysa (Hedinichthgs) yarkandensis (Day). Journal of Shanghai Ocean University, 2014, 23(3): 374-381 [陈生熬, 姚娜, 王智超, 等. 塔里木河叶尔羌高原鳅摄食和生长的研究. 上海海洋大学学报, 2014, 23(3): 374-381] |
CHEN S A, YAO N, XIE C X, et al. Complete mitochondrial genome of the Triplophysa (Hedinichthys) yarkandensis (Day). Taylor and Francis, 2016, 11(5): 64-89 |
DEWOODY J A, AVISE J C. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. Journal of Fish Biology, 2000, 56(3): 461-473 DOI:10.1111/j.1095-8649.2000.tb00748.x |
DING H Y, ZHU M, PENG C, et al. Preliminary analysis of the microsatellite diversity of Chinese loach (Misgurnus anguillicaudatus). Journal of Fisheries of China, 2015, 39(10): 1433-1442 [丁海燕, 朱明, 彭冲, 等. 我国泥鳅微卫星遗传多样性初步分析. 水产学报, 2015, 39(10): 1433-1442] |
GONZáLEZ C, VECCHIONACCE H, ARAQUE H, et al. Extraction of antimicrobial peptides from epidermis of Triplophysa yarkandensis and bacteriostasis experiment. Chinese Journal of Veterinary Medicine, 2013, 15(5): 399-404 |
GUO Y. Fishes of Xinjiang. Urumqi: Xinjiang Science and Technology Press, 2012: 34-35 [郭焱. 新疆鱼类志. 乌鲁木齐: 新疆科学技术出版社, 2012: 34-35]
|
HE C C. Two species of fish with the highest elevation and their distribution. Sichuan Animal, 1996, 15(3): 116-117 [何长才. 两种海拔最高的鱼类及其分布. 四川动物, 1996, 15(3): 116-117] |
IBRAHIM I O, ILHAN D, ERTUGRUL F. In silico analysis of simple sequence repeats (SSRs) in chloroplast genomes of Glycine species. Plant Omics, 2015, 8(1): 24-29 |
LEI M H, ZHAO Y P, JIANG R J. Acute toxicity of four drugs to Triplophysa(Hedinichthgs) yarkandensis(Day). Journal of Aquaculture, 2010, 31(9): 43-45 [雷曼红, 赵艳斐, 姜仁军. 4种药物对叶尔羌高原鳅的急性毒性试验. 水产养殖, 2010, 31(9): 43-45] |
LI D M, TANG S K, LIU Y S, et al. Genetic diversity and population structure of four Neosalanx taihuensis populations in Jiangsu Province. Progress in Fishery Sciences, 2019, 40(6): 1-10 [李大命, 唐晟凯, 刘燕山, 等. 江苏省4个太湖新银鱼种群遗传多样性和遗传结构分析. 渔业科学进展, 2019, 40(6): 1-10] |
LI J F, CAI K J, GONG C P, et al. Microsatellite analysis of the genetic characteristics of Pelteobagrus fulvidraco, Pelteobagrus vachelli, and its hybrid generation. Jiangsu Agricultural Sciences, 2018, 46(7): 159-162 [李景芬, 采克俊, 公翠萍, 等. 普通黄颡鱼、瓦氏黄颡鱼及其杂种一代遗传特征的微卫星分析. 江苏农业科学, 2018, 46(7): 159-162] |
LI W, LI J X, JING H F, et al. Development of microsatellite loci for Dabryxs sturgeon (Acipenser dabryanus) using high- throughput sequencing. Chinese Journal of Zoology, 2017, 52(3): 449-457 [李薇, 李久煊, 荆慧芳, 等. 基于高通量测序的达氏鲟微卫星标记筛选. 动物学杂志, 2017, 52(3): 449-457] |
LI W S, LIU C, LU C Y, et al. Isolation and characterization of tri- and tetranucleotide repeat microsatellites in grass carp (Ctenopharyngodon idellus). Journal of Fishery Sciences of China, 2011, 18(4): 742-750 [李文升, 刘翠, 鲁翠云, 等. 草鱼三、四核苷酸重复微卫星标记的分离与特征分析. 中国水产科学, 2011, 18(4): 742-750] |
LI X P, LUAN S, CAO B X, et al. Genetic background analysis of six groups of white shrimp Litopenaeus vannamei in China using SSR markers. Progress in Fishery Sciences, 2020, 41(3): 103-110 [李旭鹏, 栾生, 曹宝祥, 等. 凡纳滨对虾6个国内群体的遗传背景分析. 渔业科学进展, 2020, 41(3): 103-110] |
LIU J, GAO F Y, LU M X, et al. Microsatellite genetic diversity of loach (Misgurnus anguillicaudatus) populations in China. Journal of Agricultural Biotechnology, 2019, 27(12): 2101-2109 [刘洁, 高风英, 卢迈新, 等. 我国7个泥鳅群体遗传多样性的微卫星分析. 农业生物技术学报, 2019, 27(12): 2101-2109] |
LIU Z H. An optimized method to extract genomic DNA from animal whole blood by phenol method. Inner Mongolia Science Technology and Economy, 2019(20): 77-78 [刘忠浩. 苯酚法提取动物全血中基因组DNA的优化方案. 内蒙古科技与经济, 2019(20): 77-78] |
Lü Z M, HOU L, GONG L, et al. Isolation and analysis on EST microsatellites of Sepiella japonica by de novo high- throughput ranscriptome sequencing. Oceanologia et Limnologia Sinica, 2017, 48(4): 877-883 [吕振明, 侯龙, 龚理, 等. 基于de novo高通量测序的曼氏无针乌贼(Sepiella japonica) ESTs中微卫星位点筛选与特征分析. 海洋与湖沼, 2017, 48(4): 877-883] |
MA X Y, WANG W, PANG YZ, et al. Microsatellite marker analysis of genetic diversity in the two populations of yellow River carps. Heilongjiang Animal Science and Veterinary Medicine, 2016(23): 22-26 [马秀英, 王伟, 庞有志, 等. 两个黄河鲤鱼群体遗传多样性的微卫星标记分析. 黑龙江畜牧兽医, 2016(23): 22-26] |
MAO S K, MA A J, DING F H, et al. Analysis of genetic structures of four wild geographic populations of mullet Liza haematocheila by using microsatellite marker technique. Progress in Fishery Sciences, 2016, 37(2): 68-75 [毛守康, 马爱军, 丁福红, 等. 梭鱼4个野生地理群体遗传多样性的微卫星分析. 渔业科学进展, 2016, 37(2): 68-75] |
SHI J J, SHI L Y, LI C T, et al. Study on the germplasm identification technique of four species of Carp by microsatellite markers. Genomics and Applied Biology, 2018, 37(2): 748-755 [史君洁, 石连玉, 李池陶, 等. 微卫星标记对4种鲤鱼种质鉴定技术的研究. 基因组学与应用生物学, 2018, 37(2): 748-755] |
SONG Y, CHENG Y, LUO L L, et al. Correlation analysis between morphometrics and body weight of Triplophysa yarkandensis of wild populations. Journal of Domestic Animal Ecology, 2018, 39(11): 25-29 [宋勇, 程勇, 罗磊磊, 等. 叶尔羌高原鳅野生群体形态性状与体重的相关分析. 家畜生态学报, 2018, 39(11): 25-29 DOI:10.3969/j.issn.1673-1182.2018.00.006] |
THIEL T, MICHALEK W, VARSHNEY R K, et al. Exploiting EST databases for the development and characterization of gene derived SSR-markers in barley (Hordeum vulgare L). Theoretical and Applied Genetics, 2003, 106(3): 411-422 DOI:10.1007/s00122-002-1031-0 |
WANG L L, ZHANG X M, WANG Z, et al. Morphological characteristics and genetic differentiation of a breeding population of Sepia esculenta Qingdao. Journal of Fishery Sciences of China, 2019, 26(2): 342-352 [王林龙, 张秀梅, 王展, 等. 青岛近岸金乌贼繁殖群体形态特征及遗传分化. 中国水产科学, 2019, 26(2): 342-352] |
WANG S, SONG Y, REN D Q, et al. Effects of ingestions and digestive enzyme activity in Triplophysa (Hedinichthgs) yarkandensis (Day) of different temperature. Journal of Tarim University, 2017, 29(3): 29-34 [王帅, 宋勇, 任道全, 等. 养殖温度对叶尔羌高原鳅摄食及主要消化酶活性的影响. 塔里木大学学报, 2017, 29(3): 29-34 DOI:10.3969/j.issn.1009-0568.2017.03.005] |
WEI Y Z, YAO N, WANG S, et al. Studies on the differences of three isozymes in Triplophysa (Hedinichthgs) yarkandensis (Day). Heilongjiang Animal Science and Veterinary Medicine, 2019(1): 152-156 [魏玉众, 姚娜, 王帅, 等. 叶尔羌高原鳅三种同工酶的组织差异性研究. 黑龙江畜牧兽医, 2019(1): 152-156] |
WU S X, JIANG X T, WANG W, et al. Genetic diversity among 6 populations of Hexagrammos otakii based on microsatellite analysis. Chinese Fishery Quality and Standards, 2018, 8(3): 52-60 [武世雄, 姜欣彤, 王伟, 等. 大泷六线鱼6个群体遗传多样性的微卫星分析. 中国渔业质量与标准, 2018, 8(3): 52-60 DOI:10.3969/j.issn.2095-1833.2018.03.007] |
YAN Y J, CAO Z M, DING W D, et al. On genetic diversity analysis of 6 populations of Siniperca chuatsi in Jiangsu Province. Marine Fisheries, 2019, 41(1): 25-33 [颜元杰, 曹哲明, 丁炜东, 等. 江苏省6个翘嘴鳜群体的遗传多样性分析. 海洋渔业, 2019, 41(1): 25-33 DOI:10.3969/j.issn.1004-2490.2019.01.004] |
YAO N, SONG Y, WANG S, et al. Toxicity of salinity and alkalinity to Triplophysa(Hedinichthgs)yarkandensis(Day)of Tarim River. Southwest China Journal of Agricultural Sciences, 2018, 31(2): 423-428 [姚娜, 宋勇, 王帅, 等. 盐度和碱度对塔里木河叶尔羌高原鳅毒性的研究. 西南农业学报, 2018, 31(2): 423-428] |
ZHAI Y, WU R X, NIU S F, et al. Isolation and characterization of dinucleotide microsatellite loci for Lateolabrax maculates using next generation sequencing technology. Genomics and Applied Biology, 2020, 39(2): 507-513 [翟云, 吴仁协, 牛素芳, 等. 采用高通量技术开发花鲈二碱基重复微卫星标记. 基因组学与应用生物学, 2020, 39(2): 507-513] |
ZHANG Z, YU D, LIU F, et al. Isolation of microsatellite loci and diversity analysis of Sinogastromyzon sichangensis. Acta Hydrobiologica Sinica, 2019, 43(6): 1224-1230 [张智, 俞丹, 刘飞, 等. 西昌华吸鳅的微卫星引物筛选及赤水河四个地理种群的遗传多样性分析. 水生生物学报, 2019, 43(6): 1224-1230] |



