2. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业资源可持续发展重点实验室 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 山东 青岛 266071
2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao); Key Laboratory of Sustainable Utilization of Marine Fisheries Resources, Ministry of Agriculture and Rural Affairs, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao, Shandong 266071, China
肌肉生长抑制素(Myostatin, Mstn)基因,也称为生长和分化因子-8(GDF-8),是动物肌肉发育和生长过程中的负调控因子,在脊椎动物的研究中,发现Mstn主要在肌肉组织中表达,并负向抑制肌肉生长和调节发育(Kambadur et al, 1997; McPherron et al, 1997)。Mstn对肌肉生长发育的作用为促进动物生产、增加市场利润提供了新途径,随着对Mstn研究的不断深入,其肌肉调控机制和相关基因功能也越来越完善。Mstn作为胞外信号分子,可与成肌细胞膜上的受体结合引起受体自身的磷酸化,启动细胞内一系列信号传导过程,作用于生肌分化抗原(MyoD)靶基因的调控区,进而调控肌肉的形成和分化(Morikawa et al, 2016)。脊椎动物Mstn功能的发挥主要通过参与调控TGF-β/Smad通路活性而参与肌肉的生长和发育。目前,公认的Mstn的信号传导途径是Mstn-ActRⅡB型受体(蛋白激酶受体)-Smad蛋白信号传导通路。Smads是细胞质内TGF-β信号转导分子,可直接与DNA结合,它们可以将TGF-β信号直接由细胞膜导入细胞核内。Langley等(2003)研究发现,Mstn对成肌细胞分化的抑制正是通过Smad3抑制MyoD的活性与表达实现的,其能够使成肌细胞不能分化形成肌管,阻碍了肌肉的形成。除了典型的Smad介导的信号途径外,Mstn也通过MAPK途径,包括p38、Erk 1/2和JNK途径参与肌肉发育的信号转导,如成熟的Mstn可分别通过TAK1-MKK6、Ras-MEK1和TAK1- MKK4级联激活p38 MAPK、Erk 1/2和JNK通路,参与调控肌肉发育(Huang et al, 2011)。
Mstn对肌肉生长发育的作用,不仅仅局限于脊椎动物,对无脊椎动物Mstn的研究也发现其参与并调控无脊椎动物肌肉生长(Li et al, 2016; Zhuo et al, 2017),但与脊椎动物Mstn调控的肌肉发育机制相比,无脊椎动物的研究相对滞后,目前其调控模式和相关基因功能仍不清楚。转录组分析是揭示分子机制的主流方法,其可以分析特定条件下基因表达情况,挖掘生物网络调控和分子机理,为基因功能研究提供新的思路。RNA干扰可以有效地沉默靶基因的表达,进而影响通路中基因表达量的变化,RNA干扰后从转录水平检测基因的表达变化,可以为阐释基因功能及其调控网络提供基础。
中国对虾(Fenneropenaeus chinensis)作为无脊椎动物甲壳类的典型代表,具有较高的经济价值,但其海捕量极低,基本依靠养殖供给市场,而养殖产量远远满足不了市场消费的需求,加之群体的生长性状选育进展缓慢且规格整齐度较差,利用分子手段来提高产量已迫在眉睫。我们前期对中国对虾的初步研究表明,Mstn可能与脊椎动物类似,负向调控中国对虾肌肉生长(Kong et al, 2020)。因此,本研究拟通过生物信息学的方法发掘Mstn调控体系中与肌肉生长发育有关的基因与通路,为探索无脊椎动物Mstn的作用机制及肌肉发育调控模式提供更多证据。本研究通过RNA干扰方法沉默中国对虾Mstn基因,采用高通量测序技术对其进行转录组测序分析,根据分析结果,参考脊椎动物肌肉发育调控模式和差异基因的功能,初步筛选出受Mstn调控、与肌肉发育相关的重要基因,了解其在脊椎动物和无脊椎动物中的异同之处,为解析对虾肌肉发育调控机制提供理论依据。
1 材料与方法 1.1 实验地点与材料实验所用中国对虾取自山东省青岛市即墨区鳌山卫镇黄海水产遗传育种中心第14代选育群体。养殖期间温度范围为21℃~24℃,每日换水量30%,每日投喂配方颗粒饲料和新鲜的菲律宾帘蛤(Ruditapes philippinarum),日投喂总量为中国对虾湿重的5%。将体重无显著差异的中国对虾分成2组,分别置于500 L养殖箱中暂养7 d。暂养结束后,分别对实验虾的肌肉尾部注射PBS (对照组)和Mstn-siRNA [生工生物工程(上海)股份有限公司设计合成](实验组),于注射后12 h取肌肉组织,每组取6尾,每尾取2份相同的肌肉组织,一份用于抑制效果检测,另一份用于转录组测序分析。所有样品于液氮中保存。
1.2 实验方法 1.2.1 Mstn基因RNA干扰中国对虾Mstn基因干扰实验所用干扰链siRNA (F: GCUGAAUACCCUA AUGAAATT; R: UUUCAUUAGGGUAUAGCTT),由生工生物工程(上海)股份有限公司设计并合成。以1 μg/g的剂量通过尾部肌肉注射至中国对虾体内,于注射后12 h取肌肉组织,每组取6尾,通过qRT-PCR (n=6)分别检测相对表达量,计算抑制效率。
1.2.2 总RNA提取和反转录用Trizol (TaKaRa)法提取中国对虾肌肉组织总RNA,用分光光度计(NanoDrop 2000, 美国)测定A260 nm和A280 nm值,检测RNA纯度和浓度,用1.5%的琼脂糖电泳检测RNA完整性。使用PrimeScript RT reagent kit (TaKaRa)试剂盒,按照说明书进行总RNA的反转。反转录完成后,将cDNA保存于–20℃,以进行下一步qRT-PCR。
1.2.3 实时相对荧光定量(qRT-PCR)根据Zhang等(2008)的方法,以18S作为内参基因,根据中国对虾Mstn (FcMstn) ORF序列(NCBI登录号: MG437236)设计Mstn基因实时相对定量特异性引物(表 1),引物由生工生物工程(上海)股份有限公司合成。
|
|
表 1 qRT-PCR所需引物 Tab.1 Primers used in the qRT-PCR |
为了验证转录组表达数据,根据转录组测序结果查找到相关基因的序列,通过PCR成功扩增并验证ActRⅡ、Smad1/5、Smad3和Smad6基因的编码区序列,根据编码区序列,分别设计引物用于qRT-PCR的特异性引物(表 1)。所有引物通过PCR检测并测序,确保目的片段特异性扩增和序列的准确性。使用SYBR PrimeScript™ RT试剂盒(TaKaRa, 日本)置于ABI QuantStudio 3系统中进行qRT-PCR分析。FcMstn的相对表达量用2–ΔΔCt法计算。
1.2.4 转录组测序分析通过qRT-PCR分析对照组和实验组相对表达量,确保实验组中FcMstn基因表达被有效抑制后,取出液氮中保存的样品,使用RNA提取试剂盒(TaKaRa)进行总RNA的提取,用1.5%的琼脂糖电泳检查RNA完整性,NanoDrop2000超微量分光光度计检测RNA浓度,所有指标经检测合格后,根据qRT-PCR的检测结果选取抑制效果最好的3个样品,交送至广州基迪奥生物科技有限公司进行cDNA文库的构建以及高通量测序,产出样品测序原始数据(raw reads),对原始数据进行接头及低质量reads去除处理,获得高质量的clean数据,使用短reads组装软件Trinity对高质量clean数据进行de novo组装(Liang et al, 2013)得到unigene。
组装得到的高质量unigene按照Nr、Swissprot、KOG和KEGG (E-value < 0.000 01)的优先级进行Blast比对,得到与已有unigene具有最高相似性的同源序列,从而得到该unigene的功能注释信息。基因表达量通过FPKM(Mortazavi et al, 2008)方法进行标准化,差异表达基因的阈值设置为Padj(矫正后P-value) < 0.05和log2Fold≥1 (C组和E组),使用DEGseq (Wang et al, 2010)软件对差异表达unigene进行分析。
2 结果与分析 2.1 RNA干扰效果分析通过qRT-PCR方法分析检测C组和E组FcMstn的相对表达水平,结果显示,E组的FcMstn相对表达量显著低于C组,抑制率达到76% (图 1)。
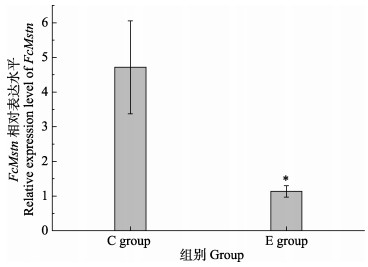
|
图 1 FcMstn在对照组(C)和实验组(E)肌肉组织中的相对表达水平 Fig.1 Relative expression level of FcMstn in muscle in the control (C) and experimental (E) groups "*"表示两组间具有显著性差异(P < 0.05)。下同 "*" indicates significant difference between two groups (P < 0.05). The same as in Fig.4 |
将符合测序标准的C组和E组的肌肉组织总RNA送至广州基迪奥生物科技有限公司进行高通量测序和质量控制,结果显示,共产生36, 605个unigenes,Q30碱基为94.05%,N50位1107 bp,表明测序质量良好,组装完整性较高,可用于后续的分析(表 2)。对获得的全部新基因与Nr、Swissprot、KOG和KEGG数据库进行比对,获得注释信息的基因总数为19, 628个,占新基因总数的53.62% (表 3)。在对照组和实验组样品中,共筛选到1657个差异表达基因,包括805个上调基因和852个下调基因(图 2)。
|
|
表 2 转录组组装数据的特性 Tab.2 Characteristics of assembly data |
|
|
表 3 四大数据库注释统计 Tab.3 Annotation statistics of four databases |
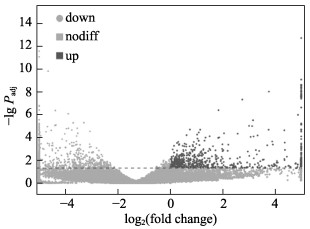
|
图 2 中国对虾Mstn基因RNA干扰转录组差异基因火山图 Fig.2 Volcano plot of differentially expressed gene distribution of FcMstn RNA interference transcriptome Padj < 0.05为差异基因筛选标准。有显著性差异表达的基因用红色点(上调)和黄色点(下调)表示,无显著性差异表达的基因用蓝色点表示 The screening standard of differential genes is Padj < 0.05. Red dots indicate the up-regulated genes with significant differences, yellow dots indicate the down regulated genes with significant differences, and no significant difference in the expression of genes with blue dots |
参考脊椎动物肌肉经典信号通路调控模式和差异基因的功能,初步筛选出肌肉发育相关的基因(表 4和表 5)。从中国对虾转录组数据中查找到Mstn,实验组表达显著降低(log2fc=–4.31),和定量结果一致,进一步证明了RNA干扰的有效性。参考脊椎动物肌肉经典信号通路调控模式,从中国对虾转录组数据中共筛选到16个参与其调控通路的基因。其中,参与TGF-β/Smad信号通路的筛选到12个基因,除Mstn外,其他分别为3个卵泡抑素(follistatin, FST)家族成员(FST、FST Like 1和FST Like 5)基因,3个激活素受体(activin receptor, ActR)成员(ActRIA、ActRIB和ActRⅡ)基因,4个Smad蛋白家族成员(Smad1/5、Smad3、Smad4和Smad6)基因和1个骨形态发生蛋白(bone morphogenetic protein, BMP)家族成员(BMP-7)基因。参与MAPK信号通路的筛选到4个基因,分别为p38、Erk、Ras和MAP3K7基因。通过基因的功能筛选到的非经典信号通路的与肌肉生长、蜕皮过程等密切相关的基因有14个,其中包括α-骨骼肌肌动蛋白(α-skeletal muscle actin)、肌球蛋白重链(myosin heavy chain)和肌钙蛋白C (troponin-C)基因,涉及蜕皮过程的蜕皮羧肽酶A(molting fluid carboxypeptidase A)、蜕皮激素受体HR3、蜕皮激素受体HR38、几丁质结合蛋白(chitin-binding protein) PCP20和CP14.6型、几丁质酶(chitinase 1)、表皮蛋白(cuticle protein)基因,参与肌肉生长的锌指蛋白MLP84B (muscle LIM protein 84B, MLP 84B)、谷氨酰胺合成酶2 (glutamine synthetase 2)、胰岛素样生长因子结合蛋白(IGF-binding protein)以及其参与脂肪代谢的脂肪酸结合蛋白7 (fatty acid binding protein 7)基因。这些基因在Mstn被抑制后,表现出不同程度的上调或下调表达模式(表 5)。在这些参与信号通路的相关基因中,除ActRIB基因表现略微上调外,其他基因均显示出不同程度的下调,其中,Erk、Wnt6、FSTL5、Mstn这4个基因下调表达变化较大。在非经典信号通路涉及的其他相关基因中,检测到8个基因(谷氨酰胺合成酶2、胰岛素样生长因子结合蛋白、α-骨骼肌肌动蛋白、2种几丁质结合蛋白、蜕皮激素受体3、蜕皮羧肽酶A和脂肪酸结合蛋白7)上调,其中,几丁质结合蛋白CP14.6、蜕皮羧肽酶A、蜕皮激素受体3这3个与蜕皮相关的基因上调变化较大;其他6个基因表现下调趋势,其中,肌钙蛋白C、肌球蛋白重链、几丁质酶ChiA和表皮蛋白下调变化较大。
|
|
表 4 筛选到的参与Mstn调控的TGF-β/Smad和MAPK信号通路的差异基因 Tab.4 Genes screening of TGF-β/Smad和MAPK signal pathway regulated by Mstn |
|
|
表 5 筛选到的Mstn调控的肌肉生长发育相关的差异基因 Tab.5 Screening of other genes related to muscle development regulated by Mstn |
为了验证转录组数据,本研究根据转录组数据相关基因的序列,PCR成功扩增并测序验证了ActRⅡ、Smad1/5、Smad3和Smad6的编码区序列,并进一步设计特异性引物检测了这些基因在PBS对照组和RNA实验组12 h的表达。结果显示,ActRⅡ、Smad1/5、Smad3和Smad6的表达量均显著下调,这与转录组数据的下调趋势是一致的,但下调程度有所差异。
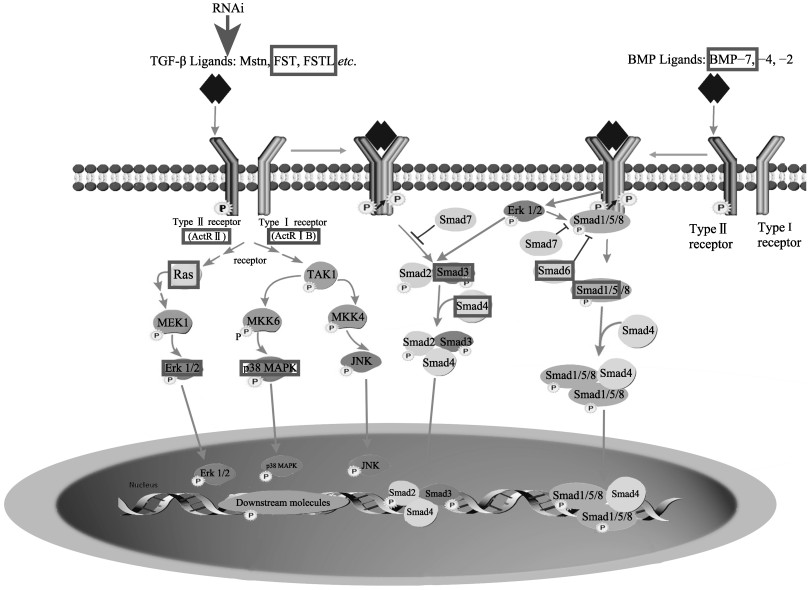
|
图 3 脊椎动物肌肉发育TGF-β/Smad信号通路 Fig.3 TGF-β/Smad signaling pathway related to muscle development in vertebrates 在中国对虾转录组数据中查找的基因用方框框出,其中,绿色框表示下调,红色框表示上调 The genes from the transcriptome data of F. chinensis were boxed, in which the green box indicated down regulation and the red box indicated up regulation |
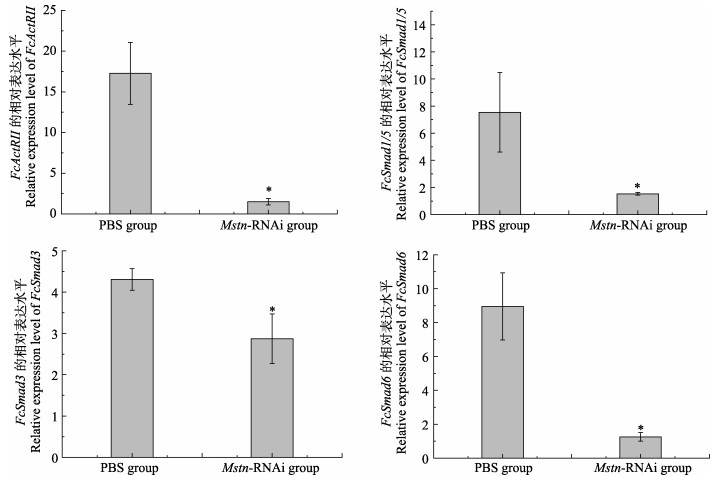
|
图 4 对照组和实验组肌肉中FcActRⅡ、Smad1/5、Smad3和Smad6 mRNA的相对表达 Fig.4 Relative expression of FcActRⅡ, Smad1/5, Smad3, and Smad6 mRNA in muscle in control and experimental groups |
前期研究表明,中国对虾Mstn可能与脊椎动物类似,负向调控中国对虾肌肉生长(Kong et al, 2020)。鉴于目前尚不清楚在无脊椎动物中Mstn调控的肌肉生长相关的基因,本研究以中国对虾为材料,利用转录组分析的方法筛选了Mstn调控的肌肉生长相关的重要基因。
在脊椎动物中,肌肉生长发育主要涉及到TGF-β/ Smad (Thomas et al, 2000)、MAPK (Freudenberg et al, 2004)、Hedgehogs(Hh) (Munsterberg et al, 1995)、Wnt (Logan et al, 2004)等经典信号通路。在脊椎动物Mstn介导的Smad信号通路中,Mstn能够结合多种蛋白,如其前肽、FST、FSTL,以及能阻断Mstn信号转导的其他物质,如ActRⅡB等拮抗剂,从而调节自身的激活,随后通过Smad2和Smad3参与的Smad信号通路激活细胞核内相关靶基因发挥对肌肉的调控作用(Huang et al, 2011)。在脊椎动物BMP介导的Smad信号通路中,BMP家族成员配体通过Smad1/5/8参与的Smad信号通路进入细胞核内,最终启动靶基因转录。除了典型的Smad介导的信号途径外,MAPK途径包括p38、Erk1/2和JNK途径,也参与了肌肉发育的信号转导。在本研究中,参考脊椎动物肌肉生长发育较为经典的TGF-β/Smad和MAPK通路筛选到包含Mstn在内的16个相关信号通路基因,其中只有ActR IB表现出轻微上调,其他基因均表现出不同程度的下调,表明Mstn被抑制后,存在的上下游关系可能与脊椎动物类似,进一步说明中国对虾肌肉生长涉及的TGF-β/Smad信号通路和MAPK信号通路可能与脊椎动物大体类似。脊椎动物中,FST和FSTL具有促进肌肉生长的功能,但其功能的发挥依赖于其与Mstn蛋白的C端配基结合,从而抑制Mstn与其受体结合,负性调节Mstn的功能(Matzuk et al, 1995; Lee et al, 2001; Li et al, 2011),因此,本研究Mstn基因被抑制也可能导致FST家族蛋白的下调。
不同于脊椎动物生长,中国对虾的肌肉生长发育是经过多次蜕皮实现的,蜕皮过程伴随着旧表皮的脱落、新表皮的形成和肌肉的收缩舒张等过程。除了参与经典的肌肉发育信号通路基因外,本研究也筛选出一些与蜕皮、肌肉生长和肌肉收缩等相关的基因,可见这些基因对肌肉的生长发育和蜕皮也起着至关重要的作用。本研究根据差异基因的功能初步筛选出14个与肌肉生长、肌肉运动和蜕皮相关的基因。其中,锌指蛋白MLP84B、谷氨酰胺合成酶2、胰岛素样生长因子结合蛋白与肌肉发育密切相关,促进肌肉生长和发育的谷氨酰胺合成酶和胰岛素样生长因子结合蛋白基因被检测到上调,提示Mstn的抑制可能会促进肌肉的生长。蜕皮羧肽酶A、蜕皮激素受体HR3、蜕皮激素受体HR38、几丁质结合蛋白、几丁质酶、表皮蛋白可能与蜕皮过程密切相关,甲壳动物通过周期性蜕去旧的表皮,产生新表皮来进行生长发育。甲壳类蜕皮过程是一个有众多基因互相作用协同的复杂过程,中国对虾Mstn被抑制后,蜕皮相关的基因也出现了不同程度的上下调表达,表明由Mstn抑制引起的相关肌肉应答系统的改变也能引起蜕皮相关基因的变化,而具体这些基因如何作用于蜕皮过程还需要更多的研究。脊椎动物中研究报道α-骨骼肌肌动蛋白、肌球蛋白重链和肌钙蛋白C参与肌肉收缩和放松(Rayment et al, 1993; Polyakov et al, 1997; Farah et al, 1999),中国对虾的生长发育、运动觅食和蜕皮等生命活动都离不开肌肉收缩与舒张,中国对虾Mstn的抑制能够引起这些基因的差异表达,表明这些基因很可能在中国对虾肌肉生长和蜕皮中发挥着关键的作用。
综上所述,本研究通过RNA干扰有效沉默Mstn基因,利用转录组测序分析,参考脊椎动物肌肉发育调控模式和差异基因功能,首次在无脊椎动物中筛选出了Mstn调控的与肌肉生长发育相关的基因,这些基因将为后续深入解析无脊椎动物肌肉调控网络和调控机制提供有价值的信息,关于这些基因在对虾中的生理功能及其Mstn调控的肌肉发育调控模式还需开展更多探索。
FARAH C S, REINACH C F. The troponin complex and regulation of muscle contraction. FASEB Journal, 1999, 9: 755-767 |
FREUDENBERG J A, CHEN D, CHEN W T. Stable suppression of membrane type 1 matrix metalloproteinase impairs MMP-2 activation, cell invasion, and tumorigenesis. Cellular, Molecular, and Tumor Biology, 2004, 64(S7): 529-530 |
HUANG Z, CHEN X, CHEN D. Myostatin: A novel insight into its role in metabolism, signal pathways, and expression regulation. Cellular Signalling, 2011, 23(9): 1441-1446 DOI:10.1016/j.cellsig.2011.05.003 |
KAMBADUR R, SHARMA M, SMITH T P L, et al. Mutations in myostatin (GDF8) in double-muscled Belgian Blue and Piedmontese cattle. Genome Research, 1997, 7(9): 910 DOI:10.1101/gr.7.9.910 |
KONG J, YAN Y, LU X, et al. Integrative phenotypic and gene expression data identify myostatin as a muscle growth inhibitor in Chinese shrimp Fenneropenaeus chinensis. Scientific Reports, 2020, 10(1): 5985 DOI:10.1038/s41598-020-61382-8 |
LANGLEY B, THOMAS M, BISHOP A, et al. Myostatin inhibits myoblast differentiation by down-regulating MyoD expression. Journal of Biological Chemistry, 2003, 277(51): 49831-49840 |
LEE S J, MCPHERRON A C. Regulation of myostatin activity and muscle growth. Proceedings of the National Academy of Sciences, USA, 2001, 98(16): 9306-9311 DOI:10.1073/pnas.151270098 |
LI S, ZHOU Z, DONG Y, et al. Molecular characterization, expression analysis of the myostatin gene and its association with growth traits in sea cucumber (Apostichopus japonicus). Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2016, 201: 12 DOI:10.1016/j.cbpb.2016.06.005 |
LI X, NIE F, YIN Z, et al. Enhanced hyperplasia in muscles of transgenic zebrafish expressing Follistatin1. Science China Life Sciences, 2011, 54(2): 65-71 |
LIANG C, LIU X, YIU S M, et al. De novo assembly and characterization of Camelina sativa transcriptome by paired- end sequencing. BMC Genomics, 2013, 14(1): 146 DOI:10.1186/1471-2164-14-146 |
LOGAN C Y, NUSSE R. The Wnt signaling pathway in development and disease. Annual Review of Cell and Developmental Biology, 2004, 20(1): 781-810 DOI:10.1146/annurev.cellbio.20.010403.113126 |
MATZUK M M, LU N, VOGEL H, et al. Multiple defects and perinatal death in mice deficient in follistatin. Nature, 1995, 374(6520): 360-363 DOI:10.1038/374360a0 |
MCPHERRON A C, LEE S J. Double muscling in cattle due to mutations in the myostatin gene. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(23): 12457-12461 DOI:10.1073/pnas.94.23.12457 |
MORIKAWA M, DERYNCK R, MIYAZONO K. TGF-β and the TGF-β family: Context-dependent roles in cell and tissue physiology. Cold Spring Harbor Perspectives in Biology, 2016, 8(5): a021873 DOI:10.1101/cshperspect.a021873 |
MORTAZAVI A, WILLIAMS B A, MCCUE K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Natural Methods, 2008, 5(7): 621-628 DOI:10.1038/nmeth.1226 |
MUNSTERBERG A E, KITAJEWSKI J, BUMCROT D A, et al. Combinatorial signaling by sonic hedgehog and Wnt family members induces myogenic bHLH gene expression in the somite. Genes and Development, 1995, 9(23): 2911-2922 DOI:10.1101/gad.9.23.2911 |
POLYAKOV A A, GUSEV N B. Utilization of troponin C as a model calcium-binding protein for mapping of the calmodulin-binding sites of caldesmon. Biochemical Journal, 1997, 321(3): 873-878 DOI:10.1042/bj3210873 |
RAYMENT I, HOLDEN H, WHITTAKER M, et al. Structure of the actin- myosin complex and its implications for muscle contraction. Science, 1993, 261(5117): 58-65 DOI:10.1126/science.8316858 |
THOMAS M, LANGLEY B, BERRY C, et al. Myostatin, a negative regulator of muscle growth, functions by inhibiting myoblast proliferation. Journal of Biological Chemistry, 2000, 275(51): 40235-40243 |
WANG L, FENG Z, WANG X, et al. DEGseq: An R package for identifying differentially expressed genes from RNA-seq data. Bioinformatics, 2010, 26(1): 136-138 DOI:10.1093/bioinformatics/btp612 |
ZHANG Q, LI F, ZHANG X, et al. cDNA cloning, characterization and expression analysis of the antioxidant enzyme gene, catalase, of Chinese shrimp Fenneropenaeus chinensis. Fish and Shellfish Immunology, 2008, 24(5): 584-591 |
ZHUO R Q, ZHOU T T, YANG S P, et al. Characterization of a molt-related myostatin gene (FmMstn) from the banana shrimp Fenneropenaeus merguiensis. General and Comparative Endocrinology, 2017, 248: 55-68 |



